毛竹LTR反转录转座子PHRE8的鉴定与转录模式分析
2020-03-13周明兵蒋政勤徐芷馨
季 航 周明兵 蒋政勤 郑 浩 徐芷馨
(1浙江农林大学亚热带森林培育国家重点实验室,浙江杭州 311300;2 浙江农林大学,浙江省竹资源与高效利用协同创新中心,浙江 杭州 311300)
植物LTR反转录转座子主要由长末端重复序列(long terminal repeat,LTR)、引物结合位点(primer binding site,PBS)、种属特异抗原(retrotransposon gag protein,GAG)基因、聚合酶(polymerase,POL)基因、多嘌呤序列(polypurinetrait,PPT)等构成[1]。其中LTR为末端反向重复序列,包含了转录的起始和终止信号,通常含有较为丰富的顺式调控元件,不编码蛋白质,PBS位于5′端LTR 下游,当转座子发生转座时,与tRNA 结合引导cDNA 负链的生成,GAG、POL基因属于编码区基因,编码与转座子复制和转座有关的酶类,结构与反转录的病毒结构相似,PPT位于3′端LTR上游,作为引物引导第二条cDNA 链的合成[1-3]。POL基因中包含蛋白质水解酶基因(pepsin-like aspartate proteases,PR)、反转录酶基因(reverse transcriptases,RT)、RNA 酶基因(ribonuclease H,RH)及整合酶基因(integrase,INT)[4]。依据POL基因中所编码的蛋白质顺序,LTR反转录转座子又分为Ty1-copia和Ty3-gypsy 2个超家族[5]。这2 类转座子的区别主要体现在INT基因的位置上[6]。
研究表明,转座子长末端重复序列便于设计引物且易于跟踪,插入位点的多态性提供了生物突变的可能,基于反转录转座子插入的分子标记技术不断被开发应用[7]。Woodrow 等[8]根据香桃木基因组数据与Ty1-copia 家族Tmc1 转座子的特征设计SSAP 标记引物,很好地区分了4个香桃木的供试品种。烟草中反转录转座子Ttn1 通过农杆菌介导转化至大豆植物中,利用插入突变的特性,将Ttn1 作为基因标签,研究大豆植物中编码重要农艺性状的基因[9]。但有研究发现,大多数已知的LTR反转录转座子均处于静默的状态,一部分由于在漫长的植物基因进化过程发生基因突变,丧失了转座功能,因此被称为“退化的转座子”;另一部分则是由于生物为维持基因的稳定性通过DNA 甲基化的修饰调控手段而导致了转座子沉默[10]。然而,当植物所处环境条件发生改变时,一些具备转座条件的反转录转座子会被激活,发生转座并插入靶基因中,且会影响插入位点相邻基因的表达[11]。如Butelli 等[12]发现反转录转座子Copia-like可在血橙处于低温条件下提升转录活性,影响调控花青苷相关基因的表达,促进花青苷的合成,使果实呈红色;蔡华等[13]研究小麦远缘杂交不亲和的主效基因Kr1时发现,玉米转座子的插入导致在小麦及其与玉米的杂交组合中Kr1基因失活。
毛竹(Phyllostachys heterocyclacv.Pubescens)为禾本科刚竹属单轴散生型常绿乔木状竹类植物,是我国栽培历史最悠久、面积最广、经济价值最高的竹种之一,因此探寻毛竹中的丰富基因资源具有重要意义[14]。目前已有关于毛竹基因表达调控方面的研究报道,王思雨等[15]在毛竹转录因子NF-Y 家族基因的鉴定和表达分析中发现,该家族基因参与了毛竹根的生长、笋的发育、开花的调控、光合作用与叶绿体发育的调控。LTR反转录转座子在毛竹基因组中含量丰富,在基因表达调控发挥了重要作用[16]。但关于毛竹LTR反转录转座子转座激活机制的研究尚鲜见报道。因此,获得活性LTR反转录转座子,解析LTR反转录转座子转座激活机制,可作为竹子诱变育种的遗传工具。通过对毛竹基因组上的LTR反转录转座子筛选,克隆1条典型的LTR反转录转座子序列,命名为PHRE8。本试验通过对PHRE8的结构、分布进化以及转录激活模式进行系统研究,旨在探讨PHRE8的活性激活机制,为挖掘出更多的毛竹活性转座子资源奠定理论基础。
1 材料与方法
1.1 材料
试验所用毛竹种子均取自广西灵川县同一株开花毛竹,从中选取大小一致、发育良好的种子并播种培育成实生苗。
1.2 试验设计
1.2.1 对照(CK)处理 将野生型毛竹种子先用蒸馏水冲洗1遍,再用70%酒精消毒30 s,无菌水冲洗3次。用无菌蒸馏水浸种24 h。随后在黑暗条件(25℃)下置于无菌的双层滤纸上萌发,并保持充足的水分。种子萌发出胚芽后移栽至温室的营养土中培养,保持水分充足。营养土的配比为珍珠岩∶泥炭土∶蛭石=1 ∶1 ∶1。待长至5~6片叶子后取该处理实生苗的叶片迅速用液氮速冻,-80℃保存备用。
1.2.2 DNA 甲基化抑制剂处理 将野生型毛竹种子先用蒸馏水冲洗1遍,再用70%酒精消毒30 s,无菌水冲洗3次。用浓度分别为50、150和250 μmol·L-1的5-氮杂胞苷浸种24 h。随后在黑暗条件(25℃)下置于无菌的双层滤纸上萌发,期间持续使用5-氮杂胞苷处理。种子萌发出胚芽后移栽至温室培养,条件同对照处理。待长至5~6片叶子后取该处理实生苗的叶片迅速用液氮速冻,-80℃保存备用。
1.2.3 辐射、高盐、高温、低温处理 选取长势一致、发育良好的毛竹实生苗分别作以下处理:在辐射剂量率为1 Gy·min-1,辐射剂量分别为30、50和70 Gy的137Cs-γ射线下进行辐射处理;用浓度分别为0.1、0.2和0.3 mol·L-1NaCl 溶液浇灌72 h;42℃培养箱中培养4 h;4℃培养箱中培养16 h。分别取每处理实生苗的叶片迅速用液氮速冻,-80℃保存备用。
1.3 PHRE8的生物信息学分析
1.3.1 结构鉴定与元件分析 在http://www.bamboogdb.org/中下载毛竹基因组数据,利用LTRharvest 软件[17]鉴定毛竹基因组LTR反转录转座子;利用LTRdigestion 软件[18]解析毛竹LTR反转录转座子结构,并剔除假阳性;利用cd-hit 软件[19]分析毛竹LTR反转录转座子的拷贝数。根据毛竹LTR反转录转座子结构的完整性和拷贝数,选取一个结构完整的LTR反转录转座子作为研究对象,将其命名为PHRE8。PHRE8 各个结构域由LTRdigestion 软件鉴定,并由此画出PHRE8 及拷贝序列的结构图,由编码区序列翻译的氨基酸序列通过NCBI 上的BLAST 比对确认。转座子的转座诱导与启动子有关,有活性的转座子5′端LTR的U3 结构域存在较多的顺式调控元件,利用PlantCARE 在线软件分析PHRE8的LTR 序列上的顺式作用元件。
1.3.2 插入时间的估算 通过LTR反转录转座子LTR 部分的相似性与宿主的核酸替换速率估算插入时间,在最初的LTR反转录转座子进行反转录时,其两端的L-LTR和LTR-R 由同一个模版合成,因此LTR 部分的同源性为100%,但是随着植物基因组的变化,LTR 部分会发生不同程度的分化,导致同源性下降,根据宿主核酸的替换速率,计算插入时间[20]。利用MEGAX 软件[21]比对PHRE8 两端LTR 序列的同源性,求分化度K。插入时间计算公式如下:

式中,T为插入时间;K为分化度;r为LTR 序列的平均替换率,约1.3×10-8bp/年[22]。
1.3.3 进化关系分析 在Gypsy Database(http://www.gydb.org/index.php/Main_Page)下载Ty3-gypsy 家族(Reina 亚家族的Gloin、Gimli和Reina,CRM 亚家族的Beetle1和Cereba,G-Rhodo 亚家族的G-Rhodo,Galadriel 亚家族的Tntom1和Galadriel,REM1 亚家族的REM1,Del 亚家族的Tma、Del和Retrosat-2)转座子RT 氨基酸序列(表1),与PHRE8的RT 氨基酸序列比对,利用MEGA6 软件中的邻接法(Neighbor-Joining)构建进化树,分析其所属转座子家族。

表1 植物典型LTR反转录转座子信息Table1 The information of typical plant LTR retrotransposon
1.4 PHRE8的表达量检测
1.4.1 毛竹基因RNA的提取 在低温条件下,取各处理实生苗叶片,采用Trizol 法[23]提取RNA,利用PrimeScriptTM 1st Strand cDNA Synthesis Kit 试剂盒[TaKaRa(日本)公司]反转录成cDNA,-20℃保存备用。
1.4.2 实时荧光定量PCR 分析 对PHRE8的POL基因部分的RT、RH、INT保守区域段设计引物(表2)。以毛竹PheACT2-1[24]作为内参基因,对在不同逆境胁迫下PHRE8的RT、RH和INT3个编码域进行实时荧光定量PCR 分析。荧光定量PCR 反应体系(10 μL):SYBR® Premix Ex TaqTMⅡ5 μL,cDNA 0.8 μL,上下游引物各0.4 μL,无菌水3.4 μL。反应程序:95℃预变性7 min;95℃变性10 s,58℃退火10 s,72℃延伸15 s,30个循环。采用2-△△ct法[25]计算3个结构域在不用处理下的基因相对表达量。
2 结果与分析
2.1 PHRE8的序列分析
2.1.1PHRE8 及其拷贝的结构PHRE8 全长5 297 bp,包括两端的LTRs区域和1个较长的开放阅读框(open reading frame,ORF)。右端LTR 长度为388 bp,左端LTR 长度为394 bp,结构顺序依次为5′-GAG-PR-RT-RH-INT-3′,属于Ty3-copia 家族成员。编码区含有4 244 bp的ORF,共编码1 414个氨基酸。1 450~1 558 bp为GAG 核心区,1 738~1 969 bp为PR核心区,2 286~2 955 bp为RT 核心区,3 099~3 444 bp为RH 核心区,3 681~4 590 bp为INT 核心区。PBS 序列为5′-AATCTGGTATCAGAGCTAGATTCGG-3′,位于5′端LTR 下游。PPT的序列为5′-ACCGTACAGG TATCCTCAGTTGCAA-3′,位于3′端LTR上游。利用cd-hit 软件鉴定出PHRE8的其他5个拷贝,将各个拷贝进行命名(表3)。LTRdigestion 鉴定5个拷贝均具有完整的结构(图1)。
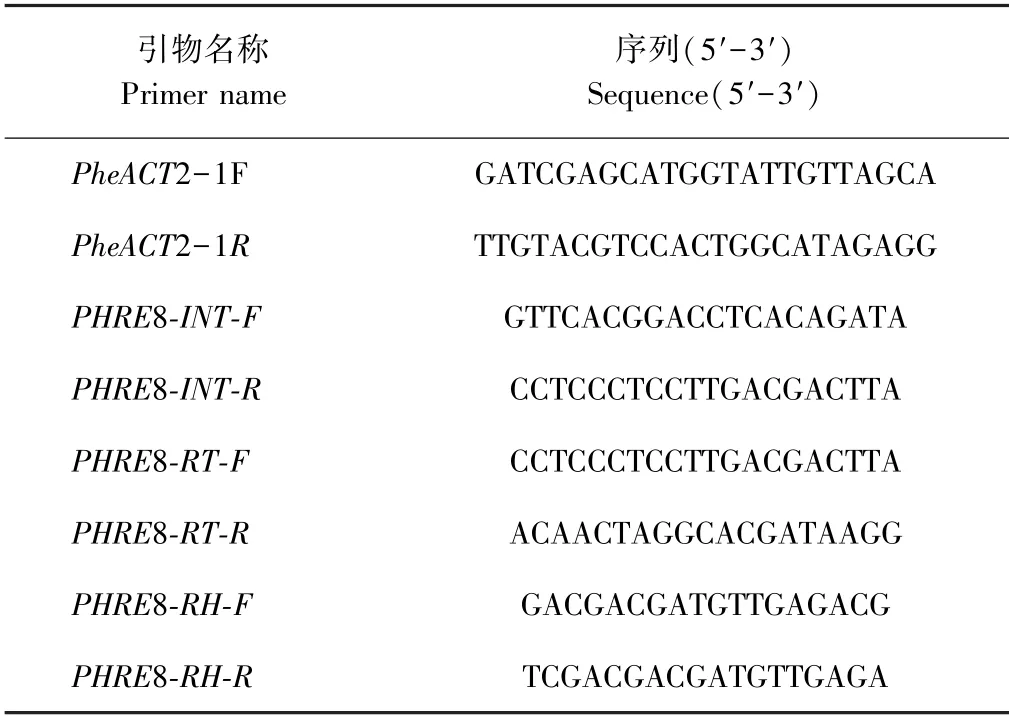
表2 引物序列Table2 The sequences of primers

表3 PHRE8 及其拷贝的命名Table3 Naming of PHRE8 and its copies
1.2.2PHRE8 及其拷贝的插入时间对比 比对PHRE8 两端LTR 序列的同源性得到分化度K为0.032。利用相同方法得到PHRE8 其他5个拷贝的分化度K 依次为0.039(PHRE8-5)、0.038(PHRE8-4)、0.016(PHRE8 - 3)、0.047 (PHRE8 - 2)和0.051(PHRE8-1)。通过式(1)计算PHRE8的插入时间为123.07万年前,PHRE8 其他5个拷贝插入时间依次为150.00万年前(PHRE8-5)、146.15万年前(PHRE8-4)、146.15万年前(PHRE8 - 3)、180.77万年前(PHRE8-2)和196.15万年前(PHRE8-1)。比对其他拷贝PHRE8的插入时间较晚,因此PHRE8 理论上存在转录活性的可能性更高(图2)。
1.2.3PHRE8的顺式作用元件分析 通过在线软件PlantCARE 分析了LTR 部分所包含的顺式作用元件(图3)。LTR 部分富含顺式作用元件,如启动子和增强子区域中常见的顺式作用元件CAAT-box,光响应顺式作用元件GA-motif,光响应顺式作用元件TCCC-motif 等。

图1 PHRE8 及其拷贝的结构Fig.1 Structure of PHRE8 and its copies

图2 PHRE8 及其拷贝的插入时间Fig.2 Insertion time of PHRE8 and its copies

图3 PHRE8 LTR 序列的顺式作用元件分析Fig.3 The analysis of cis-regulatory motifs in LTR sequences of PHRE8
1.2.4PHRE8 进化关系分析为分析PHRE8 与其他转座子之间的相似性及进化关系,利用其他已知典型的LTR反转录转座子的RT 区域氨基酸序列(表1)与PHRE8的RT 区域氨基酸序列进行比对,构建进化树。结果表明,转座子PHRE8 属于Reina分支,其与Gloin的同源性最高(图4)。

图4 PHRE8的进化关系分析Fig.4 Analysis of the evolutionary relationship of PHRE8
2.2 PHRE8 在不同胁迫处理下3个结构域的表达模式
2.2.1 DNA 甲基化抑制剂处理对结构域表达量的影响 由图5可知,INT、RH和RT3个结构域的表达量相对于野生型实生苗均显著提升。RH与INT结构域相对表达量在各浓度梯度均低于RT结构域,且随着DNA 甲基化抑制剂浓度的增加,RH和INT处理组间相对表达量均差异不显著。随着DNA 甲基化抑制剂浓度的增加,RT结构域相对表达量增幅较大,在250 μmol·L-1浓度下的相对表达量显著高于其他两个浓度。

图5 DNA 甲基化抑制剂处理下PHRE8结构域的表达水平Fig.5 Expression level of domains of PHRE8 under DNA methylation inhibitor treatments
2.2.2 不同辐射处理对结构域表达量的影响 由图6可知,与野生实生苗相比,不同辐射处理各个结构域的相对表达量均有所提升。其中INT和RT结构域的相对表达量均在30 Gy处理下达到最大值,继续增加辐射剂量其相对表达量呈下降趋势,尤其在70 Gy处理下,相对表达量与野生实生苗均无显著差异。RH结构域的相对表达量随着辐射剂量的增加呈缓慢上升的趋势,但各辐射剂量下无显著差异。

图6 辐射处理下PHRE8 结构域的表达水平Fig.6 Expression levels of domains of PHRE8 under irradiation treatments
2.2.3 低温和高温胁迫处理对结构域表达量的影响 由图7可知,在4℃低温与42℃高温胁迫处理下,3个结构域相对表达量均显著高于野生实生苗。在INT与RT结构域中,42℃高温胁迫处理下的相对表达量显著高于4℃低温胁迫处理。INT结构域相对表达量在4℃低温与42℃高温胁迫处理下均高于RH和RT结构域。此外,4℃低温和42℃高温处理下RH结构域的相对表达量均最低,且处理间无显著差异。
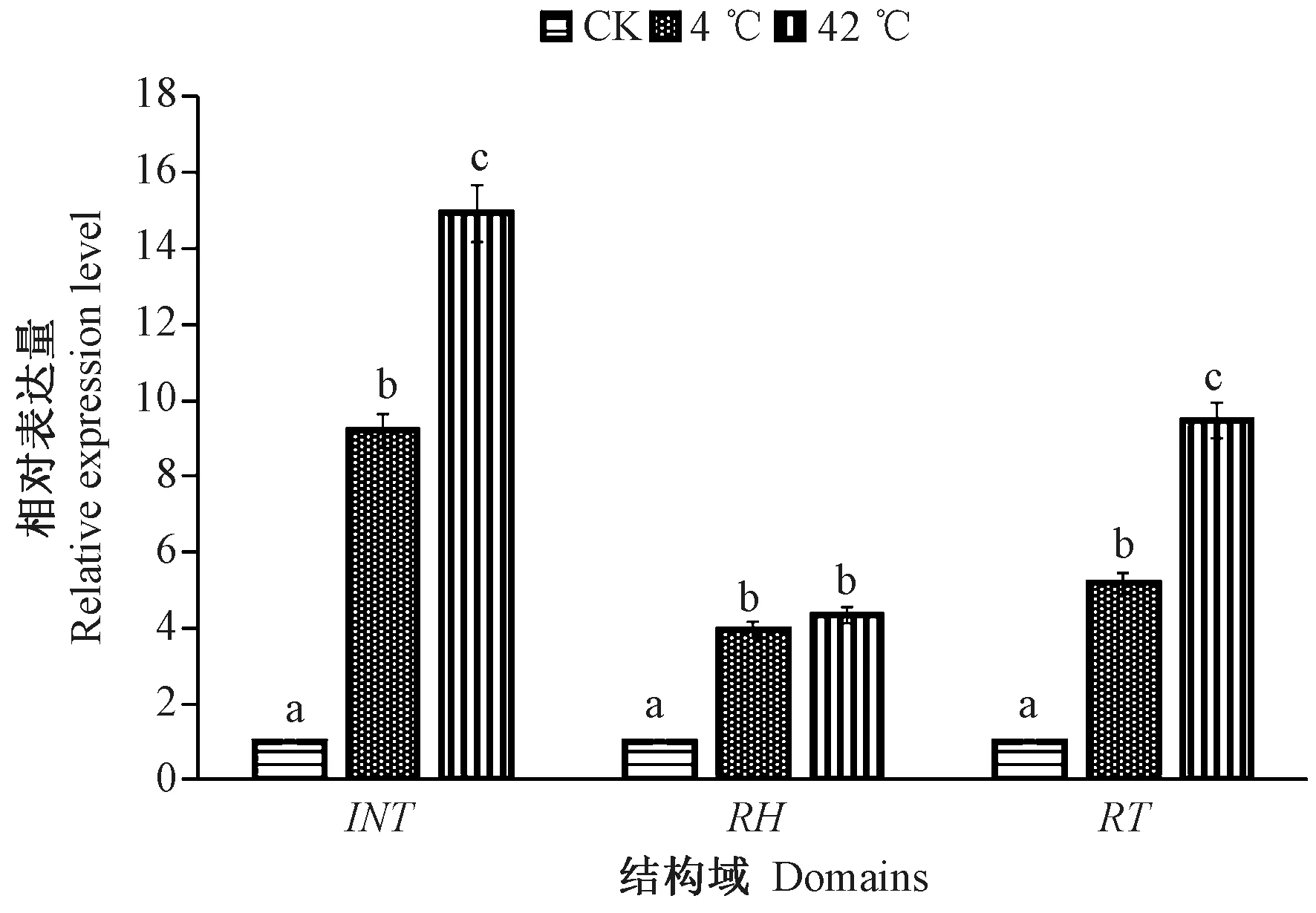
图7 高温和低温处理下PHRE8 结构域的表达水平Fig.7 Expression levels of domains of PHRE8 under high and low temperature treatments
1.2.4 高盐胁迫处理对结构域表达量的影响 由图8可知,在高盐处理下,3个结构域的相对表达量与野生型实生苗相比都有显著提升。INT结构域相对表达量随着NaCl 浓度的增加呈先增加后降低的趋势,在0.2 mol·L-1NaCl处理下达到最大值。RH结构域的相对表达量的变化趋势与INT结构域基本一致,在0.1 mol·L-1NaCl处理下,相对表达量达到最大值,之后随着浓度增加,其相对表达量降低,但处理组间差异不显著。RT结构域相对表达量在0.1、0.2 mol·L-1NaCl处理下均显著高于0.3 mol·L-1NaCl处理。

图8 高盐处理下PHRE8 结构域的表达水平Fig.8 Expression levels of domains of PHRE8 under high salt treatment
3 讨论
PHRE8是一个拥有完整结构的LTR反转录转座子,包括长末端重复序列、GAG 蛋白核心区、PR 蛋白酶核心区、RT 核心区、RH 核心区、INT 结构域、PBS 多嘌呤序列与PPT 引物结合位点[1],根据POL基因中PR、RT、RH、INT核心区的位置顺序,推测PHRE8 属于Ty3-gypsy 家族成员。PHRE8的插入时间为123.07万年,与插入时间为700万年的活性Tos17 反转录转座子相比,PHRE8是一个相对年轻的转座子,考虑到该转座子及其拷贝的结构均完整,PHRE8 很可能是一个具有潜在活性的反转录转座子。
本研究发现PHRE8的3个结构域(INT、RH和RT)在DNA 甲基化抑制剂、辐射、高盐、低温和高温处理后,其核心结构域呈现出不同情况的表达量变化,说明PHRE8是一个具有转录活性的LTR反转录转座子,在受到胁迫时通过改变其转录水平适应环境改变。
研究表明,在一些非生物胁迫下如高温、低温、高盐、重金属等,植物能够通过复杂的基因调控机制适应各种环境变化,如引起DNA 甲基化水平的变化[26]、激活转座子[27]等。激光辐射、高压诱导、远缘杂交等均可导致水稻中较常见转座子mPing的激活[28-30]。在植物中,DNA 甲基化作为一种修饰调控手段,可直接干扰转录因子与启动子识别序列结合,也可形成特异的转录抑制物调控基因表达,还可通过改变染色质的结构抑制基因的转录表达,在基因表达调控中发挥重要的作用[31]。毛竹基因组中非编码基因存在着多个具备转座能力的转座子,DNA 甲基化会影响转座子的活性表达。如庄婷婷[32]采用NO处理水稻基因组DNA,发现其能够定向诱导水稻基因发生一定程度DNA 去甲基化变异,同时有一些转座子的转座活性被激活,说明转座子的激活与DNA 去甲基化有一定的联系。本研究采用不同浓度的甲基化抑制剂对实生苗进行处理,结果发现植物发生了不同程度的DNA 去甲基化变异,PHRE8的3个结构域的相对表达量均增加,因此降低DNA 甲基化程度可能会激发PHRE8的潜在转座活性。
王静子等[33]利用γ射线在分子水平上辐射水稻基因组,结果表明其可诱导全基因组低DNA 甲基化,还能激活转座子mPing及其转座伙伴Pong[34]。本研究中,与野生实苗相比,毛竹经30、50、70 Gy 辐射处理后PHRE8 相对表达量均增加,说明PHRE8的转座活性被激发。其原因可能是由于辐射处理引起了PHRE8 DNA 甲基化变异,导致其相对表达量升高。随着辐射剂量的增加,PHRE8 相对表达量有所下降,其原因可能是过强的辐射导致植物细胞结构破坏或物化活性损伤,影响了植物的胁迫反应机制[35]。
温度是调控植物生长发育和分布最重要的因素之一[36-37]。本研究中,毛竹实生苗在高温42℃与低温4℃处理下,PHRE8 中3个结构域的相对表达量均有所升高,且PHRE8 在高温处理下相对表达量更高,说明毛竹实生苗对高温更敏感。当植物受到高温、重金属等刺激时,可诱导调节应激反应,保护机体防止细胞损伤的热激蛋白质的产生[38]。PHRE8 中含有与温度有关的顺式作用元件,且毛竹中也含有热激蛋白HSP20、HSP70、HSP90基因[39],猜测在极端温度下,PHRE8的激活会影响毛竹热激蛋白的合成与运输,是植物极端温度应答的一部分。
王萌[40]在小麦盐胁迫应答机制的研究中发现,参与盐胁迫下的应答基因,受到了DNA 甲基化的修饰调控。也有研究表明,在盐胁迫条件下,转座子会发生转座插入,影响邻近基因的表达[41]。本研究中,毛竹实生苗经0.1、0.2、0.3 mol·L-1NaCl处理后,PHRE8 中3个结构域的相对表达量均有所上升,说明该转座子参与了盐胁迫的应答机制,猜测PHRE8是通过插入影响相邻的盐胁迫应答基因,改变基因表达量以抵御高盐环境胁迫。面对逆境胁迫时,植物基因会发生一些复杂的变化来提高植物在胁迫下的耐受能力。大量研究表明,DNA 甲基化的修饰与调控作用在整个应答机制中占据了主导地位[42-43]。但逆境胁迫下植物LTR反转录转座子转录活性的变化与DNA 甲基化程度改变的关系和相关调控机制还需进一步更深入的研究。
4 结论
本研究结果表明,毛竹PHRE8是一个结构域完整、插入时间较晚、富含顺式作用元件的LTR反转录转座子,属于Ty3-gypsy 超家族;PHRE8 属于Reina分支,其与Gloin的同源性最高;PHRE8 在DNA 甲基化抑制剂(5-氮杂胞苷)、辐射、高温、低温、高盐胁迫处理下,其3个结构域的相对表达量都有不用程度的增加,说明PHRE8 有可能参与了毛竹在逆境胁迫下的应答机制。该试验结果为研究毛竹基因组中转座子功能奠定了一定的理论基础。
