基于群智能算法分类模型的番茄病害识别
2020-03-12阎园园
阎园园, 陈 华, 姜 波
(新疆大学电气工程学院,新疆乌鲁木齐 830047)
针对作物病害的图像处理技术研究及科技农业发展给农业生产带来的巨大帮助。在1989年穗波信雄已经提出,图像分析技术与模式识别技术将成为诊断作物病害的研究发展趋势[1-2]。作物病害在颜色、形状、大小等一些外在特征上存在差异性,通过视觉可以直观地获取这些差异。如今在作物病害类型辨别方面,分类识别技术的研究已经取得不错的成果。基于分类器的识别技术主要包括主成分分析(principal component analysis,简称PCA)、BP(back propogation)统计分类模型识别、粒子群优化(particle swarm optimization,简称PSO)算法等。田有文等提取玉米和葡萄叶片的色度矩特征信息,选择不同核函数对支持向量机(support vector machine,简称SVM)模型进行训练,结果表明,当样本数量在几十之间,径向基函数(radial basis function,简称RBF)下的SVM分类器对病害分类效率较高,样本集数量较大或作物种类发生变化时,该方法实用性降低[3-4];张建华等用RBF-SVM模型对棉花病害进行识别,将小波变换图像在HIS(hue-saturation-intensity)颜色空间的特征值作为分类器的输入,结果证明,RBF-SVM分类器的分类率达88%,为分类器最优核函数的选择提供了参考[5];房俊龙等用BP算法识别番茄正常果实与变异果实,遗传算法(genetil algorithm,简称GA)算法优化BP网络结构,根据番茄果实病害图像中的果实形状、圆度值、色度等之间的差异,实现了对番茄空洞果实、变形果实的识别[6]。
本研究以番茄常见病害类型如早疫病、晚疫病、灰霉病为识别对象,在对3种病害分类识别过程中将病害的特征值作为群智能分类器的输入信号。在样本有效特征提取时,一般选择作物颜色、形状、纹理3个方面的特征参数作为研究参数,以番茄早疫病为例,提取早疫病这3方面的有效特征值,并计算均值与方差,包括RGB(red-green-blue)、HSV(hue-saturation-value)空间颜色通道的均值与方差、7个形状不变矩、病斑周长、面积、形状复杂性、矩形度、对比度、相关性、能量、熵的均值与方差共31个特征参数,晚疫病和灰霉病的特征参数提取方法同早疫病,分别对3种病害的31个特征参数选择不同分类模型进行训练,检验不同分类器的分类效果。
1 材料与方法
1.1 试验材料
试验材料来自中粮新疆屯河吉木萨尔番茄产业种植基地,由新疆大学-中粮屯河产学研联合培养研究生示范基地提供。采用MATLAB Version 8.0(R2012b)对数据进行分析,图片处理工具选择(Image Processing Toolbox)Version 8.1(R2012b)以及IBM SPSS Statistics 25.0。
1.2 试验方法
1.2.1 PCA方法 PCA方法是一种用较少变量代替原样本中较多变量的线性变换方式,变换后的较少变量被称作新变量,新变量在一定程度上包含原变量较多信息量,又称降维。降维后的新变量之间保持线性互不相关,并且变量之间满足正交特性[7]。PCA方法的新变量自动按照Var(Fm)由大到小从高维空间映射。假设样本有p个特征,分别为X1,X2,…,Xp,将p个特征的综合反映记作Fm,F1代表原变量第1个主成分综合指标,即F1=a11X1+a21X2+…+ap1Xp,方差Var(Fm)的大小代表主成分包含信息量的多少。以此类推构造F1,F2,…,Fm为原变量指标下X1,X2,…,Xp第1、第2、…、第m个主成分表达如下:
F1=a11X1+a12X2+…+a1pXp;
F2=a21X1+a22X2+…+a2pXp;
⋮
Fm=am1X1+am2X2+…+ampXp。
(1)
对样本的31个特征参数值进行PCA降维,根据主成分贡献率的大小,通常选择主成分累积贡献率大于85%的成分作为样本数据集的研究依据,若贡献率太小则不足以显示样本信息,会对样本分类造成影响。以早疫病为例共采集30个样本,每个样本提取31个特征参数,其他病害成分分析及降维方法同早疫病,早疫病样本的PCA降维分析结果和主成分贡献率分别如图1、表1所示。

由图1可知,成分1~7的特征值变化较快,第8个成分之后变化平缓,7个成分的累积贡献率达到86.077%,所以把前7个成分作为样本的主成分,成分因子不受影响。通过不断改变样本数量分析发现,对样本特征参数以及主成分数进行PCA降维不会导致成分因子变化,以7个主成分中的31个成分因子作为SVM算法的输入,建立PCA-SVM的番茄病害分类模型。
1.2.2 PCA-SVM分类模型参数分析 SVM是一种分类建模的方法,在样本容量小、数据非线性、数据纬度较高的特征空间被经常用到,算法的关键是核函数的选择,核函数在高维特征数据空间获得分类函数相对比较容易,本研究选择核函数RBF作为分类函数,公式如下[8]:

表1 番茄早疫病样本数据7个主成分的累计贡献率
(2)
核函数RBF中σ2(核函数参数)与γ(惩罚系数)参数的选择最重要,最终参数的确定要经过不断调试。核函数参数σ与γ的取值范围通常在10-3~105之间[9]。在PCA-SVM分类模型中,以30个早疫病样本为训练样本,对优化后的样本特征向量进行训练,另外建立30个早疫病测试样本,样本分类训练模型结果见图2,可以看出60个样本在基于核函数RBF的PCA-SVM分类器下的分类效果,早疫病与非早疫病分类中有1个样本被错分,早疫病的识别率达到96.7%。
对测试样本识别时考虑到样本集数量可能会对识别率造成影响,试验中不断改变病害样本集数量,比较不同数量样本集下PCA-SVM模型的识别率,进而判断模型是否具有可行性,不同数量样本集下PCA-SVM模型对3种病害类型的识别结果如表2所示。
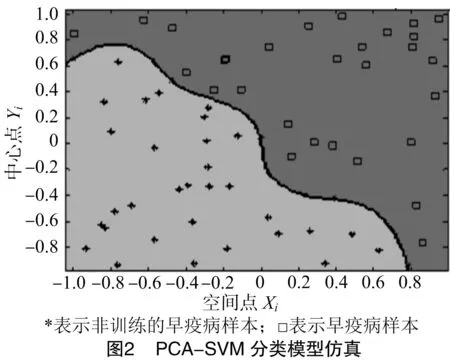

表2 PCA-SVM模型下的早疫病及番茄其他病害识别结果
1.2.3 PCA-SVM分类模型分类结果分析 表2显示,样本数为30~50个时,PCA-SVM模型对测试集的病害识别率全部达到90%及以上,尤其当早疫病测试集样本数为20个时,识别率达到100%,随着样本量的增加,早疫病识别效率相应降低,早疫病训练样本量为60个时,测试集的识别率仅为88%,结果表明,识别率大小受样本集大小影响,也证实PCA方法对样本数量有一定要求,在灰霉病训练样本数为60个时,测试集的识别率仅为80%,这种情况下要寻找新的分类模型解决大样本下的识别率问题。
1.3 遗传算法
遗传算法以种群为基准,通过计算隐藏在种群中的解集来描述实质性的问题,每个个体的染色体携带特征不同,导致个体之间存在差异,多个个体之间的差异性组成一个个体种群,解集计算就是对种群适应度、交叉概率(Pc)、变异概率(Pm)等参数的选择,通常在算法执行过程中要对个体进行编码产生新个体,通过不同方式的交叉、变异产生不同的Pc、Pm,选择子代中适应度最大的个体替代父代中适应度最小的个体,称作迭代寻优[10-11]。目前GA参数取值有一定的范围,种群大小在20~100个之间,Pc为0.40~0.99,Pm为0.001~0.100。分类器的输入同样是样本数据的特征参数,但分类模型中的输入信号为原始特征参数,不用对数据降维,这是GA-SVM模型与PCA-SVM模型的不同之处,这既保留了原始信息,又能够自动定参。
1.3.1 GA-SVM分类模型的参数分析
1.3.1.1 种群大小(M) 根据试验要求及样本对结果的影响程度决定初始种群大小(M),数目较大时,隐藏解的信息量相对较大,样本集的全局最优解相对更容易获得,运行时间要比小种群运行时间长。
1.3.1.2 交叉概率(Pc)Pc是个体之间交叉获得新个体的概率,其中个体自身作为父代,新个体称作子代,交叉方式有单点、多点、两点、洗牌等。Pc高低决定算法收敛速度,一般选择较大的Pc,本研究选择单点交叉方式,该方法简单计算量小[12]。
1.3.1.3 变异概率 新个体的产生在交叉过程中出现变异个体,这种个体的产生导致样本多样性,所以Pm大小决定样本的稳定性,通常选择Pm最小数值来减少对种群大小和Pc的影响。
1.3.1.4 进化代数 进化代数(Gm)反映样本寻找最优解的次数,Gm越大迭代时间越长,最优解获得时间也相对较长,Gm通常在100~500代范围内[13]。
为更快获得参数之间的关系来提高寻优效率,本研究引入常见的寻优函数De Jong函数,由De Jong函数确定参数之间的最优组合,对De Jong函数参数确定选用旋转正交变换法,该方法可以提高参数搜索速度。分类模型算法流程如下[13]:首先编码SVM中r与σ2;对样本数据即种群进行初始化,让样本随机产生1组个体;输入种群个体参数值作为SVM的输入参数,数据样本集对模型进行训练;通过计算样本数据集正确分类数与样本集总量的比值获得适应度;根据适应度大小用轮盘赌法对样本子代个体进行选择;重复上述操作完成GA的训练。GA-SVM分类模型在不同参数下分类结果如表3所示。

表3 GA-SVM分类器在不同参数指标下的分类效果
1.3.2 GA-SVM分类器分类结果分析 由表3可知,测试组第5组在GA-SVM分类器中的分类效果最好,3种病害识别率平均值达到96.67%,此时M=40,样本数=40,Pc=0.6,pm=0.001,早疫病识别率达到100%,灰霉病识别率 97.5%,晚疫病识别率为92.5%;当M=80,样本量=60,Pc=0.8,Pm=0.01时,分类器平均识别率最低,早疫病仅识别出52个,总体上讲Pm越小,样本量在30~60个之间识别率越高。就样本量而言,GA-SVM模型比PCA-SVM模型对样本量识别范围大。
1.4 粒子群算法
为考察遗传算法中的交叉变异计算是否会对分类器参数产生影响,选择经PSO算法优化的参数作为分类器的输入来验证交叉变异的影响。PSO算法摆脱了智能算法中的交叉、变异、进化等算子操作,将种群个体看作一个粒子,每个粒子都有对应的适应度值,用粒子每一速度下的个体最佳极值和整个群体的最佳极值进行比较,或选择种群中一部分粒子的最佳极值进行比较。当粒子适应度值大于全局极值时,用适应度值代替全局极值,在优化过程中粒子速度、位置不断变化,但都是朝着粒子最优解和全局最优解的位置搜索。粒子群算法在寻优过程中减少了交叉、变异计算,相应的运算时间较短。
1.4.1 PSO-SVM分类模型参数分析 PSO算法也会受到参数大小的影响,例如惯性权重(ω)、群体规模(m)、学习因子(c1、c2)、种群结束条件、最大速度(vmax)等。粒子群算法中有2个学习因子——认知学习因子和社会学习因子,学习因子能够加速算法收敛性以及提高粒子搜索能力,两者取值一般在 0~4 之间,通常学习因子与ω之间存在一定关系,c1参数取值一般为1.494、2.000、2.050、2.800;c2为1.300、1.494、2.000、2.050。ω在PSO算法中主要来决定粒子的局部搜索能力和全局搜索能力,Eberhart等对ω范围作出总结,在0.9~1.2之间时算法的优化效果和搜索能力显著提高。粒子群训练结束后,把种群中具有最大适应度函数值的r和σ2作为支持向量机模型的最优参数来完成模型搭建。
1.4.1.1 粒子初始化 假设种群大小用Z={Z1,Z2,…,Zm}表示,其中m为粒子数大小,粒子大小用来确定SVM算法中参数的可行解,r的取值在0~1 000 之间,σ2=[0,1]。
例如,在D维的目标空间,粒子数为N个的一个群体中,假设其中的一个粒子记作i,它的D维向量表示为Xi=(xi1,xi2,…,xiD),i=1,2,…,N,该粒子的速度也是一个D维的向量,记作Vi=(vi1,vi2,…,viD),i=1,2,…,N,粒子搜索到的最优位置也称作个体的极值,记作pbest=(pi1,pi2,…,piD),i=1,2,…,N,整个粒子群搜索到最优位置,即全局极值的表达式为gbest=(pg1,pg2,…,pgD),在找到这2个最优值的过程中,粒子会根据以下公式来时刻更新自己的速度和位置[15-16]:
vid=ω×vid+c1r1(pid-xid)+c2r2(pgd-xid);
(3)
xid(t+1)=xid(t)+vid。
(4)
式中:t表示时间。vid∈[-vmax,vmax],vmax是常数,根据需求设定,用来限制粒子的速度,r1和r2大小则是介于0~1之间的随机数。
1.4.1.2 适应度函数的选择 粒子群算法的适应度函数确定方式与GA适应度函数选择方式相同,都是测试样本中正确分类个数(ano)与测试样本总数(tno)的比值,记作f1=ano/tno。
1.4.1.3 粒子群更新 按照公式(3)、(4)找到个体极值与全局极值时,粒子会按照以上公式更新位置与速度,每1个粒子在寻优的过程中都会自动寻找2个极值。
1.4.2 PSO-SVM分类器在不同参数下的分类结果分析 PSO算法中粒子的位置和速度受参数影响较大,以ω、c1、c2、群体规模作为研究参数,用“1.3.1”节中的正交变换法对不同参数下的模型进行验证,寻找参数与分类模型识别率之间的关系,正交试验法中的参数选择4个,按照正交规则一共要产生42个试验组,从中选择最佳参数组合建立PSO-SVM分类模型,正交试验下PSO参数及 PSO-SVM 分类模型对病害分类结果如表4所示。由表4可以看出,测试组7中的平均识别率达到96.67%,样本量=40,种群规模=35,c1=2.05,c2=2.00;相对样本量较大的测试组12、16,学习因子对识别率的影响效果不明显,而样本量和种群规模对识别率的影响较大,相对于前2种分类模型PSO-SVM分类模型在样本量为30~40个,种群规模在25~35个之间时,平均识别率普遍在90%以上。说明参数对识别率的影响较大,要想获得更高的识别率要不断寻找最优参数。较大的c1、c2对识别率影响也较大,16组试验中2个因子较大的试验组平均识别率普遍偏高。
2 试验分析
为验证PCA-SVM分类模型的分类效果,增加对比试验,其中试验样本以及试验样本数据特征均来自PCA-SVM试验,2组对比试验模型分别为 GA-SVM 群智能分类模型、PSO-SVM群智能分类模型。根据3个分类器模型在不同参数下的分类效果,保留识别率大于90%的试验组作为评判分类模型性能的指标,对目标试验组数的认别率分别求平均值,综合比较3种分类模型分别对番茄早疫病、晚疫病、灰霉病识别效果,不同分类模型的分类试验分析结果如表5所示。

表4 正交试验下PSO参数及番茄病害分类结果

表5 不同分类模型试验分析结果
3 结论
综上所述,在3种分类模型中,总体上PSO-SVM分类模型的分类效果最好,但在PCA-SVM模型中,早疫病和晚疫病在3种分类模型中识别率最高,而该模型对灰霉病的识别效果相对略差一些;在PSO-SVM分类模型中,灰霉病的识别率在3种分类模型中最高,达到93.87%,从平均值来看,3种分类模型中PCA-SVM识别率最高,这也证明了作物识别模型不具普遍适用性的特点。
