基于土壤宏基因组的新颖木聚糖酶基因克隆及生物信息学分析
2020-03-07魏琦超王亚丽张琛赵加迪
魏琦超,王亚丽,张琛,赵加迪
(河南科技学院生命科技学院,河南新乡 453003)
木质纤维素(lignocellulose)主要包括纤维素(cellulose)、半纤维素(hemicellulose)和木质素(lignin),是自然界含量最丰富的生物质原料。其中,纤维素的丰度最高,约占其总量的30%~50%;半纤维素的丰度次之,约占其总量的15%~35%[1-2]。半纤维素的主链由葡萄糖(glucose)、甘露糖(mannose)和木糖(xylose)等β-D-吡喃糖(pyranose)残基通过β-1,4-糖苷键连接,可分为甘露聚糖(mannan)、木聚糖(xylan)和木葡聚糖(xyloglucans)3大类[3]。木聚糖由β-D-吡喃木糖通过β-1,4-糖苷键连接形成主链,是植物半纤维素的主要组分,约占植物干重的1/3[4]。
木聚糖酶(xylanase,EC.3.2.1.8)是一类能够将木聚糖降解成低聚木糖和少量木糖、阿拉伯糖(arabinose)的一组酶系的总称,在饲料、食品、化工等领域的用途非常广泛[5]。王佳玉等[6]的研究表明,在全麦粉中木聚糖酶的添加量达到0.03%,经过90min酶解反应,面筋结构可得到明显改善。班志彬等[7]在肉鸡的不同类型饲料中添加木聚糖酶,研究了对其生长性能的影响,发现木聚糖酶可提高平均日增重并降低料重比。王阳等[8]研究了木聚糖酶预处理对檀皮纤维漂白的影响,认为预处理可有效疏松纤维结构,提高后期处理过程中漂白剂的可及性,进而提高檀皮纤维白度。
虽已在真菌、细菌、酵母菌、海藻和植物种子、甲壳类动物、蜗牛等物种中发现了木聚糖酶[9-10],但普遍认为,真菌和细菌是该酶的主要来源[11]。利用基因工程异源表达木聚糖酶,可在一定程度上解决木聚糖酶原始来源微生物目标酶表达量低、纯化困难等酶生产和应用领域的限制性因素。木聚糖酶基因传统发掘方式为分离自然环境中的产酶微生物,经纯培养后克隆目标基因。然而,就土壤微生物而言,其可培养微生物仅占1%~10%[12-13],因此通过传统方式分离木聚糖酶产酶菌株、发掘新颖木聚糖酶基因较为困难。
宏基因组最初是指土壤细菌混合基因组[14]。Rondon 等[15]以 细菌人工染色体(bacterial artificial chromosome,BAC)载体构建了土壤宏基因组DNA文库,文库克隆的表型包括脂肪酶、淀粉酶、核酸酶等,该研究为基于土壤宏基因组发掘新颖工业、医药用酶基因奠定了基础。本研究拟以土壤宏基因组DNA为聚合酶链式反应(PCR)模板,使用CODEHOP方法[16]设计木聚糖酶基因核心序列的简并引物,采用hiTAILPCR技术[17]获悉其侧翼序列,并最终克隆非培养木聚糖酶基因全长序列,为有效利用土壤微生物资源,发掘新颖木聚糖酶基因奠定基础。
1 材料与方法
1.1 土壤样品及采样方法
土壤样品采自河南科技学院校园内常年潮湿的花坛土。除去地表植被和枯枝落叶,铲除表面5cm左右的表土,取样深度距土壤表面5~10cm,置于无菌采样袋后带回实验室。
1.2 生化及分子生物学试剂
土壤基因组DNA提取试剂盒购自天根生化科技(北京)有限公司,大肠杆菌(Escherichia coli)感受态细胞、微柱浓缩DNA凝胶回收试剂盒购自北京庄盟国际生物基因科技有限公司,2×Taq PCR MasterMix、DNA分子量标准品购自北京博迈德基因技术有限公司,pMD™19-T Vector Cloning Kit、PrimeSTAR®GXL DNA Polymerase 购 自 Takara Bio,pEASY®-Blunt Zero Cloning Kit购自北京全式金生物技术有限公司,琼脂糖、50×TAE缓冲液,购自北京索莱宝科技有限公司。
1.3 土壤宏基因组DNA的提取与质量检测
剔除土壤中的石砾和植物残根等杂物,按照土壤基因组DNA提取试剂盒说明书提取其DNA,经琼脂糖凝胶电泳检测判断所提取DNA样品的完整性。使用微生物16S rRNA通用引物进行PCR扩增,检验DNA样品是否满足PCR扩增的纯度要求。使用引物为 27F(5’AGAGTTTGATCCTGGCTCAG)/1525R(5’AAGGAGGTGATCCAGCC)[18]。反应程序为:95℃,5min;95℃,30s;55℃,30s;72℃ 45s,30个循环;72℃,5min。
1.4 木聚糖酶基因核心序列的克隆与分析
1.4.1 简并引物设计
在UniProt网站(https://www.uniprot.org/)以检索词“xylanase”检索并下载登录号为Q8GJ44、Q9HFH0、B3A0S5等的多条已公开木聚糖酶氨基酸序列,使用CODEHOP设计基因核心区简并引物[19]。Xyn-GH10(F):CTACGACTGGGAYGTNIBSAAYGA;Xyn-GH10(R):GTGACTCTGGAWRCCIABNCCRT。下划线为核心序列的简并区域,Y=C/T、N=A/T/G/C、I为次黄嘌呤、B=C/G/T、S=C/G、W=A/T、R=A/G。
1.4.2 核心序列扩增
以经27F/1525R检测正常的土壤宏基因组DNA为模板,使用Xyn-GH10(F)/Xyn-GH10(R)进行PCR扩增。鉴于核心序列扩增用引物为简并引物,反应程序采用降落PCR的方式进行,即以66℃为首个循环的退火温度;前15个循环,每循环的退火温度降低1℃;以51℃为退火温度循环25轮。
1.4.3 核心序列克隆与分析
将PCR产物进行琼脂糖凝胶电泳和切胶回收,胶回收产物与pMD™19-T克隆载体连接,连接产物转化DH5α感受态细胞,随机挑取单克隆转化子经PCR鉴定后送测序。测序结果通过以下流程进行分析:(1)测序结果去除简并引物序列;(2)多条测序结果使用Vector NTI的Alignment子程序进行序列比对,如发现一致率为100%的序列,则仅保留一条序列用于后续分析;(3)序列提交至NCBI,使用blastx程序进行同源序列检索;(4)综合考虑检索到同源序列的注释信息及序列一致率,筛选相关核心序列进行侧翼序列的扩增。
1.5 候选核心区侧翼序列的克隆
如表1所示,根据候选基因核心区测序结果,按照hiTAIL-PCR特异性引物设计规则,设计3条特异性上游引物和3条特异性下游引物,分别与hiTAILPCR通用引物配合使用,扩增核心区3’端和5’端侧翼序列。
1.6 木聚糖酶基因编码区全长序列的克隆
将前期克隆的木聚糖酶基因核心区及其5’端、3’端侧翼序列进行拼接,根据拼接序列,设计2对引物(表2),以前期相应的土壤宏基因组DNA为模板,通过巢式PCR扩增木聚糖酶基因编码区全长序列。

表1 侧翼序列扩增用引物
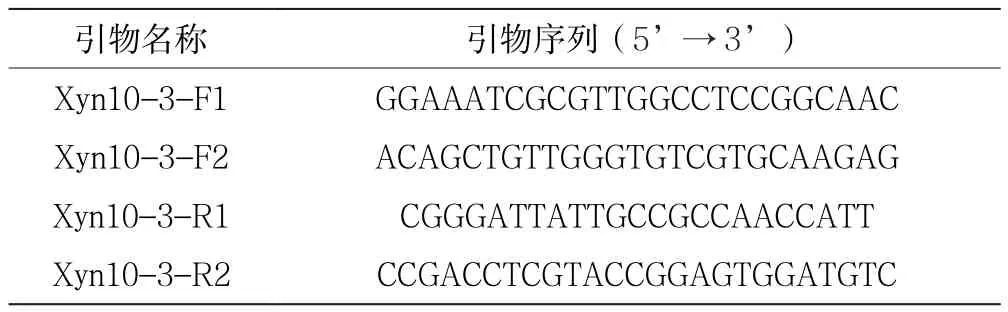
表2 编码区全长序列扩增用引物
1.7 木聚糖酶基因编码区序列的生物信息学分析
克隆序列经DNAMAN软件翻译为氨基酸序列后,使用各工具网站(表3)进行同源序列的比对和理化性质、蛋白结构等的分析。

表3 生物信息学分析用工具网站
2 结果与分析
2.1 土壤宏基因组DNA的质量检测
土壤中因富含腐殖酸、多酚等杂质,其高质量微生物总DNA的提取颇有困难困难较大。本研究使用试剂盒法提取总DNA。所提样品经琼脂糖凝胶电泳检测,结果如图1所示。发现其条带单一,泳道内无弥散、降解现象,初步认为可满足PCR扩增对模板质量的要求。为了进一步验证模板质量,以微生物16S rRNA通用引物27F/1525R扩增,PCR产物经电泳检测,结果如图2所示。可以看到扩增产物条带清晰、符合预期大小,因此所提取DNA可满足后续实验要求。

图1 土壤宏基因组DNA的电泳检测
2.2 木聚糖酶基因核心序列的PCR扩增
目标基因编码区核心序列的扩增,是获得其全长的必要条件。本研究使用CODEHOP方式设计的简并引物、通过降落PCR的方式开展上述工作。PCR产物经琼脂糖凝胶电泳检测,获得长度约为250bp(base pairs,碱基对)的电泳条带(图3),认为其符合CODEHOP简并引物设计的预期大小。PCR产物经切胶回收后,连入T载体,随机挑取15个单克隆经PCR检测后送武汉金开瑞生物工程有限公司进行序列测定工作。

图2 16S rRNAPCR 检测

图3 木聚糖酶基因核心序列PCR产物的电泳检测
2.3 木聚糖酶基因核心序列的新颖性分析
将核心序列测序结果去除简并引物及其外侧序列,使用blastx程序进行同源序列检索,发现有3条序列与非全长的未培养微生物GH10家族(glycosyl hydrolase family 10)木聚糖酶氨基酸序列具有较高的一致率,认为其具有一定的新颖性。编号为Xyn10-3的核心序列,其一致率最高(70.59%)的序列为未培养微生物来源木聚糖酶(ACR24800),将其作为目标序列,开展编码区全长序列的克隆工作。
2.4 Xyn10-3编码区全长序列的克隆
根据Xyn10-3核心序列设计特异性引物,采用hiTAIL-PCR方式克隆其5'端和3'端侧翼序列。根据获悉的侧翼序列信息,设计巢式PCR引物进行编码区全长序列的克隆。经过两轮PCR,克隆到Xyn10-3 的完整开放阅读框(open reading frame,ORF),全长为 1 284bp(表4)。
2.5 Xyn10-3编码区序列的生物信息学分析
将Xyn10-3编码区核苷酸序列进行blastx检索,其编码的氨基酸序列与海洋放线菌(Actinoalloteichuscyanogriseus)β-1,4-内 切 木 聚糖酶的一致率最高,且仅为51.43%,表明所克隆的Xyn10-3编码区为未知的新序列。NCBI的结构域预测结果(图4)显示:Xyn10-3的295-1272位氨基酸为β-1,4-内切木聚糖酶结构域,E值(E-value)为8.22E-61,认为其预测可靠。
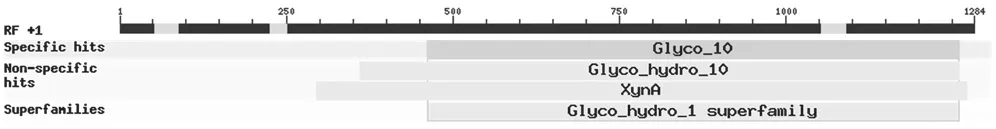
图4 Xyn10-3的结构域预测
将Xyn10-3编码区核苷酸序列翻译为氨基酸序列,发现其共有427aa,分子量为47398.91Da,理论等电点为8.99。各氨基酸位点的亲疏水系数如图5所示,平均亲水系数(GRAVY)为-0.287,可判断为亲水蛋白。蛋白质跨膜区域分析结果(图6)表明,该蛋白可能不存在跨膜区域。
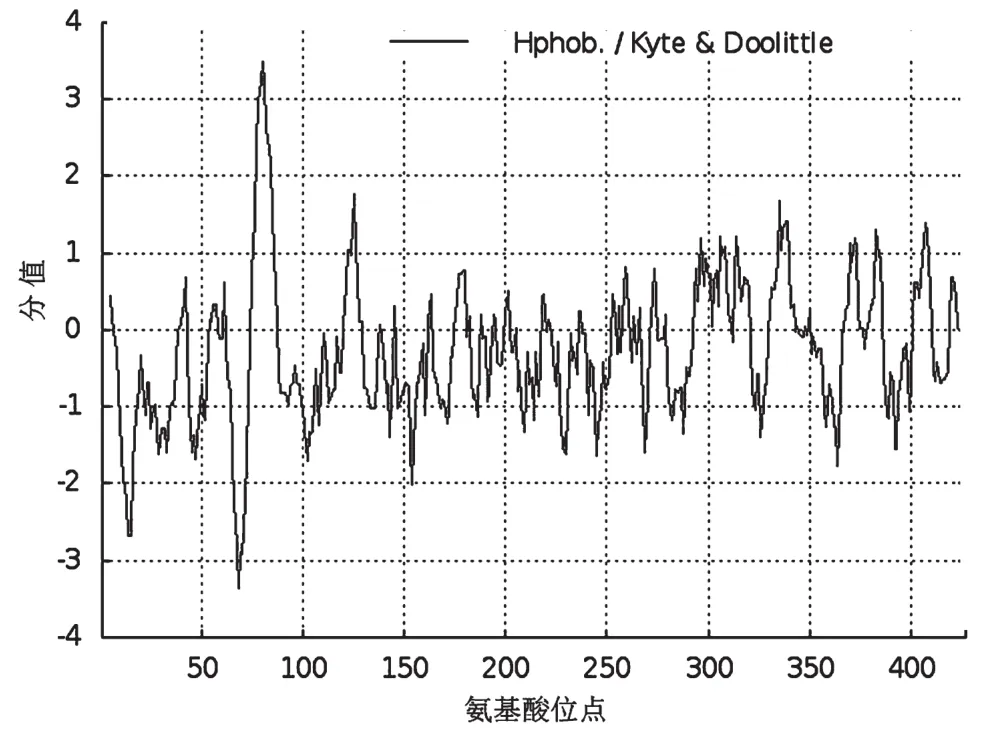
图5 氨基酸疏水性分析

图6 蛋白质跨膜区域分析

表4 Xyn10-3编码区核苷酸序列信息
3 结论
传统的新酶发掘技术方案,通常以目标性状微生物的分离、筛选为起始环节。为了充分利用土壤未培养微生物蕴藏的海量基因资源,本研究尝试使用CODEHOP方法设计的简并引物先行克隆目标酶基因的编码区核心序列,继而使用hiTAIL-PCR方式获悉其旁侧DNA序列信息,最终通过巢式PCR的方式克隆全长的编码区DNA序列。所克隆Xyn10-3经生物信息学分析,认为其是一个未报道的木聚糖酶全长基因。后续将构建pGAPZαA-Xyn10-3重组质粒,并进行毕赤酵母(Pichia pastoris)工程菌株的构建和目标蛋白的表达、纯化等工作,以进一步检测其酶活。
