趋化因子受体4与胃癌患者预后的相关性及潜在机制探索
2020-02-27张椿钰侯世科
张椿钰,侯世科,吴 超, 程 明
胃癌是全球范围内常见的恶性肿瘤之一,在所有恶性肿瘤中,其发病率排在第5位,病死率居第3位[1]。因此,进一步寻找能提示其预后的指标并探索胃癌发生发展的相关分子机制,对早期制定治疗策略并提高疗效具有重要意义[2]。研究表明,趋化因子受体4(chemokine receptor 4, CXCR4)相关通路能够激活肿瘤干细胞,可促进胃癌细胞的增殖,并通过激活肿瘤细胞逃逸宿主免疫机制以抑制肿瘤细胞凋亡[3]。但多数临床研究仅限于肿瘤组织中CXCR4表达高低与基础临床数据的相关性分析,长期随访数据鲜有报道。本研究拟通过利用公开的癌症基因组图谱(the cancer genome atlas, TCGA)数据库和生物信息学方法,分析CXCR4对胃癌患者肿瘤分期及远期生存率的影响,并探讨潜在的相关机制,从而为胃癌的预后判断和治疗方案制定提供一定的参考。现报告如下。
1 资料和方法
1.1一般资料及生存曲线比较 从包含TCGA数据网站(http://www.linkedomics.org)下载胃癌患者数据集TCGA_STAD RNASeq和TCGA_STAD Clinical,筛选出同时包含生存时间数据信息和CXCR4基因表达数据的患者共375例,比较CXCR4基因表达水平与年龄、种族、放疗状况的相关性。按照CXCR4表达水平由高到低排序,将排在前50%的患者设为高表达组(188例),其余患者设为低表达组(187例),绘制2组患者的生存曲线。
1.2不同肿瘤分期的CXCR4比较 根据肿瘤的病理学分期及TNM分期,将患者分别归入相应的各组,比较不同分期CXCR4的基因相对表达水平。
1.3CXCR4相关基因筛选 利用Omisc软件,将检测的所有基因表达水平逐一与CXCR4表达水平进行Pearson相关性检验,根据相关系数,选取出与CXCR4表达水平相关性最高的50个基因,并绘制基因热图[4]。
1.4富集分析 将筛选出的50个CXCR4相关基因导入DAVID在线数据库(https://david.ncifcrf.gov/)进行基因本体(gene ontology, GO)富集分析和京都基因与基因组百科全书通路(the kyoto encyclopedia of genes and genomes pathway, KEGG通路)富集分析。
1.5蛋白相互作用网络分析 将筛选出的差异基因导入String在线数据库(http://string-db.org/),获得相关基因所编码的蛋白质之间的相互作用,并进行聚类分析,从而找出处于网络核心的关键蛋白。

2 结果
2.1CXCR4基因表达水平与年龄、种族及放疗状况的相关性 CXCR4表达水平与胃癌患者年龄呈显著相关性(P<0.01);放疗状况及不同种族间胃癌患者的CXCR4表达水平比较差异无统计学意义(P>0.05)。见图1。
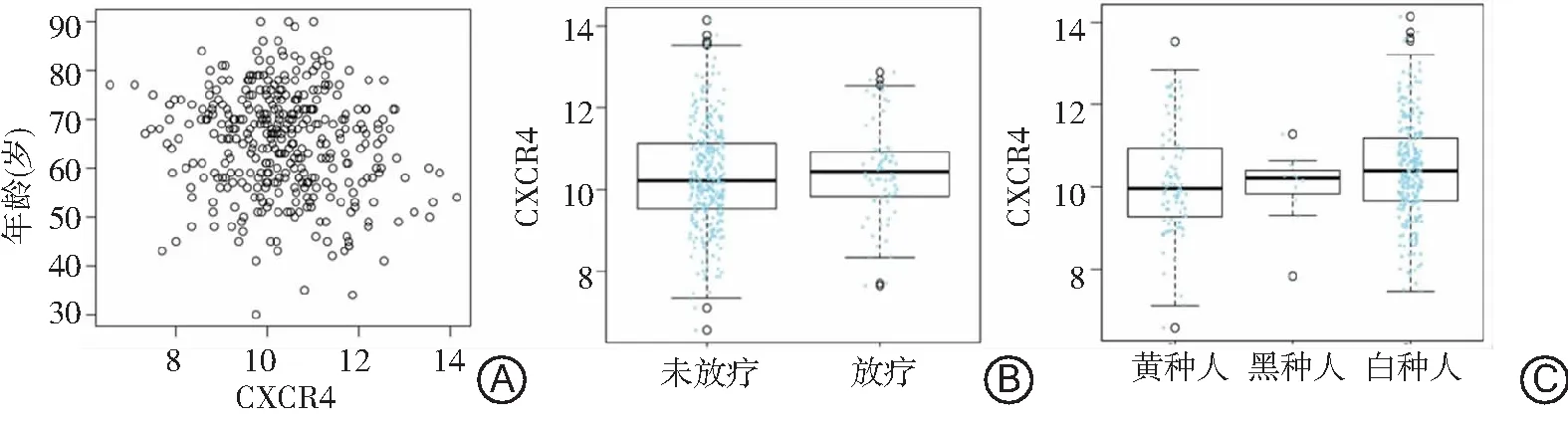
图1 CXCR4基因表达水平与胃癌患者年龄、放疗状况及种族的相关性
A.CXCR4基因表达水平与年龄相关性;B.放疗状况对CXCR4基因表达水平的影响;C.不同种族CXCR4基因表达水平情况;CXCR4为趋化因子受体4
2.2CXCR4表达差异对生存时间的影响 胃癌患者CXCR4高表达组的中位生存时间低于CXCR4低表达组(P<0.01)。见图2。
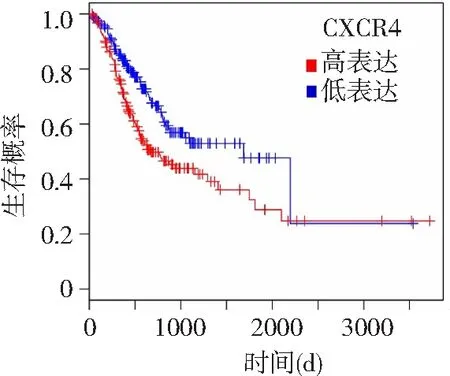
图2 2组胃癌患者生存曲线比较CXCR4为趋化因子受体4
2.3不同分期胃癌患者CXCR4表达差异 病理分期Ⅰ期患者CXCR4表达水平显著低于Ⅱ~Ⅳ期的患者(P<0.05),见图3A;T1期患者CXCR4表达水平显著低于T2~T4期患者(P<0.05),见图3B;N分期各期患者间CXCR4表达水平比较差异无统计学意义(P>0.05),见图3C;M0期患者CXCR4表达水平低于M1期患者(P<0.05),见图3D。

图3 不同分期胃癌患者CXCR4表达水平差异
A.不同病理分期胃癌患者CXCR4表达;B.T分期胃癌患者CXCR4表达;C.M分期胃癌患者CXCR4表达;D.N分期胃癌患者CXCR4表达;CXCR4为趋化因子受体4
2.4CXCR4相关基因的筛选 在检测的20 225个基因中,有11623个基因与CXCR4存在相关性(P<0.05)。见图4A。选取与CXCR4表达相关系数最高的前50个基因,并绘制基因热图。见图4B。其中氢电压门控通道1(hydrogen voltage gated channel 1, HVCN1)和RCSD结构域1(RCSD domain containing 1, RCSD1)与CXCR4表达水平的相关系数最高(r=0.749,0.738,均P<0.001)。见图4C和图4D。

图4 CXCR4相关基因筛选结果
A.CXCR4与各基因的相关性分析结果;B.与CXCR4表达相关系数最高的前50个基因;C.HVCN1与CXCR4表达的相关系数;D.RCSD1与CXCR4表达的相关系数;CXCR4为趋化因子受体4,HVCN1为氢电门控制通道,RCSD1为RCSD结构域1
2.5GO富集分析和KEGG通路富集分析 对筛选的50个基因进行GO富集分析可见,主要涉及的生物学过程包括对刺激的反应、生物调节、细胞通讯、多细胞生物过程和发育过程;主要定位的细胞组分为膜、囊泡、细胞质、细胞外空间和内膜系统;主要涉及的分子功能为蛋白质结合、分子换能器活动、酶调节剂活性、离子结合和脂质结合。见图5。KEGG通路富集分析可见主要涉及的信号通路为负性胸腺T细胞选择、B细胞活化参与免疫反应、免疫球蛋白介导的免疫反应、调节适应性免疫反应和T细胞分化。见图6。

图5 GO富集分析

图6 KEGG通路富集分析
2.6编码蛋白相互作用分析 相关基因所编码的蛋白之间的相互作用网络。见图7。酪氨酸蛋白激酶BTK(tyrosine-protein kinase BTK,BTK)、胞质分裂蛋白2脱除剂(dedicator of cytokinesis protein 2,DOCK2)、受体型酪氨酸蛋白磷酸酶C(receptor-type tyrosine-protein phosphatase C,PTPRC)是处于网络核心的蛋白。见图7。
3 讨论
CXCR4由352个氨基酸构成,其分子量为48 000,基因定位于2q21,在肿瘤形成和转移中起重要作用[5]。CXCR4活化后可影响肿瘤细胞增殖、血管生成、侵袭和远处转移[3]。陈佳栋等[6]通过对300例胃癌患者病理标本研究发现,CXCR4的转录及表达水平在胃癌组织中明显升高,可能与胃癌患者的临床分期和淋巴结转移有关。Nikkhoo等[7]报道,CXCR4在胃癌细胞中表达增加,且细胞核优势表达患者较细胞质优势表达患者的预后更佳。但多数研究缺乏长期随访数据,CXCR4对于胃癌患者长期生存率的影响仍有待进一步探讨。本实验通过分析公开数据库中的患者信息发现,胃癌患者CXCR4高表达组的中位生存时间显著低于CXCR4低表达组,表明CXCR4高表达与胃癌患者预后不良相关。进一步对CXCR4表达与肿瘤分期的相关性进行分析发现,肿瘤分期级别越高,CXCR4的表达越高。这与Nikzaban等[8]结果一致,其发现CXCR4水平在胃癌晚期显著升高,CXCR4水平与疾病的分期和淋巴侵袭有关。但CXCR4可参与多条信号通路,其造成胃癌患者预后不良的相关机制尚未完全阐明,为此本研究进一步探索并分析了与CXCR4表达相关系数较高的基因,以期寻找潜在相关机制。

图7 相关基因编码蛋白的相互作用网络
本研究结果显示,HVCN1和RCSD1与CXCR4表达水平的相关系数最高,其中HVCN1基因编码电压门控蛋白质通道蛋白,其可介导高选择性氢离子外向电流,能在短时间内补偿酸负荷。肿瘤细胞由于耗能增加可造成细胞内酸性物质聚积,若酸性物质聚积过量则会诱导发生细胞凋亡,而HVCN1的过表达则可缓解癌细胞内酸性物质的聚积,促进肿瘤细胞的增殖[9-10]。RCSD 1又称为CapZ相互作用蛋白(capZ-interacting protein, CAPZIP),在应激条件下诱导的磷酸化可调节F-肌动蛋白加帽蛋白重塑肌动蛋白丝组装的能力。CAPZIP主要高表达于免疫细胞和骨骼肌,当细胞暴露于外界应激时,可被多个应激蛋白激酶磷酸化,导致CapZIP中CapZ的分离,从而进一步影响CD28胞浆内结构域的磷酸化[11]。CD28的磷酸化是激活T 细胞发挥抗肿瘤免疫效应时重要的共刺激信号,其磷酸化程度降低将有利于肿瘤细胞的免疫逃逸[12]。CAPZIP在肿瘤细胞中高表达或可与免疫细胞中的CAPZIP竞争性结合应激蛋白激酶,从而促进肿瘤细胞的免疫逃逸。
本研究对相关基因进行富集分析和相互作用分析可见,无论是对刺激的反应功能、蛋白质结合功能,还是主要定位于细胞膜和囊泡,这都与通路富集分析中的负性胸腺T细胞选择、B细胞活化参与免疫反应、免疫球蛋白介导的免疫反应、调节适应性免疫反应和T细胞分化等结果相一致。这提示CXCR4促进胃癌患者预后不良的机制可能与对应激后免疫系统的调控有关。蛋白相互作用分析也同样再次印证了这一点。相互作用分析结果可见BTK、DOCK2和PTPRC处于网络核心,其中BTK对B淋巴细胞的发育、分化和信号传导是必不可少的[13],DOCK2可参与趋化因子响应的淋巴细胞迁移所需的细胞骨架重排[14],PTPRC与DPP4结合后可作为T细胞共激活的正调节剂[15]。
综上所述,CXCR4在提示胃癌患者预后中发挥重要作用,这可能与其通过调控免疫系统而促进肿瘤细胞免疫逃逸有关,这为胃癌的机制和药物治疗研究提供了一定参考。
