南京地区圈养黄颊长臂猿核心种群的遗传多样性分析
2020-02-21程家求傅兆水
陈 蓉 吕 冉 程家求 傅兆水
(南京市红山森林动物园管理处,南京,210028)
黄颊长臂猿(Nomascusgabriellae)隶属长臂猿科(Hylobatidae),冠长臂猿属,仅分布于老挝南部、柬埔寨东部、越南中部和南部的低地森林,主要以植物的果实、树叶、嫩枝、花朵为食,也兼食少量的昆虫、鸟卵。因受栖息地丧失和狩猎因素的威胁,野生黄颊长臂猿数量不断减少,已被IUCN列为濒危种。目前,关于黄颊长臂猿微卫星的研究尚未见报道,多数圈养黄颊长臂猿种群存在亲缘关系模糊、谱系不精确等问题,不利于迁地保护,更不利于迁地保护向就地保护转变。本研究通过对黄颊长臂猿微卫星位点进行筛选,对南京红山森林动物园圈养的14只黄颊长臂猿进行亲缘关系分析,了解个体间的亲缘关系和群体的遗传多样性情况,从而促进黄颊长臂猿物种保护的有效、科学、可持续地发展。
1 材料与方法
1.1 实验对象
实验对象为南京市红山森林动物园14只经物种鉴定的黄颊长臂猿,其基本信息见表1。采集其毛发,并保存于-20 ℃。
表1 14只黄颊长臂猿基本信息

Tab.1 The basic information table of 14 Nomascus gabriellae individuals
注:22、25、26、40、43、48、51、110均为谱系号
Note:Numbers 22,25,26,40,43,48,51 and 110 are pedigree numbers
1.2 主要试剂与仪器
微量基因组提取试剂盒、TaqPlus MasterMix、D2000 DNA Marker、GeneGreen核酸染料购自天根生化科技(北京)有限公司。普通引物均有金斯瑞生物科技有限公司合成。荧光引物合成和测序均有上海英潍捷基生物技术公司。
梯度PCR仪购自杭州龙扬仪器公司。凝胶成像系统购自上海欧翔仪器公司。水平电泳仪购自北京君意东方有限公司。高速冷冻仪离心机购自上海沪粤明科学仪器有限公司。
1.3 基因组DNA提取
按微量基因组提取试剂盒说明书提取长臂猿毛囊中的基因组DNA。提取后的基因组DNA样本于-20 ℃保存,备用。
1.4 引物筛选与PCR
以微卫星标记的序列重复单位、杂合度、多态信息含量和片段大小等特性为依据,从相近物种[1-2]和NCBI数据库里筛选25个位点。梯度PCR反应体系为:Mix 12.5 μL,上游引物 0.5 μL,下游引物0.5 μL,双蒸水6.5 μL,DNA模板5 μL。梯度PCR反应条件为:94 ℃ 5 min;94℃ 1 min,49 ℃—60 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min。
以黄颊长臂猿大黑个体 DNA 为模板来检测每个微卫星位点的最佳PCR 扩增条件,挑选出多态性较高、扩增效果最好的微卫星位点用以开发荧光PCR体系。
1.5 荧光引物的筛选
将多态性高、扩增条件好的微卫星位点,相应引物的上游引物5′端分别用FAM,HEX,TAMRA 3种荧光染料进行标记。参考普通PCR的反应体系与反应条件,进行荧光标记引物的PCR扩增。
将PCR扩增产物避光保存于-20 ℃,依事先组合等量混合同一个体的PCR产物,低温运输,分3次送至上海英潍捷基生物技术公司进行SSR分型检测。根据SSR分型检测报告,根据每个图谱峰值的位点位置和大小、片段大小,同时判断纯合子(单峰)或杂合子(双峰),挑选出适合的微卫星位点。
1.6 统计分析
通过Cervus 3.0软件进行遗传多样性和亲缘系数分析数据分析,统计参数主要包括等位基因数(analyze the number of alleles,Na),观测杂合度(observed heterozygosity,Ho),期望杂合度(expected heterozygosity,He),多态信息含量(polymorphic information content,PIC),无效等位基因频率(null allele frequency,F(Null)),哈迪温伯格平衡(Hardy-Weinberg equilibrium,HWE)检测,亲缘系数等。
2 结果
2.1 微卫星位点的筛选
25对微卫星引物的梯度PCR扩增结果表明,有17对扩增出目的片段。对这17对引物进行荧光标记的PCR扩增,并进行SSR分型检测,结果显示仅11对荧光引物扩增出来的图谱峰值的位点位置和大小、片段大小是正确的,其余6对出现错峰、位点较多不符合孟德尔定律等情况。因此,采用这11对微卫星引物进行黄颊长臂猿的亲子鉴定。具有多态性的微卫星引物序列、反应条件见表2。
表2 11对黄颊长臂猿微卫星特征
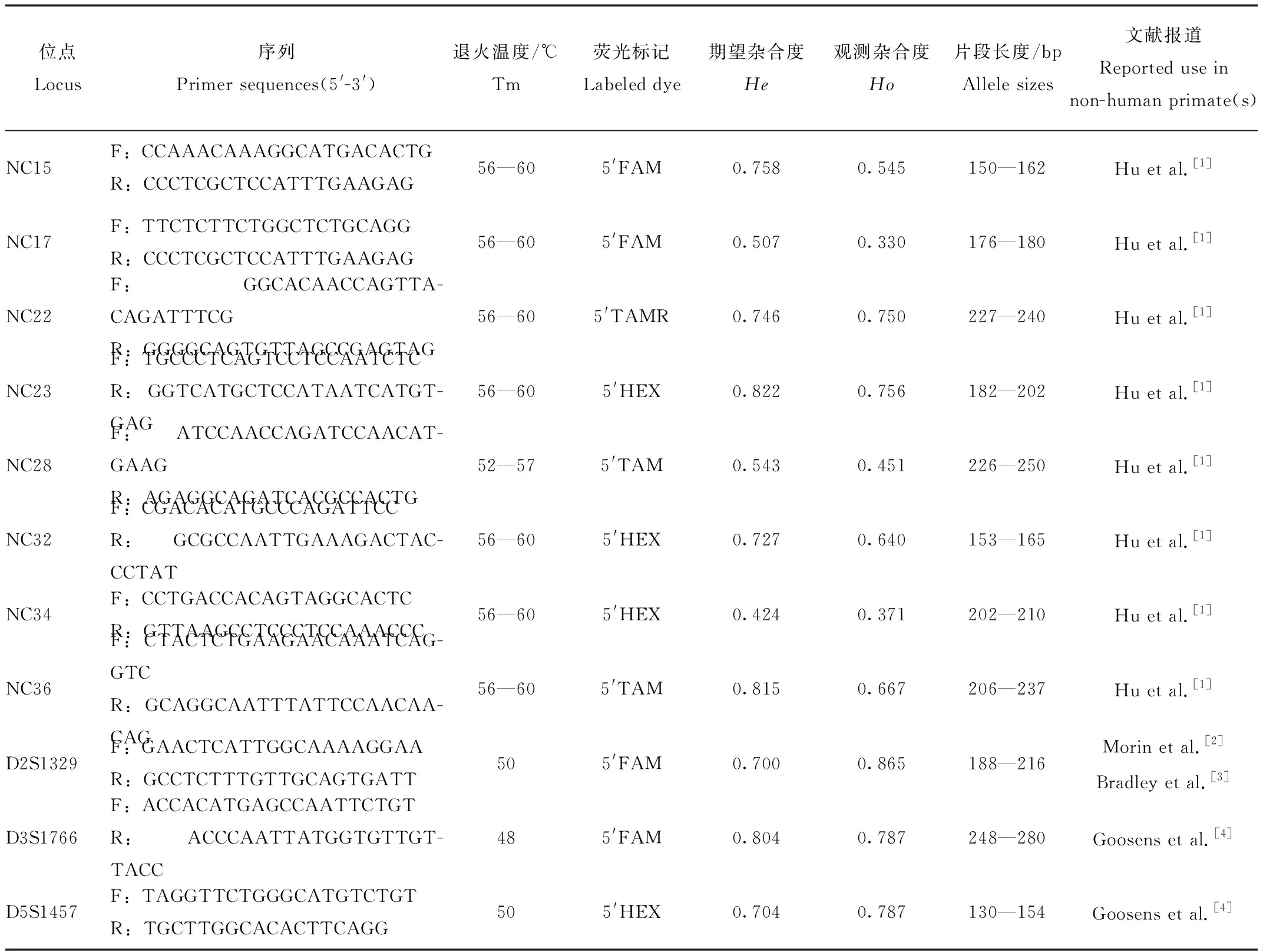
Tab.1 Characterization of 11 polymorphic microsatellite loci for Nomascus gabriellae
2.2 遗传多样性分析
将11对英光引物的PCR产物送至上海英潍捷基生物技术公司进行检测,结果11个微卫星位点,共检测出41个等位基因。运行Cervus 3.0中的Allen Frequentcy Analysis程序,得到11个微卫星位点在14只个体中的遗传信息参数(表3)。结果表明,平均等位基因数(K)为3.7,多态信息含量(PIC)为0.175—0.701,平均多态信息含量为0.463,属于中度多态位点(0.25 表3 11个微卫星位点在14只黄颊长臂猿中的遗传信息参数分析 Tab.3 Analysis of genetic information parameters of 11 microsatellite loci in 14 Nomascus gabriellae individuals 注:K为位点等位基因数;N为基因分型的个体数:Ho为位点观测杂合度;He为位点期望杂合度;PIC为位点多态信息含量;NE-1P为第一个亲本的排除概率;NE-2P为第二个亲本的排除概率;NE-PP为双亲的排除概率;F(Null)为无效等位基因频率 Note:Kis allele number of loci,Nis number of genotyping,Hois observed beterozygosity,Heis expected heterozygosiry,PICis polymorphic information content,NE-1Pis exclusion probability of the first parent,NE-2Pis exclusion probability of the second parent,NE-PPis exclusion probability of the parent pair,F(Null)is frequency with null 通过Cervus 3.0软件进行亲缘系数结果见表4。已知大黄家族中,父本为大黑,子一代分别为园园、冬至、乐乐、夏天。其大黑与园园、冬至、乐乐、夏天的亲缘系数分别为0.476 1,0.476 5、0.556 8、0.506 2;其子代间亲缘系数为0.261 9—0.754 5。另一已知家族中,母本为黄婆,父本为园园,子代为恩奇。其母子亲缘系数为0.452 6,父子亲缘系数为0.452 6,亲本间亲缘系数为0.133 2。这些数值符合已知的谱系记载情况。 通过亲缘系数分析,小黄与黄婆(0.326 7)、六华(0.422 7)亲缘系数大于0.25,但均为雌性。迪克与冬至(0.453 3)、小小(0.310 7)、小四(0.353 1)、园园(0.267 0)亲缘系数大于0.25,但均为雄性。黄婆与三兮(0.462 8)、小四(0.438 3)亲缘系数大于0.25,其中与小四为异性。三兮除与黄婆外,还与九九(0.599 9)、恩奇(0.266 6)、大黑(0.360 2)亲缘系数大于0.25。小小除与迪克外,还与冬至(0.391 5)、恩奇(0.319 7)亲缘系数大于0.25。大黑与小四的亲缘系数为0.712 9。小四除了小黄、六华外,与其他黄颊长臂猿的亲缘系数都大于0.25。 PIC可以作为衡量基因变异程度高低的指标,其值越大,说明这个群体中在该座位上杂合子比例越大,能提供的遗传信息就越多。双亲在该位点杂合度越高,则其后代中越能够清楚地观察到等位基因的分离。在遗传连锁分析中,当该位点的PIC大于0.7时,说明该位点为理想的选择标记。本研究所采用的11个座位的多态信息含量(PIC)为0.175—0.701,平均多态信息含量为0.463。其中,4个位点为高度多态,4个位点为中度多态,3个位点为低度多态。由此可见,这11个微卫星位点可以作为评判这群黄颊长臂猿群体遗传多样性的有效遗传标记。 表4 14只黄颊长臂猿的亲缘系数(r)结果 Tab.4 Affinity coefficient(r)results of 14 Nomascus gabriellae individuals 该群体11个微卫星位点共获得等位基因41个,平均等位基因数(K)为3.7,多态信息含量(PIC)为0.175—0.701,平均多态信息含量为0.463,属于中度多态位点(0.25 此次,个体之间的亲缘关系较近,追踪原有的谱系记录,发现大部分黄颊长臂猿来自同一地区,同时我国圈养种群存在数量较少、配对少、繁殖难等问题,导致该圈养黄颊长臂猿小种群的亲缘关系较近。在濒危小种群物种管理中,奠基者效应造成的遗传漂变较为常见,如我国的朱鹮(Nipponianippon)种群[5]、华南虎(Pantheratigrisamoyensis)种群[6]等。但是在有条件的情况下,尽量选择亲缘较远的配偶进行繁殖,同时在引进新的血缘时,要结合遗传学检测,从而更加科学地进行物种管理。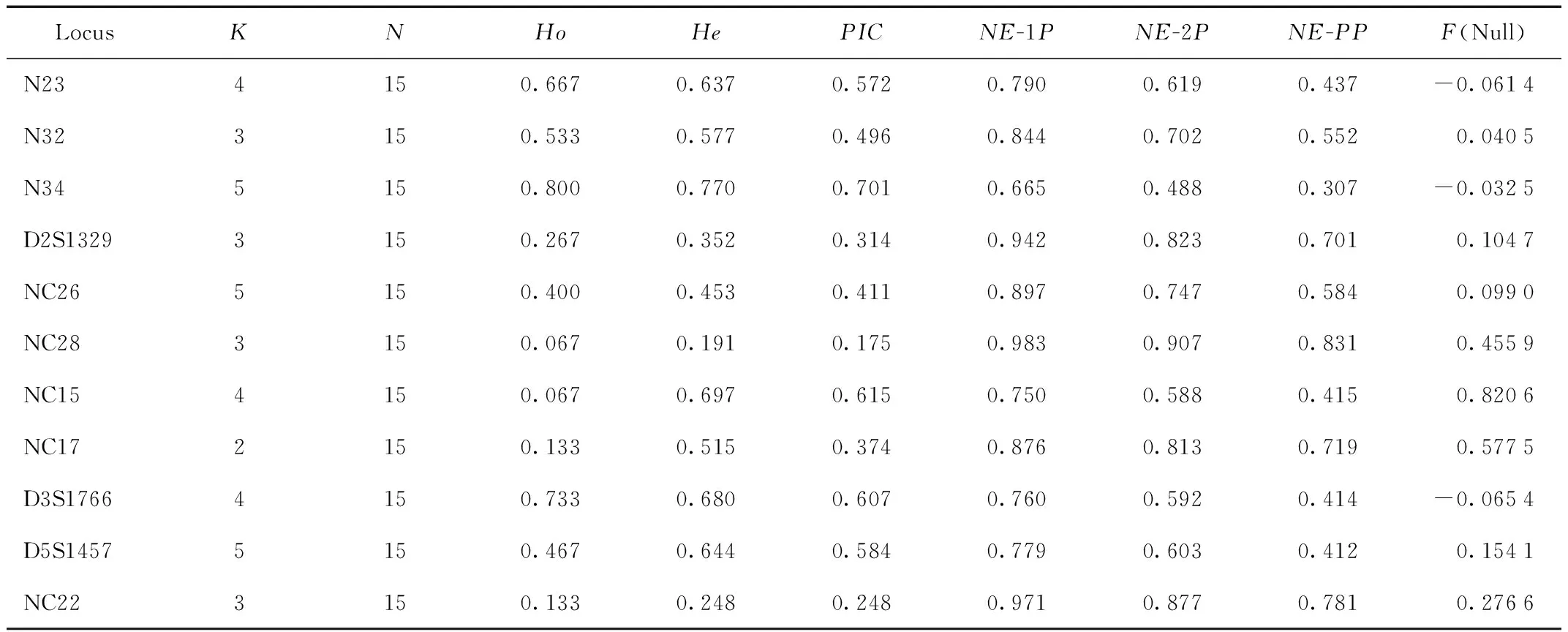
2.3 亲缘系数
3 讨论

