1 株来源于茅台酒酿造过程宛氏拟青霉MTDF-01的全基因组测序及分析
2020-01-07王和玉刘延峰张巧玲堵国成李江华
王和玉,刘延峰,张巧玲,堵国成,杨 帆,李江华,*
(1.贵州香台酒股份有限公司,贵州 仁怀 564501;2.江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122)
白酒发酵过程中复杂的微生物种群是决定白酒风味特征的重要因素[1-3]。霉菌能够分泌多种酶类分解原料为发酵过程微生物提供代谢底物,同时其自身可生成各种代谢产物,对白酒风味形成有重要贡献[4-6]。在白酒生产中主要的功能霉菌包括毛霉、曲霉等。在酱香型白酒的酿造过程制曲、堆积发酵和窖池发酵中,宛氏拟青霉(Paecilomyces variotii)是优势霉菌之一[7],研究表明该菌具有高糖化酶活力,能够降解原料中的淀粉[8],并且在制曲工艺、堆积工艺以及兼性条件下分别进行纯种固态发酵,能代谢生成酸、醇、醛、酮、酯及芳香族化合物等多种物质,发酵产生多种风味物质和风味前体物质,包括苯乙醇、苯乙酸等,是酱香型白酒中众多呈香呈味物质中的组成部分[9]。同时P. variotii也能够分泌降解糠醛的酶,可有效降低酒醅中的糠醛[10]。
目前,在白酒酿造行业,针对酿造微生物功能研究已从传统的发酵分析手段转方代谢组学结合宏基因组、转录组进行全方面的解析[11-14],迄今为止,白酒酿造微生物全基因组测序报道有地衣芽孢杆菌(Bacillus licheniformis)GMCC3963[15]、华根霉(Rhizopus chinensis)CTCCM201021[16]、耐高温放线菌(Thermoactinomyces daqus)H-18[17]以及窖泥中分离得到的丁酸梭菌(Clostridium butyricum)JKY6D110[18]等。P. variotii作为酱香型白酒酿造过程的优势微生物之一,在制曲及制酒阶段活动活跃,目前对其研究主要在于产酶特性及代谢产物分析[8],P. variotii全基因组分析鲜见报道,对P. variotii发酵代谢机制及生理生化等微生物特性缺乏足够的认识和了解,限制了深入开发和利用该微生物。
本研究通过PacBio RS II测序平台,对香台酒酿造过程中分离得到的P. variotii MTDF-01进行全基因组测序,根据测序获得的数据进行基因组分析、基因功能注释和比较基因组学分析,为今后进一步深入了解P. variotii在酱香型白酒生产过程中的代谢机理以及为调控菌株代谢能力提供重要的生物信息学基础。
1 材料与方法
1.1 材料与试剂
菌株MTDF-01分离自香台酒酿造过程。
真菌基因组DNA提取试剂盒 北京索莱宝科技有限公司;其他化学试剂均为国产分析纯。
1.2 仪器与设备
ACB-401超净工作台 新加坡Esco科技有限公司;spx-250B-z生化培养箱 上海福马实验设备有限公司;MaxQ 6000轨道摇床 赛默飞世尔科技公司;5804R台式高速冷冻离心机 德国Eppendorf公司;GelDoc XR凝胶成像系统 美国Bio-Rad公司。
1.3 方法
1.3.1 菌体培养与回集
菌体培养:拟青霉划线PDA琼脂斜面培养基37 ℃培养72 h后通过无菌生理盐水洗下孢子,制备成孢子悬浮液。
菌体摇瓶培养:吸取450 µL孢子悬浮液(孢子浓度数量级107)接种至20 mL/250 mL的PDA液体培养基中,摇床培养30 ℃、转速200 r/min。将培养好的菌体通过过滤回集。
1.3.2 菌株总DNA提取
采用真菌基因组DNA提取试剂盒提取基因组DNA,操作步骤参照试剂盒说明书。提取得到的基因组通过0.8%琼脂糖凝胶电泳检测。
1.3.3 全基因测序组装
本次测序委托武汉生物技术研究院完成。样本质检合格后,用Covarisg-TUBE对基因组DNA随机打断,进行文库构建。利用磁磁富集、纯化大片段DNA,对片段化的DNA进行损伤修复、末端修复;在DNA片段两端连接茎环状测序接头,并利用外切酶去除连接失败的片段,纯化后,采用Agilent 2100 Bioanalyzer HighSensitivity Kit进行文库质量检测。采用第3代测序仪PacBioRS II对DNA进行非扩增长片段测序,测序完成后去除adapter序列及截掉reads部分区域的低质量碱基,采用HGAP[19]流程进行组装,挑选长reads作为种子序列,其他的较短reads通过BLASR软件[20]比对到种子序列,进行校正。最终以校正后的高质量种子reads采用Celera assembler组装软件[21]用OLC组装算法进行组装。利用Quiver软件[22]进行组装结果的优化和校正。在Quiver校正结果的基础上,去除低覆盖度的重叠群及冗余的重叠群。
1.3.4 基因预测与注释
通过EVM(EvidenceModeler)软件[23]整合Augustus软件[24]、SNAP软件[25]、GeneWise软件[26]基因预测结果获得基因结构预测,将获得的基因提交COG(cluster of orthologous groups of proteins)[27]、GO(gene ontology)[28]、KEGG(kyoto encyclopedia of genes and genomes)[29-30]、NR(non-redundant protein database)、Swiss-Prot数据库[31]、TrEMBL数据库[31]进行比对,获得功能注释信息。
1.3.5 物种进化树构建及分析
挑选菌株进行系统发育树构建,运用MEGA 7.0软件(https://www.megasoftware.net/)Neighbor-Joining法进行分析,以18S rRNA基因进行比较分析构建系统进化树。
1.3.6 CAZy糖酶分析
通过dbCAN HMMs 3.0在线工具[32](http://csbl.bmb.uga.edu/dbCAN/index.php)将P. variotiiMTDF-01的CDS序列与碳水化合物活性酶(CAZy)数据库(http://www.cazy.org/)进行比对分析。比对时dbCAN的阈值参数设置如下:如果比对的序列长度大于80 aa,阈值<1×10-5,否则阈值>1×10-3。
1.3.7 次级代谢产物合成基因簇分析
利用antiSMASH 3.0[33](https://fungismash.secondarymetabolites.org)对MTDF-01菌株中次生代谢物合成基因组簇进行预测。
1.3.8 基因组共线性分析
利用Mummer程序(https://sourceforge.net/projects/mummer/postdownload,版本3.23)对MTDF-01及NCBI中已测序的菌株P. variotiiNo.5进行分析比较,并用MUMmerplot将结果可视化。菌株P. variotiiNo.5序列从NCBI网站下载(ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCA/000/497/085/GCA_000497085.1_PVAR5_assembly01/)。
2 结果与分析
2.1 MTDF-01全基因组基本特征及初步分析
PacBio RS II是首个商业化应用的第3代测序平台,其采用特有的单分子实时技术使得测序读长较第2代测序技术显著增加(半数测序读长大于20 kb),同时减弱GC性[19]。通过PacBio RS II测序平台对菌株MTDF-01进行全基因组测序,获得菌株MTDF-01全基因组数据。测序数据过滤后总数据量为2.58 G,平均测序读长为14.64 kb,最长的测序读长达到 46.94 kb,测序深度为83.81×,DNA靶序列平均长度为7 823 bp。对原始数据进行组装后得到参考序列由19 个重叠群组成,形成19 个基因组骨架,总长度为30 833 540 bp,其中最长重叠群为7 588 950 bp,GC平均含量为47.46%。MTDF-01基因组测序数据提交至NCBI,GenBank登录号为SAMN09987769。
2.2 系统进化树构建
为进一步确认MTDF-01的种属关系,本研究以18S rRNA基因进行比较分析,构建系统发育树(图1)。结果表明MTDF-01与P. variotiiCBS 102.74、P. variotiiCBS 101075聚为一支,可以确定为P. variotii,同时与P. variotiiNo.5也聚为一支,两者物种亲缘关系较近。

图1 通过MEGA Neighbor-Joining法构建的真菌分子系统发育树Fig. 1 Molecular phylogenetic tree of fungi constructed by Neighbor-Joining method with MEGA software
2.3 基因结构预测
基于ab initio预测软件(Augustus、SNAP、GeneMark-ES)及同源比对预测两种方法对P. variotiiMTDF-01测序拼接片段进行基因预测与编码区分析,共预测出基因8 815 个,基因总长度为13 530 750 bp,平均基因长度为1 740 bp,平均编码序列长度为478 bp。预测基因序列采用多个数据库(KOGGOKEGGNRSwiss-ProtTrEMBL)进行比对,获得相应功能注释信息,最终注释基因8 662 个,详细统计数据见表1。在基因组序列上共找到5 044 个简单重复序列,分别采用同源比对和de novo预测的方式对基因组的重复序列进行注释,结果显示该基因组中含有3.80%的重复序列。在基因序列上共找到tRNA数量为221 个,rRNA为49 个。

表1 基因功能注释统计Table 1 Gene function annotation statistics of MTDF-01
2.4 基因功能分类
2.4.1 COG聚类分析
NCBI创建并维护的蛋白数据库COG是根据细菌、藻类和真核生物完整基因组的编码蛋白系统进化关系分类构建而成。真核生物的序列功能可通过真核基因组数据库KOG进行比对及预测。对P. variotiiMTDF-01基因功能进行初步分析,选择利用COG进行基因注释与功能分类,共注释基因1 972 个,占总蛋白数的22.37%,可分为25 个功能组,3 大类分别为细胞过程及信号、信息储存及加工、代谢过程,分别注释基因数为556、336 个和822 个。从图2可看出,分类为R(普通功能预测)的基因数量最多为377 个,其余基因序列主要集中在O(蛋白翻译后修饰)、C(能量产生与转换)、I(脂质转运和代谢)及Q(次级代谢物合成、转运和代谢)等分类单元。

图2 MTDF-01预测蛋白COG功能分类Fig. 2 COG cluster analysis of MTDF-01 proteins
2.4.2 GO聚类分析
基于基因本体数据库GO功能分类,该数据库按照细胞学组件、分子功能、生物学过程对蛋白进行分类注释。本研究将MTDF-01菌株进行蛋白质功能分析,共预测得到5 490 个蛋白,注释统计结果如表2所示。可看出菌株的GO功能聚类在细胞、细胞组分、连接、催化活性、细胞过程、代谢过程等条目的基因数占据优势,其中细胞和细胞组分各有1 187 个基因,分子功能中的连接和催化活性功能分别有2 929 个和2 922 个,生物学过程中的细胞过程和代谢过程分别注释到2 077 个及2 914 个基因,表明在菌株的蛋白功能主要为细胞组成、代谢和酶催化。
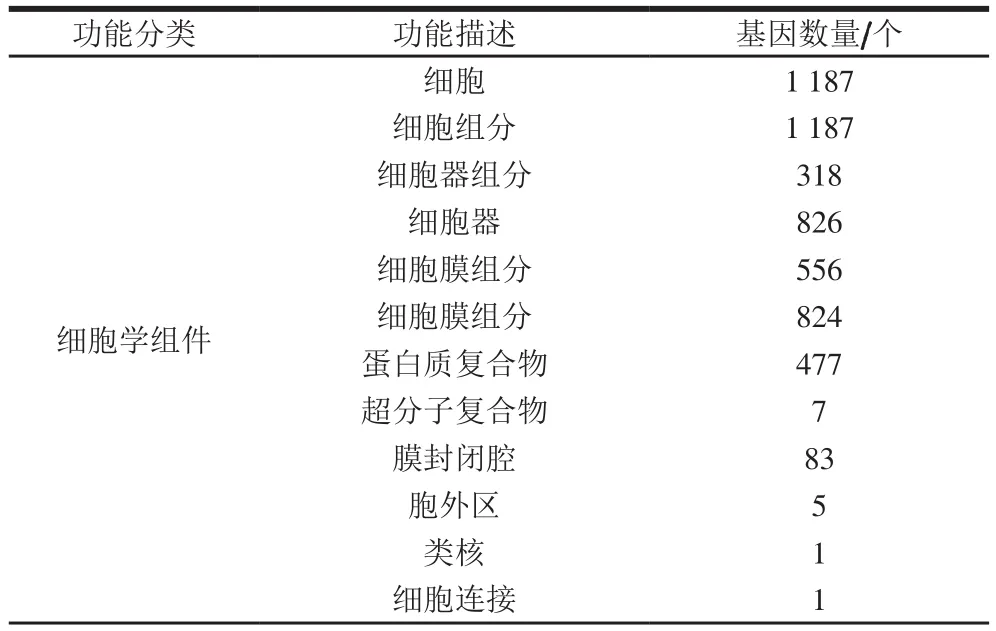
表2 MTDF-01预测蛋白GO分类Table 2 Gene ontology classi fication of MTDF-01 proteins

续表2
2.4.3 KEGG代谢途径预测分析
本研究通过将P. variotiiMTDF-01与KEGG数据库进行比对分析,共注释得到物质代谢通路274 个。MTDF-01中预测代谢通路(图3)在新陈代谢、遗传信息加工、细胞过程3 个大类中涉及基因较多,遗传信息加工(转录、翻译、蛋白质的折叠和加工等)共有566 个基因,细胞过程(转运代谢、细胞运动、细胞生长与死亡细胞通讯等)共有382 个基因,而涉及代谢的这类基因最多,行使的功能较多,既有细胞本身合成氨基酸的基因,也有氨基酸代谢、膜蛋白基因以及能量系统基因等,共有1 506 个。在代谢中涉及基因最多的通路主要有嘌呤代谢途径(ko00230)、氧化磷酸化途径(ko00190)、嘌呤代谢途径(ko00240)、糖酵解途径(ko00010)、淀粉与蔗糖代谢途径(ko00500)、精氨酸与脯氨酸代谢途径(ko00330)。
通过比对结果发现,MTDF-01中涉及氨基酸代谢的代谢途径有20 条共462 个基因,包含了苯丙氨酸、色氨酸和酪氨酸等芳香族氨基酸代谢途径,芳香族氨基酸可为苯乙醇、苯乙酸等风味物质代谢提供前体物质,但缺失精氨酸合成途径的相关基因。进一步对白酒特征风味物质相关通路分析发现MTDF-01具有苯乙醇、苯乙酸、亚麻油酸、苯甲酸、肉桂酸乙酯、乙酸、乳酸等风味物质的代谢途径。其中苯乙醇和苯乙酸可通过苯丙氨酸、酪氨酸和色氨酸生物合成途径将苯丙氨酸在氧化氢-过氧化物酶作用下生成2-苯乙酰胺,进一步在酰胺酶作用下生成苯乙酸,苯乙酸可在乙醛脱氢酶和和芳基乙醇脱氢酶作用下生成苯乙醇。苯丙氨酸也可通过芳香族L-氨基酸/L-色氨酸脱羧酶作用生成苯乙胺,苯乙胺在芳基乙醇脱氢酶作用下生成苯乙醇。在P. variotiiMTDF-01中有多条通路可合成乙酸,该菌株具有乙醇脱氢酶和乙醛脱氢酶相关基因能够将乙醇氧化成乙酸,还可丙酮酸代谢途径、乙醛酸和二羧酸盐代谢途径代谢生成乙酸。注释结果表明P. variotiiMTDF-01有亚麻油酸、油酸、花生四烯酸、二十二碳六烯酸等多种有机酸合成的关键酶。有文献报道,P. variotii具有降解糠醛的功能[10],但在注释结果中并未发现糠醛降解途径。根据预测信息可推测该菌株在白酒发酵过程中可能对白酒风味代谢具有重要贡献。
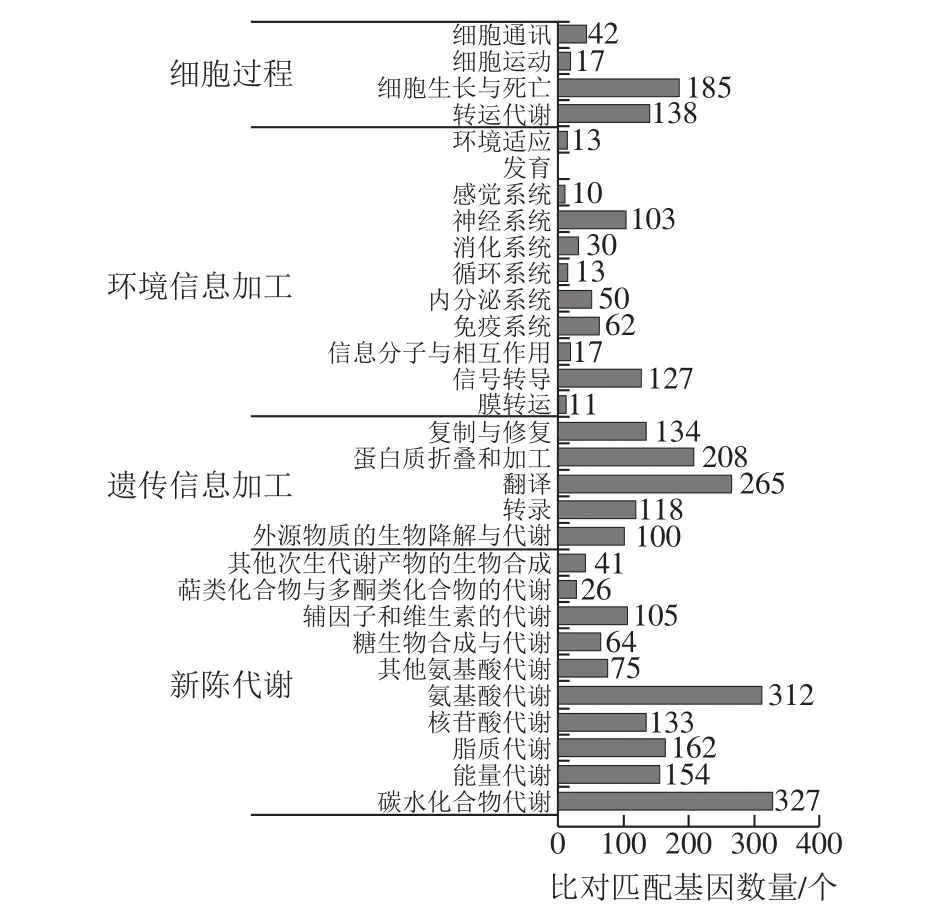
图3 P. variotiiMTDF-01 KEGG功能分类Fig. 3 KEGG cluster analysis of P. variotii MTDF-01 proteins
2.4.4 CAZy糖酶分析
在白酒酿造过程中,淀粉酶能够将原料中的淀粉降解生成葡萄糖等利于微生物利用的还原糖,为发酵进程提供营养底物,促进酵母等微生物生长代谢。原料中的纤维成分较多,纤维素酶能够对纤维素进行降解,释放淀粉,有利于糖化酶的作用,同时纤维素酶能够将原料淀粉中3%左右的纤维素和半纤维素转化成可发酵性糖,有利于酵母的利用[34],此外,纤维素酶也会影响白酒的风味[35]。
将基因组序列与CAZy数据库进行比对,在P. variotiiMTDF-01的基因组中共有907 个基因编码的蛋白质结构域属CAZy家族,包括425 个糖苷水解酶(glycoside hydrolases,GHs)、307 个糖苷转移酶、75 个碳水化合物酯酶、5 个多糖裂解酶、213 个碳水化合物结合组件(carbohydrate-binding modules,CBMs),其中194 个基因含有多种组分结构域,表明MTDF-01含有丰富的碳水化合物代谢酶。CBMs作为一些GHs所含有的非催化活性结构的结构域,使得GHs能靠近底物,以增加GHs锚定底物的能力,从而影响GHs的酶催化活性。通过预测MTDF-01的基因组中共有108 个GHs基因含有CBMs结构域。
淀粉降解的酶主要为α-淀粉酶、α-葡萄糖苷酶(糖化酶)等,纤维素酶主要为α/β-葡聚糖酶、β-葡萄糖苷酶,木聚糖是半纤维素中最主要的碳水化合物,因此木聚糖酶也成为了半纤维素降解中的主要酶类。为进一步探索P. variotiiMTDF-01对淀粉及纤维素的降解能力,本研究对在P. variotiiMTDF-01基因组中淀粉/糖原、纤维素等降解酶相关酶进行统计,见表3。结果表明在P. variotiiMTDF-01基因组中含有多个淀粉及纤维素水解酶基因,分别为60 个及165 个,可推测P. variotiiMTDF-01具有降解淀粉和纤维素的潜力。在白酒生产过程中,纤维素酶来源广泛,大曲中的细菌、真菌和放线菌都有产纤维素酶的能力。在曲霉[36]、Paenibacillussp.、Acinetobactersp.[37]等微生物中均有较强的纤维素降解力,但对P. variotii纤维素降解方面研究较少,P. variotii纤维素降解能力仍需进一步进行实验验证。

表3 P. variotii MTDF-01淀粉及纤维素降解酶CAZy预测结果Table 3 Predicted starch and cellulose degrading-enzymes of P. variotiiM TDF-01 from CAZy database
2.4.5 次级代谢产物合成基因簇分析

表4 MTDF-01次级代谢产物合成基因簇Table 4 Secondary metabolic gene clusters of MTDF-01
聚酮合酶基因合成酶途径、非核糖体多肽合成酶途径与聚酮合酶基因合成酶-非核糖体多肽合成酶混合代谢是大多数真菌毒素合成中的关键代谢途径。同时在真菌的萜类化合物代谢过程中,也能产生多种真菌毒素[16]。用anti-SMASH对基因组进行次级代谢产物合成分析预测,基因组中共预测得到23 个次级代谢产物基因簇,主要为聚酮合酶(polyketide synthase,PKS)基因簇、非核糖体多肽合成酶(non-ribosomal peptide synthase,NRPS)基因簇和萜烯类,具体预测结果如表4所示。在预测到的基因簇中,pks和Nrps基因簇共15 个,占预测总基因簇的65.21%,T1pks有7 个,Nrps有6 个,T1pks-Nrps有2 个,萜烯类合成途径基因簇有5 个。但所有基因簇与已知次级代谢产物进行BLAST比对发现,P. variotiiMTDF-01的基因组中PKS途径、NRPS途径与PKS-NRPS混合代谢途径、萜烯类途径中的基因簇并未匹配及注释到相似性高的基因信息,相似度最高的仅为33%。因此可认为P. variotiiMTDF-01中不存在PKS、NRPS、PKSNRPS混合代谢途径、萜烯类等途径合成代谢能力,P. variotiiMTDF-01不具备合成该类物质的能力。
通过KEGG对萜类化合物合成途径分析时也发现,在P. variotiiMTDF-01中注释到参与萜类化合物代谢途径的基因仅有26 个,其中萜类化合物合成途径注释到18 个基因,主要为萜类物质骨架结构的真菌甲羟戊酸合成途径中注释到相对完整的代谢途径,共16 个基因,但并未注释到特异性后修饰相关蛋白序列,因此,根据基因组预测信息可表明P. variotiiMTDF-01不具备真菌毒素合成能力,是白酒酿造过程的发全菌株。
2.5 与P. variotii No.5比较基因组学分析
2.5.1P. variotiiNo.5全基因组概况
目前已报道的关于P. variotii的全基因组测序的菌株为P. variotiiNo.5(NBRC109023)。P. variotiiNo.5是1 株分离于土壤中的耐甲醛的真菌,能够在20 min内降解2%的甲醛[38]。将P. variotiiMTDF-01与P. variotiiNo.5菌株进行比较基因组学研究,以研究不同环境P. variotii基因组信息的差异,为研究不同来源P. variotii基因组框架、组成及蛋白差异提供理论基础及研究方方。两个菌株基本信息统计结果如表5所示。

表5 P. variotiiMTDF-01与P. variotii No.5基本特征比较分析Table 5 Comparative analysis of general features of P. variotii MTDF-01 and P. variotii No.5
2.5.2 共线性分析
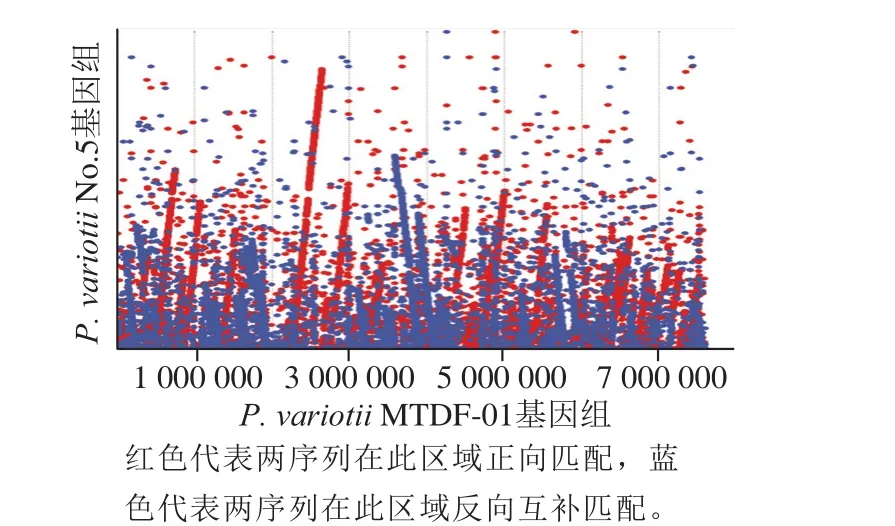
图4 P. variotii MTDF-01与P. variotii No.5基因组序列线性结构比较Fig. 4 Global alignment between P. variotii MTDF-01 and No.5
通过比较P. variotiiMTDF-01与P. variotiiNo.5两个基因组的同源性,绘制共线性关系点阵图(图4)以观测结构性的差异。结果表明P. variotiiMTDF-01与P. variotiiNo.5在基因组水平上存在较大差异。从图4可看出,两者基因组中有多条大片段能够比对上,两株菌基因组存在一定相似性,但是存在大量倒位、重排等现象。对单核苷酸多态性进行分析发现P. variotiiMTDF-01与P. variotiiNo.5两个基因组共存在16 414 处单核苷酸碱基突变,其中有525 个碱基缺失,335 个碱基插入,15 554 个单碱基替换。
进一步对蛋白编码序列进行分析发现:P. variotii MTDF-01共预测CDS序列8 815 个,P. variotii No.5共预测CDS序列8 877 个,总匹配蛋白数(相似度大于50%)为7 743 个,其中仅有26 个基因完全匹配上,其余7 717 个基因存在氨基酸层面的突变、插入或缺失,相似度在90%以上的基因有5 803 个,占总匹配蛋白数的74.94%。两者在氨基酸水平上既有相似性又有各自的独特性。通过KEGG注释结果表明No.5存在大量环境信息处理相关基因,主要涉及信号转导。由于P. variotii MTDF-01和P. variotii No.5两株菌株来源环境差异较大,P. variotii No.5来源于土壤环境,而P. variotii MTDF-01在白酒酿造环境中经过高温高酸等环境胁迫条件长期驯化,因此这也可能导致两株菌在基因组及功能的差异。白酒酿造环境的长期驯化使P. variotii MTDF-01显示出独特性。
3 讨论与结论
Oka等[38]将从土壤中分离得到的P. variotii No.5进行全基因组测序,并根据其具有降解甲醛的功能,对参与甲醛代谢的相关途径进行了分析,发现了7 个相关蛋白。Radwan等[39]从喷气燃料中分离得到Paecilomyces sp. BYSS01,预测发现共有334 种真菌酶参与碳水化合物代谢,并且能够参与芳香烃和正烷烃的降解。但在白酒酿造过程中关于P. variotii的基因组研究鲜见报道。本研究通过对白酒酿造过程中分离得到P. variotii MTDF-01进行全基因组测序,拼接得到基因组总长度为30 833 540 bp,GC平均含量为47.46%。通过COG、GO、KEGG等数据库比较分析发现MTDF-01菌株基因主要涉及遗传信息加工、细胞转运及代谢过程,在代谢过程中主要参与碳水化合物、氨基酸、核苷酸、能量代谢等代谢通路,这为进一步了解P. variotii代谢机理提供了理论基础。基于KEGG和全基因组测序数据代谢网络注释发现,P. variotii MTDF-01具有苯乙醇、苯乙酸、苯甲酸、肉桂酸乙酯、乙酸、乳酸等代谢途径并具有合成多种有机酸的关键酶,此外,通过碳水化合物预测和次级代谢产物分析表明P. variotii MTDF-01含有多个淀粉和纤维素水解酶,具有降解淀粉和纤维素的潜力,不具备真菌毒素合成能力,是白酒酿造过程的发全菌株。对P. variotii MTDF-01与P. variotii No.5基因组进行比较基因组学分析发现,两者存在显著差异,存在翻转及异位等基因组重排现象,P. variotii MTDF-01具有独特性。
本研究利用全基因组测序对白酒酿造过程中分离得到的P. variotii MTDF-01进行分析,在P. variotii MTDF-01中发现了参与酿酒原料代谢及风味物质合成代谢的基因及代谢通路,为解析P. variotii的酿造功能提供了新思路,从基因组水平对P. variotii代谢途径的挖掘,为深入了解P. variotii在白酒生产过程中的代谢机理提供了参考信息及研究方方,对以后P. variotii的相关研究具有重要意义。
