小麦抗麦红吸浆虫基因标记的开发与验证
2020-01-02郝志明耿妙苗温树敏闫桂军王睿辉刘桂茹
郝志明 耿妙苗 温树敏 闫桂军 王睿辉,* 刘桂茹,*
小麦抗麦红吸浆虫基因标记的开发与验证
郝志明1耿妙苗1温树敏1闫桂军2王睿辉1,*刘桂茹1,*
1河北农业大学 / 华北作物种质资源研究与利用教育部重点实验室, 河北保定 071000;2西澳大学农业与环境学院, 澳大利亚尼德兰兹 6009
麦红吸浆虫(Géhin)严重影响小麦产量和品质, 选育和使用抗虫品种是降低虫害损失最安全有效的途径, 利用抗虫性连锁或功能标记对提高小麦抗虫分子育种效率具有重要意义。在前期从转录组数据中挖掘到抗虫性主效QTL () 6个相关差异基因的基础上, 依据这些基因序列中存在的InDel和SNP, 分别开发了2个EST标记和6个KASP标记, 并在抗虫性不同的一套重组近交系(RIL)和一套小麦品种中进行了标记的有效性验证。所开发的8个标记在抗、感虫小麦亲本间均表现出较好的多态性, 在RIL株系中的检测有效率均达到90%左右; 除外, 这些标记在供试高抗(56.3%~86.7%)和高感(85.7%~100.0%)小麦品种中的检测有效率均较高, 可用于小麦种质资源的抗虫性筛选。同时发现, 11个具备所有抗虫标记位点的抗虫小麦品种, 多为审定时间较早或已停止使用的品种, 这使得结合标记辅助选择等手段鉴定和创新小麦抗虫种质资源的工作日益紧迫。
小麦; 麦红吸浆虫; 抗虫相关基因; 功能标记; SNP; EST; KASP
麦红吸浆虫(Géhin)是严重威胁小麦生产的重要害虫, 吸浆虫以幼虫吸吮灌浆期小麦籽粒内含物, 使籽粒秕瘦、粒重降低, 更易受病原菌侵染, 导致小麦籽粒品质下降, 商品性受到严重影响。受害麦田产量损失在10%~60%之间, 严重时甚至颗粒无收[1-2]。在加拿大、美国、英国、波兰、德国等小麦主产国, 均有吸浆虫为害的报道[3-8]。在20世纪50年代和80年代, 我国小麦生产曾两度遭遇吸浆虫大面积为害, 损失巨大[9-10]。21世纪以来, 吸浆虫的危害依然严重。2013—2018年, 小麦主产区年均虫害发生面积超过2.0×106hm2(http://www. agri.gov.cn/), 成为影响华北、黄淮和西北等地小麦生产的重要虫害(https://www.natesc.org.cn/)。生产上, 可通过药物(农药)、农艺措施(深翻、轮作等)、生物(性引诱剂、天敌等)和抗虫品种防治麦红吸浆虫[4,11-12], 而以抗虫品种的选育和利用最为经济有效[13]。在20世纪, 西农6028等抗虫品种的使用对抑制虫害起到了关键作用, 而抗虫品种的缺乏也是当前我国麦田吸浆虫为害面积居高不下的重要原因[9]。但吸浆虫危害的隐蔽性、毁灭性以及小麦对吸浆虫抗性遗传的复杂性, 使得小麦抗虫育种进展缓慢[14-15]。传统的抗性育种单纯依赖于表型鉴定结果, 极易受环境和人为因素的影响, 而且费时费力, 抗虫品种的选育效率十分低下。分子标记辅助选择(marker-assisted selection, MAS)技术在小麦等主要作物抗性育种中的应用, 能够显著降低外界因素对抗虫鉴定结果的干扰, 也有利于实现作物抗虫性与农艺性状的同步改良, 进而提高育种效率[16-17]。开发与抗麦红吸浆虫基因/QTL紧密连锁的分子标记, 尤其是根据抗虫候选基因或抗虫基因序列上的突变位点开发成的目的基因标记(gene-targeted markers, GTMs)和功能标记(functional markers, FMs)[18-22], 不仅能够提高抗虫品种筛选及抗虫性选择的效率, 而且也有利于辅助实现不同来源抗虫基因的聚合, 使小麦的抗虫性更持久和稳定[23]。
是最早发现的小麦抗麦红吸浆虫基因[24], 位于2BS上SSR标记和与SCAR标记之间2.5 cM的遗传区间内[3]。和已在北美和英国的小麦抗虫性MAS育种中得以应用[25]。Kassa等[26]利用含有基因的小麦亲本组合构建的DH群体, 又发现了7个由单核苷酸多态性(SNP)位点开发的KASP标记与抗虫性紧密连锁, 可用于鉴定抗/感虫单倍型。也是当前加拿大、英国和美国的抗虫小麦品种中唯一被广泛利用的抗虫基因[27](http://midgetolerantwheat.ca/; http://www. bcpc.org/; https://www.usda.gov/)。过度依赖单一抗虫基因, 大大增加了新毒性吸浆虫生物型出现的风险, 也促使人们寻找新的抗虫基因资源[28]。
中国小麦品种可能携带了不同的麦红吸浆虫抗性基因。我国抗吸浆虫小麦品种多具有南大2419、西农6028和洛夫林10号等的血缘[9,14,29-32], 晋麦31、咸农151、武农99、郑州8号、冀麦23、冀麦24、河农215、荆麦66等抗虫品种的系谱中都或多或少携带有上述品种的血缘[9,14,30-31], 与国外抗虫小麦品种的系谱不同[3,24,27-28,33-34]。北美等地小麦的抗虫基因或QTL被定位于2B ()[24]和1A ()[28]染色体上, 以为主要抗源。我们发现, 来自我国小麦品种的抗虫性主要被定位于4A染色体()[14,35]。基因的连锁标记(、和)虽已被应用于当地小麦抗虫品种的培育, 但这些标记在我国小麦品种中要么不具多态性, 要么检测结果与抗虫性不吻合。阿魏酸等多酚类物质曾被认为是小麦籽粒中主要的抗虫成分[36-37], 与基因的抗性有关[3,26]。然而, 我们测定了4A主效QTL近等基因系的阿魏酸含量, 未能发现这种差异的存在。咖啡酸--甲基转移酶(COMT)是阿魏酸合成途径中的关键酶[38](https://www.kegg.jp/)。我们从吸浆虫胁迫下的小麦转录组数据中鉴定出3个COMT同源基因, 但在抗、感小麦亲本间均不存在SNP。上述研究表明, 我国小麦品种可能存在新的吸浆虫抗性遗传机制。鉴于此, 开发我国小麦种质中与抗虫位点紧密连锁甚至共分离的分子标记和功能标记, 不但能更好地满足我国抗虫性MAS育种的需要, 也有助于对我国小麦种质资源抗虫性的准确鉴定。
随着测序技术的改进、成本的降低, 以及多个物种参考基因组序列(包括中国春小麦参考基因组序列)的公开, 对基因组中大量序列变异和结构变异的挖掘成为可能, 对直接利用转录组数据或结合集群分离分析法(bulked segregant analysis, BSA)的思路进行标记开发变得更加便利[39]。王智兰等[40]对抗旱相关基因进行序列检测和分析, 根据该基因在不同小麦品种中存在的多态性位点开发了功能标记。Wu等[41]从转录组和芯片测序数据中挖掘到235个染色体特异性SNP位点, 并开发成KASP标记, 成功用于基因的精细定位。徐晓丹[42]利用BSR-Seq方法开发了SNP标记, 其中2个SNP标记对和D检测的有效率达97%和100%。
在前期研究中, 我们采用BSR-Seq[43]方法和QTL-Seq分析[44]的思路, 结合基因功能注释信息及qRT-PCR表达谱分析, 从抗、感虫小麦样本(含双亲、极端混池、重组近交系、近等基因系)的转录组数据中, 获得了位于4AL主效QTL[14,35]区间的6个抗虫性相关差异基因, 其中基因和不仅在全部抗、感RIL株系及NIL株系间的表达水平差异显著, 而且显著富集于与抗虫性相关的异黄酮生物合成代谢通路(isoflavonoid biosynthesis pathway)中[45-46]。因此, 本研究以上述6个抗虫相关基因及其SNP和插入缺失位点(InDel)为基础, 设计和开发目的基因标记, 并利用抗、感虫小麦RIL系和小麦品种验证了这些标记在小麦吸浆虫抗性鉴定中的可用性。
1 材料与方法
1.1 植物材料及DNA提取
用于标记验证的植物材料包括小麦亲本6218 (感虫)、冀麦24 (抗虫), 从6218/冀麦24重组近交系(RIL)群体中选择的92个抗、感吸浆虫株系以及具有不同抗性水平的95个小麦品种。其中, RIL株系来自小麦亲本6218与冀麦24的F2代群体的单粒传自交后代, 经多年抗虫鉴定, 表型稳定。供试小麦材料分别在2014—2015年度、2015—2016年度和2016—2017年度种植于河北农业大学育种中心虫圃。每个材料种植1行, 每行40粒, 行长20 cm, 行距20 cm[14]。完全随机区组设计, 3次重复。供试材料的名称及表型见表1。采用CTAB法[47]提取各供试材料的基因组DNA。
1.2 供试小麦材料的抗虫性鉴定
利用抗性指数(resistance index, RI)评价供试小麦材料的抗虫性[14]。在小麦灌浆至乳熟期, 取每重复、每株系10~15个穗子, 密封于纸袋中, 带回室内计数籽粒上的虫子。根据存在的吸浆虫数目, 将待鉴定小麦籽粒抗性分为5级。0级为籽粒上无虫; 1级为籽粒上有虫1头; 2级为籽粒上有虫2头; 3级为籽粒上有虫3头; 4级为籽粒上的虫数≥4头。根据以下公式计算每个株系的估计损失率()[2]。
(%) = Σ() / 4Σ× 100%
式中,为相应籽粒的抗性级别,为各级别的籽粒数目。
用每个株系的估计损失率(L)除以全部参试株系(品种)的平均估计损失率(ML), 计算出抗性指数(RI)来确定株系的抗性分级。当同一株系在不同重复间的鉴定结果存在较大差异时, 以最重重复数据进行L、RI值的估算[2]。基于RI值将供试小麦材料的抗性分为免疫(RI=0) (immune, I)、高抗(0.01≤RI<0.19) (highly resistant, HR)、中抗(0.20≤RI<0.49)(moderately resistant, MR)、低抗(0.50≤RI<0.99) (lowly resistant, LR)、感虫(1.00≤RI<1.50) (susceptible, S)和高感(RI≥1.50)(highly susceptible, HS)。
1.3 抗虫差异基因序列检测
以前期研究中获得的6个抗虫相关基因、、、、和进行抗虫标记的开发。根据与中国春参考序列(IWGSC RefSeq v1.0, https://wheat-urgi.versailles.inra.fr/)的比对结果, 提取上述基因的侧翼序列。用Primer5软件设计引物。用高保真酶在小麦亲本冀麦24和6218中扩增目的基因。10 μL PCR体系包括模板DNA 50 ng、正反引物各0.4 μmol L–1、1× Pfu PCR MasterMix (天根生化科技(北京)有限公司)。PCR条件为: (1) 94℃, 预变性3 min; (2) 94℃, 30 s; 55℃, 30 s; 72℃, 2 min; 共35个循环; (3) 72℃终延伸5 min。扩增产物经1%琼脂糖凝胶电泳分离, 回收目的条带, 送生工生物工程(上海)股份有限公司进行双向测序。将测序结果与中国春(IWGSCv1.0)和百农AK58 (v4.24)的基因组序列进行比对。
1.4 抗虫标记的开发
1.4.1 EST (expressed sequence tag, 表达序列标签)标记 根据测序结果, 分析目标基因在小麦亲本间的InDel, 并根据InDel两侧翼各200 bp的序列设计引物。采用普通DNA聚合酶(2× EsMasterMix)进行PCR扩增。10 μL PCR体系包括模板DNA 50 ng、正反引物各0.4 μmol L–1、1× EsMasterMix (北京康为世纪生物科技有限公司)。反应程序为: (1) 94℃, 预变性5 min; (2) 94℃, 30 s; 57℃, 30 s; 72℃, 1 min; 共35个循环; (3) 72℃终延伸7 min。扩增产物在30%的聚丙烯酰胺凝胶上电泳, 银染显色。
1.4.2 KASP (kompetitive allele-specific PCR, 竞争性等位特异PCR)标记 以抗虫相关基因在冀麦24 (高抗吸浆虫)与6218 (高感吸浆虫)、百农AK58 (高感吸浆虫)间均表现出多态性的SNP位点及其侧翼序列与小麦参考序列(IWGSCv1.0)进行比对, 挑选出在基因组中具有特异序列或其中一种SNP等位型(突变类型)在参考基因组中不存在的SNP, 进行KASP标记的设计与开发。提取选定SNP在参考基因组中的侧翼序列(>200 bp), 由艾吉析科技(上海)有限公司(LGC Science Shanghai Ltd.)进行引物的设计与合成, 在两条KASP正向引物序列的5′端加上FAM (6-carboxy-fluorescein)或HEX (Hexachlorofluorescein)荧光基团的接头序列。参照公司提供的程序进行KASP标记的分型试验。反应体系总体积为3 μL, 含KASP (V4.0) 2× Mastermix 1.5 μL, 引物mix (72×) 0.0417 μL, DNA模板50~100 ng左右。PCR为Touchdown反应程序, 94℃变性15 min; 94℃变性20 s, 68℃退火60 s, 每循环一次下降0.6℃, 10个循环; 94℃变性20 s, 62℃退火60 s, 26个循环。PCR扩增完毕后, 利用LGC系统的SNPline平台进行SNP分型检测, 利用Kraken (v16.3.16.16288)软件分析扫描数据。
2 结果与分析
2.1 基因序列分析
6个抗虫相关基因在抗、感小麦亲本冀麦24和6218中均能扩增到目的片段。除外, 其他5个基因在抗、感小麦亲本中的序列与参考基因组序列(中国春和百农AK58)相符;在抗虫亲本冀麦24的序列与参考基因组序列相符, 而在感虫亲本6218的序列与参考基因组序列差异较大。
上述基因在抗、感小麦亲本间均存在SNP, 且都包含使氨基酸发生改变的非同义突变位点(图1)。由于6218和百农AK58均高感麦红吸浆虫, 因此那些同时存在于冀麦24与6218之间、冀麦24与百农AK58之间的多态性SNP, 被视为高可信度SNP。基因中存在4个非同义突变SNP, 其中3个为可信度较高的SNP。基因中存在6个非同义突变SNP, 其中4个为可信度较高的SNP。存在1个高可信度的非同义突变的SNP。存在SNP和插入/缺失(InDel)各1个, 其中抗虫品种(冀麦24)中存在一个12 bp的插入, 而感虫品种(系)(6218和百农AK58)则缺失了这12 bp。由于未能获得在感虫亲本(6218)中的扩增序列, 故根据该基因在抗虫亲本冀麦24与感虫品种百农AK58间存在的2个SNP位点进行标记开发。在抗、感小麦亲本中分别存在1个4 bp和1 bp的插入, 由于这种插入可能导致其后的读码框发生改变(移码突变), 因此重新预测了该基因在抗、感虫小麦亲本中的所有开放阅读框(https://web.expasy.org/), 分别获得在抗、感小麦亲本中使编码蛋白仍具有丝氨酸蛋白酶抑制剂(SPI)结构域的编码序列, 发现在编码区序列上存在11个非同义突变位点, 其中1个为可信度较高的SNP; 此外, 该基因在冀麦24的基因下游处存在一个29 bp的插入, 而在感虫亲本则缺失了这29 bp的序列。
以这些高可信度的SNP位点及其侧翼序列与小麦参考基因组序列(IWGSCv1.0)的比对结果, 对基因组特异性或突变等位型特异性(即仅在目的基因序列中的特定碱基位置上具有抗虫亲本的突变类型, 而在参考基因组中全部同源序列对应的碱基位点上均不存在该突变类型)的SNP, 进行KASP标记开发。根据比对结果, 选择基因上存在的2个特异性位点,、、和上存在的各1个特异性位点, 共6个可信度较高的特异性SNP用于KASP标记的开发, 选择基因和序列中存在的InDel, 用于EST标记的开发。
2.2 抗虫标记的开发及多态性检测
根据上述基因中存在的SNP和InDel, 分别设计出6个KASP标记(、、、、和)和2个EST标记(和), 用这些标记检测了抗、感虫小麦亲本、RIL株系和小麦品种。2个EST标记在小麦亲本间均表现出多态性, 其中在抗、感虫亲本中分别扩增出173 bp和161 bp的片段,分别扩增出211 bp和182 bp的片段(图2)。6个KASP标记在抗、感虫小麦亲本间亦具有多态性(图3)。标记、、和在小麦亲本中均为纯合分型, 在抗虫亲本中分别为A:A、T:T、T:T和G:G, 在感虫亲本中分别为C:C、G:G、A:A和A:A。和在抗虫小麦亲本中为杂合分型(分别为T:G和C:G), 在感虫亲本中表现纯合分型(均为G:G)。将和的2条正向引物序列与小麦参考基因组序列(IWGSCv1.0)比对后发现, 感虫位点对应序列在参考基因组中均存在多个匹配(数据未列出)。因此, 在抗、感虫小麦亲本中, 这2个标记均能检测出感虫等位型; 而抗虫位点对应序列与参考基因组间不存在3′末端匹配, 即存在于抗虫小麦品种冀麦24中的突变型位点在参考基因组中不存在, 因此在不具有该突变的材料中无法检测到抗虫标记位点。综上, 这些标记可用于检测小麦品种或株系中4A染色体抗虫QTL。
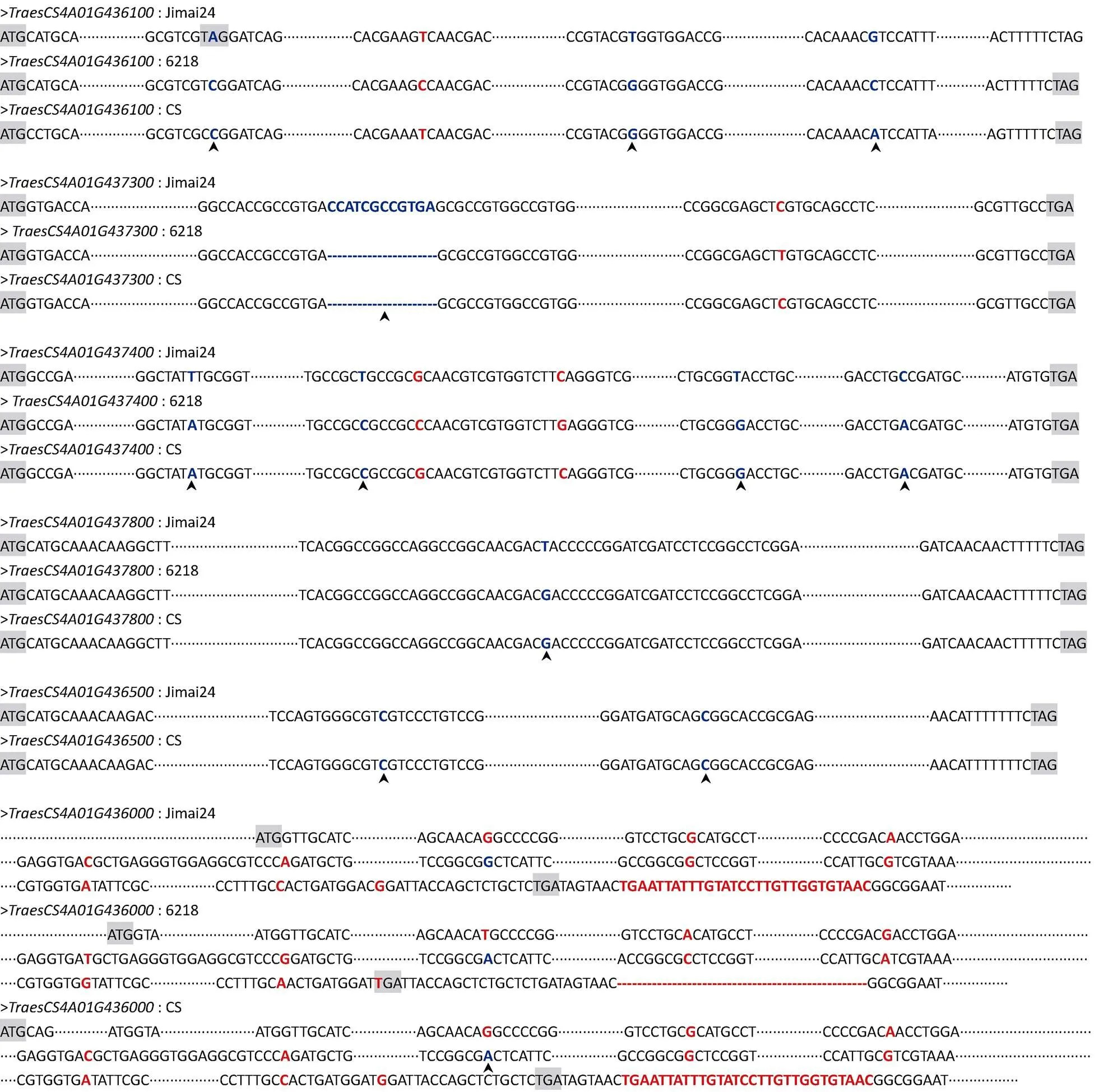
图1 6个差异基因在抗(冀麦24)、感(6218)小麦亲本及中国春参考序列之间的比对结果
蓝色或红色碱基代表非同义突变的SNP位点或插入缺失, 其中蓝色碱基为可信度较高的SNPs, 并以箭头标注。
Red or blue font letters represent non-synonymous SNPs and InDels present in the genes. Blue letters with arrowheads represent the SNPs with high confidence.
2.3 EST、KASP标记在小麦抗、感虫RIL系中的检测结果
用这些标记检测了47个抗虫(RI= 0.0330~ 0.4673)、45个感虫(RI= 1.2325~6.3697) RIL系(表1)。所有KASP标记和EST标记在抗、感虫RIL株系中的检测结果(带型或分型)与相应抗、感小麦亲本的吻合度较高。KASP标记能够在85%以上的抗虫株系中检测出抗虫标记位点(其中标记和为杂合分型), 在95%以上的感虫株系中检测出感虫标记位点(表2)。两个EST标记, 均能够在85%以上的抗虫株系中扩增出与抗虫亲本一致的带型, 在超过90%的感虫株系中扩增出与感虫亲本一致的带型(表2)。如果忽略个别标记在个别株系中的缺失情况(标记、、和均分别在2个RIL株系中缺失, 标记在4个RIL株系中存在缺失), KASP标记在所检测RIL株系间的抗、感分型结果完全一致, 说明这些标记均应位于同一个染色体交换区段内, 并且可能与抗虫基因紧密连锁或共分离。
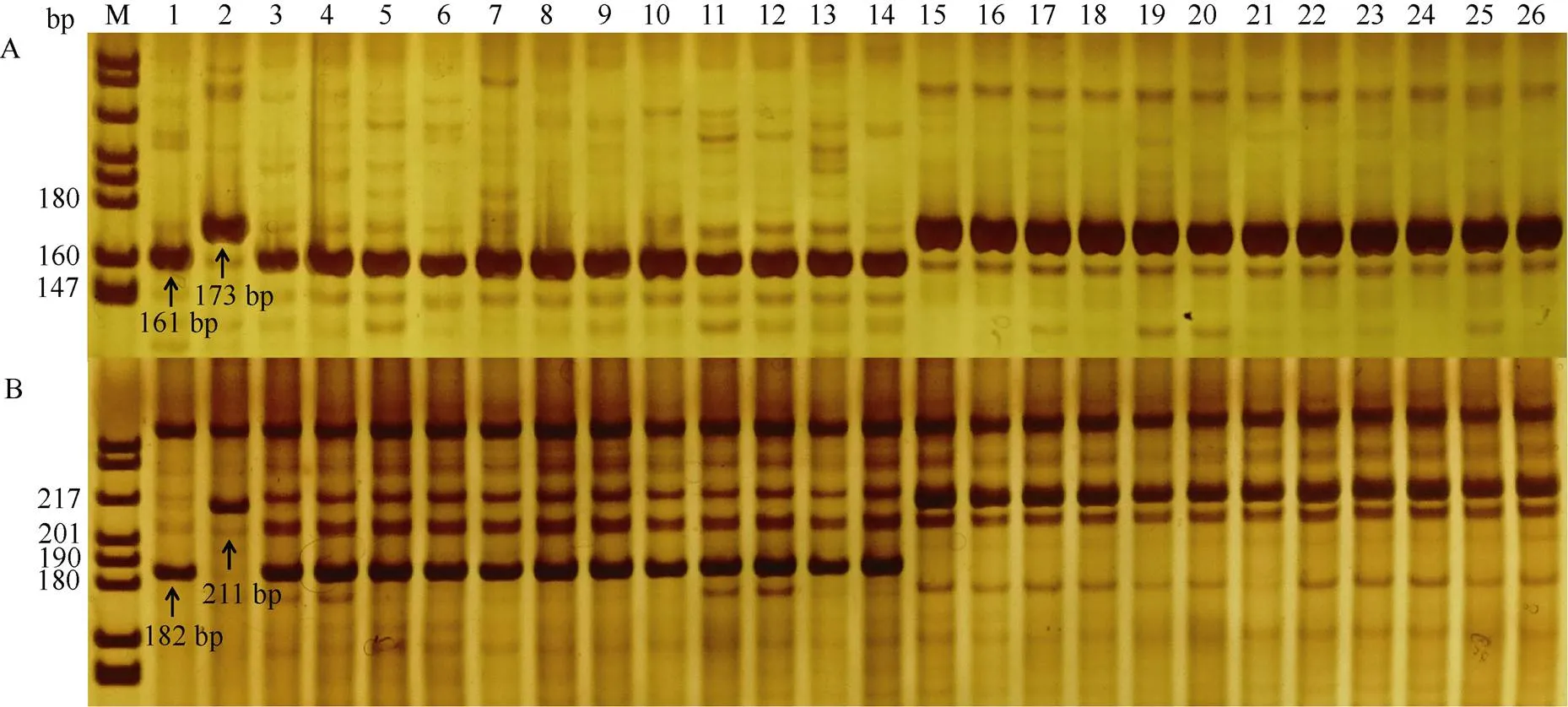
图2 EST标记E1-2 (A)和E10-10 (B)在抗、感虫小麦亲本及部分RIL系中的扩增结果
M: 分子量marker (pBR322/I酶切片段); 1: 感虫小麦亲本6218; 2: 抗虫小麦亲本冀麦24; 3~14: 感虫株系(RIL-16、RIL-19、RIL-20、RIL-21、RIL-25、RIL-29、RIL-45、RIL-102、RIL-113、RIL-119、RIL-125和RIL-139); 15~26: 抗虫株系(RIL-7、RIL-12、RIL-18、RIL-23、RIL-64、RIL-73、RIL-91、RIL-115、RIL-134、RIL-156、RIL-169和RIL-170)。
M: Molecular marker (restriction fragments of plasmiddigested withI endonuclease); 1: Susceptible wheat parent ‘6218’; 2: Resistant parent ‘Jimai 24’; 3–14: Susceptible lines (RIL-16, RIL-19, RIL-20, RIL-21, RIL-25, RIL-29, RIL-45, RIL-102, RIL-113, RIL-119, RIL-125, and RIL-139); 15–26: Resistant lines (RIL-7, RIL-12, RIL-18, RIL-23, RIL-64, RIL-73, RIL-91, RIL-115, RIL-134, RIL-156, RIL-169, and RIL-170).
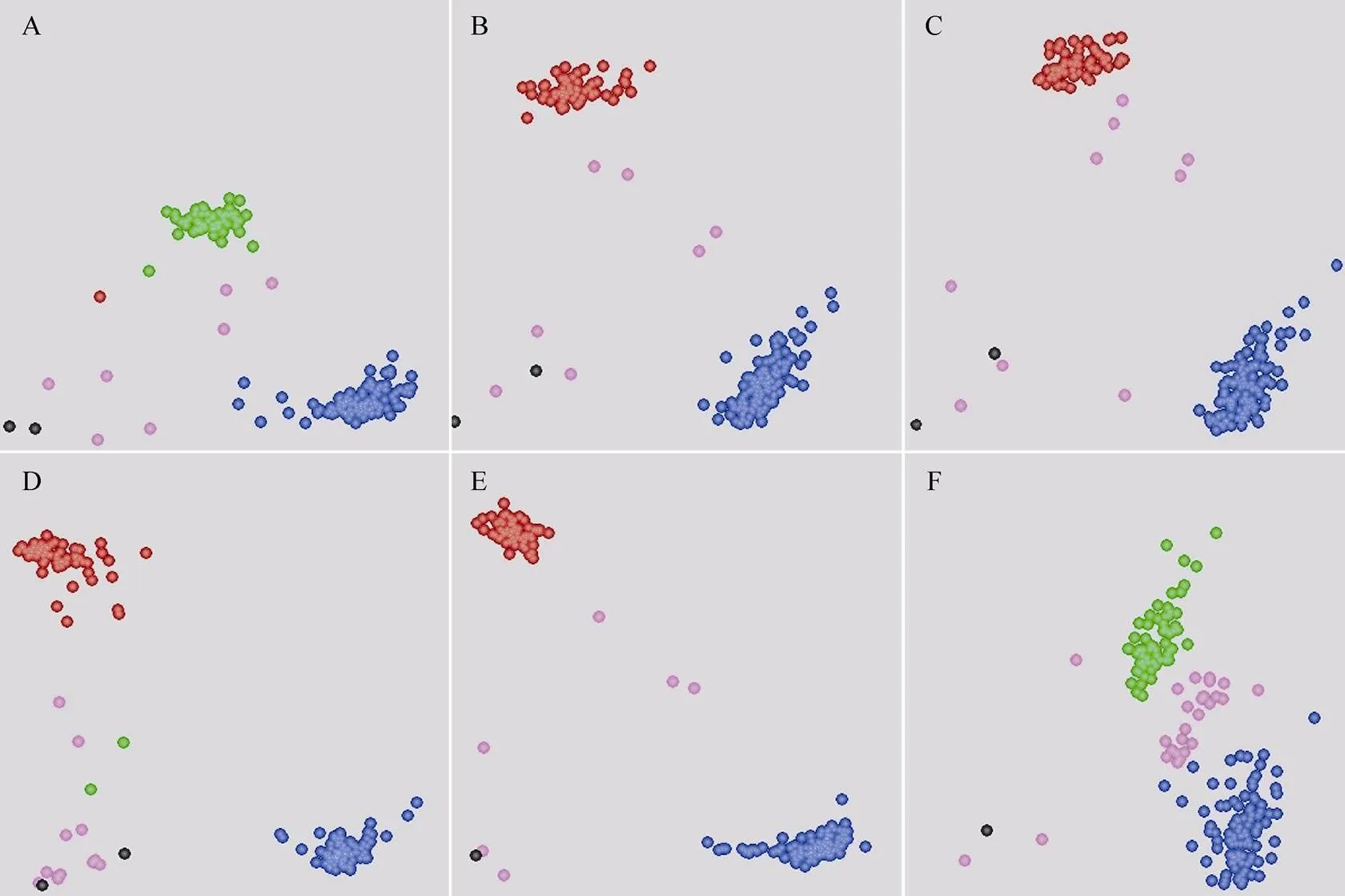
图3 KASP标记在抗、感虫小麦亲本、92个RIL系和95个小麦品种中的分型结果
红色点, HEX基因型; 蓝色点, FAM基因型; 绿色点, 杂合基因型; 粉色点, 缺失; 黑色点, 对照. 图中A~F分别为KASP标记、、、、和的分型结果。
Red dots represent the HEX-type allele; blue dots, the FAM-type allele; green dots, the heterozygous-type allele; pink dots, undetected; black dots, the NTC (non-template control); A to F refer to genotypes for,,,,, and, respectively.

表1 供试小麦亲本与RIL株系的表型

表2 EST标记和KASP标记在供试RIL株系中的标记基因型及比例
a: 物理位置为标记在4A染色体上的物理位置; A: 与抗虫亲本冀麦24一致的基因型; B: 与感虫亲本6218一致的基因型; H: 杂合基因型。
a: Physical position refers to the physical position of EST or KASP markers on chromosome 4A; A: genotype identical to the resistant wheat parent ‘Jimai 24’; B: genotype identical to the susceptible wheat parent ‘6218’; H: heterozygous genotype.
2.4 EST、KASP标记在不同抗性小麦品种中的检测结果
利用95个不同抗性的小麦品种为材料(表3), 对上述标记的适用性进行了检测。标记能够在37.2%的抗虫小麦品种中扩增出纯合抗虫带型, 在80%的感虫品种中扩增出纯合感虫带型(表4)。由于在感虫小麦品种中的检测有效率较高(为80.0%), 因此该标记对具有抗虫标记位点小麦品种的鉴定准确率为80%左右。在对小麦品种进行检测时, 则只要在测试品种中检测出抗虫带型, 那么该品种为抗虫品种(即具有抗虫位点)的可能性在80%左右, 可作为筛选抗虫种质资源的标记。而标记在抗、感虫小麦品种中所检测出的抗、感基因型所占比例相近(表4), 不能区分抗、感虫小麦品种, 因此该标记不能用于鉴定供试小麦品种的抗虫性。
KASP标记、和在超过30% (分别为30.8%、31.6%和37.5%)的抗虫小麦品种中检测到纯合抗虫标记位点, 在85%以上(分别为92.3%、92.3%和87.2%)的感虫小麦品种中检测到纯合感虫标记位点(表4)。这说明, 如果能在品种中检测到这些抗虫标记位点, 则该供试品种为抗虫品种(即具有抗虫位点)的可能性高于85%、为感虫品种(基因型与表型不相符)的概率低于15%, 因此这3个标记可以较好地筛选具有位点的抗虫小麦种质。
标记, 能够在62.5%的抗虫品种中检测出纯合抗虫标记位点, 在76.9%的感虫品种中检测出纯合感虫标记位点(表4), 检测有效率为70.4%, 因此该标记亦可作为品种抗虫性的筛选标记。
标记和在抗虫小麦亲本中为杂合分型。分型结果表明, 2个标记能够在超过30% (分别为35.0%和38.5%)的抗虫小麦品种中检测到抗虫标记位点, 在超过75% (分别为92.3%和79.2%)的感虫品种中检测到纯合感虫标记位点(表4), 即, 如果和检测到抗虫标记位点, 则该供试品种为抗虫品种(即具有抗虫位点)的可能性在75%以上, 而为感虫品种(基因型与表型不相符)的概率不到10%和25%。因此标记比能更好地检测出具有位点的抗虫小麦种质。
进一步分析7个标记、、、、、和在各不同抗性小麦品种的分型结果(不计检测缺失的品种)表明, 这些标记在高抗小麦品种中的平均检测有效率为64.7% (56.3%~86.7%), 在中抗品种中的平均检测有效率为19.1% (13.0%~41.2%), 在感虫品种中为66.7% (50.0%~75.0%), 在高感品种中为94.1% (85.7%~100.0%) (图4)。同样, 如果只考虑高抗和高感两类极端类型品种, 则这7个标记对抗性品种的平均检测有效率为64.7% (56.3%~86.7%), 低于对抗虫RIL系的检测有效率(平均86.8%, 范围85.1%~ 88.6%); 对感虫品种的平均检测有效率为94.1% (85.7%~100.0%), 与对感虫RIL系的检测有效率接近(平均95.5%, 范围95.5%~95.6%)(图4)。标记在RIL系间检测率较高, 与RIL系具有较为一致的遗传背景密不可分, 而标记在高抗RIL系间、高抗品种间的检测有效率低于对应高感RIL系和高感品种的事实, 再次说明小麦对麦红吸浆虫抗性的数量遗传特征。

表3 供试小麦品种的表型
根据3个检测率较高的标记、和在95个小麦品种中的分型结果可以看出, 这3个标记在11个抗虫小麦品种(RI = 0.0235~0.4798)中的分型与抗虫亲本冀麦24的分型完全一致(表5), 这11个品种分别是中农28 (1932年引进种质)、西农6028 (1956年审定, 下同)、丰产2号(1968)、晋麦33 (1985)、冀麦23 (1986)、河农215 (与冀麦23、冀麦24互为姊妹系)、邯麦9号(2003)、石家庄11号(2003)、石麦12号(2004)、河农4198(2005)和河农6049 (2008)。
综合EST和KASP标记的检测结果, 共有20个小麦品种[包括14个抗虫品种, 5个感虫或中间型(低抗)品种, 1个无表型鉴定结果的品种(咸农39)]都被检测出具有较多的抗虫标记位点(分型与带型同抗虫亲本冀麦24), 且这20份品种在任何标记位点上均未被检测出感虫标记位点, 因此说明这些材料在4AL抗虫标记位点附近的基因组区段可能是相同的。而在一些感虫小麦品种被检测出具有文中所有抗虫位点的现象, 也间接说明了基因组内可能有与4AL抗虫主效位点互作的基因(如抑制效应的上位基因等)存在。

表4 EST标记和KASP标记在供试小麦品种中的标记基因型及比例
A: 与抗虫亲本冀麦24一致的基因型; B: 与感虫亲本6218一致的基因型; H: 杂合基因型。
A: genotype identical to the resistant wheat parent ‘Jimai 24’; B: genotype identical to the susceptible wheat parent ‘6218’; H: heterozygous genotype.

表5 在7个抗性标记位点上完全一致的11个小麦品种的抗虫性及其标记基因型
a: 与抗虫亲本冀麦24一致的基因型。
a: genotype identical to the resistant wheat parent ‘Jimai 24’.

图4 抗(47个)、感(45个)虫小麦RIL株系、高抗品种(19个)、中抗品种(24个)、低抗品种(12个)、感虫品种(12个)和高感品种(28个)中的标记基因型所占比例
3 讨论
3.1 功能标记的检测有效性
功能标记是一类可通过对个体基因型的间接检测来反应目标性状(表型)的分子标记, 理论上对表型的检测率应达到或接近100%, 能够准确检测作物种质资源是否含有目的基因[18,20,22]。但由于影响表型变异的基因或功能位点可能不止一个, 实际开发出的功能标记不一定均能在全部参试材料中获得基因型与相应的表型完全一致的结果, 这对复杂性状尤为明显。伍玲等用//位点的功能标记[48]和、、、、[49], 在273份CIMMYT来源的小麦材料中获得高达96.7%的标记检测有效率; 但将这些标记用于检测中国小麦地方品种时却发现, 25.8%的标记阳性品种却表现完全感病[50]。张帆等以抗旱相关基因开发出的4个显性互补标记, 虽然能够对我国150份小麦品种的种子萌发期抗旱性进行检测, 但仍有6%的弱抗旱性材料被检测为抗旱基因型, 2%的抗旱性较强材料被检测为弱抗旱基因型[51]。在用功能标记检测小麦多酚氧化酶(PPO)、面粉黄色素含量时, 也发现功能标记检测与表型不完全一致的现象[52-55]。这些现象可归因于目的位点上其他变异的存在(包括互作位点)影响到目的基因的表达[50-51], 同时也不能排除品种遗传背景差异和环境因素的影响[56]。本研究基于转录组数据挖掘到的抗吸浆虫差异基因, 开发出8个抗虫性基因标记(2个EST和6个KASP标记)。这些标记在遗传背景较近的RIL系中能够达到很高的检测有效率(~90%), 在遗传背景差异较大的小麦品种中, 这些标记(不包括)在感虫品种中的检测有效率(76.9%~92.3%)远高于在抗虫品种中的检测有效率(30.8%~62.5%), 这说明抗虫位点()之外的其他抗虫基因、互作因子[50-51]和遗传背景的差异均可能对标记的检测效率产生影响。因此, 本研究开发的这7个标记可应用于选择小麦种质资源位点的抗虫等位基因型, 为提高小麦吸浆虫抗性的重要手段。同时, 鉴定、开发小麦基因组中存在的其他遗传因子及标记, 对于揭示小麦抗吸浆虫分子机制和提高小麦抗虫分子标记的检测效率也将十分重要。
3.2 抗虫相关标记的应用前景及展望
本研究将RNA-Seq技术和BSA分析思路应用于小麦抗吸浆虫差异基因发掘及标记开发工作, 从多组抗、感吸浆虫小麦材料转录组数据中发掘的差异基因, 经qRT-PCR、KEGG Pathway及基因功能注释等分析过程, 初步证实了6个差异基因可能与抗虫性相关; 并根据这些基因在小麦抗、感亲本之间存在的序列差异, 开发出8个标记, 能够较好地分辨抗/感RIL株系。其中7个标记、、、、、和在供试感虫小麦品种中的检测有效率较高(76.9%~92.3%, 表4), 如果只考虑高感品种, 检测有效率则更高(85.7%~ 100.0%, 图4)。理论上说, 感虫品种不应携带任何抗虫等位基因, 如果在某些感虫品种中存在抗性位点, 那么该位点就不能完全准确预测品种的抗虫性。因此, 能够准确预测小麦品种抗虫性、并可应用于MAS育种中的标记, 应在感虫品种中的检测有效率高(尽可能达到100%)。本研究中, 标记、和在供试抗虫小麦品种中的检测有效率为30%左右(30.8%~35.0%), 在供试感虫小麦品种中的检测有效率超过90% (为92.3%), 如果用这3个标记从小麦品种中检测出全部抗虫标记位点, 则该品种为抗虫品种(具有)的可能性应超过90%, 因此这3个标记可较好地作为筛选具有抗虫位点小麦种质的标记。同时, 从图4也可看出, 小麦品种的吸浆虫抗性越好, 所用标记的抗虫等位型(resistant alleles)所占比例相对越高, 因此这3个标记可以很好地用于品种是否具有抗虫位点的检测, 这对我们鉴别抗虫种质以及早代育种材料的抗虫性具有重要意义。本研究中, 供试小麦品种中有11个抗虫品种被检测到携带了这3个标记的全部抗虫等位基因(表5), 这些品种多为生产上已经停止推广的老品种, 比较近的品种其审定时间也在10年以上, 因此如何更有效地使用比较老的小麦种质创新小麦抗虫资源, 任务十分迫切。
这种基于混池转录组测序(BSR-Seq)并结合生物信息学工具、qRT-PCR、基因测序和遗传定位信息等的方法, 不但可快速挖掘用于开发标记的候选基因及功能位点, 提升潜在功能标记开发速度, 还能缩小差异基因范围、提升标记与目标性状紧密相关的几率。虽然我们目前的工作尚未实现对小麦抗虫主效QTL的图位克隆, 但利用该方法所获得潜在候选基因的抗虫相关标记, 在不同作图群体中被证明是有用的, 不仅能被用于对小麦种质资源抗虫性的辅助鉴定, 同时可对小麦抗吸浆虫主效QTL的精细定位和图位克隆提供重要帮助。
4 结论
基于6个小麦抗吸浆虫相关基因序列中存在的差异位点, 成功设计并开发了8个基因标记, 这些标记在抗、感小麦RIL株系间的分型结果与对应株系的抗虫性水平相符度在90%左右, 在不同小麦品种中的检测有效率变异较大。标记、和能够从超过30% (30.8%~35.0%)抗虫小麦品种中检测到抗虫等位型, 从90% (92.3%)以上的感虫品种中检测到感虫等位型, 对于那些均能检测到抗虫标记等位型的小麦品种, 其具有4AL抗虫主效位点的可能性超过90% (为92.3%), 可用于小麦抗虫性种质资源筛选和分子标记辅助育种。此外, 包含全部抗虫等位标记的11个抗虫小麦品种, 多已或即将停止推广, 这为如何有效利用老品种的抗虫性创新小麦新种质提出了十分迫切的要求。
致谢: 感谢中国农业科学院杨欣明研究员提供了部分小麦种质, 感谢中国农业科学院作物科学研究所贾继增研究员、赵光耀研究员提供了百农AK58 (v4.24)参考基因组序列并进行的相关分析, 感谢本课题组张力菁、章悦等在抗虫鉴定等方面的大力帮助。
[1] 屈振刚, 温树敏, 屈赟, 刘桂茹. 小麦品种抗麦红吸浆虫鉴定与抗性分析. 植物遗传资源学报, 2011, 12: 121–124. Qu Z G, Wen S M, Qu Y, Liu G R. Evaluation and identification of wheat varieties resistant to., 2011, 12: 121–124 (in Chinese with English abstract).
[2] 温树敏, 赵玉新, 屈振刚, 刘桂茹, 王琳琳, 王金耀. 小麦品种抗麦红吸浆虫鉴定及抗性评价. 河北农业大学学报, 2007, 30(5): 71–74. Wen S M, Zhao Y X, Qu Z G, Liu G R, Wang L L, Wang J Y. The utilization and evaluation of resistance in wheat varieties to., 2007, 30(5): 71–74 (in Chinese with English abstract).
[3] Thomas J, Fineberg N, Penner G, McCartney C, Aung T, Wise I, McCallum B. Chromosome location and markers of: a gene of wheat that conditions antibiotic resistance to orange wheat blossom midge., 2005, 15: 183–192.
[4] Bruce T J, Hooper A M, Ireland L, Jones O T, Martin J L, Smart L E, Oakley J, Wadhams L J. Development of a pheromone trap monitoring system for orange wheat blossom midge,, in the UK., 2007, 63: 49–56.
[5] Flodrops Y, Taupin P. Soft wheat: combating the orange blossom midge., 2010, 365: 46–50.
[6] Jacquemin G, Chavalle S, De P M. Forecasting the emergence of the adult orange wheat blossom midge,(Géhin) (Diptera: Cecidomyiidae) in Belgium., 2014, 58: 6–13.
[7] Gaafar N, Volkmar C, Cöster H, Spilke J. Susceptibility of winter wheat cultivars to wheat ear insects in central Germany., 2011, 62: 107–115.
[8] 赵中华, 王强, 朱晓明. 2015年全国小麦病虫害发生新特点与防治新思路. 中国植保导刊, 2016, 36(8): 33–36. Zhao Z H, Wang Q, Zhu X M. New patterns of wheat pest and disease infection in 2015 and new ideas for control in China., 2016, 36(8): 33–36 (in Chinese).
[9] 尹青云, 郑王义, 谢咸升, 李峰, 范绍强, 郑晓玲. 小麦品种对麦红吸浆虫的抗性及抗性种质资源创新应用研究进展. 麦类作物学报, 2003, 23(2): 88–91. Yin Q Y, Zheng W Y, Xie X S, Li F, Fan S Q, Zheng X L. Advances in the study on wheat variety resistance to wheat midge and innovation and application of resistance germplasm resources., 2003, 23(2): 88–91 (in Chinese with English abstract).
[10] 段云, 蒋月丽, 苗进, 巩中军, 李彤, 武予清, 罗礼智. 麦红吸浆虫在我国的发生、危害及防治. 昆虫学报, 2013, 56: 1359–1366. Duan Y, Jiang Y L, Miao J, Gong Z J, Li T, Wu Y Q, Luo L Z. Occurrence, damage and control of the wheat midge,(Diptera: Cecidomyiidae), in China., 2013, 56: 1359–1366 (in Chinese with English abstract).
[11] Olfert O, Elliott R H, Hartley S. Non-native insects in agriculture: strategies to manage the economic and environmental impact of wheat midge,, in Saskatchewan., 2009, 11: 127–133.
[12] Gaafar N, El-Wakeil N, Volkmar C. Assessment of wheat ear insects in winter wheat varieties in central Germany., 2011, 84: 49–59.
[13] 屈振刚, 温树敏, 赵玉新, 路子云, 孙会. 河北省麦红吸浆虫为害逐年加重的原因及防治对策. 河北农业科学, 2006, 10(1): 102–104. Qu Z G, Wen S M, Zhao Y X, Lu Z Y, Sun H. Reason analysis and control strategies on the gradually increasedGehin damage in Hebei province., 2006, 10(1): 102–104 (in Chinese with English abstract).
[14] 郝燕冉, 温树敏, 王睿辉, 安雪娇, 刘桂茹. 小麦品种冀麦24抗麦红吸浆虫QTL定位. 植物遗传资源学报, 2017, 18: 933–938. Hao Y R, Wen S M, Wang R H, An X J, Liu G R. QTL analysis for midge resistance in wheat cultivar Jimai 24., 2017, 18: 933–938 (in Chinese with English abstract).
[15] 黎丹. 河北省麦红吸浆虫发生规律、成虫测报方法及品种抗虫性鉴定的研究. 河北农业大学研究生院硕士学位论文, 河北保定, 2012. Li D. Study on Occurrence Regularity, the Method of Prediction of Adults, Analysis and Evaluation of Resistance of Wheat Midge,Géhin. MS Thesis of Agricultural University of Hebei, Baoding, Hebei, China, 2012 (in Chinese with English abstract).
[16] 仇松英, 史晓芳, 史忠良, 张松令, 谢福来, 高炜, 逯腊虎. 小麦抗麦红吸浆虫品种遗传多样性的表型和SSR标记分析. 麦类作物学报, 2011, 31: 1050–1056. Qiu S Y, Shi X F, Shi Z L, Zhang S L, Xie F L, Gao W, Lu L H. Genetics diversity of wheat midge resistant varieties by phenotype and simple sequence repeat (SSR) markers analysis., 2011, 31: 1050–1056 (in Chinese with English abstract).
[17] 瞿华香, 张玉烛, 张岳平, 曾翔, 屠乃美. 分子标记辅助选择育种研究进展. 作物研究, 2008, 22: 355–358. Qu H X, Zhang Y Z, Zhang Y P, Zeng X, Tu N M. Progress on marker-assisted selection., 2008, 22: 355–358 (in Chinese).
[18] 王昊龙, 韩俊杰, 李淼淼, 南富波, 李卫华. 功能标记的开发及在禾谷类作物中的应用. 核农学报, 2014, 28: 1963–1971. Wang H L, Han J J, Li M M, Nan F B, Li W H. Development and application of functional markers in cereal crops., 2014, 28: 1963–1971 (in Chinese with English abstract).
[19] Li G, Quiros C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica., 2001, 103: 455–461.
[20] 刘国圣, 张大乐. 功能性分子标记在小麦育种中的应用. 生物技术通报, 2016, 32(11): 18–29. Liu G S, Zhang D L. The application of the functional molecular marker in wheat breeding., 2016, 32(11): 18–29 (in Chinese with English abstract).
[21] Andersen J R, Lübberstedt T. Functional markers in plants., 2003, 8: 554–560.
[22] 贺道华, 雷忠萍, 邢宏宜. 功能标记的开发、特点和应用研究进展.西北农林科技大学学报(自然科学版), 2009, 37(1): 110–116. He D H, Lei Z P, Xing H Y. Development progress, characteristics and application of functional marker.(Nat Sci Edn), 2009, 37(1): 110–116 (in Chinese with English abstract).
[23] 沈新莲, 张天真. 作物分子标记辅助选择育种研究的进展与展望. 高技术通讯, 2003, (2): 105–110. Shen X L, Zhang T Z. Advances on molecular marker-assisted selection in crop., 2003, (2): 105–110 (in Chinese with English abstract).
[24] McKenzie R I H, Lamb R J, Aung T, Wise I L, Barker P, Olfert O O, McIntosh R I. Inheritance of resistance to wheat midge,, in spring wheat., 2002, 121: 383–388.
[25] Randhawa H S, Asif M, Pozniak C, Clarke J M, Graf R J, Fox S L, Humphreys D G, Knox R E, DePauw R M, Singh A K, Cuthbert R D, Hucl P, Spaner D, Gupta P. Application of molecular markers to wheat breeding in Canada., 2013, 132: 458–471.
[26] Kassa M T, Haas S, Schliephake E, Lewis C, You F M, Pozniak C J, Krämer I, Perovic D, Sharpe A G, Fobert P R, Koch M, Wise I L, Fenwick P, Berry S, Simmonds J, Hourcade D, Senellart P, Duchalais L, Robert O, Förster J, Thomas J B, Friedt W, Ordon F, Uauy C, McCartney C A. A saturated SNP linkage map for the orange wheat blossom midge resistance gene., 2016, 129: 1507–1517.
[27] Fox S L, Lamb R J, McKenzie R I H, Wise I L, Smith M A H, Humphreys D G, Brown P D, Townley-Smith T F, McCallum B D, Fetch T G, Menzies J G, Gilbert J A, Fernandez M R, Despins T, Lukow O, Niziol D. Registration of ‘Fieldstar’ hard red spring wheat., 2012, 6: 161–168.
[28] Blake N K, Stougaard R N, Weave D K, Sherman J D, Lanning S P, Naruoka Y, Xue Q, Martin J M, Talbert L, E. Identification of a quantitative trait locus for resistance to(Géhin), the orange wheat blossom midge, in spring wheat., 2011, 130: 25–30.
[29] 李建军, 李修炼, 成卫宁. 小麦吸浆虫研究现状与展望. 麦类作物, 1999, 19(3): 52–55. Li J J, Li X L, Cheng W N. Status and prospect on wheat blossom midge., 1999, 19(3): 52–55 (in Chinese).
[30] 成卫宁, 李修炼, 李建军, 辛转霞. 小麦品种抗麦红吸浆虫的研究现状与展望. 麦类作物学报, 2003, 23(3): 132–135. Cheng W N, Li X L, Li J J, Xin Z X. Status and prospect of studies on wheat blossom midge resistance of wheat varieties., 2003, 23(3): 132–135 (in Chinese with English abstract).
[31] 赵菊香, 任芝英. 小麦品种资源对麦红吸浆虫的抗性鉴定. 山西农业科学, 1990, (4): 5–6. Zhao J X, Ren Z Y. Evaluation of resistance in wheat cultivars and germplasms to(Géhin)., 1990, (4): 5–6 (in Chinese).
[32] Zheng D S. Use of Italian wheat varieties in China., 1993, 40: 137–142.
[33] Lamb R J, McKenzie R I H, Wise I L, Barker P S, Smith M A H, Olfert O O. Resistance to(Diptera: Cecidomyiidae) in spring wheat (Graminae)., 2000, 132: 591–605.
[34] Berzonsky W, Ding H, Haley S D, Harris M O, Lamb R J, McKenzie R I H, Ohm H W, Patterson F L, Peairs F B, Porter D R, Ratcliffe R H, Shanower T G. Breeding wheat for resistance to insects., 2003, 22: 221–297.
[35] 张哲. 小麦种质资源抗吸浆虫鉴定及分子标记筛选. 河北农业大学研究生院硕士学位论文, 河北保定, 2018. Zhang, Z. Screening for Midge-resistance Wheat Germplasm and Their Linked DNA-based Molecular Markers. MS Thesis of Agricultural University of Hebei, Baoding, Hebei, China, 2018 (in Chinese with English abstract).
[36] Ding H, Lamb R J, Ames N. Inducible production of phenolic acids in wheat and antibiotic resistance to., 2000, 26: 969–985.
[37] Abdel-Aal E S M, Hucl P, Sosulski F W, Graf R, Gillott C, Pietrzak L. Screening spring wheat for midge resistance in relation to ferulic acid content., 2001, 49: 3559–3566.
[38] Wu X, Wu J, Luo Y, Bragg J, Anderson O, Vogel J, Gu Y Q. Phylogenetic, molecular, and biochemical characterization of caffeic acid-methyltransferase gene family in., 2013, 423189.
[39] 李小白, 向林, 罗洁, 胡标林, 田胜平, 谢鸣, 孙崇波. 转录组测序(RNA-seq)策略及其数据在分子标记开发上的应用. 中国细胞生物学学报, 2013, 35: 720–726. Li X B, Xiang L, Luo J, Hu B L, Tian S P, Xie M, Sun C B. The strategy of RNA-seq, application and development of molecular marker derived from RNA-seq., 2013, 35: 720–726.
[40] 王智兰, 毛新国, 李昂, 昌小平, 刘惠民, 景蕊莲. 小麦蛋白磷酸酶2A结构亚基基因的功能标记作图. 中国农业科学, 2011, 44: 2411–2421. Wang Z L, Mao X G, Li A, Chang X P, Liu H M, Jing R L. Functional marker mapping of protein phosphatase 2A structural subunit genein common wheat., 2011, 44: 2411–2421 (in Chinese with English abstract).
[41] Wu J H, Zeng Q D, Wang Q L, Liu S J, Yu S Z, Mu J M, Huang S,·Sela H, Distelfeld A, Huang L L, Han D J, Kang Z S. SNP-based pool genotyping and haplotype analysis accelerate fine-mapping of the wheat genomic region containing stripe rust resistance gene., 2018, 131: 1481–1496.
[42] 徐晓丹. 5份小麦农家品种的抗白粉病基因分析及定位. 中国农业大学博士学位论文, 北京, 2017. Xu X D. Identification and Mapping of Powdery Mildew Resistance Genes from Five Wheat Landraces. PhD Dissertation of China Agricultural University, Beijing, China, 2017 (in Chinese with English abstract).
[43] Liu S, Yeh C T, Tang H M, Nettleton D, Schnable P S. Gene mapping via bulked segregant RNA-seq (BSR-seq)., 2012, 7: e36406.
[44] Takagi H, Abe A, Yoshida K, Kosugi S, Natsume S, Mitsuoka C, Uemura A, Utsushi H, Tamiru M, Takuno S, Innan H, Cano L M, Kamoun S, Terauchi R. QTL-seq: rapid mapping of quantitative trait loci in rice by whole genome resequencing of DNA from two bulked populations., 2013, 74: 174–83.
[45] Sutherland O R W, Russell G B, Biggs D R, Lane G A. Insect feeding deterrent activity of phytoalexin isoflavonoids., 1980, 8: 73–75.
[46] Caballero P, Smith C M, Franczek F R, Fischer N H. Isoflavones from an insect-resistant variety of soybeans and the molecular structure of afrormosin., 1986, 49: 1126–1129.
[47] Saghai-Maroof M A, Soliman K M, Jorgensen R A, Allard R W. Ribosomal DNA spacer-length polymorphisms in barley: Mendelian inheritance, chromosomal location, and population dynamics., 1984, 81: 8014–8018.
[48] Lagudah E S, McFadden H, Singh R P, Huerta-Espino J, Bariana H S, Spielmeyer W. Molecular genetic characterization of the/slow rusting resistance gene region in wheat., 2006, 114: 21–30.
[49] Lagudah E S, Krattinger S G, Herrera-Foessel S, Singh R P, Huerta-Espino J, Spielmeyer W, Brown-Guedira G, Selter L L, Keller B. Gene-specific markers for the wheat gene//which confers resistance to multiple fungal pathogens., 2009, 119: 889–898.
[50] 伍玲, 夏先春, 朱华忠, 李式昭, 郑有良, 何中虎. CIMMYT 273个小麦品种抗病基因//的分子标记检测. 中国农业科学, 2010, 43: 4553–4561. Wu L, Xia X C, Zhu H Z, Li S Z, Zheng Y L, He Z H. Molecular characterization of//in 273 CIMMYT wheat cultivars and lines using functional markers., 2010, 43: 4553–4561 (in Chinese with English abstract).
[51] 张帆, 蒋雷, 鞠丽萍, 金秀锋, 王轩, 张晓科, 王宏礼, 付晓洁. 一个普通小麦Trx 超家族新基因的克隆与抗旱相关标记开发. 作物学报, 2014, 40: 29–36. Zhang F, Jiang L, Ju L P, Jin X F, Wang X, Zhang X K, Wang H L, Fu X J. Cloning a novel geneof Trx superfamily and developing its molecular markers related to drought resistance in common wheat., 2014, 40: 29–36 (in Chinese with English abstract).
[52] 肖永贵, 何心尧, 刘建军, 孙道杰, 夏先春, 何中虎. 中国冬小麦品种多酚氧化酶活性基因等位变异检测及其分布规律研究. 中国农业科学, 2008, 4: 954–960. Xiao Y G, He X Y, Liu J J, Sun D J, Xia X C, He Z H. Molecular identification and distribution of the polyphenol oxidase genes in Chinese winter wheat cultivars., 2008, 4: 954–960 (in Chinese with English abstract).
[53] He X Y, Zhang Y L, He Z H, Wu Y P, Xiao Y G, Ma C X, Xia X C. Characterization of phytoene synthase 1 gene () located on common wheat chromosome 7A and development of a functional marker., 2008, 116: 213–221.
[54] 王亮, 穆培源, 徐红军, 庄丽, 桑伟, 聂迎彬, 韩新年, 邹波. 新疆小麦品种黄色素含量基因()等位变异的分子检测. 麦类作物学报, 2009, 29: 782–786. Wang L, Mu P Y, Xu H J, Zhuang L, Sang W, Nie Y B, Han X N, Zou B. Allelic variations ofgene for yellow pigment content in Xinjiang wheat cultivars., 2009, 29: 782–786 (in Chinese with English abstract).
[55] 胡凤灵, 何中虎, 葛建贵, 姜文武, 时萍, 夏先春. 小麦品种黄色素含量和多酚氧化酶活性基因的分子标记检测. 麦类作物学报, 2011, 31: 47–53. Hu F L, He Z H, Ge J G, Jian W W, Shi P, Xia X C. Identification of genes for yellow pigment content and polyphenol oxidase activity in common wheat using molecular markers., 2011, 31: 47–53 (in Chinese with English abstract).
[56] 张学林, 梅四伟, 郭天财, 王晨阳, 朱云集, 王永华. 遗传和环境因素对不同冬小麦品种品质性状的影响. 麦类作物学报, 2010, 30: 249–253. Zhang X L, Mei S W, Guo T C, Wang C Y, Zhu Y J, Wang Y H. Effects of genotype and environment on winter wheat qualities., 2010, 30: 249–253 (in Chinese with English abstract).
Development and validation of markers linked to genes resistant toin wheat
HAO Zhi-Ming1, GENG Miao-Miao1, WEN Shu-Min1, YAN Gui-Jun2, WANG Rui-Hui1,*, and LIU Gui-Ru1,*
1Hebei Agricultural University / North China Key Laboratory for Crop Germplasm Resources of Education Ministry, Baoding 071000, Hebei, China;2School of Agriculture and Environment, the University of Western Australia, Nedlands 6009, Australia
Orange wheat blossom midge (OWBM) (Géhin) has seriously reduced wheat production and processing quality. Breeding midge-resistant wheat has been considered the most effective way to reduce kernel losses caused by OWBM, and marker-assisted selection (MAS) strategy in crop breeding using linked or functional markers of target trait of interest is of great importance in improving breeding efficiency. Based on the InDels and SNPs within the sequences of six midge resistance-related genes discovered from transcriptome data in the previously mapped major QTL () region, we developed and validated two EST and six KASP markers in a panel of recombinant inbred lines (RILs) and a panel of wheat cultivars with different OWBM resistance levels. These markers were polymorphic between the resistant and susceptible wheat parents, and approximately 90% of RIL lines showed the corresponding marker-based genotypes with their phenotypes. Except for, the other seven markers had higher detection efficiency in highly resistant (56.3%−86.7%) and in highly susceptible (85.7%−100.0%) wheat cultivars, thus making them applicable for screening midge-resistant wheat germplasm with locus. Among the eleven midge-resistant wheat cultivars with all resistance alleles for the seven markers developed, most were historical wheat cultivars, and rarely used in the present production, which suggests how to use old cultivars in wheat germplasm identification and enhancement on midge-resistance through MAS is urgent.
bread wheat (L.);; resistance-related genes; functional markers; SNP; EST; KASP
本研究由国家自然科学基金项目(31371617)资助。
This study was supported by the National Natural Science Foundation of China (31371617).
10.3724/SP.J.1006.2020.91029
王睿辉, E-mail:wangrh@hebau.edu.cn, Tel: 0312-7528408; 刘桂茹, E-mail: nxlgr@hebau.edu.cn
E-mail: haozhiming0730@hotmail.com
2019-04-08;
2019-09-26;
2019-10-16.
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20191015.1444.004.html
