基于SNP标记揭示我国小麦品种(系)的遗传多样性
2020-01-02刘易科朱展望佟汉文何伟杰张宇庆高春保
刘易科 朱展望 陈 泠 邹 娟,2 佟汉文 朱 光 何伟杰 张宇庆 高春保,2,
研究简报
基于SNP标记揭示我国小麦品种(系)的遗传多样性
刘易科1朱展望1陈 泠1邹 娟1,2佟汉文1朱 光1何伟杰1张宇庆1高春保1,2,*
1湖北省农业科学院粮食作物研究所 / 农业部华中地区小麦病害生物学科学观测实验站 / 湖北省小麦工程技术研究中心, 湖北武汉 430064;2主要粮食作物产业化湖北省协同创新中心 / 长江大学, 湖北荆州 434025
为了解我国主要小麦品种(系)的遗传多样性, 为亲本组配提供参考, 利用90K SNP芯片技术对国内为主的240个小麦品种(系)进行全基因组扫描, 分析其遗传多样性和遗传基础。结果表明, 多态性SNP位点在B基因组最多, D基因组最最少, 尤其4D上最少; 在全基因组范围内PIC平均值为0.26。参试品种间平均遗传相似系数为0.656, 变幅为0.133~0.998, 且87.05%品种间遗传相似系数在0.60~0.78之间; 国内西南麦区和长江中下游麦区小麦品种(系)间的平均相似系数较高, 分别为0.718和0.712, 国外品种(系)间的相似系数最低, 为0.552。聚类分析将参试品种(系)划分为7个类群, 大部分类群含有来自不同区域育成品种(系), 主成分分析显示各区域育成的品种(系)相互交集, 表明我国各省市间种质资源交流较为频繁, 但部分单位育成的品种(系)遗传基础不够丰富, 部分品种(系)间遗传相似性较高, 在育种中亟待引入新的种质, 拓宽遗传基础。
小麦; 品种; 遗传多样性; SNP
遗传多样性一般指种内个体之间或一个群体内不同个体的遗传变异总和。丰富的植物遗传资源多样性为育种工作者改良品种和培育理想特性新品种提供了种质基础, 理想特性包括农民偏好的产量潜力大、大粒等性状和育种家偏好的抗病虫和光敏性等性状[1]。20世纪60年代中期, 在绿色革命的引领下, 培育出对高产和肥料有严格要求的矮秆品种, 这些品种大面积种植满足了人类对粮食不断增加的需求, 然而也导致绝大多数具有某些优良性状的地方品种被淘汰, 原始和广适性基因灭绝, 遗传多样性急剧下降[1]。
我国是世界上最大的小麦生产国和消费国, 其安全生产对我国粮食安全具有重要意义[2]。在小麦遗传改良中, 突破性遗传资源鉴定和利用与小麦品种选育取得突破性进展密切相关。20世纪60年代矮秆基因的利用, 使小麦在降低株高的同时, 单产也大幅度提高; 与抗病性相关小麦1B/1R易位系种质的应用, 培育出许多抗病高产的小麦品种[3]; 在我国, 优秀骨干亲本如碧蚂4号、南大2419和小偃6号等的利用, 使我国小麦单产得到大幅度提高[4], 促成了我国主要麦区小麦品种数次更新换代。然而, 与其他作物一样, 小麦的遗传多样性随着驯化和现代育种选择压力不断下降, 导致遗传差异较大、适应当地环境的地方品种数量减少和遗传变异性降低[5,6]。
研究小麦种质资源遗传多样性, 可为小麦育种的亲本选配提供理论支撑, 有利于突破性种质和新品种的选育。SNP (Single Nucleotide Polymorphism)标记能较真实反映小麦品种间的亲缘关系[7-9], 可用于小麦品种遗传多样性研究[10]。Würschum等[9]利用小麦9K SNP芯片和91对SSR标记对172份欧洲优异冬小麦品系进行遗传多样性和群体结构研究表明, 小麦SNP可以作为基因组学研究的有效方法, 并可作为小麦改良的重要工具。曹廷杰等[10]利用小麦高密度90K芯片对2000—2013年间河南省审品种和部分大面积种植的国审定品种进行遗传多样性分析, 表明参试品种间遗传相似度较高, 而且品种间遗传多样性有逐渐降低的趋势。Baloch等[11]利用39,568个DArTseq和20,661个SNP标记对91个硬粒小麦品种的遗传特性研究表明, 无意识的农民选择和商业品种的缺乏可能导致了遗传物质的交换, 这在土耳其和叙利亚的硬粒小麦的遗传结构中表现得尤为明显。宋晓朋等[12]利用小麦90K SNP芯片对黄淮麦区144个小麦品种遗传多样性分析表明黄淮麦区小麦品种可以分为5个类群, 品种的分类与其生态种植区域和亲缘关系存在一定程度的相关。Joukhadar等[13]利用小麦90K SNP芯片对澳大利亚1840—2011年间育成的482份小麦品种进行遗传多样性、种群结构和祖先起源分析阐明了澳大利亚小麦育种的进化历史, 并为小麦基因组如何适应当地的生长条件做出了解释。Turuspekov等[14]利用小麦高密度90K芯片对哈萨克斯坦已育成或有潜力的90份小麦品种进行遗产多样性分析, 表明小麦品种的分类与其地理位置有显著关联。Müller等[15]利用小麦高密度15K芯片对瑞士502份栽培品种、地方品种和育种品系的遗产多样性分析, 表明现代小麦品种遗传上与地方品种不同, 可能与早期育种中地方品种的组配丢失有关。
目前在我国小麦育种过程中, 越来越多的杂交组合选配围绕部分大面积推广应用的高产品种[2]进行, 我国幅员辽阔、生态环境多样, 不同麦区育成的品种众多, 而利用高通量SNP芯片对我国主要麦区小麦品种的系统性遗传多样性研究尚未见报道。本研究选取我国主要小麦产区育成的部分主要小麦品种(系)和少量国外品种, 利用小麦高通量SNP芯片技术揭示我国小麦品种(系)的遗传多样性和亲缘关系, 以期为小麦新品种选育的亲本选配、遗传研究以及种质资源创制提供理论依据。
1 材料与方法
1.1 参试品种
供试品种(系)有240个(附表1), 包含我国小麦主产区育成的一些主要小麦品种(系), 其中超过50个小麦品种在2000—2016年间的最大年种植面积超过10万公顷, 包括骨干亲本济麦22号、宁麦9号、小偃6号、良星99、矮抗58和周麦18等。按育成区域划分, 包括西南麦区17个(代表品种为川麦和绵麦系列品种等)、长江中下游麦区50个(代表品种为扬麦、宁麦和鄂麦系列品种等)、黄淮南片94个(代表品种为郑麦、周麦和西农系列品种等)、黄淮北片47个(代表品种为济麦、山农和衡观系列品种等)、北部麦区品种(系) 21个(代表品种为中麦和兰天系列品种等)和国外品种11个(代表品种为CIMMIT高代品系等)。所有材料由粮食作物种质创新与遗传改良湖北省重点实验室保存。
1.2 SNP芯片分型
选取小麦幼苗叶片, 按Saghai-Maroof等[16]的CTAB法提取基因组DNA。利用Illumina 90K iSelect SNP标记对240个小麦品种进行全基因组扫描[17], 由美国农业部农业研究服务小谷物基因分型实验室(美国北达科他州立大学)采用Illumina’s GenomeStudio Software (https://www.illumina.com/ techniques/microarrays/array-data-analysis-experimentaldesign/ genomestudio.html)进行样本的原始SNP分型。
1.3 SNP位点的多态性信息含量分析
利用多态性信息含量(polymorphism information content, PIC)对SNP位点的遗传多样性进行评价。按照Powermarker v3.25软件的格式要求对芯片分型数据进行整理并导入计算, 单个SNP位点的PIC值按照公式PIC = 1–∑P2计算,P表示第个标记第个等位变异出现的频率。
1.4 品种(系)的遗传多样性及聚类分析
将SNP分型数据转换为0、1二元数据格式, 建立原始矩阵。用NTSYS-PC2.1统计软件计算品种(系)间遗传相似系数。采用非加权配对算术平均法(UPGMA)构建遗传关系树状图, 结合iTOL (https://itol.embl.de/tree/)网站进行聚类图的修饰和亚群区分。
1.5 主成分分析
利用TASSEL5.2.51对供试品种进行主成分分析(principle component analysis, PCA), 利用ggplot2工具绘制主成分分析图。
2 结果与分析
2.1 多态性SNP位点的分布
芯片中分布于21条染色体的81,587个SNP位点中, 22,904个具备多态性, 多态性比率为28.07%, 其中18,741个被定位于明确的染色体上并用于后续分析。每条染色体分布76~2038个多态性位点(图1-A), 差异较大, 4D上差异位点最少, 而1B上差异位点最多。同时, 多态性位点在A、B、D基因组上分布不均, 以B基因组最多, 为9376个, 占50.03%, A基因组上多态性位点为7672个, 占40.94%, 6A较多, 4A最少; D基因组上多态性位点最少, 仅有1693个, 占9.03%。
多态性SNP位点在7个部分同源群中分布不均, 在第1和第2部分同源群上分布最多, 在第4部分同源群最少(图1-B)。
2.2 SNP位点的多态性信息含量分析
利用18,741个SNP分子标记, 在240份小麦材料中检测到37,482个等位变异, 每个位点均检测到2个等位变异。SNP分子标记多态性信息含量即PIC值的变幅为0.01~0.38, 平均值为0.26, 主要分布于0.10~0.38之间(图2)。

图1 多态性SNP位点在染色体(A)和同源群(B)上的分布
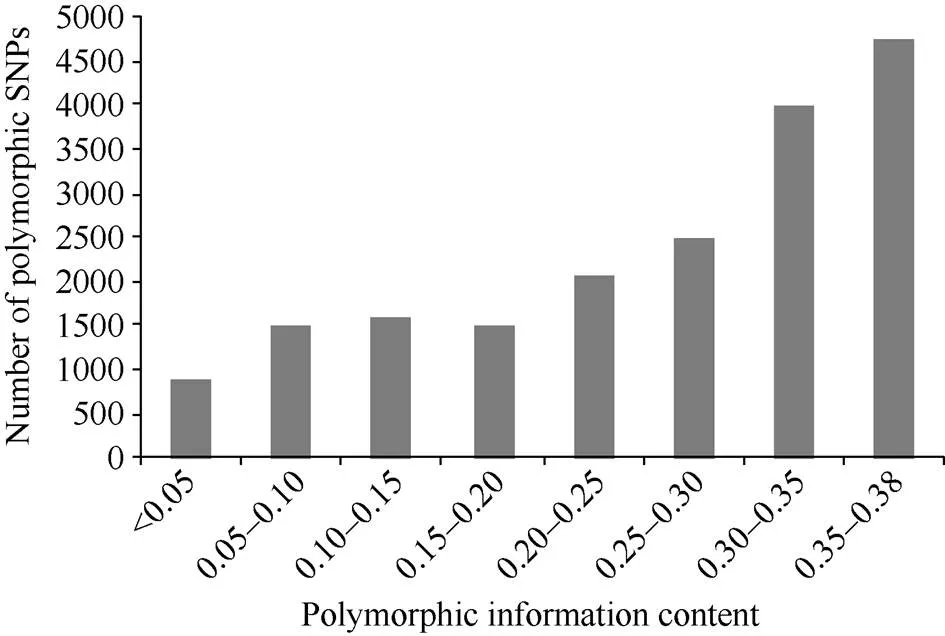
图2 SNP标记在240份小麦材料中多态性信息含量(PIC)的分布
2.3 小麦品种(系)间的遗传相似系数
利用NTSYS-PC2.1统计软件, 对240个品种(系)相互间的相似系数进行计算, 共获得28,680个遗传相似系数, 其平均变异系数为0.656, 变异范围为0.133~0.998 (表1)。品种(系)间的遗传系数主要集中在0.60~0.78之间, 有24,965个, 占87.05%, 而小于0.6的有2974个, 占10.37%, 而大于0.78的最少, 只有741个, 占2.58% (图3)。
为了探讨我国不同麦区小麦品种(系)遗传多样性, 将240个品种(系)按育成区域分组, 在组内进行品种(系)间的遗传相似性分析。结果表明(表1), 西南麦区品种(系)间的平均相似系数最高, 为0.718, 其次为长江中下游麦区, 为0.708; 北部麦区、黄淮北片和黄淮南片品种(系)间的平均相似系数相对最低, 分别0.660、0.680和0.686; 国外品种(系)间的平均相似系数最低, 为0.552。
2.4 聚类分析
为探明240个品种(系)之间的亲缘关系, 根据品种间遗传相似系数, 采用非加权配对算术平均法(UPGMA)构建遗传关系树状图。聚类分析结果表明(图3), 240个品种(系)被划分为7个类群。
第I类有56个品种(系), 主要包含长江中下游麦区品种(系) 38个和西南麦区品种(系) 14个。鄂麦11号是鄂麦18的亲本之一, 两者遗传相似系数较高为0.998; 扬麦11号与扬麦12号有共同的亲本扬麦158和85-853, 两者遗传相似系数较高为0.989; 宁麦16号与镇麦5号有共同的亲本宁麦9号, 两者遗传相似系数较高为0.986。
第II类和第III类分别有13个和7个品种(系), 第II类主要包含黄淮南片麦区品种(系) 9个和黄淮北片麦区品种(系)2个, 第III类主要包含黄淮南片麦区品种(系) 5个。
第IV类有34个品种(系), 主要包含黄淮南片麦区品种(系) 31个。其中偃展4110与新麦208有共同的亲本豫麦18, 两者遗传相似系数较高, 为0.992。
第V类有41个品种(系), 包含黄淮北片麦区品种(系) 29个、黄淮南片麦区品种(系) 7个和北部麦区品种(系) 5个。其中宁冬10号与宁冬11号均有国外品种血缘和共同的国内亲本北农2号, 两者的遗传相似系数较高, 为0.989。
第VI类含有17个品种(系), 包含国外品种(系)11个和宁春47号、宁春4号、宁春43号、宁麦13号、小偃6号鄂07901六个国内品种(系)。其中国内品种中宁春系列品种含有CIMMYT品种Sonoro-64的血缘, 宁麦13号含有日本品种西风的血缘, 小偃6号具有意大利小麦品种郑引4号(St2422/464)的血缘, 鄂07901含有CIMMYT品种的血缘。国外小麦品种Mayoor是SYN1的亲本之一, 两者遗传相似系数较高, 为0.861。
第VII类有72个品种(系), 该类品种分布比较广泛, 包含黄淮南片麦区品种(系) 35个、黄淮北片麦区品种(系) 13个、北部麦区品种(系) 11个、长江中下游麦区品种(系) 6个和西南麦区品种(系) 3个。豫麦70和豫麦70-36优系的相似系数为0.971, 豫麦49-197和豫麦49-168两个豫麦49改良优系的相似系数为0.998。
2.5 主成分分析
为反映不同群体间的遗传关系, 基于SNP标记对供试品种分组进行主坐标分析。结果表明(图5), 前2个坐标能够解释的遗传变异分别为31.59%和23.67%; 长江中下游麦区和西南麦区的大部分品种(系)聚在一起, 侧面验证了这2个地区的小麦品种(系)的遗传相似性较高(表1); 黄淮南片、黄淮北片和北部麦区的品种(系)在坐标图中分布广泛, 侧面验证了这些地区的小麦品种(系)的遗传相似性相对较低(表1); 供试材料中国外品种(系)的样品量虽少, 但分布也较为广泛, 故遗传相似性相对较低(表1)。

表1 不同地区育成品种(系)的遗传相似系数
品种(系)间的遗传相似性在主成分分析图中也得到了验证, 例如鄂麦11号(167)和鄂麦18号(175)、扬麦11号(185)和扬麦12号(14)、偃展4110 (3)和新麦208 (224)以及宁冬11号(24)和宁冬10号(50)等相似性较高的品种(图4), 在主成分分析图中也聚集在一起(图5)。
3 讨论
遗传多样性分析对植物种质资源的评价与利用、新品种培育以及新基因的定位克隆具有重要意义, 是植物遗传学、育种、保护和进化的重要组成部分[5]。以往的研究也发现半个多世纪的育种选择使中国小麦品种的遗传基础日趋狭窄[18]。由于品种审定制度和商业化育种对多出快出品种的要求, 小麦育种家过多关注产量、抗病性、品质等性状, 越来越多的杂交组合选配围绕部分大面积推广应用的高产品种[2]。以山东省2018年审定的小麦新品种为例, 在29个小麦新品种中, 单交育成品种有25个, 24个品种至少有一个亲本为大面积推广应用的高产品种, 其中矮抗58和良星99作为亲本之一育成的品种多达8个, 9个品种的亲本均为已育成品种。在小麦种质资源创新未取得突破性进展的情况下, 以现有品种(系)为亲本配制杂交组合的育种手段, 短期看来难以改观。本研究利用小麦高通量SNP芯片技术对包括矮抗58和良星99在内的我国主要小麦产区育成的代表性小麦品种(系)进行遗传多样性和亲缘关系研究, 可为现有条件下小麦新品种培育的亲本选配提供参考, 尽可能避免亲缘关系过近品种(系)间杂交, 有助于丰富育成品种的遗传多样性, 提高品种抵抗不良环境的能力。
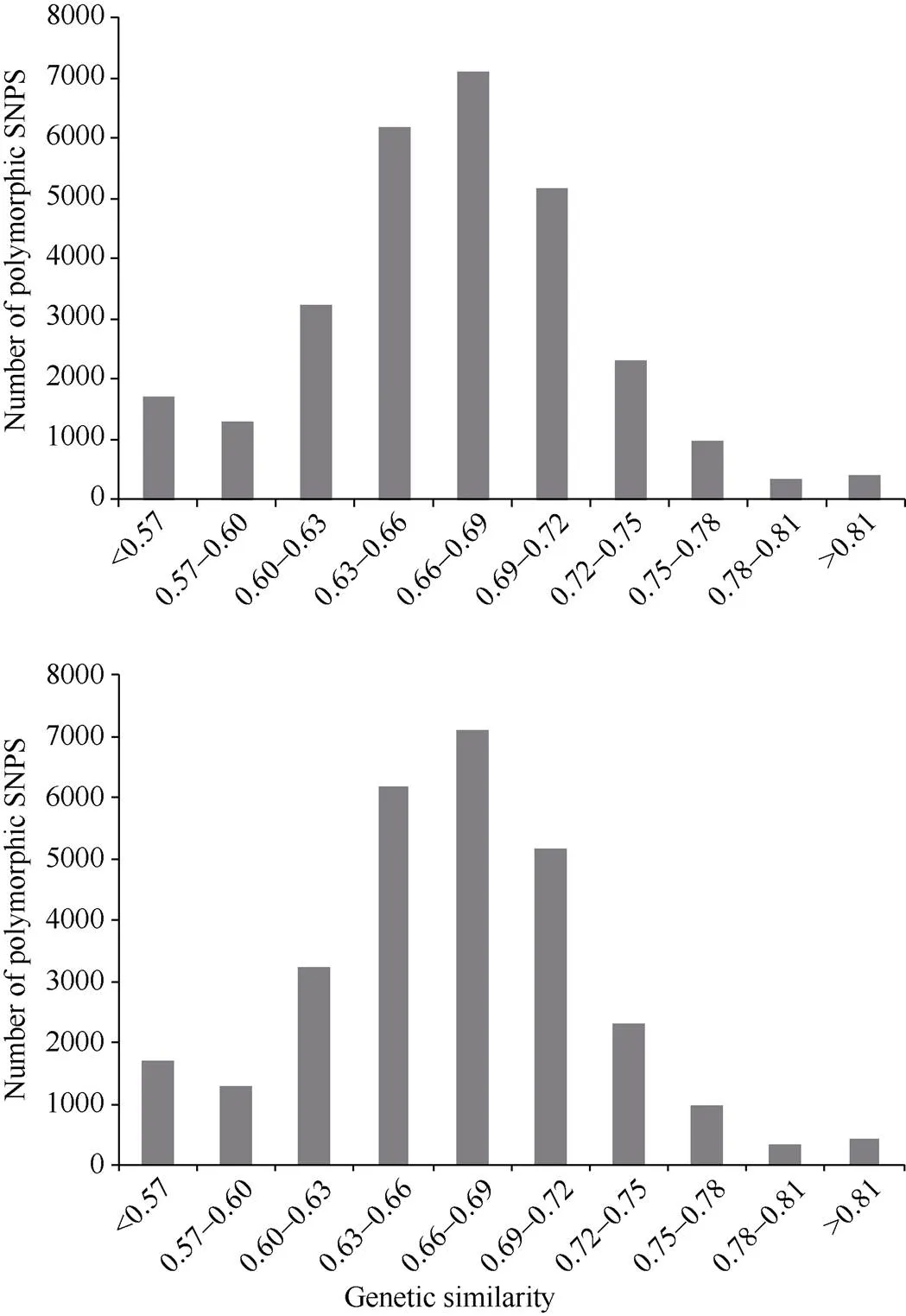
图3 240个品种(系)间遗传相似系数的次数分布
研究表明, 我国不同区域的小麦品种都不同程度存在亲缘关系较近和遗传多样性不足的情况[10,12,19-23]。蒲艳艳等[19]利用68对SSR引物对44个山东小麦品种进行遗传多样性分析品种间的平均遗传相似系数为0.694, 变幅0.593~0.918; 潘玉朋等[23]利用31对SSR引物对黄淮麦区大面积推广的25个小麦品种进行遗传多样性分析, 表明品种间的遗传相似系数为0.61, 变幅0.34~0.85; 刘丽华等[24]利用21对SSR对2009—2014年参加国家冬小麦区域试验的430个品系进行遗传多样性分析, 结果表明北部冬麦区旱地组参试品系的遗传多样性为0.73, 黄淮海冬麦区南片春水组参试品系的遗传多样性为0.63; 本研究利用高密度SNP芯片, 对国内的229份小麦品种(系)进行遗传相似性研究, 结果表明各大区品种(系)间遗传相似系数在0.660~0.718之间(表1), 与前人利用SSR标记做类似研究的结果相似[19,23-24]。本研究中, 西南地区因样品数量不足且均为四川小麦品种(系)外, 长江中下游麦区小麦品种(系)的相似性系数最高(0.712), 可能与长江中下游麦区小麦育种单位相对较少, 也可能与该区域育种单位引进利用其他地区种质资源相对有限、育成小麦品种(系)的类型较少有关。黄淮南片麦区、黄淮北片麦区和北部麦区的小麦品种(系)的相似性系数相对较低(0.660~0.686), 可能与这些区域小麦育种单位相对较多实力雄厚有关, 也可能与该区域育种单位积极引入外源优异种质资源有关, 如陕西育种过程中引入外源种质资源长穗偃麦草和地方品种[25], 相对丰富的遗传多样性也造就了陕西品种特别是西农系列品种相对较广的适应性。
我国幅员辽阔、生态环境多样, 不同麦区的品种经过长期演变和选择形成了相对独立的遗传特点, 与此同时本研究也发现(图4), 不同类组内大多含有来自不同区域或省份的育成品种(系), 且不同区域小麦品种(系)在主成分分析图中分布较广(图5), 说明我国各育种单位间种质资源交流较为频繁。国外品种(系)聚集在第VI类之中, 宁春系列的3个品种也属于第VI类(图4), 可能与宁春系列品种含有国外小麦品种的遗传物质有关[26]; 周麦系列品种(系)主要分布在第VII类, 新麦系列主要分布在第VI类, 郑麦系列品种广泛分布在第VII、IV、II和I类中, 可能与郑麦系列品种的遗传基础相对较广有关。江苏品种中扬麦和宁麦系列品种主要聚集在第I类中, 说明扬麦和宁麦系列的遗传关系相对较近。山东品种中济麦、烟农和山农系列品种交集一起聚集在第V类中, 说明3种系列的小麦品种遗传关系相对较近。湖北品种中鄂恩系列品种聚集在第VII类, 与陕西品种遗传关系较近; 鄂麦和襄麦系列品种属于第I类, 与江苏品种的遗传关系相对较近。陕西的西农和小偃系列在聚类图中分布较广, 说明两个系列的品种遗传基础较为丰富。四川的绵麦和川麦系列品种在聚类图中分布较广, 说明两个系列的品种遗传基础较为丰富。河北品种在聚类图中分布较广, 衡观系列品种(系)聚集分布在第VII类, 说明该系列品种的遗传关系相对较近。
本研究结果表明, 大部分品种(系)的聚类结果(图4)与其系谱来源及地域分布较为吻合, 也与笔者观测的田间性状相符。但研究中也发现, 部分品种遗传相似系数很高, 但系谱分析发现这些品种的亲本并无直接的亲缘关系。究其原因, 可能存在以下情况, 一是种子收集、繁殖、保存时出现偏差, 造成品种名称与实际材料不符的情况; 二是在同一生态区内, 育种家的长期定向选择, 造成选择的方向往往集中在某些位点上, 而造成另一些位点的丢失; 三是存在系谱记载不准确或不完善的可能[10]。下一步, 在确认种子无误的情况下, 可对这些品种(系)进行更为精细的田间观测, 也可利用其他分子标记或更高密度的SNP芯片对其进行分析, 已确定这些品种(系)的遗传相似性。
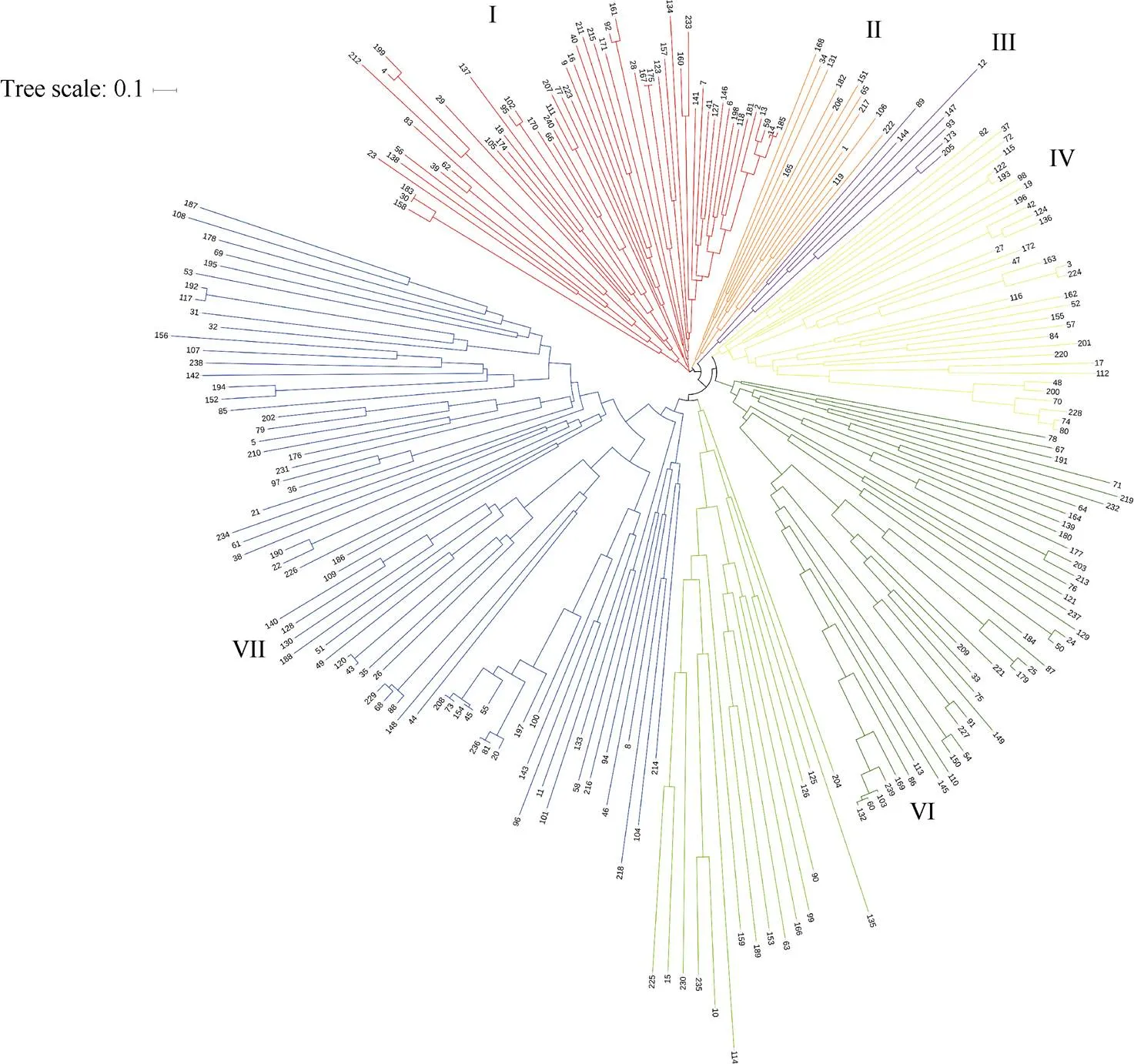
图4 基于SNP标记的240个小麦品种(系)的聚类图

图5 基于SNP标记的240个小麦品种(系)主成分分析
A为西南麦区品种, B为长江中下游麦区品种, C为黄淮南片品种, D为黄淮北片品种, E为北部麦区品种, F为国外品种。
A:wheat varieties (lines) from Southwest China; B: wheat varieties (lines) from the middle and lower reaches of Yangtze River; C: wheat varieties (lines) from South Huang-Huai; D: wheat varieties (lines) from North Huang-Huai, China; E: wheat varieties (lines) from Northern China; E: wheat varieties (lines) from Foreign.
附表 请见网络版: 1) 本刊网站http://zwxb.chinacrops. org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http:// c.wanfangdata. com.cn/Periodical-zuowxb.aspx。
[1] Govindaraj M, Vetriventhan M, Srinivasan M. Importance of genetic diversity assessment in crop plants and its recent advances: An overview of its analytical perspectives., 2015, 2015: 1–14.
[2] 刘志勇, 王道文, 张爱民, 梁翰文, 吕慧颖, 邓向东, 葛毅强, 魏珣, 杨维才. 小麦育种行业创新现状与发展趋势. 植物遗传资源学报, 2018, 19: 430–434. Liu Z Y, Wang D W, Zhang A M, Liang H W, Lyu H Y, Deng X D, Ge Y Q, Wei X, Yang W C. Current status and perspective of wheat genomics, genetics and breeding., 2018, 19: 430–434 (in Chinese with English abstract).
[3] 何中虎, 夏先春, 陈新民, 庄巧生. 中国小麦育种进展与展望. 作物学报, 2011, 37: 202–215. He Z H, Xia X C, Chen X M, Zhuang Q S. Progress of wheat breeding in China and the future perspective., 2011, 37: 202–215 (in Chinese with English abstract).
[4] 徐鑫, 李小军. 小麦骨干亲本研究进展. 河南农业科学, 2012, 41(2): 5–8. Xu X, Li X J. Research progress of founder parents in wheat., 2012, 41(2): 5–8 (in Chinese with English abstract).
[5] Peterson G W, Dong Y, Horbach C, Fu Y B. Genotyping-by-sequencing for plant genetic diversity analysis: a lab guide for SNP genotyping., 2014, 6: 665–680.
[6] Royo C, Elias E M, Manthey F A. Durum wheat breeding. In: Carena M J, ed. Cereals-Handbook of Plant Breeding. Berlin: Springer, 2009. pp 199–266.
[7] Ren J, Chen L, Sun D, You F M, Wang J, Peng Y, Nevo E, Beiles A, Sun D, Luo M C, Peng J. SNP-revealed genetic diversity in wild emmer wheat correlates with ecological factors., 2013, 13: 169.
[8] Chao S, Dubcovsky J, Dvorak J, Luo M C, Baenziger S P, Matnyazov R, Clark D R, Talbert L E, Anderson J A, Dreisigacker S, Glover K, Chen J, Campbell K, Bruckner P L, Rudd J C, Haley S, Carver B F, Perry S, Sorrells M E, Akhunov E D. Population- and genome-specific patterns of linkage disequilibrium and SNP variation in spring and winter wheat (L.)., 2010, 11: 727.
[9] Wurschum T, Langer S M, Longin C F, Korzun V, Akhunov E, Ebmeyer E, Schachschneider R, Schacht J, Kazman E, Reif J C. Population structure, genetic diversity and linkage disequilibrium in elite winter wheat assessed with SNP and SSR markers., 2013, 126: 1477–1486.
[10] 曹廷杰, 谢菁忠, 吴秋红, 陈永兴, 王振忠, 赵虹, 王西成, 詹克慧, 徐如强, 王际睿, 罗明成, 刘志勇. 河南省近年审定小麦品种基于系谱和SNP标记的遗传多样性分析. 作物学报, 2015, 41: 197–206. Cao Y J, Xie Q Z, Wu Q H, Chen Y X, Wang Z Z, Zhao H, Wang X C, Zhan K H, Xu R Q, Wang J R, Luo M C, Liu Z Y. Genetic diversity of registered wheat varieties in Henan province based on pedigree and single-nucleotide polymorphism., 2015, 41: 197–206 (in Chinese with English abstract).
[11] Baloch F S, Alsaleh A, Shahid M Q, Çiftçi V, Miera L E S, Aasim M, Nadeem M A, Aktaş H, Özkan H, Hatipoğlu R. A whole genome DArTseq and SNP analysis for genetic diversity assessment in durum wheat from central fertile crescent., 2017, 12: e0167821.
[12] 宋晓朋. 黄淮麦区小麦品种遗传多样性及分子标记与性状的关联分析. 西北农林大学硕士学位论文, 陕西杨凌, 2016. Song X M. The Analysis of Genetic Diversity and Study Markers Associated with Traits in Huang-Huai Winter Wheat. MS Thesis of Northwest A&F University, Yangling, Shaanxi, China, 2016 (in Chinese with English abstract).
[13] Joukhadar R, Daetwyler H D, Bansal U K, Gendall A R, Hayden M J. Genetic diversity, population structure and ancestral origin of Australian wheat., 2017, 8: 2115.
[14] Turuspekov Y, Plieske J, Ganal M, Akhunov E, Abugalieva S. Phylogenetic analysis of wheat cultivars in Kazakhstan based on the wheat 90 K single nucleotide polymorphism array., 2017, 15: 29–35.
[15] Müller T, Schierscher-Viret B, Fossati D, Brabant C, Schori A, Keller B. Unlocking the diversity of GeneBanks: whole-genome marker analysis of Swiss bread wheat and spelt., 2018, 131: 407–416.
[16] Saghai-Maroof M A, Soliman K M, Jorgensen R A, Allard R W. Ribosomal DNA spacer-length polymorphisms in barley: Mendelian inheritance, chromosomal location, and population dynamics.1984, 81: 8014–8018.
[17] Wang S C, Wong D, Forrest K, Allen A, Chao S M, Huang B E, Salvi S, Milner S G, Cattivelli L, Mastrangelo A M, Whan A, Stephen S, Barker G, Wieseke R, Plieske J. Characterization of polyploid wheat genomic diversity using a high-density 90 000 single nucleotide polymorphism array., 2014, 12: 787–796.
[18] 郝晨阳, 王兰芬, 张学勇, 游光霞, 董玉琛, 贾继增, 刘旭, 尚勋武, 刘三才, 曹永生. 我国育成小麦品种的遗传多样性演变. 中国科学C辑, 2005, 35: 408–415 Hao C Y, Wang L F, Zhang X Y, You G X, Dong Y C, Jia J Z, Liu X, Shang X W, Liu S C, Cao Y S. Genetic diversity in Chinese modern wheat varieties revealed by microsatellite markers.:, 2005, 35: 408–415 (in Chinese with English abstract).
[19] 蒲艳艳, 程凯, 李斯深. 山东省近期育成小麦品种遗传多样性的SSR分析. 分子植物育种, 2011, 9: 443–449. Pu Y Y, Cheng K, Li S S. Genetic diversity of recent wheat cultivars in Shandong province using SSR markers., 2011, 9: 443–449 (in Chinese with English abstract).
[20] 李志波, 王睿辉, 张茶, 梁虹, 马峙英, 赵玉欣, 王静华. 河北省小麦品种基于农艺性状的遗传多样性分析. 植物遗传资源学报, 2009, 10: 436–442. Li Z B, Wang R H, Zhang C, Liang H, Ma Z Y, Zhao Y X, Wang J H. Genetic diversity analysis of bread wheat (L.) cultivars in Hebei province based on agronomic traits., 2009, 10: 436–442 (in Chinese with English abstract).
[21] 傅体华, 王春梅, 任正隆.四川育成小麦品种的SSR 遗传多态性及系谱关系. 四川农业大学学报, 2007, 25: 1–7. Fu T H, Wang C M, Ren Z L. SSR genetic diversity among modern advanced wheat cultivars (L.) in Sichuan and its relationships with their pedigree., 2007, 25: 1–7 (in Chinese with English abstract).
[22] 李学军, 潘玉朋, 王小利, 李立群, 王培, 冯毅, 王辉. 陕西育成小麦品种的遗传多样性演变. 西北农林科技大学学报(自然科学版), 2011, 39: 48–54. Li X J, Pan Y P, Wang X L, Li L Q, Wang P, Feng Y, Wang H. Evolution of genetic diversity of the winter varieties grown at Shaanxi.(Nat Sci Edn), 2011, 39: 48–54 (in Chinese with English abstract).
[23] 潘玉朋, 李立群, 郑锦娟, 王培, 冯毅, 李学军. 黄淮麦区近年大面积推广小麦品种的遗传多样性分析. 西北农业学报, 2011, 20: 47–52. Pan Y P, Li L Q, Zheng J J, Wang P, Feng Y, Li X J. Analysis of genetic diversity of the large-scale promoted wheat varieties grown at Huang-Huai area in recent years., 2011, 20: 47–52 (in Chinese with English abstract).
[24] 刘丽华, 庞斌双, 刘阳娜, 邱军, 李宏博, 张欣, 王娜, 赵昌平. 2009–2014年国家冬小麦区域试验品系的遗传多样性及群体结构分析. 麦类作物学报, 2016, 36: 165–171.Liu L H, Pang B S, Liu Y N, Qiu J, Li H B, Zhang X, Wang N, Zhao C P. Genetic diversity and population structure analysis of winter wheat lines from recent national regional trials in China.2016, 36: 165–171.
[25] 孙道杰, 张玲丽, 冯毅, 陈春环, 张荣琦, 奚亚军, 何心尧, 王辉, 宋哲民. 西农系列小麦骨干新品种赤霉病抗源浅析. 麦类作物学报, 2016, 36: 822–823.Sun D J, Zhang L L, Feng Y, Chen C H, Zhang R Q, Xi Y J, He X Y, Wang H, Song Z M. Analysis of FHB resistance sources for newly released Xinong varieties.2016, 36: 822–823 (in Chinese with English abstract).
[26] 袁汉民, 裘志新, 陈东升, 袁海燕, 王晓亮, 张富国, 赵桂珍, 汤忠. 小麦种质资源宁春4号的研究和利用. 麦类作物学报, 2009, 29: 160–165.Yuan H M, Qiu Z X, Chen D S, Yuan H Y, Wang X L, Zhang G F, Zhao G Z, Tang Z. Study and utilization of wheat germplasm ‘Ningchun 4’., 2009, 29: 160–165 (in Chinese with English abstract).
Revealing the genetic diversity of wheat varieties (lines) in China based on SNP markers
LIU Yi-Ke1, ZHU Zhan-Wang1, CHEN Ling1, ZOU Juan1,2, TONG Han-Wen1, ZHU Guang1, HE Wei-Jie1, ZHANG Yu-Qing1, and GAO Chun-Bao1,2,*
1Food Crops Institute, Hubei Academy of Agricultural Sciences / Wheat Disease Biology Research Station on Central China, Ministry of Agriculture, China / Hubei Engineering and Technology Research Center of Wheat, Wuhan 430064, Hubei, China;2Hubei Collaborative Innovation Center for Grain Industry / Yangtze University, Jingzhou 434025, Hubei China
In order to understand the genetic diversity of major wheat cultivars (lines) in China and provide a reference for parent selection, we selected 240 domestically dominated wheat cultivars (lines) for SNP genotyping using the Illumina 90K iSelect SNP chip. The SNP loci were the most in the B genome, and were the least in the D genome, especially in 4D. The mean value of the polymorphism information content (PIC) was 0.26. The genetic similarity of the 240 cultivars (lines) ranged from 0.133 to 0.998 with an average value of 0.656. However, genetic similarity of 0.60 to 0.78 was found in 87.05% of the tested cultivars (lines). The genetic similarity was up to 0.718 between Southwest Wheat Region cultivars (lines), 0.712 between Middle and Low Yangtze Valleys Wheat Region, and 0.552 between foreign cultivars (lines). The 240 wheat cultivars (lines) were classified into seven groups by UPGMA analyses, most of them contained varieties (lines) bred from different regions. Principal component analysis (PCA) showed that the cultivars (lines) bred in different regions intersected together. The exchange of germplasm resources among different regions in China was frequent, but the genetic basis of the cultivars (lines) bred by some units was not rich enough and the genetic similarity between some varieties (lines) was higher. It is urgent to introduce new germplasm and broaden the genetic basis in breeding.
wheat; cultivars; genetic diversity; SNPs
本研究由国家重点研发计划项目(2017YFD0100802), 湖北省技术创新专项(2018ABA085), 国家小麦产业技术体系“武汉综合试验站”(CARS-03)和湖北省农业科技创新中心项目资助。
The work was supported by the National Key Research and Development Program (2017YFD0100802), the Hubei Technical Innovation Project (2018ABA085), the Modern Agro-Industry Technology Research Systems (CARS-03), and the Hubei Agricultural Science and Technology Innovation Center Project.
10.3724/SP.J.1006.2020.91039
高春保, E-mail: gcbgybwj@163.com
E-mail: hbliuyk@foxmail.com
2019-06-03;
2019-08-09;
2019-09-11.
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20190910.1511.012.html
