基于宏转录组学技术解析传统酸面团中微生物代谢机理
2019-12-13张国华张纬珍何国庆
张国华 王 伟 涂 建 张纬珍 何国庆
(山西大学生命科学学院1, 太原 030006) (浙江大学生物系统工程与食品科学学院2, 杭州 310058)
传统酸面团,也称为老面、酵子、面肥或起子,是传统用于发酵面制品的发酵剂,由酵母菌、乳酸菌及霉菌等多菌群组成的混合发酵体系[1,2]。据报道,传统酸面团中的乳酸菌超过60种,其中乳杆菌属为主,旧金山乳杆菌为优势菌种[3];有20多种酵母菌[6],其中酿酒酵母为优势菌种。传统酸面团中酵母菌是利用原料中糖类物质转化为CO2及醇、醛类等物质,形成发酵面制品特有的面筋结构[4];改善面团特性,如体积,质地和风味,增加营养价值等[5-8]。乳酸菌则可在发酵过程中产生乳酸、醋酸及丙酸等有机酸,与酵母菌相互作用,形成传统发酵面制品独特的风味品质[9]。
国外对面包用的传统酸面团的研究已有100多年的历史,按照制作工艺的不同,国外将酸面团分为3种[10],Ⅰ型酸面团主要是利用传统制作工艺生产的发酵剂,类似我国传统酸面团,是以旧金山乳杆菌和酿酒酵母菌为优势乳酸菌[11],Ⅱ型酸面团主要是一种液态发酵剂,以同型发酵乳酸菌菌种为优势乳酸菌[12],Ⅲ型酸面团是一种干燥的粉状发酵剂[13],在面包制作过程中常作为酸味剂或增香剂使用。国外研究发现酵母菌和乳酸菌之间存在代谢产物互补机制,酵母菌在发酵过程中为乳酸菌的生长提供了营养因子,而乳酸菌的代谢产物又为酵母菌提供了能量来源[14]。目前,国内酸面团的研究相对薄弱,集中在对我国部分地区的酸面团中微生物多样性进行了系统研究,研究发现我国酸面团也是以酿酒酵母菌与旧金山乳杆菌为优势菌种[15,16],酵母菌和乳酸菌混合发酵面团与活性干酵母发酵相比,总酸度较高,麦芽糖、果糖等可溶性糖变化差异显著[17]。
宏转录组学技术是以样品中的全部微生物RNA信息为分析对象,从整体水平上研究微生物基因表达情况及转录调控规律的技术[18]。本研究基于宏转录组学技术,以传统酸面团Sx样品为研究对象,对面团发酵前后时期中优势微生物的变化、功能基因表达及代谢途径等进行探究,以期阐明馒头发酵过程中微生物代谢机理,为我国传统发酵面食工业化的研究提供参考。
1 材料与方法
1.1 材料与试剂
传统酸面团Sx样品:由山西省临汾市农家提供;惠宜低筋面粉:市售。
建库试剂: Ribo-Zero Magnetic Kits; TruSeqTMRNA Sample Prep Kit;
测序试剂: cBotTruseq PE Cluster Kit v3-cBot-HS;Truseq SBS Kit v3-HS(200 cycles);
1.2 仪器与设备
NanoDrop2000分光光度计;安捷伦2100;Illumina Hiseq测序平台;
1.3 方法
1.3.1 样品的制备
称取50.0 g的Sx酸面团样品,加入45 mL蒸馏水,浸泡10~15 min(室温),然后混入70.0 g的小麦面粉,搅拌均匀;在温度28~30 ℃,相对湿度70%~75%左右条件下,发酵2.5~3 h;(取样品,标记为Sx-1)再补入30.0 g的小麦粉,反复揉面团10~15 min,至面团不粘手、表面光滑;然后定量分割和搓圆,在温度28~30 ℃、相对湿度70%~75%条件下,醒发40~60 min(二次发酵,取样品,标记为Sx-2)。
1.3.2 RNA的提取及检测
对Sx-1和Sx-2样品进行RNA抽提(试剂盒:TruSeqTMRNA Sample Prep Kit),然后采用NanoDrop2000和1.5 %琼脂糖凝胶电泳检测RNA的浓度、纯度以及RNA的完整性和质量,利用安捷伦2100检测RIN值。
1.3.3 构建转录组文库并测序
将检测合格的RNA进行构建转录组文库,采用Illumina Hiseq测序平台进行测序。
1.3.4 生物信息分析
1.3.4.1 生信分析流程
Sx-1和Sx-2两个样品的原始数据经过质控以及去除rRNA后,得到优化数据,进行组装得到转录本信息。对转录本信息进行差异表达基因分析:差异表达基因筛选、差异基因模式聚类分析、差异基因的基因本体(GO)注释、差异基因京都基因与基因组百科全书(KEGG)注释、GO显著性富集分析及KEGG显著性富集分析。
1.3.4.2 物种注释
用BLASTP(BLASTVersion2.2.28+)将Sx-1和Sx-2两个样品基因集与非冗余蛋白数据库(NR)数据库进行比对(BLAST设置期望值e-value:1e-5),获得参与发酵的物种在属、种水平上的注释。
1.3.4.3 功能注释
GO功能注释:利用GO数据库,对Sx-1和Sx-2两个样品基因按照其参与的生物过程、分子功能及细胞组分三个方面进行分类注释。使用 blast2go对基因集进行GO注释,然后使用GO对应的基因丰度总和计算该GO的丰度。
COG功能注释:利用基因进化谱系:非监督直余同源群(eggNOG)数据库,对Sx-1和Sx-2两个样品基因集进行比对(BLAST设置期望值e-value:1e-5),获得基因对应的直系同源序列聚类,然后使用COG对应的基因丰度总和计算该COG的丰度。
KEGG功能注释:利用KEGG的基因数据库,对Sx-1和Sx-2两个样品基因信息进行比对(BLAST设置期望值e-value:1e-5),使用基于KEGG直系同源的注释系统(KOBAS 2.0)进行注释。
差异基因注释:得到两个样品的基因表达量之后,使用edgeR 软件对两个样品的基因表达量进行比较,以筛选不同发酵时期显著差异表达基因,筛选标准为:FDR<0.05且 |log2FC|>1。筛选的差异表达基因进行GO、KEGG注释,处理方法与GO、KEGG注释的方法一致。
采用R语言对Sx-1和Sx-2两个样品在物种、基因、COG、KEGG和GO功能等层次上进行热图聚类分析。
2 结果与分析
2.1 样品总RNA质量检测
采用NanoDrop2000对提取的2个样品的总RNA检测的纯度(OD260/280和OD260/230分别在1.95)和浓度(>50 ng/μL)均较高,达到RNA-seq建库的要求(表1)。1.5%的琼脂糖凝胶电泳检测其完整性较好,2个样品的RIN值均大于8(表1),完整性较好,可进行后续生物信息分析。

表1 样品总RNA质量信息
2.2 测序序列统计与质控
在测序实验中采用了样品平行混合测序,各样品中的序列均引入了一段标示其样本来源信息的标签序列(约200 bp)。将原始序列进行统计,见表2。Sx样品发酵前后的原始序列相比,序列条数增加了1.09倍。

表2 原始序列统计信息
序列优化后,数据统计表见表3, clean reads约占raw reads的80 %左右,而clean bases约占raw bases的70%左右,序列Sx样品发酵后(Sx-2)的Clean reads约为发酵前(Sx-1)1.14倍。

表3 优化序列统计信息
将优化序列进行组装拼接,组装结果见表4。两个样品(Sx-1,Sx-2)用于宏转录分析的序列条数Seqs分别为15 659、15 670, N50与N90数值表明序列拼接效果较好,可以进一步分析。
表4 序列组装拼接统计信息
注:N50(N90):将各转录本序列按长度大小排序,从大至小逐一扫描各条序列的长度值,进行累加,当该累加值第一次超过所有序列总长度的50%(90%)时,此时扫描到的序列,其长度值即为N50(N90);相比序列平均长度,N50(N90)更能准确表示此次的序列拼接效果。
发酵前后Sx-1和Sx-2样本的序列长度主要分布在1~700 bp以及700~1 400 bp之间,这与表4中N50和N90值相符合,选取这几个区间段序列长度,可以准确表示此次序列的拼接效果。Sx-2样本中序列长度在700~1 400 bp之间的数量要明显小于Sx-1样本,这可能与发酵前后的差异基因有关,进行下一步生物信息分析。
2.3 物种注释结果
使用BLASTP将基因集与NR数据库进行比对获得不同水平物种注释信息,有210个种水平物种注释信息,其中属水平、种水平的群落分布见图1与图2。
图1和图2中可以看出,接入Sx酸面团后,在发酵开始阶段,以乳杆菌属所占比例大约为55.47%,而旧金山乳杆菌占菌群的44.41%,说明Sx这个传统酸面团发酵前微生物中乳酸菌属中有80.06 %是旧金山乳杆菌。酵母属所占比例大约39.81 %,而酿酒酵母所占总群落的39.64%,即酿酒酵母占Sx酸面团中99.57 %。而发酵高峰时期, 乳杆菌属在菌群中所占比例增大到63.51%,而旧金山乳杆菌在整个菌群中所占比例为51.71%,因此,发酵高峰时期,乳酸菌中有81.42%是旧金山乳杆菌。酵母菌属相对比例降低至32.06%,酿酒酵母在总群落中所占比例为31.91%,即酿酒酵母菌占总酵母属的99.53%。发酵高峰时期,乳酸菌比例明显上升,酵母菌比例相对下降,其原因不仅是由于细菌繁殖速度明显高于真菌,还因为在面团发酵过程中乳酸菌会产生乙酸、乳酸等有机酸,抑制了酵母菌的生长[19]。

图1 属水平微生物群落结构图

图2 种水平微生物群落结构图
2.4 GO功能注释结果
利用GO数据库,对Sx-1,Sx-2样品中基因参与的生物过程、分子功能及细胞组分三个方面进行分类注释。与发酵起始阶段(Sx-1)相比较,发酵高峰时期(Sx-2)在催化活性与单一生物过程的基因丰度变化最大,基因丰度增加超过了20%。其次为抗氧化活性、细胞过程、发展过程、代谢过程及转运活性方面,基因丰度增加了约为10%左右。说明Sx样品中微生物在这些方面的基因过表达,可能相关的代谢呈现了正调节作用,而在细胞连接、酶调节剂活性、鸟苷酸交换因子活性、大分子复合物等方面,基因丰度呈现降低趋势,降低比例超过10%。
催化活性方面的基因丰度超过20%,细胞过程、发展过程、代谢过程转运活性方面的基因丰度超过10%,可能是由于乳酸菌和酵母菌之间存在代谢产物的互补机制,酵母菌在发酵过程中产生的代谢产物可为乳酸菌的生长提供了营养因子,而旧金山乳杆菌生成的葡萄糖可成为酵母菌的能量来源。当发酵体系中乳酸菌生长需要的营养因子过多时,乳酸菌会释放信号,加速其生长发育速率,缩短生长发育周期,代谢产物增多,又会为酵母菌生长提供更多能量来源,促进酵母菌生长。因此在发酵高峰期,调控催化活性、细胞发育过程以及代谢过程等的基因丰度会相对增加[14]。
四类参与生物过程的差异基因呈极显著富集(P<0.001),分别为通过UDP-半乳糖的半乳糖分解代谢过程、碳水化合物代谢过程、碳水化合物转运和半乳糖代谢过程,主要是参与碳水化合物及半乳糖的代谢过程的相关基因。
参与碳水化合物过程的相关基因变化显著,主要集中在糖酵解、TCA循环、半乳糖代谢过程中,这些显著变化会对酸面团风味品质带来一定影响:其中,麦芽糖是旧金山乳杆菌在发酵过程中优先利用的碳水化合物,参与糖酵解代谢途径的己糖激酶基因上调表明在酸面团混合发酵过程中旧金山乳杆菌将麦芽糖分解为葡萄糖和葡萄糖-1-磷酸,使得酿酒酵母对葡萄糖的利用率增加[20],此外,果糖也能作为额外的电子受体被旧金山乳杆菌接收,促进了旧金山乳杆菌对它的吸收利用,上述原因使得旧金山乳杆菌与酵母菌可以同时稳定存在于酸面团中,不会由于对碳水化合物的竞争而互相抑制生长[8,21]
单一生物碳水化合物分解代谢过程,碳水化合物分解代谢过程,单一生物碳水化合物代谢过程,己糖代谢过程,单糖代谢过程,半乳糖转运和半乳糖跨膜转运蛋白活性,七个具有显著差异(P<0.01)富集的基因也主要是碳水化合物及半乳糖参与的生物代谢过程。此外,己糖分解代谢过程,单糖分解代谢过程,丙酮酸代谢过程和麦芽糖代谢过程,即单糖相关的代谢过程具有显著富集差异(P<0.05)。
旧金山乳杆菌与酿酒酵母混合发酵会使酿酒酵母增加葡萄糖的利用,加强氨基酸代谢,减少乙酸、乙醇的合成。这是由于发酵过程中丙酮酸代谢相关差异基因呈显著富集,其中丙酮酸脱羧酶参与酒精发酵,转化丙酮酸为乙醛,乙醛再代谢生成乙醇,与丙酮酸代谢的相关基因表达下调导致乙醇含量减少。己糖激酶基因上调表明酿酒酵母对葡萄糖利用增加[23]。
旧金山乳杆菌菌株是以麦芽糖、果糖、核糖和葡萄糖酸盐作为碳源。当使用果糖、柠檬酸或α-酮戊二酸作为替代电子受体时,通常会加速以麦芽糖为碳源的旧金山乳杆菌的生长,且旧金山乳杆菌的基因组只编码不完全柠檬酸循环,因为在旧金山乳杆菌中只含有马酸水合酶、苹果酸脱氢酶及柠檬酸裂解酶[24]。发酵期间的碳水化合物代谢过程极其活跃,因此参与此过程的差异基因呈极显著性富集。
转录组的结果表明在面团发酵过程中,起主要代谢作用的是酿酒酵母和旧金山乳杆菌,具体菌种参与的具体代谢过程需要进一步进行单菌种转录及互作转录分析。
对Sx-1与Sx-2两个发酵阶段,微生物差异基因表达进行 GO功能显著性富集分析,在基因功能水平阐明两个样品间的差异。
2.5 COG功能注释结果
通过与eggNOG数据库比对,得到基因对应的COG功能分类注释,其丰度盒装图见图3。
数据库共注释了25个COG功能分类,其中与翻译,核糖体结构和生物学相关的基因丰度值最高,其次为碳水化合物的运输和新陈代谢的相关基因丰度值较高,且面团发酵前后基因丰度值变化最大。随着发酵的进行,伴随的能量代谢相关基因也呈现较大的变化,即能源生产和转换。这与GO注释结果也基本一致。
2.6 差异基因KEGG富集分析
传统酸面团中的各菌种基因表达产物之间存在代谢产物互补机制,并非单独的发挥作用,不同微生物的代谢产物之间相互协调来实现菌种相互作用。对Sx-1与Sx-2样品所获得的差异表达基因进行KEGG注释,在差异表达基因中显著富集的KEGG通路(见图4)。
半乳糖代谢途径的通路中基因表达富集差异最为显著(P<0.001),这与GO与COG注释结果一致。参与糖酵解、糖异生与氯代烷烃和氯代烯烃降解的代谢途径的相关基因表达也呈现较显著的差异(P<0.01)。此外,丙酮酸代谢,芳香化合物的降解,脂肪酸降解和萘降解的相关代谢途径中基因表达也呈现较为显著的差异(P<0.05)。
混合发酵过程中,氨基酸代谢过程差异基因也呈显著性富集,发酵初始阶段,由于乳酸菌与酵母菌的生长消耗了大量的氨基酸致使氨基酸的含量有所下降,随着发酵过程的进行,乳酸菌产生大量的乳酸以及乙酸导致酸面团pH下降,蛋白酶的活性上升,分解蛋白质产生氨基酸,导致酸面团中氨基酸的含量上升[25],芳香族氨基酸通过转氨和脱氨途径产生酸面团中的风味物质[24]。后期阶段,苯丙氨酸在酸面团中含量明显上升,说明乳酸菌与酵母菌混合发酵导致的芳香族氨基酸含量上升对酸面团风味物质的形成具有重要作用。
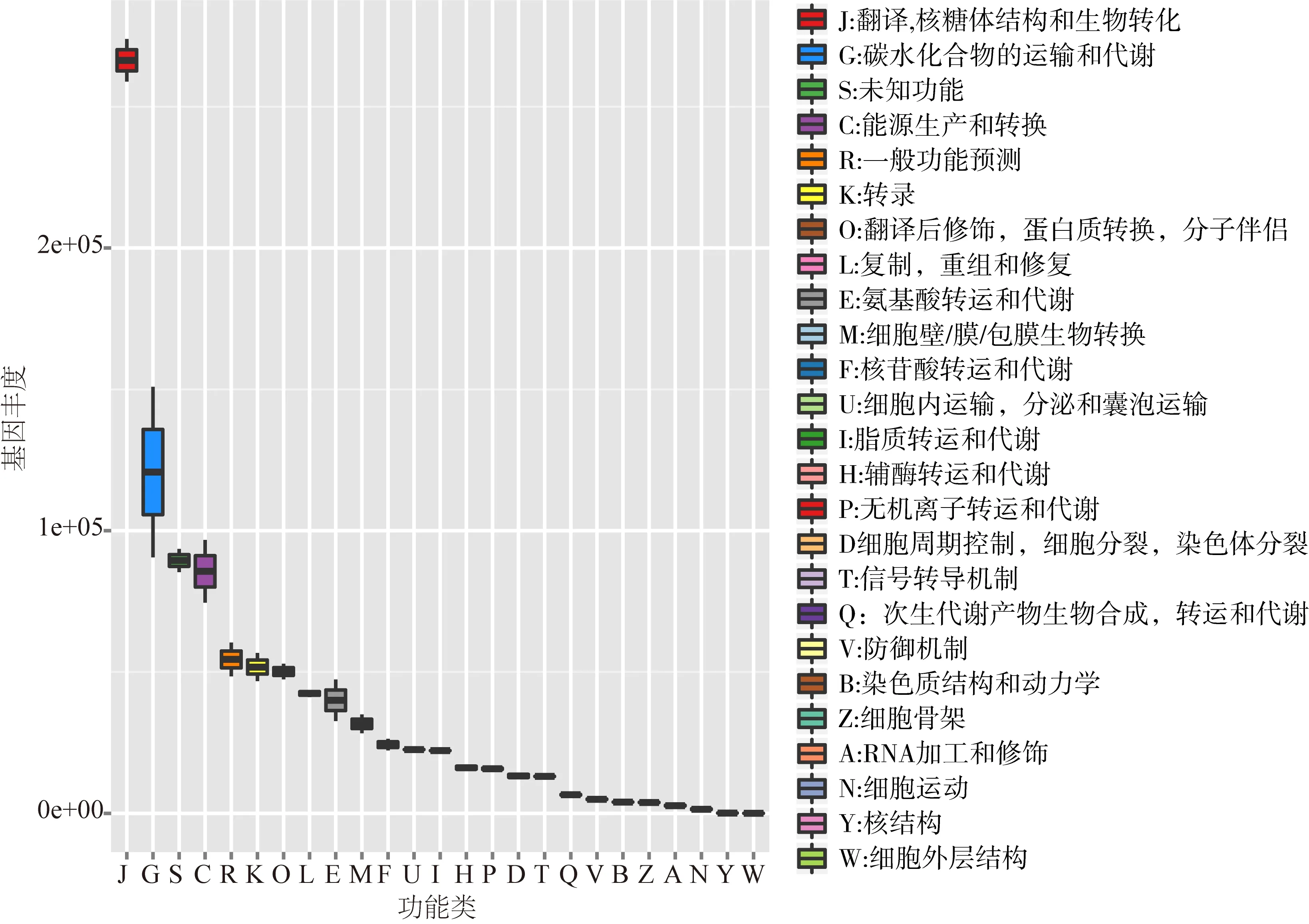
图3 Sx样品不同发酵时期基因表达的COG注释结果

注:P<0.001的标记为***,P<0.01的标记为**,P<0.05的标记为*,右侧颜色梯度表示P值大小。图4 Sx样品不同发酵时期基因表达的KEGG富集分析结果
丙酮酸途径中,由于丙酮酸氧化酶基因发生移码,旧金山乳杆菌内丙酮酸直接转化为乙酰磷酸是无法实现的,因此旧金山乳杆菌首先通过乳酸将丙酮酸转化为乙酸,然后从乙酸生成乙酰磷酸。丙酮酸盐可由旧金山乳杆菌生产,除了通过磷酸酮醇酶途径从糖和葡萄糖酸盐形成外,还可由天冬酰胺和丙氨酸通过转氨基作用产生以及苹果酸脱氢酶催化的苹果酸产生。在发酵过程中,丙酮酸代谢过程相对活跃,因此参与丙酮酸代谢途径的相关基因表达富集呈现较为显著差异[22]。
2.7 差异物种表达模式聚类分析
对Sx-1与Sx-2两个样品中物种进行热图聚类分析,筛选显著差异物种进行表达模式聚类分析。Sx-1与Sx-2两个样品间群落结构模式聚类图表明Sx两个发酵阶段中,相对丰度高的物种都是酿酒酵母和旧金山乳杆菌,这说明这两个菌种是Sx发酵过程中起着关键作用的物种,这与先前的研究结果相一致。肠系膜明串珠菌、干酪乳杆菌等菌种在发酵后,相对丰度有所增加,而类谷糠乳杆菌的菌种在发酵后,相对丰度略有降低。
经过传统酸面团Sx发酵后的面团,不同物种的基因表达呈现一定的差异,但是总体而言,两个样品的群落结构相似性更大,差异性相对较小。
3 结论
通过宏转录组学技术,对传统酸面团Sx样品发酵不同阶段中优势菌种的变化、关键功能基因注释及代谢途径等进行了分析,结果表明Sx样品发酵过程中是以乳杆菌属与酵母菌属为优势菌属,而其中主要以旧金山乳杆菌与酿酒酵母为关键优势菌种。在Sx酸面团发酵过程中,参与碳水化合物及半乳糖代谢过程相关的差异基因呈极显著富集,基因丰度超过20%,说明在酸面团发酵过程中,这两种代谢途径占据重要地位。本实验对传统酸面团Sx样品在面团发酵前后微生物菌群结构、功能基因表达及代谢途径的研究,探讨面团发酵过程中起关键作用的菌种,为进一步研究传统发酵面食的品质影响因素提供参考。
