云纹石斑鱼、鞍带石斑鱼及其杂交F1的DNA甲基化分析*
2019-12-06李炎璐陈建国翟介明
李炎璐 陈 超 陈建国 翟介明
云纹石斑鱼、鞍带石斑鱼及其杂交F1的DNA甲基化分析*
李炎璐1陈 超1①陈建国1翟介明2
(1. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业可持续发展重点实验室青岛市海水鱼类种子工程与生物技术重点实验室 青岛 266071;2. 莱州明波水产有限公司 莱州 261400)
为探讨石斑鱼杂种优势形成过程中基因组DNA甲基化水平的变化,本研究采用甲基化敏感扩增多态性(Methylation-sensitive amplification polymorphism, MSAP)技术检测云纹石斑鱼()、鞍带石斑鱼()及云纹石斑鱼(♀)×鞍带石斑鱼(♂)杂交F13个群体的基因组DNA甲基化水平,分析杂交F1与亲本基因组DNA甲基化水平的差异。结果显示,云纹石斑鱼和鞍带石斑鱼的基因组DNA属于甲基化程度较高的类群;云纹石斑鱼、鞍带石斑鱼及其杂交F1的DNA总甲基化率分别为60.62%、59.38%和55.78%,DNA全甲基化率分别为31.37%、30.67%和29.27%,DNA半甲基化率分别为29.25%、28.71%和26.51%;杂交F1的DNA总甲基化率、全甲基化率和半甲基化率均低于双亲,并存在极显著差异(<0.01),3个群体的全甲基化率均大于半甲基化率。研究表明,云纹石斑鱼(♀)×鞍带石斑鱼(♂)杂交F1DNA甲基化水平与杂种优势呈负相关,杂交F1DNA甲基化水平的降低可能是形成快速生长等杂种优势的原因之一。
云纹石斑鱼;鞍带石斑鱼;杂交F1;DNA甲基化
杂种优势是不同基因型的亲本杂交产生的杂交子代在生长、成活、抗逆性和品质等方面均优于双亲的现象。表观遗传学是指基于非基因序列改变所致基因表达水平变化,主要包括DNA甲基化、组蛋白修饰、染色体空间结构的调控和非编码RNA调控等。其中,DNA甲基化是最重要的基因表观修饰方式之一,尤其是胞嘧啶甲基化,能够保持整个基因组DNA的稳定,并调控特定组织基因的表达(Cheng, 2008)。Cedar(1988)研究表明,基因组DNA的甲基化水平及分布与基因表达率显著相关。
用于分析DNA甲基化的方法很多,包括甲基化胞嘧啶分离分析技术、甲基化生物亲和检测技术、甲基化特异性扩增技术、甲基化测序检测分析技术、甲基化基因芯片及图谱检测技术和甲基化传感器检测分析技术等。其中,甲基化敏感扩增多态性(MSAP)技术因简便、高效、可靠等特点而得到广泛应用,可在全基因组水平检测CCGG位点的胞嘧啶甲基化程度,能够准确分析不同基因组DNA甲基化差异。目前,该技术已应用于水稻() (Xiong, 1999)、猪()(蒋曹德, 2004)、草鱼()(曹哲明等, 2007)、湿地松(, ♀)×加勒比松(, ♂)杂交F1(李义良等, 2018)、栉孔扇贝(, ♀)×虾夷扇贝(, ♂)杂交F1(于涛等, 2010)、橙色莫桑比克罗非鱼(, ♀)×荷那龙罗非鱼(, ♂)杂交F1(尚慧文等, 2017)等多种生物以及杂交F1与亲本间的基因组DNA甲基化研究中。
云纹石斑鱼()和鞍带石斑鱼()均为暖温性礁栖鱼类,云纹石斑鱼主要分布于日本、韩国以及我国的东海和南海,具有个体大、生长快、肉质鲜美的特性(郭明兰等, 2008; 宋振鑫等, 2012);鞍带石斑鱼主要分布于东南亚、大洋洲海域,在我国主要分布于南海诸岛,数量较少,是石斑鱼类中体型最大的种类,具有生长速度快、抗病性强、营养价值高等优点(Heemstra, 1993; Sadovy, 2000)。前期研究表明,云纹石斑鱼(♀)×鞍带石斑鱼(♂)杂交F1(张梦淇等, 2016)在生长、抗逆性(邵彦翔等, 2017)、营养成分(王林娜等, 2018)等方面均具有明显的杂种优势,是值得开发的优良新品种。本研究运用MSAP技术检测云纹石斑鱼、鞍带石斑鱼及云纹石斑鱼(♀)×鞍带石斑鱼(♂)杂交F13个群体的基因组DNA甲基化水平,分析杂交F1与亲本基因组DNA甲基化水平的差异,从而研究基因组DNA甲基化水平的变化与杂种优势的关系,为从表观遗传学方面揭示石斑鱼杂种优势的形成机制提供理论基础。
1 材料与方法
1.1 实验材料
实验材料均取自山东莱州明波水产有限公司,选取云纹石斑鱼自交子代幼鱼群体、鞍带石斑鱼自交子代幼鱼群体以及云纹石斑鱼(♀)×鞍带石斑鱼(♂)杂交F1幼鱼群体为样本,每个群体分别随机选取30尾,取尾鳍于无水乙醇中,-20℃保存备用。
1.2 DNA提取
DNA的提取采用天根海洋动物组织基因组DNA提取试剂盒,参照试剂盒说明书进行操作。用1%琼脂糖凝胶电泳和紫外分光光度计检测DNA质量与浓度。
1.3 甲基化扩增敏感多态性(MSAP)分析
MSAP分析参照Xiong等(1999)的方法,并对实验条件进行优化。
1.3.1 双酶切及连接反应 对各基因组DNA分别用RⅠ+Ⅱ和RⅠ+Ⅰ进行双酶切反应。反应体系包括200 ng基因组DNA、2 UⅡ/Ⅰ、2 URⅠ、2.5 μl 10×Buffer Tango,加ddH2O至20 μl,37℃水浴酶切6 h,再65℃酶切变性10 min。连接体系包括5 μl酶切产物、1 μl HM接头、l μl E接头、5 U T4DNA连接酶、4 μl 5×T4DNA连接酶缓冲液,加ddH2O至20 μl,16℃连接过夜,产物稀释l0倍用于预扩增。实验所用接头与引物组合序列如表1所示。
表1 所用接头和引物序列

Tab.1 Sequence of adapters and primers used for the MSAP analysis
1.3.2 预扩增反应 预扩增体系包括2 μl稀释连接产物、1 μl E0和HM0预扩引物、0.5 μlDNA Polymerase、2.5 μl 10×Buffer、0.5μl dNTPs,加ddH2O至25 μl。预扩增程序设置为94℃预变性2 min、94℃变性30 s、56℃退火30 s、72℃延伸80 s,共进行30个循环,72℃ 5 min。产物稀释20倍用于选择性扩增反应。
1.3.3 选择性扩增反应 选择扩增体系包括2 μl 稀释预扩增产物、1 μl HMn和En选扩引物、0.5 μlDNA Polymerase、2.5 μl 10×Buffer、0.5 μl dNTPs,加ddH2O至25 μl。选择性扩增反应设置为94℃预变性2 min;第1轮扩增参数为94℃ 30 s、65℃ 30 s、72℃ 80 s,之后每轮循环,温度递减0.7℃,共进行12个循环;第2轮扩增参数为94℃ 30 s、55℃ 30 s、72℃ 80 s,共进行23个循环,72℃延伸10 min,4℃保存。
1.3.4 产物检测 用ABI Prism 377型测序仪电泳检测片段大小,通过GENESCAN软件进行数据收集和分析,输出凝胶电泳图谱。
1.3.5 数据统计与分析 对同一样品DNA分别用RⅠ+Ⅱ和RⅠ+Ⅰ识别并切割CCGG序列,扩增出的每条片段代表1个CCGG识别位点,在MSAP图谱上,每个样品分别对应有H和M两个泳道,同一位点上有条带出现计为“1”,无条带计为“0”,根据扩增产物在2个泳道内有无的情况,将甲基化模式分为4种类型。I型,2个泳道均有带,即“H、M”带型为“1、1”,为去甲基化位点;Ⅱ型,H泳道有带,M泳道无带,即带型为“1、0”,为单链外甲基化,代表半甲基化位点;Ⅲ型,H泳道无带,M泳道有带,即带型为“0、1”,为双链内甲基化,为全甲基化位点;Ⅳ型,H和M泳道均没有带“0、0”,为双链外甲基化的位点,Ⅳ型位点不进行统计。统计50~500 bp的条带,计算各群体的半甲基化率、全甲基化率和总甲基化率,并利用SPSS 19.0进行方差分析。计算方法为:半甲基化率(%)=半甲基化条带数(Ⅱ)/总扩增条带数(Ⅰ+Ⅱ+Ⅲ)×100%,全甲基化率(%)=全甲基化条带数(Ⅲ)/总扩增条带数(Ⅰ+Ⅱ+Ⅲ)×100%,总甲基化率(MSAP)(%)=甲基化总条带数(Ⅱ+Ⅲ)/总扩增条带数(Ⅰ+Ⅱ+Ⅲ)×100%。
2 结果
2.1 引物组合
云纹石斑鱼、鞍带石斑鱼和云纹石斑鱼(♀)×鞍带石斑鱼(♂)杂交F1的基因组DNA经酶切、连接和预扩增后,由7个E和8个HM引物组成的引物组合进行选择性扩增,根据所有扩增结果,筛选8对具有多态性好、重复性好、条带清晰的选扩引物组合(E1HM6、E3HM5、E3HM6、E4HM7、E4HM8、E5HM4、E5HM7和E5HM8)进行基因组DNA的MSAP分析。MSAP扩增片段长度大多在50~1000 bp,主要集中在50~500 bp,大于500 bp条带很少,因此,本研究统计的数据为50~500 bp的条带。
2.2 3个石斑鱼群体基因组DNA甲基化MSAP图谱
3个石斑鱼群体基因组DNA的选扩产物经聚丙烯酰氨凝胶电泳后,得到的扩增结果MSAP图谱如图1所示。结果显示,DNA甲基化MSAP图谱形成了3种不同类型的条带,包括“H、M”条带为“1、1”的Ⅰ型、“H、M”条带为“1、0”的Ⅱ型和“H、M”条带为“0、1”的Ⅲ型3种甲基化类型。

图1 3个石斑鱼群体基因组DNA甲基化MSAP图谱(E4HM7)
H:RⅠ/Ⅱ双酶切;M:RⅠ/Ⅰ双酶切;I:去甲基化位点;Ⅱ:半甲基化位点;Ⅲ:全甲基化位点
H:RⅠ/Ⅱdouble digestion; M:RⅠ/Ⅰdouble digestion;I: Unmethylated sites; Ⅱ: Hemi-methylated sites;Ⅲ: Fully methylated sites
2.3 3个石斑鱼群体间DNA甲基化水平差异分析
8对引物对3个石斑鱼群体的扩增结果如表2所示。云纹石斑鱼、鞍带石斑鱼和杂交F13个群体扩增的总位点数分别为5840、5781和6246,其中,总甲基化位点分别为3540、3433和3484。云纹石斑鱼群体的总甲基化率为60.62%,其中,全甲基化率为31.37%,半甲基化率为29.25%;鞍带石斑鱼群体的总甲基化率为59.38%,其中,全甲基化率为30.67%,半甲基化率为28.71%;杂交F1群体的总甲基化率为55.78%,其中,全甲基化率为29.27%,半甲基化率为26.51%。结果显示,杂交F1的DNA总甲基化率、全甲基化率和半甲基化率均低于亲本,杂交F1与双亲之间的DNA甲基化水平均存在极显著差异(<0.01),3个群体的全甲基化率均大于半甲基化率。
3 讨论
3.1 DNA甲基化水平的差异性
不同物种间甲基化水平存在一定的差异,可能是由于在真核生物的不同物种中,DNA甲基化的调控作用所占的重要性不同而造成的,同时,不同物种相同组织以及同一物种不同组织的甲基化水平均存在差异。近年来,随着MSAP技术的不断发展,MSAP在动植物研究中得到了广泛应用。研究表明,棉花(spp.)的总甲基化水平为20%~27.1% (赵云雷, 2007);哺乳动物如猪不同组织的总甲基化水平为40.6%和44.8%,羊()为45.7%(唐韶青等, 2006);禽类如鸡()不同组织的总甲基化水平为44.6%和47.9%,鸭为40.7%(唐韶青等, 2006);在水产动物中,总甲基化水平大多集中在20%~50%之间,如栉孔扇贝为32.79%、虾夷扇贝为24.13%(于涛等, 2010)、太平洋牡蛎()为26% (Jiang, 2013)、刺参()为23.36%~56.52% (郭婷婷等, 2013; Zhao, 2015; 左之良等, 2016)、背角无齿蚌()为35.5%~56% (曹哲明等, 2009)、文蛤()为26.7%~30.0% (朱东丽, 2011)、尼罗罗非鱼()为23.74% (朱华平等, 2013)。在周莹等(2017)的报道中,棕点石斑鱼()和鞍带石斑鱼的总甲基化率分别为57.18%和63.16%。在本研究中,云纹石斑鱼和鞍带石斑鱼总甲基化率分别为60.62%和59.38%,与周莹等(2017)的研究结果较为接近,由此推测,石斑鱼群体基因组DNA属于甲基化程度较高的类群。
表2 3个石斑鱼群体间DNA甲基化水平比较
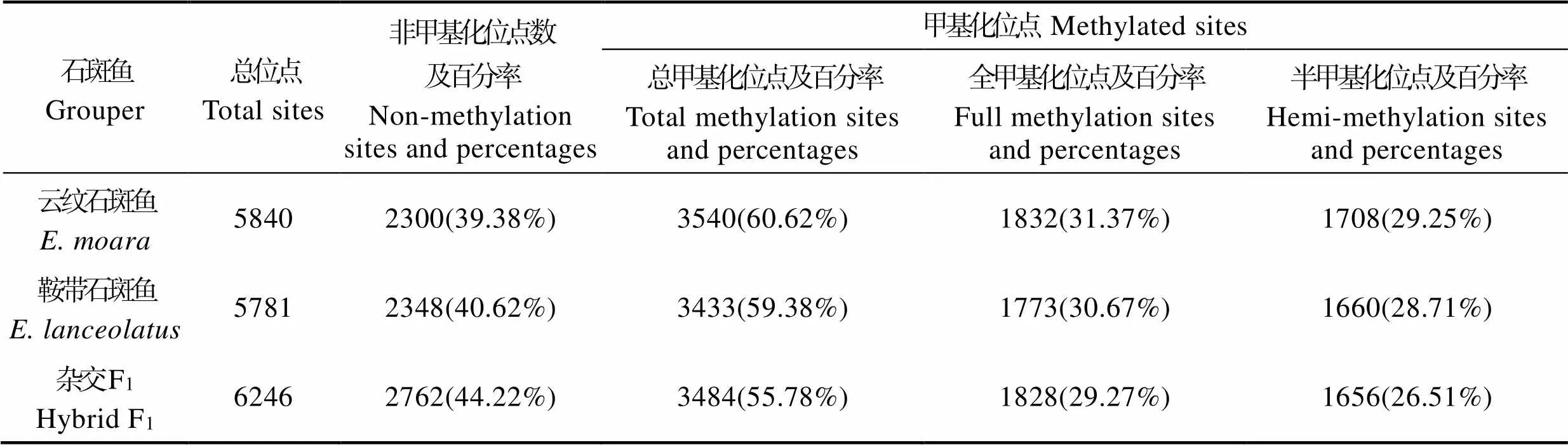
Tab.2 Comparison of DNA methylation levels of E.moara, E.lanceolatus and thehybrid F1
3.2 DNA甲基化水平差异与杂种优势的相关性分析
研究表明,DNA甲基化可以调控基因的表达 (Cedar, 1988),在杂交组合中,亲本基因组DNA甲基化模式经过复杂的调整,将大部分模式稳定遗传给后代外,基因组DNA的甲基化和去甲基化水平均发生了改变。
近年来,学者多倾向于从基因组DNA甲基化的角度分析杂交后代与亲本的差异,以及这种差异与杂种优势的关系。洪舟等(2009)研究发现,杉木杂交F1的甲基化水平呈显著减少的趋势,并且与树高、胸径和材积性状的杂种优势均呈显著负相关;重庆山地黄牛()杂交F1群体的总甲基化水平、全甲基化水平和半甲基化水平均低于双亲,并且甲基化水平与胸围、体重等杂种优势率存在显著负相关(万亚琴, 2008);栉孔扇贝(♀)×虾夷扇贝(♂)杂交F1的总甲基化率降低,显示DNA甲基化率与生长相关的性状呈负相关(吴彪等, 2012);橙色莫桑比克罗非鱼、荷那龙罗非鱼以及杂交F1莫荷罗非鱼“广福1号”的总甲基化率分别为32.21%、38.03%和29.77%,杂交F1的甲基化水平低于双亲,杂交F1幼鱼在生长速度、耐盐胁迫方面具有明显的杂种优势(刘玉姣, 2014; 李茜茜, 2016),研究推测,杂交F1基因组DNA甲基化水平的降低和甲基化模式的重调可能与耐盐胁迫的杂种优势有关(尚慧文等, 2017);棕点石斑鱼(♀)×鞍带石斑鱼(♂)杂交F1是具有抗病力强、生长速度快等较强杂种优势的杂交后代,棕点石斑鱼、鞍带石斑鱼和杂交F1的DNA总甲基化率分别为57.18%、63.16%和54.76%,杂交F1的DNA总甲基化率低于双亲,推测甲基化水平的降低有利于杂种优势的形成(周莹等, 2017)。
本研究中,云纹石斑鱼(♀)×鞍带石斑鱼(♂)杂交F1的DNA总甲基化率(55.78%)低于云纹石斑鱼(60.62%)和鞍带石斑鱼(59.38%),杂交F1的DNA全甲基化率和半甲基化率也均显著低于双亲,这与上述研究发现的杂交F1的甲基化水平降低的结果相一致。3个石斑鱼群体基因组DNA的全甲基化位点数均多于半甲基化位点数,与哺乳动物基因组全甲基化位点较多、半甲基化位点较少的研究结果相一致(唐韶青等, 2006)。杂交F1(张梦淇等, 2016)在生长、抗逆性(邵彦翔等, 2017)、营养成分(王林娜等, 2018)等方面均具有明显的杂种优势,结合本研究,杂交F1的DNA甲基化水平与杂种优势呈负相关,推测杂交F1基因组DNA甲基化水平的降低、甲基化模式的改变和重新调整可能是形成快速生长等杂种优势的诸多原因之一。
上述动植物DNA甲基化水平与杂种优势均呈负相关关系,研究证明了杂种优势的形成与杂交F1基因组DNA总甲基化水平的降低有关。一般来说,DNA甲基化与基因表达呈负相关关系,杂交后代基因组DNA甲基化水平降低,激活了许多沉默基因,基因的表达活性增强(Tsaftaris, 1997),使杂交后代表现出杂种优势。关于特异基因的甲基化水平与杂种优势之间的关系,以及这些DNA甲基化位点的遗传方式、对基因结构及功能的作用和调控机制还有待进一步研究。
Cao ZM, Ding WD, Yu JH,. Differences in methylated loci among different grass carp individuals from one pair of parents. Acta Zoologica Sinica, 2007, 53(6): 1083–1088 [曹哲明, 丁炜东, 俞菊华, 等. 草鱼全同胞鱼苗不同个体甲基化位点的差异. 动物学报, 2007, 53(6): 1083–1088]
Cao ZM, Yang J. Analysis of the methylation in genome DNA from different tissues of. Ecology and Environmental Science, 2009, 18(6): 2011–2016 [曹哲明, 杨健. 背角无齿蚌不同组织的基因组DNA甲基化分析. 生态环境学报, 2009, 18(6): 2011–2016]
Cedar H. DNA methylation and gene activity. Cell, 1988, 53(1): 3–4
Cheng XD, Blumenthal RM. Mammalian DNA methyltransferases: A structural perspective. Structure, 2008, 16(3): 341–350
Guo ML, Su YQ, Chen XF,. Comparative studies on morphology ofand. Acta Oceanologica Sinica, 2008, 30(6): 106–114 [郭明兰, 苏永全, 陈晓峰, 等. 云纹石斑鱼与褐石斑鱼形态比较研究. 海洋学报, 2008, 30(6): 106–114]
Guo TT, Sun GH, Yang JM,. MSAP analysis of genome and methylation in different tissues of. Oceanologia et Limnologia Sinica, 2013, 44(1): 77–82 [郭婷婷, 孙国华, 杨建敏, 等. 刺参()不同组织基因组甲基化状态MSAP分析. 海洋与湖沼, 2013, 44(1): 77–82]
Heemstra PC, Randall JE. Groupers of the world. FAO Fisheries Synopsis, 1993, 16 (125): 130–132
Hong Z, Shi JS, Zheng RH,. Epigenetic inheritance and variation of DNA methylation in Chinese fir() intraspecific hybrids. Molecular Plant Breeding, 2009, 7(3): 591–598 [洪舟, 施季森, 郑仁华, 等. 杉木亲本自交系及其杂交种DNA甲基化和表观遗传变异. 分子植物育种, 2009, 7(3): 591–598]
Jiang CD. Relationship between DNA methylation and hybrid performances and pig genetic diversity analysis. Doctoral Dissertation of Huazhong Agricultural University, 2004 [蒋曹德. 猪DNA甲基化与杂种表现的关系及遗传多样性分析. 华中农业大学博士研究生学位论文, 2004]
Jiang Q, Li Q, Yu H,. Genetic and epigenetic variation in mass selection populations of Pacific oyster. Genes and Genomics, 2013, 35(5): 641–647
Li QQ. Cloning and expression characterization analysis ofandgenes related to salt-alkaline tolerance in tilapia. Master´s Thesis of Shanghai Ocean University, 2016 [李茜茜. 罗非鱼耐盐碱相关基因、克隆和表达特征分析. 上海海洋大学硕士研究生学位论文, 2016]
Li YL, Zhao FC, Zhong SY,. Epigenetic analysis and DNA methylation in×and their parents. Molecular Plant Breeding, 2018, 16(1): 76–81 [李义良, 赵奋成, 钟岁英, 等. 湿加松及亲本DNA甲基化和表观遗传分析. 分子植物育种, 2018, 16(1): 76–81]
Liu YJ. A study on salt tolerance related genes of,and their hybrids. Master´s Thesis of Shanghai Ocean University, 2014 [刘玉姣. 橙色莫桑比克罗非鱼、荷那龙罗非鱼及其正反交子代耐盐相关基因的研究. 上海海洋大学硕士研究生学位论文, 2014]
Sadovy Y, Cornish AS. Reef fishes of Hong Kong. Hong Kong: Hong Kong University Press, 2000
Shang HW, Liu ZG, Zhu HP,. Analysis of DNA methylation difference between“Mohe” hybrid tilapia and its parents. Journal of Fisheries of China, 2017, 41(11): 1699–1709 [尚慧文, 刘志刚, 朱华平, 等. 莫荷罗非鱼“广福1号”与其亲本间DNA甲基化的差异分析. 水产学报, 2017, 41(11): 1699–1709]
Shao YX, Chen C, Li YL,. Effects of low temperature stress on serum biochemical indices of the juvenile hybrid of(♀) ×(♂). Progress in Fishery Sciences, 2017, 38(2): 70–76 [邵彦翔, 陈超, 李炎璐, 等. 低温胁迫对云纹石斑鱼(♀)×鞍带石斑鱼(♂)杂交后代血清生化指标的影响. 渔业科学进展, 2017, 38(2): 70–76]
Song ZX, Chen C, Zhai JM,. Embryonic development and morphological characteristics of larval, juvenile and young kelp bass,Progress in Fishery Sciences, 2012, 33(3): 26–34 [宋振鑫, 陈超, 翟介明, 等. 云纹石斑鱼胚胎发育及仔、稚、幼鱼形态观察. 渔业科学进展, 2012, 33(3): 26–34]
Sun Q, Wu L, Ni Z,. Differential gene expression patterns in leaves between hybrids and their parental inbreds are correlated with heterosis in a wheat diallel cross. Plant Science, 2004, 166: 651–657Tang SQ, Zhang Y, Xu Q,. Analysis of methylation level of genome in various tissues of different animal species. Journal of Agricultural Biotechnology, 2006, 14(4): 507–510 [唐韶青, 张沅, 徐青,等. 不同动物部分组织基因组甲基化程度的差异分析. 农业生物技术学报, 2006, 14(4): 507–510]
Tsaftaris AS, Kafka M, Polidoros A,. Epigenetic changes in maize DNA and heterosis. Cimmyt, 1997, 112
Wan YQ. Study on the relationship between DNA methylation and heterosis in beef cattle. Master´s Thesis of Southwest University, 2008 [万亚琴. DNA甲基化与肉牛杂种优势关系的初步研究. 西南大学硕士研究生学位论文, 2008]
Wang LN, Tian YS, Tang J,. Analysis and quality evaluation of nutritional components in the muscle of,and hybrid “Yunlong grouper”. Journal of Fisheries of China, 2018, 42(7): 1085–1093 [王林娜, 田永胜, 唐江, 等. 云纹石斑鱼、鞍带石斑鱼及杂交“云龙斑”肌肉营养成分分析及品质评价. 水产学报, 2018, 42(7): 1085–1093]
Wu B, Yang AG, Liu ZH,. Comparison of early growth and DNA methylation betweenhybridized families and self- fertilize families of two scallops. Marine Sciences, 2012, 36(2): 1–6 [吴彪, 杨爱国, 刘志鸿, 等. 两种扇贝杂交和自交家系早期生长及甲基化的比较分析. 海洋科学, 2012, 36(2): 1–6]
Xiong LZ, Xu CC, Saghai MA,. Patterns of cytosine methylation in an elite rice hybrid and its parental lines, detected by a methylation-sensitive amplification polymorphism technique. Molecular and General Genetics, 1999, 261(3): 439–446
Yu T, Yang AG, Wu B,. Analysis of,and their offspring using methylation-sensitive amplification polymorphism (MSAP). Journal of Fisheries of China, 2010, 34(9): 1335–1342 [于涛, 杨爱国, 吴彪, 等. 栉孔扇贝、虾夷扇贝及其杂交子代的MSAP分析. 水产学报, 2010, 34(9): 1335–1342]
Zhang MQ, Chen C, Li YL,. Effects of salinity on the hatching of the fertilized eggs of(♀) ×(♂) and the observation of the morphological development of larvae, juvenile and young fish. Progress in Fishery Sciences, 2016, 37(4): 82–89 [张梦淇,陈超, 李炎璐, 等. 盐度对云纹石斑鱼(♀)×鞍带石斑鱼(♂)受精卵孵化的影响及杂交仔稚幼鱼形态发育观察. 渔业科学进展, 2016, 37(4): 82–89]
Zhao Y, Chen MY, Storey KB,. DNA methylation levels analysis in four tissues of sea cucumber () based on fluorescence-labeled methylation- sensitive amplified polymorphism (F-MSAP) during aestivation. Comparative Biochemistry and Physiology B-Biochemistry and Molecular Biology, 2015, 181: 26–32
Zhao YL. Analysis of DNA methylation and differential gene expression between cotton hybrids and their parents. Doctoral Dissertation of Huazhong Agricultural University, 2007 [赵云雷. 棉花杂交种与亲本间DNA胞嘧啶甲基化及其基因差异表达分析. 华中农业大学博士研究生学位论文, 2007]
Zhou Y, Han YL, Luo J,. Methylation-sensitive amplification polymorphism analysis of genomic DNA methylation on,and their hybrid generation. Journal of Hainan University (Natural Science), 2017, 35(2): 145–151 [周莹, 韩玉龙, 骆剑, 等. 鞍带石斑鱼、棕点石斑鱼及其杂交子代DNA甲基化的MSAP分析. 海南大学学报(自然科学版), 2017, 35(2): 145–151]
Zhu DL. The analysis of molecular markers and DNA methylation in the different strains of. Master´s Thesis of Ocean Universityof China, 2011 [朱东丽. 文蛤()不同壳色花纹品系的分子标记及DNA甲基化分析. 中国海洋大学硕士研究生学位论文, 2011]
Zhu HP, Lu MX, Huang ZH,. Effect of low temperature on genomic DNA methylation in Nile tilapia (). Journal of Fisheries of China, 2013, 37(10): 1460–1467 [朱华平, 卢迈新, 黄樟翰, 等. 低温对罗非鱼基因组DNA甲基化的影响. 水产学报, 2013, 37(10): 1460–1467]
Zuo ZL, Tan J, Wu B,. MSAP analysis of genomic DNA in the tissues ofand white. Progress in Fishery Sciences, 2016, 37(3): 93– 100 [左之良, 谭杰, 吴彪, 等. 普通刺参()和白刺参不同组织基因组DNA的MSAP研究. 渔业科学进展, 2016, 37(3): 93–100]
DNA Methylation Analysis of,and Their F1Hybrid
LI Yanlu1, CHEN Chao1①, CHEN Jianguo1, ZHAI Jieming2
(1. Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Qingdao Key Laboratory of Marine Fish Breeding and Biotechnology, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071; 2. Laizhou Mingbo Fisheries Limited Company, Yantai 261400)
DNA methylation is one of the most important epigenetic modifications. It is vital to maintain the stability of the whole genome and regulate the expression of tissue-specific genes. Methylation level and distribution throughout genomic DNA are significantly correlated with gene expression rate. In order to investigate the changes of genomic DNA methylation level associated with heterosis ofspp., we applied methylation-sensitive amplification polymorphism (MSAP) technology to detect the genomic DNA methylation levels of longtooth grouper (), giant grouper (),and their F1hybrid[(♀) ×(♂)], and we analyzed the difference of DNA methylation level between the F1hybrid and parents. The results showed that overall genomic DNA methylation was higher inand. The total methylation rates of,, andtheF1hybrid were 60.62%, 59.38%, and 55.78%, respectively. The rates of fully methylated sites were 31.37%, 30.67%, and 29.27%, respectively, and the hemi-methylation rates were 29.25%, 28.71%, and 26.51%, respectively. The total methylation rate, the full methylation rate, and the hemi-methylation rate of DNA of theF1hybrid were all significantly lower (<0.01) than that of either parent. The full methylation rates were all higher than the hemi-methylation rates of,,andtheF1hybrid. The study also showed that the DNA methylation level of theF1hybrid is negatively correlated with heterosis. Moreover, reduced DNA methylation level of theF1hybrid may be one of the reasons for the heterosis phenomena such as rapid growth. In general, DNA methylation is negatively correlated with gene expression. The DNA methylation level of the hybrid was lower than either parent, allowing otherwise silenced genes to be activated, enhancing gene expression activity, and resulting in the hybrid progeny exhibiting heterosis. The relationship between methylation level of specific genes and heterosis, the genetic pattern of these DNA methylation sites, the effect on gene structure and function, and the regulatory mechanism require further study.
;; Hybrid F1; DNA methylation
S917.4
A
2095-9869(2019)06-0098-07
10.19663/j.issn2095-9869.20180910001
http://www.yykxjz.cn/
李炎璐, 陈超, 陈建国, 翟介明. 云纹石斑鱼、鞍带石斑鱼及其杂交F1的DNA甲基化分析. 渔业科学进展, 2019, 40(6): 98–104
Li YL, Chen C, Chen JG, Zhai JM. DNA methylation analysis of,and their F1hybrid. Progress in Fishery Sciences, 2019, 40(6): 98–104
* 山东省自然科学基金(ZR2015PC015)、中华人民共和国农业农村部农业国际合作交流项目–热带国家水产养殖科技创新合作(2130114181309)和中国水产科学研究院黄海水产研究所基本科研业务费专项(20603022018023)共同资助 [This work was supported by the Natural Science Foundation of Shandong (ZR2015PC015), Projects of International Exchange and Cooperation in Agriculture, MARA, PRC·Science, Technology and Innovation in Aquaculture with Tropical Countries (2130114181309), and Central Public-Interest Scientific Institution Basal Research Fund of Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences (20603022018023)]. 李炎璐,E-mail: ysfriliyanlu@126.com
陈 超,研究员,E-mail: ysfrichenchao@126.com
2018-09-10,
2018-10-17
CHEN Chao, E-mail: ysfrichenchao@126.com
(编辑 冯小花)
