污水地下渗滤系统微生物代谢组学分析
2019-11-28李英华李海波
杨 蕾,李英华,李海波,苏 菲
污水地下渗滤系统微生物代谢组学分析
杨 蕾,李英华*,李海波,苏 菲
(东北大学资源与土木工程学院,辽宁 沈阳 110000)
应用超高效液相色谱-质谱技术(UPLC-MS),基于代谢组学方法,筛选不同进水有机负荷下地下渗滤系统潜在生物标志物.采用偏最小二乘法(PLS-DA)和主成分分析(PCA)模式识别方法对样品进行分型处理,根据模型的变量重要性因子(VIP值)筛选潜在生物标志物,分析代谢产物蕴含的生物学信息,研究代谢通路,通过RDA分析探索代谢产物与环境因子之间的相关性.PLS-DA模型结果表明,当进水有机负荷为250mg/L、400mg/L、500mg/L时,同一高度层代谢产物间存在显著性差异,共筛选出VIP值大于1.5的230种差异代谢物,以有机酸为主;同时也存在一些醇类、酚类等微生物代谢中间产物.此外,微生物代谢产物与模拟土柱高度有显著对应性.在水力负荷为0.14m3/(m2·d),COD污染负荷为400mg/L条件下,3个高度层H2(500mm)、H4(1000mm)、H6(1500mm)筛选出53种VIP值大于1.5的差异代谢物,以酸类、酮类物质为主.RDA分析表明,随着有机负荷波动和剖面高度的变化,代谢产物受到氧化还原环境(ORP)与硝态氮(NO3-)的影响较大,呈负相关.
地下渗滤系统;代谢组学;微生物;潜在生物标志物
污水地下渗滤系统(SWIS)是一种由非均匀介质组成多相共存的污水生态处理技术,具有运行费用低,处理效果好,维护管理简单,生态效益明显等优点[1],其理论研究和工程应用受到广泛关注.
近年来,传统平板法、变性梯度凝胶电泳(PCR- DGGE)、温度梯度凝胶电泳(TGGE)等传统生物技术被广泛应用于SWIS微生物种群结构分析.通常认为利用微生物的时空协调性指示SWIS是否健康更具说服力,通过精准诊断微生物结构和丰度来判定微生物协作关系,一直被学界广为接受[2].然而,微生物空间分布虽然能够反映一段时间内基质层的菌群协作状态,但当系统因扰动偏离健康轨迹时,微生物群落并不能快速做出调整,反馈在出水上即不会立刻出现水质恶化的现象[3].因此,利用DNA分子标记、基因序列分析与核酸分子杂交等方法研究SWIS菌群结构变化仅对稳态系统有意义,对多因素扰动的系统,所获得的生物学信息仅能标定相对狭窄的时段,用以表征系统健康度,必要但不充分[4].例如,通过对水力负荷波动条件下SWIS多剖面PCR-DEEG分析,发现在低水力负荷(8cm/d)状态下获取的微生物空间结构数据仅能指示不超过24h的种群特征,当水力负荷在±2cm/d范围内波动时,同一层DNA数据变化较大[5-6].即使进一步采用DNA指纹和Real-time荧光定量技术分析,也只是在稳态下才能获得ANO、qnorB等优势硝化基因群落富集于上升流区, amoA、narG等优势反硝化基因群落富集于重力流区的结论.而当扰动发生时(即使不剧烈),同样发现同一流区基因空间分布相对紊乱(上升流区出现amoA)[7].因此,以往研究表明,利用微生物结构-水质的响应关系表征SWIS健康度在理论上存在缺陷.
高通量测序具有高准确性、高通量、高灵敏度、低运行成本等突出优势,可以同时完成传统基因组学研究以及功能基因组学研究.高通量测序技术基于转录组分析,可以从大量转录组信息中发掘差异基因,快速判断核心调控网络和关键候选基因,促进转录因子功能的进一步分析.但由于基因的补偿作用,某个基因的缺失会使得其他基因的存在而得到补偿,最后的反应净结果为零,基因组的变化不一定能够得到表达.而代谢组学可对生物体内所有代谢产物进行定性和定量分析,小分子的产生和代谢能够准确反映生物体系的状态.
微生物代谢组学是一种理解细胞功能至关重要的功能基因组学的技术,是对细胞生理状态的直接反映,也可较为详细地反映土壤微生物多样性[8].微生物代谢组学能够逆向推理微生物的代谢途径及代谢方式,在微生物种类极其繁多的现实条件下,利用该方法对微生物代谢途径进行研究,具有精准性和靶向性优势.本研究利用微生物代谢组学的宏观性,通过超高效液相色谱-质谱(UPLC-MS)分析技术对代谢产物进行数据采集,经预处理、统计分析、生物学解释获得大量数据,表达为特定的代谢指纹图谱,筛选潜在生物标志物[9];探究代谢产物蕴含的生物学信息,研究其代谢通路,分析代谢产物与环境因子的相关性.
1 材料与方法
1.1 试验装置
试验采用如图1所示的模拟装置1#、2#、3#进行研究.装置主体尺寸为180cm×30cm(高×直径).所填充的基质从下往上依次为5cm 厚的砾石、3cm厚的细砂、以及140cm厚的混合基质.混合基质由沙、炉渣和农田土按体积比10%:25%:65%混合而成,孔隙度0.55,渗透系数范围为4.167×10-5~1.389× 10-3cm/s.由蠕动泵泵送进水,并通过“十字”型穿孔布水管(表层土壤下65cm处)散水.每套设置沿纵向剖面设6组土壤取样口(从上至下依次编号为H1、H2、H3、H4、H5、H6),平时密封,只有采样时短暂开启.各取样口距离装置底部的距离及取样口编号如表1所示.
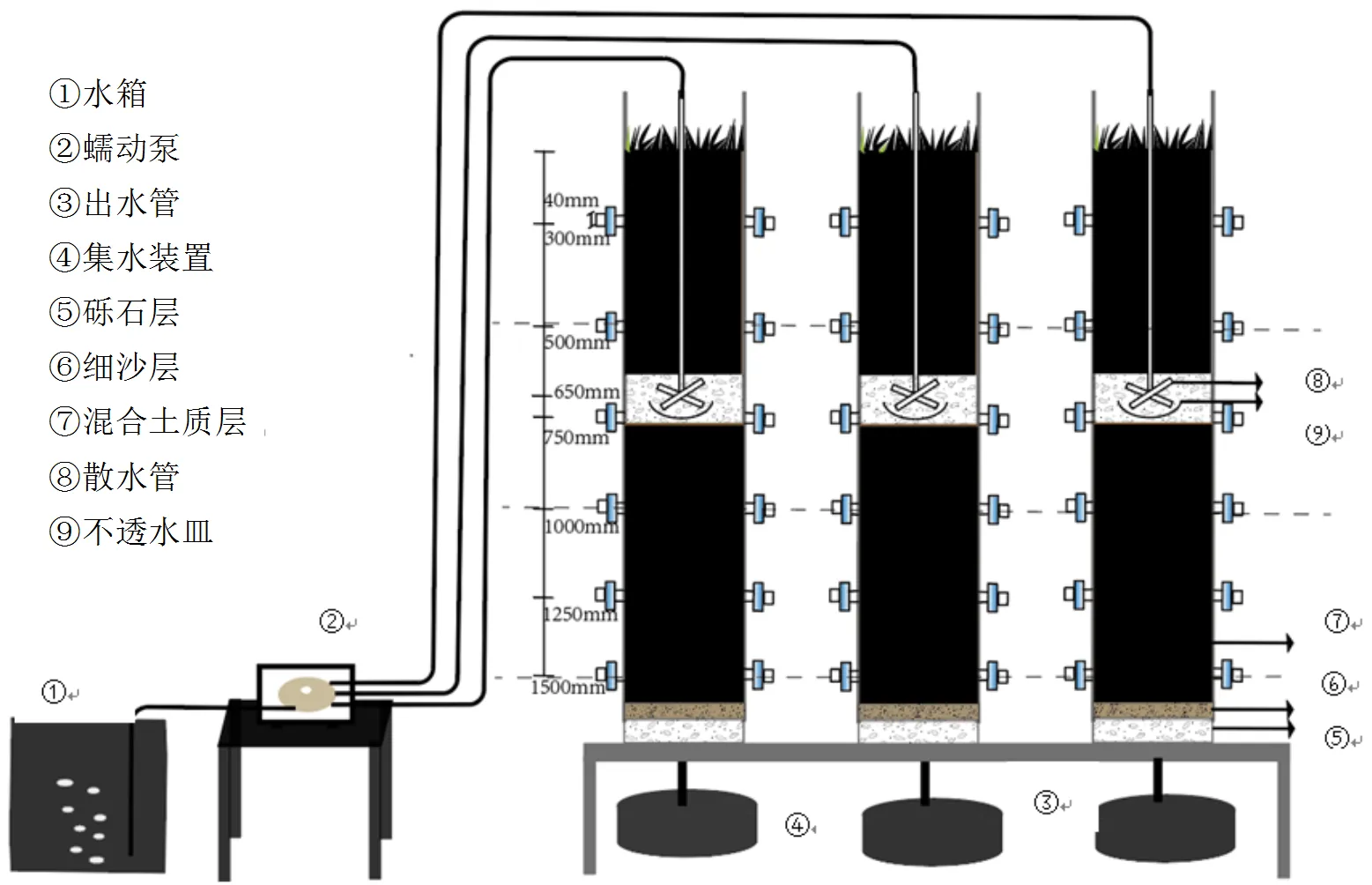
图1 SWIS模拟装置
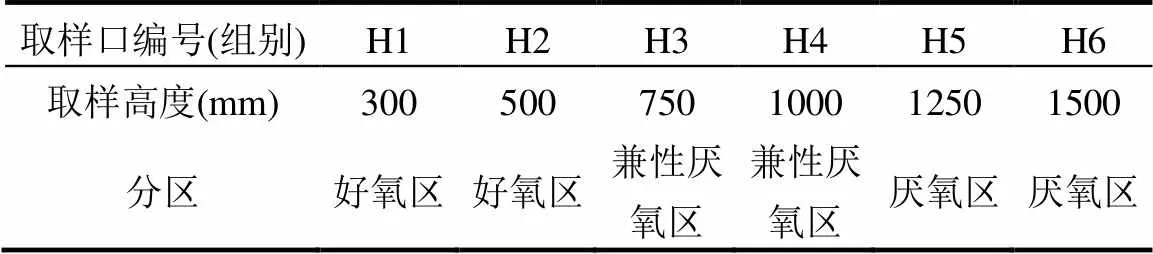
表1 地下渗滤系统取样口编号、高度及分区
1.2 试验过程
以葡萄糖(C6H12O6)、氯化铵(NH4CL)、硝酸钾(KNO3)、亚硝酸钠(NaNO2)、磷酸二氢钾(KH2PO4)按照所需比例配制成生活污水,在水力负荷为0.14m3/(m2·d)的条件下,控制有机污染负荷为250mg/L、400mg/L、500mg/L,干湿交替运行,控制布水期(12h)和落干期(12h)时间.根据前期研究结论,SWIS氧化还原电位(ORP)大于300mV时可认为是好氧环境,小于300mV时则为缺氧或厌氧环境,小于-200mV为完全厌氧环境[10].经连续监测,H1(300mm)、H2(500mm)处氧化还原电位ORP均值分别为300mV,450mV;H3(750mm)、H4(1000mm)处分别为150mV,50mV;H5(1250mm)、H6(1500mm)处ORP值均低于-380mV.因此,随着进水-落干的交替运行,H1、H2高度以上区域为好氧环境,H3、H4区域环境则缺氧-厌氧变化,H5、H6区域始终处于厌氧环境[11].因此为了减少样品分析工作量,待系统稳定运行后,分别从H2、H4、H6高度的土壤取样口取5g土壤于5mL离心管中,每一高度取样口所取土壤均制成2个平行样,样品处理完后将所有样本立即置于-80℃冰箱中保存.同时,采集出水水样并测定常规的理化指标.
1.3 样品预处理
将采集到的土壤样品,迅速用液氮灭活,用纯甲醇提取剂进行溶液提取,提取液体积10mL,提取3次,合并提取液,真空旋干,用1mL甲醇复溶,13000r/min离心机离心10min,吸取上清,装入1.5mL进样小瓶,放置于4℃冰箱,待上机检测.
1.4 试验条件
土壤样品测定采用美国安捷伦公司生产1260Infinity-6420液相色谱-三重四级杆质谱联用仪.测定条件为:流动相由A相(甲酸:水=0.1:100)和B相(乙腈)组成,流速0.2mL/min,柱温30℃.梯度洗脱条件为:0min,15%B;10min,65%B;15min,80%B; 30min, 95%B;38min,99%B;55min,99%B;56min,15%B;69min, 15%B;质谱采用ESI离子源,在正离子模式下采集数据,离子采集范围是50~1800m/z.
1.5 数据处理
采用UPLC-MS全扫描方式分析微生物代谢产物,将得到的数据文件以简单模式进行AMDIS分析,进而对代谢产物的总离子流图进行色谱峰识别与峰匹配,采用主成分分析(PCA)和偏最小二乘法判别分析(PLS-DA)模式识别对操作条件进行差异判别分析.
2 结果与讨论
2.1 SWIS代谢组学检测方法

表2 检测到的部分化合物
2.1.2 数据采集 由于土壤微生物代谢产物成分较复杂,大多数化合物为碱性化合物,TIC在ESI+模式下的响应比较好,大部分代谢产物能够在此模式下出峰[14].且正离子模式下,检测到的峰谱较集中,检测时间较短.对于SWIS代谢产物的检测,适合在质谱正离子模式(ESI+)下分析.图2是SWIS土壤样品经UPLC-MS分析得到的典型代谢指纹图谱.
图2 正离子模式识别色谱
2.1.3 数据预处理 应用一系列的化学计量学方法将色谱质谱中的波谱信号为数据信息[15],包括:去除奇异点,滤除噪音,峰识别,重叠峰分析,峰对齐,峰匹配,标准化和归一化等.
2.1.4 统计分析 经过预处理后的数据进行多变量统计分析,多元统计量的主要手段是模式识别技术.模式识别可分为非监督有监督方法[16],非监督方法中主成分分析(PCA)和监督方法中偏最小二乘法判别分析(PLS-DA)是代谢组学研究中最常用的模式识别方法,这两种方法通常以得分图获得对样品分类的信息,载荷图对分类有贡献变量及其贡献大小,从而用于筛选生物标志物.
2.2 不同有机负荷下生物标志物的筛选
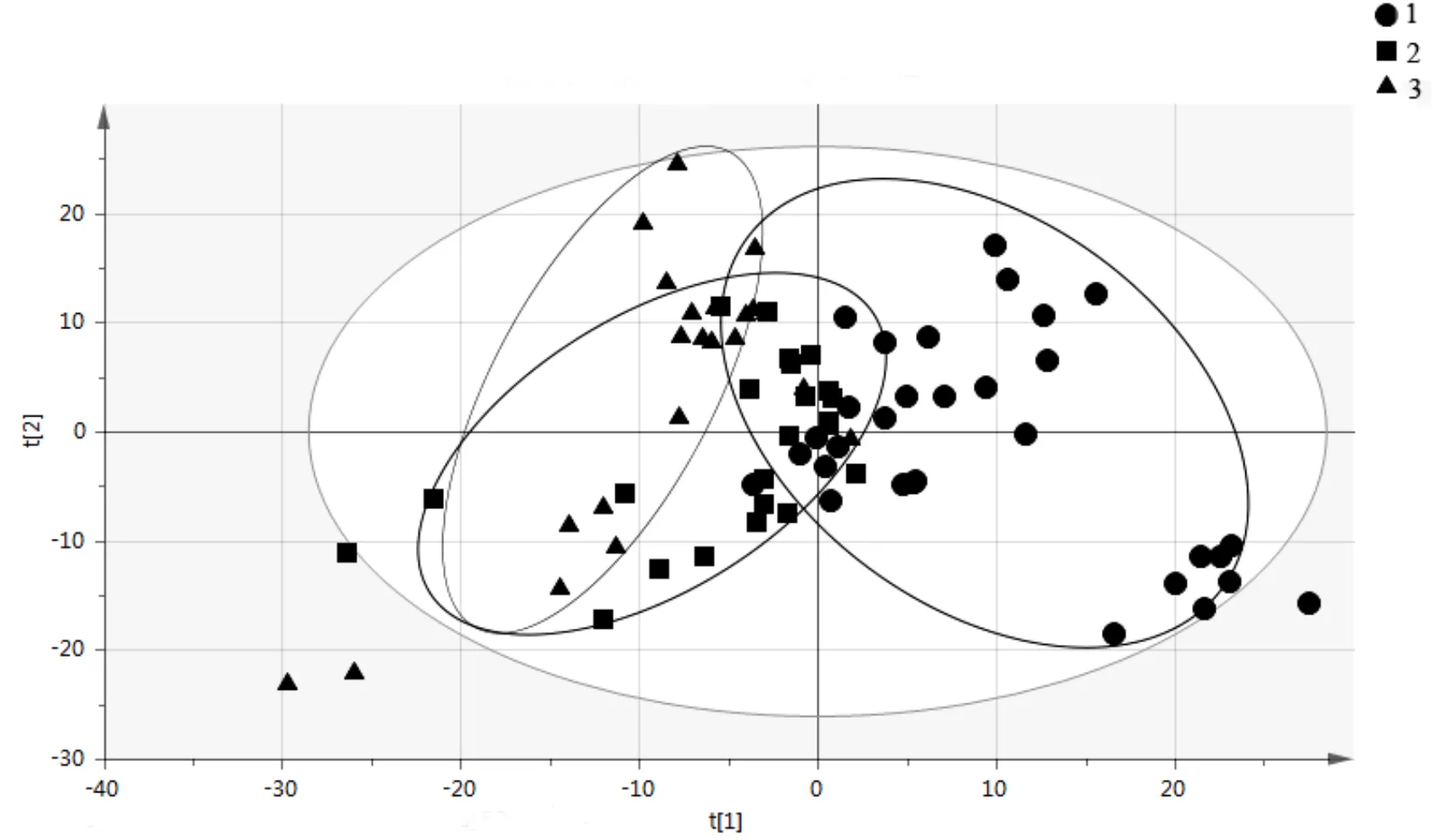
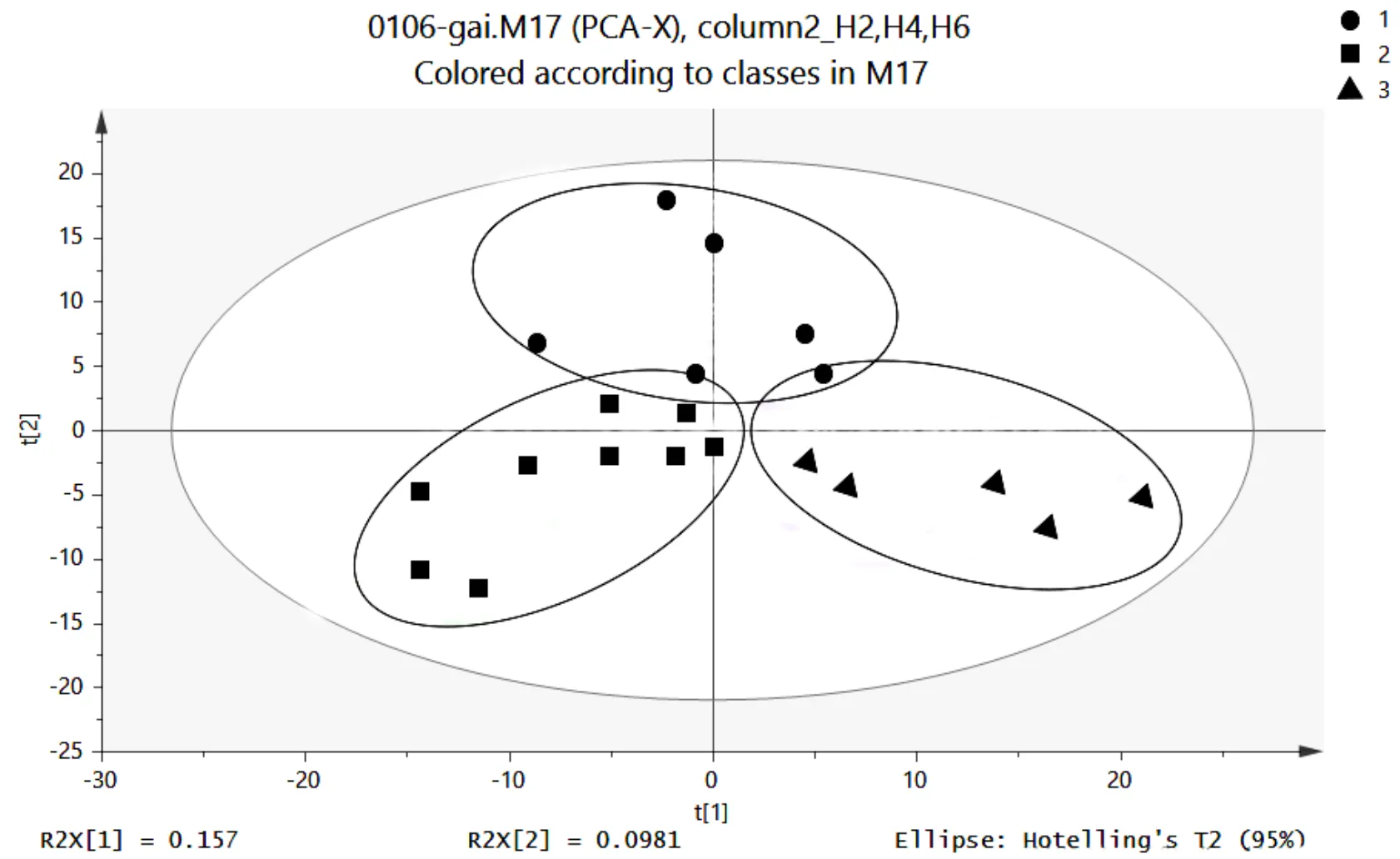
图3 不同有机负荷波动下PCA
横纵轴代表两个能最大程度反映方差的两个特征值,图中每个点代表一个样本,形状则代表不同柱子分组,即圆圈代表柱1样本、方框代表柱2样本、三角形代表柱3样本(下同);越相似的样本在图中的距离越近,反之距离越远的样本差异越大;圈外的点代表离散点,误差较大
根据模式识别模型的VIP值筛选潜在标志物.通过分析不同样品的组成可以反映样品间的差异和距离,PCA运用方差分解,将多组数据的差异反映在二维坐标图上,坐标轴为能够最大程度反映方差的两个特征值[17].如图3所示,图中每一个颜色代表一个模拟柱,即样本点分组,相同分组的点以椭圆标出,可以看出样本组与组之间存在一定的距离,这说明样本组之间的距离越大,对应的代谢产物差异越大;而图中样本点接近或重合的部分,则表示微生物代谢产物差异不明显.这说明在无监督模式识别情况下,有机负荷为250mg/L、400mg/L、500mg/L的3个模拟柱之间微生物代谢产物存在差异.
从PCA模型看出,3组地下渗滤系统模拟柱微生物代谢产物之间虽存在差异,但不明显,模型只解释了45%的原始数据,需要采用更加适合的模型来表征代谢指纹.PLS-DA是以偏最小二乘法回归模型为基础,作为一种有监督的模式识别的方法,根据给定的样本分组信息,对群落结构进行判别分析[18].从PCA图中可以看到样本的原始状态,而PLS-DA看到的是样本的分组效果.通过PLS-DA模型图找到造成组间差异的物质.为此,采用PLS-DA方法来区分3个模拟柱微生物代谢产物差异,筛选潜在标志物.PLS-DA在PCA分析的基础上,使得组分之间的区分效果更明显,具有导向性.
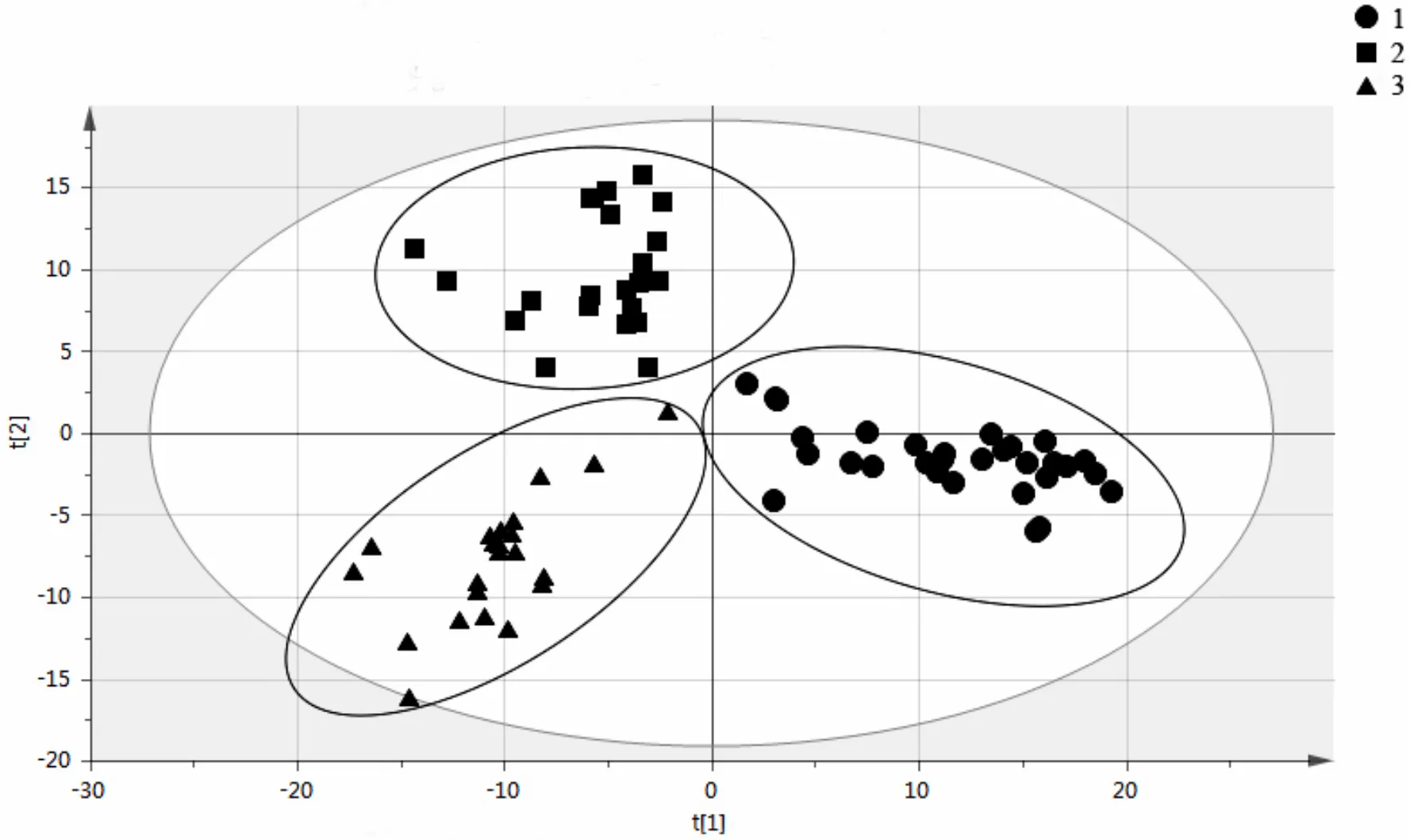
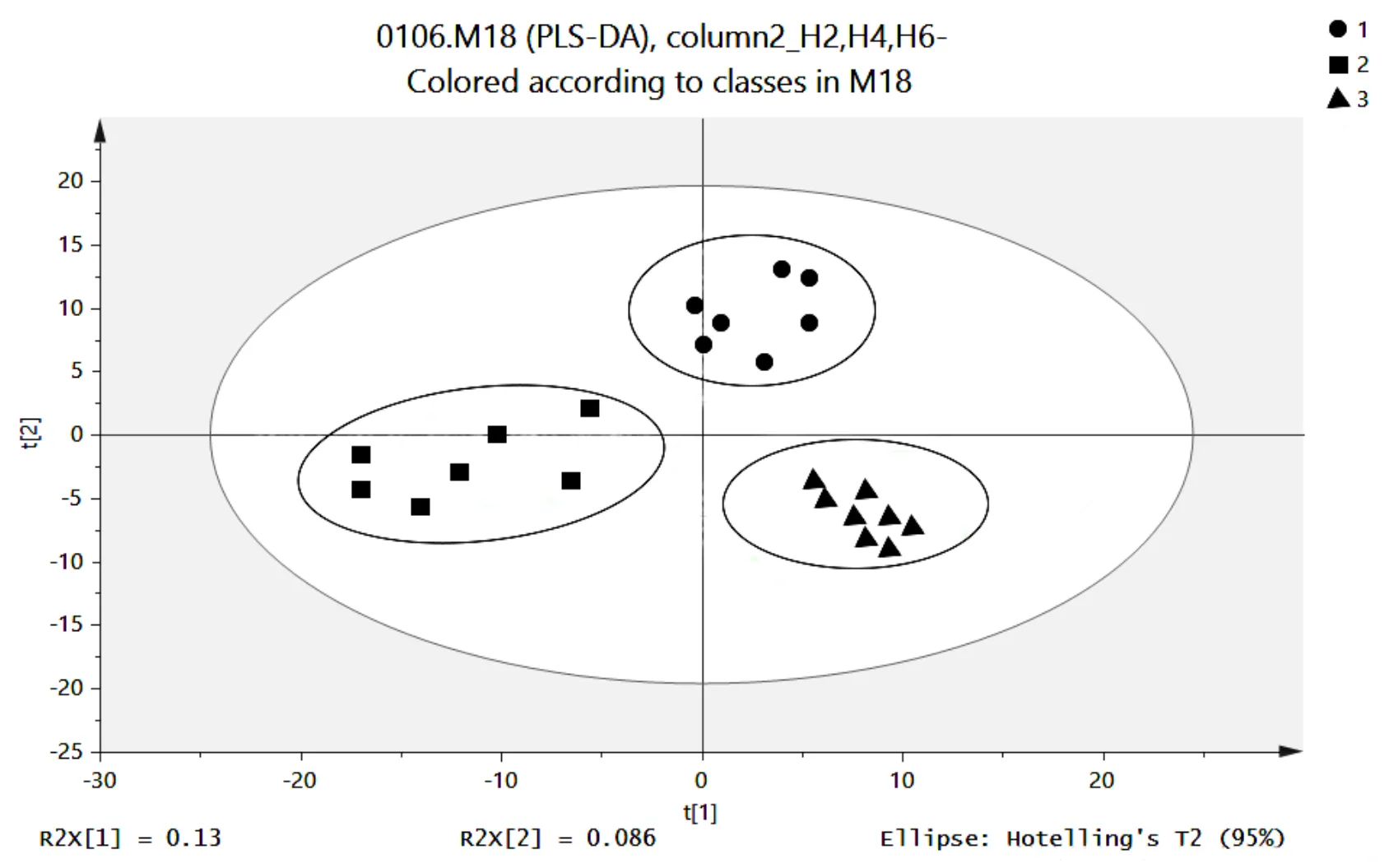
图4 不同有机负荷波动下PLS-DA
形状相同的点属于同一分组,如果属于同一分组的样本之间的距离越近,同时不同分组的样本之间的距离越远,说明分类模型效果越好
如图4所示,属于同一椭圆内的样本之间某种程度上聚合,不同分组的样本之间的距离区分比较明显,这充分说明不同有机负荷的微生物代谢产物之间存在差异.实验模拟柱中的装填基质土和取样时间点相同,而3个模拟柱有机负荷进水浓度逐渐增加,这说明进水浓度与模拟柱之间的微生物代谢产物差异存在相关性.
在污水地下渗滤系统中,营养物质是影响微生物生长分布的主要因素之一,有机物的降解主要依靠土壤等基质过滤吸附和微生物作用,污水与基质不断接触的过程中,污水中大部分有机物被土壤胶体物质吸附,继而在微生物氧化作用下分解去除[19].不同有机负荷的进水条件为微生物的生长提供必要的营养物质,有机负荷不同,相对应的微生物结构及代谢途径存在差异,进而微生物代谢产物之间存在显著性差异.
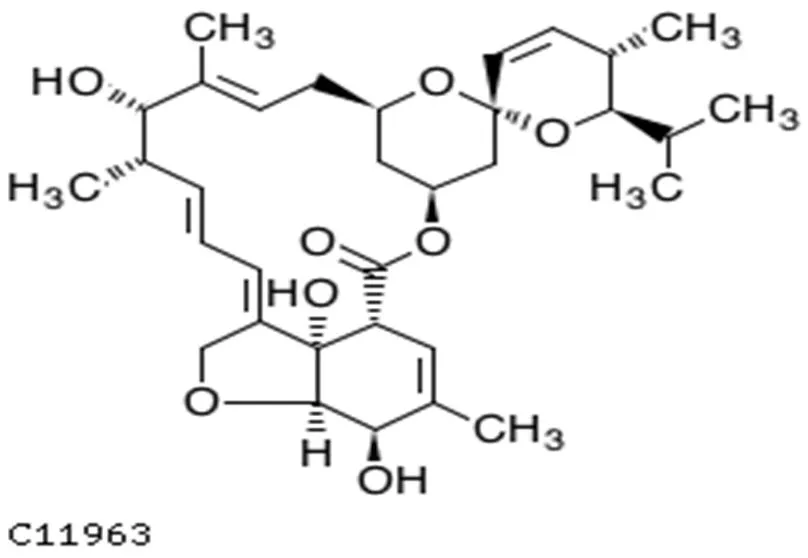
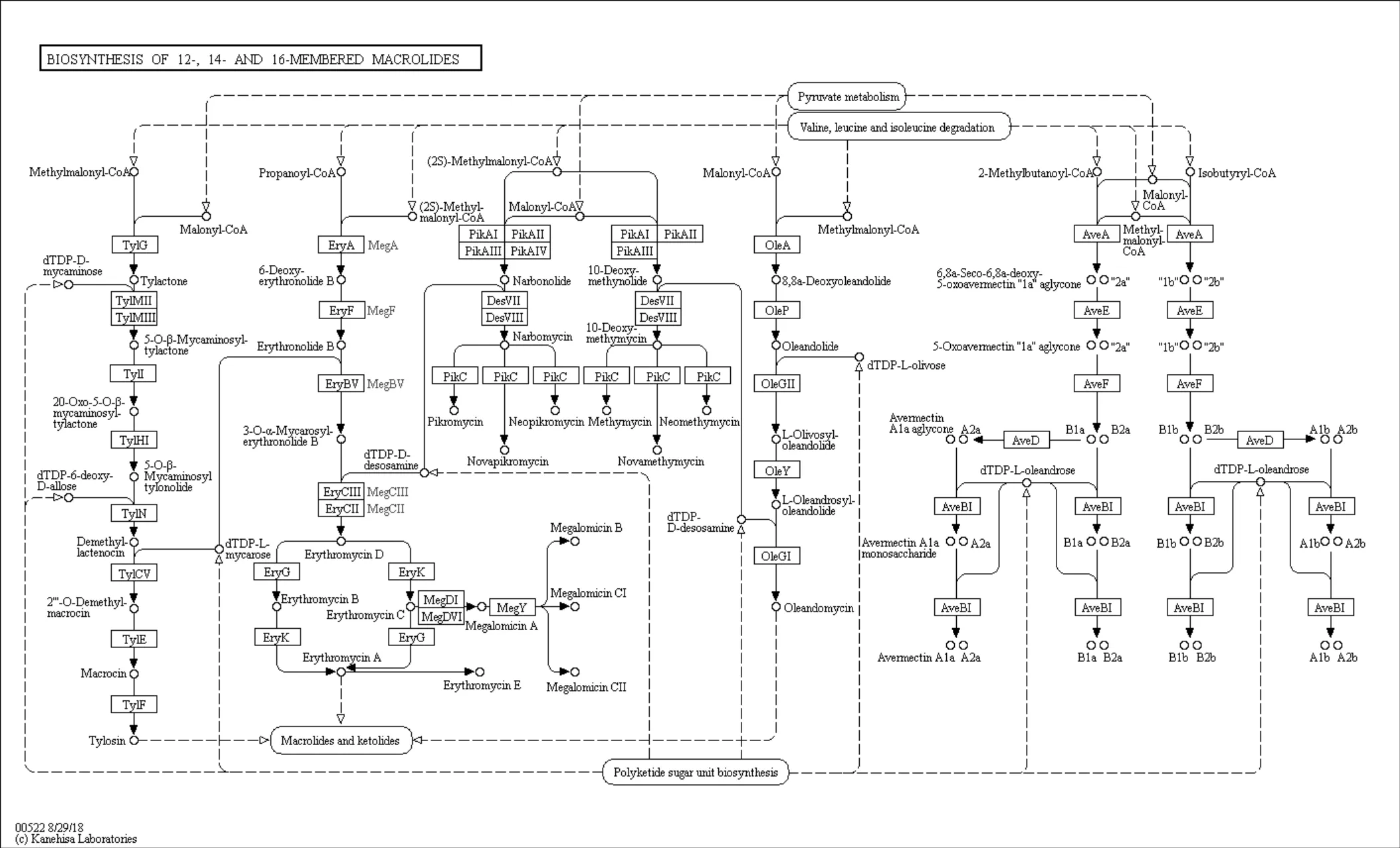
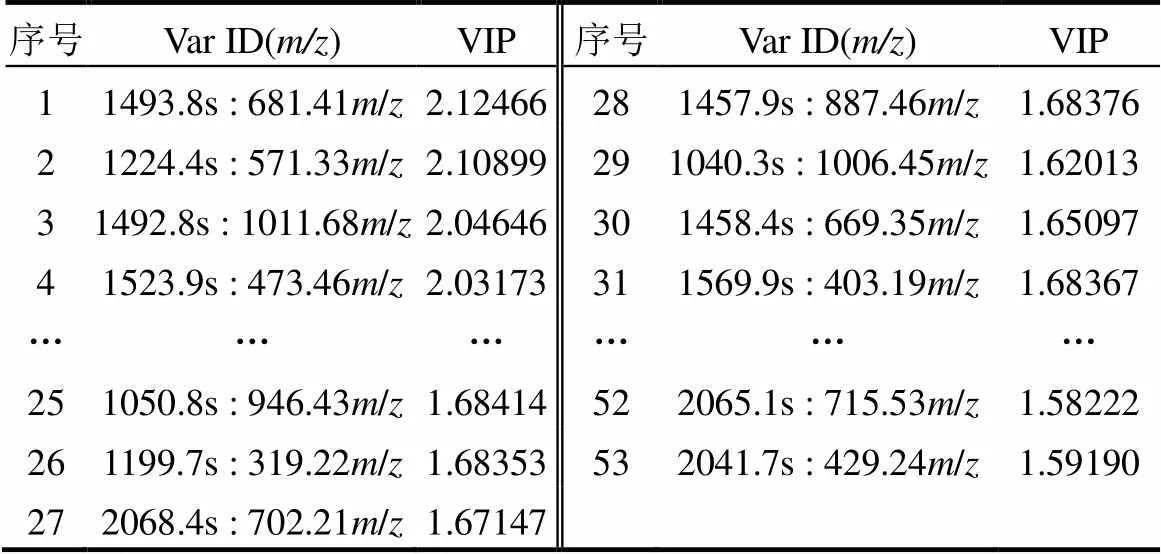
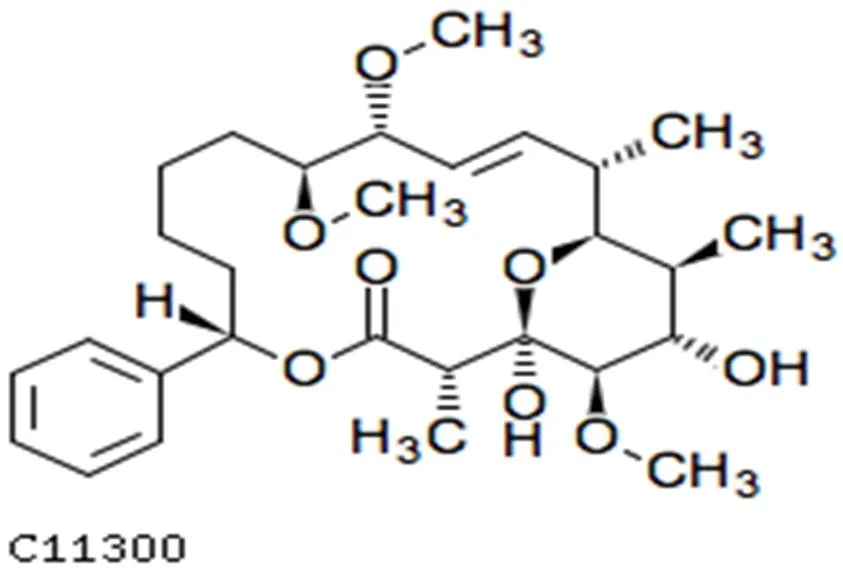
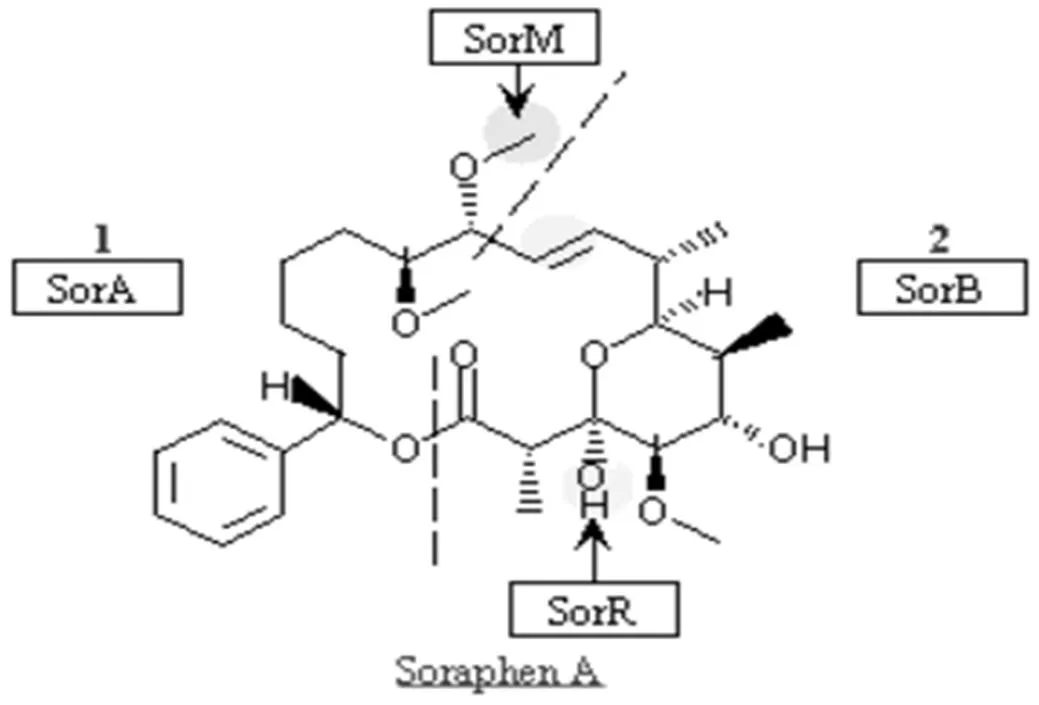
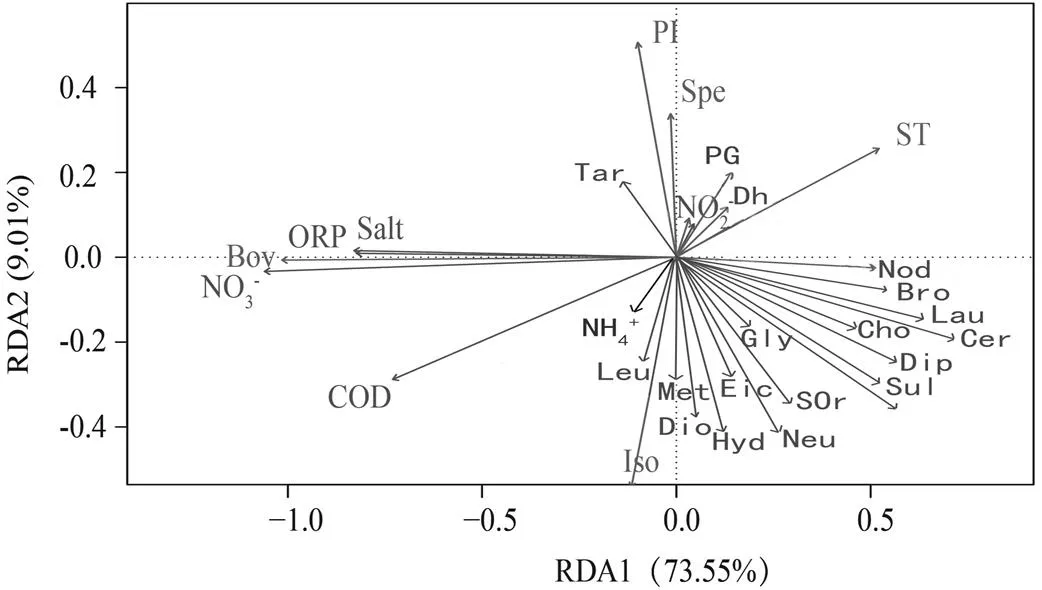
2.2.1 潜在标志物识别 经过PLS-DA分析得到所有磷脂分子的变量投影重要值(VIP值)排列图,代谢差异化合物筛选的一般原则是在两组(分批过程和连续过程组)PLS模型中的VIP值大于1[20].通过模拟柱不同有机负荷的PLD分析,找到VIP 值大于1的有500多个化合物,VIP大于1.5的化合物有230个.在此挑选具有代表性的差异物,选取1.5作为筛选的临界值.VIP值从0~2之间波动,VIP值大于2的潜在标志物约为8.4%,1.5 表3 不同有机负荷条件下的潜在标志物 注:1)Var ID代表一个物质;2)以序列号为例,1253.8s代表处理时间,292.23代表化合物的分子量, 2.93324代表VIP值. 表4 有机负荷波动下SWIS生物代谢产物定性分析 2.2.2 代谢途径分析 基于上述理论,将推定的鉴定化合物提交KEGG途径数据库.代谢通路图可以直观的展示产物代谢过程中的相互作用,可以表现受体结合的活动,蛋白质复合物,磷酸化反应,酶的活化等,将通路与生物学注释连接在一起,使得产物的代谢途径更加清晰. 图5为Gingerglycolipid C 结构示意图.糖脂是糖通过半缩醛羟基以糖苷键与脂类连接的化合物.由于脂类部分不同,糖脂可分为鞘糖脂、甘油糖脂和类固醇衍生糖脂.根据化合物结构,Gingerglycolipid C属于鞘糖脂,是细胞膜脂的主要成分.鞘糖脂分子中神经酰胺羟基被甲基取代.脂类代谢产生长链脂肪酸,长链脂肪酸首先被转化成脂酰辅酶CoA,经辅酶AveA、AveE、AveF催化,在脂肪酸氧化酶作用下脱氢,脱下的氢和O2结合成H2O2,进一步反应释出较短链脂肪酸,生成“B1b”.图6为Gingerglycolipid C代谢通路图.通过Gingerglycolipid C代谢通路图,可以逆推Gingerglycolipid C是脂类物质代谢产物,而分解代谢的初步阶段是将蛋白质、多糖以及脂类等大分子物质降为氨基酸、单糖及脂肪酸等小分子物质,这充分说明Gingerglycolipid C是微生物的代谢产物.通过已知的代谢通路逆推找出化合物和调节酶,完成微生物代谢机理研究. 图5 Gingerglycolipid C结构示意 图6 Gingerglycolipid C代谢通路 在水力负荷为0.14m3/(m2·d),有机污染负荷为400mg/L的条件下,采用PCA分析法对模拟柱高度H2、H4、H6微生物代谢产物进行分析,土壤样品微生物代谢产物的PCA图如图7所示.可以看出,模拟柱相同基质层高度的样本点距离相近,高度聚合,不同基质层高度样本代谢产物信息分离比较清晰明显.这说明模拟柱高度层之间代谢产物存在差异,这与高度层之间的耗氧量存在相关性[21].在SWIS中溶解氧含量是影响微生物生长分布的主要因素之一,土柱H2高度的ORP值为450mV,处于好氧区,区域内以好氧微生物为主;相对而言,土柱中层H4的OPR值为50mV,处于兼性厌氧区;下层H6的ORP值在-380mV波动,氧气含量逐渐减少,以厌氧微生物为主;土柱中氧气含量划分为H2好氧区~ H4兼性厌氧区~H6厌氧区,硝化反应主要发生在渗滤土层的上部好氧区,说明好氧条件刺激了好氧微生物分泌初级代谢产物;反硝化则发生在土层的下部厌氧区,刺激了厌氧微生物分泌了大量特异性代谢产物[22].氧气含量沿纵向高度发生变化,相对应的代谢产物也发生变化,存在显著差异,与结果反映的信息一致. 图7 不同高度微生物代谢产物PCA 横纵轴代表两个能最大程度反映方差的两个特征值,图中每个点代表一个样本,形状则代表不同高度分组,即方框代表高度H2样本、三角代表高度H4样本、圆圈代表高度H6样本(下同).PCA图中的大椭圆形区域称作95%置信区间,处于大椭圆圈内的样本点,具有可信性,处于圈外的样本点称作离散点.越相似的样本在图中的距离越近,反之距离越远的样本差异越大 图8是有机负荷为400mg/L,不同高度层土壤样品微生物代谢产物的PLS-DA图.PLS-DA方法是发现、提取、统计微生物代谢产物差异的有力途径之一[23].从图中看出,模拟柱相同高度样本点聚合,不同高度组分间区分效果明显,说明同一高度层代谢产物差异较小,不同高度层代谢产物差异较大.表明PLS-DA稳定且具有很好的解释能力.因此,采用PLS-DA分析对本研究进行结果分析具有可靠性、导向性.适合采用PLS-DA分析进行差异代谢物的筛选. 图8 不同高度 PLS-DA 每一个点代表一个样本,每个形状代表一个高度;如果属于同一分组的样本之间的距离越近,同时不同分组的样本之间的距离越远,说明分类模型效果越好 2.3.1 潜在标志物识别 潜在生物标志物的识别是代谢组学研究的核心内容和目的之一.本研究以PCA分析为基础,通过PLS-DA图对应VIP值(VIP>1的变量表示它对模型分组的贡献高于平均水平)找到潜在标志物.后期可以通过ANOVA分析(方差分析,t-test,<0.05)识别出不同组别的土壤微生物代谢产物含量有显著差异的代谢产物,进一步筛选潜在生物标志物[24].SWIS有机负荷为400mg/L,不同高度层土壤样品微生物代谢产物经PLS-DA分析得到VIP>1.5的代谢产物部分数据,如表5所示.模拟柱高度潜在标准物有53个,VIP>2的有5个化合物.VIP值越大,对模型的分类贡献越大,可作为筛选差异代谢物的指标之一.采用METLIN代谢组学数据库对潜在标志物进行分子式推断、定性,得到部分的化合物如表6所示. 表5 不同高度潜在标志物 表6 不同高度变化下SWIS微生物代谢产物定性分析 2.3.2 代谢途径分析 代谢途径作为一种平衡生物合成进程的机能,它主要负责揭示物质的生长过程.研究代谢产物在代谢途径中的作用已经成为目前的一大热点.为利于微生物代谢途径的系统性分析,本研究采用UPLC-MS的方法在生物系统中对特异性代谢物进行鉴定和精确定量.Soraphen A结构图如图9所示.经鉴定结果分析,Soraphens是从一种黏细菌Sorangium cellulosum的次级代谢产物中分离得到的一类聚酮类化合物.脂肪酸经ß-氧化会产生大量乙酰辅酶,部分辅酶被转化成酮体.葡萄糖供应充足时,糖分解代谢旺盛,供能充分,脂肪酸氧化分解减少,酮体生成被抑制;葡萄糖供应不足时,脂肪酸氧化分解增加,生成辅酶增加,酮体生成增多. Soraphen A代谢路径图如图10所示,在辅酶SorM和SorB的催化作用下,-CH3被氧化,经脱氢后生成酮体.经过代谢的转录和翻译,机体产生的代谢物可以成为识别代谢途径异常的关键指标.通过显著性代谢产物分析,探索何种代谢通路在该环境条件下占主导作用,实现SWIS微生物代谢产物对操作参数等响应的定性跟踪,从而通过代谢物的变化来改变环境干预条件,保持地下渗滤系统的稳定、高效运行. 图9 Soraphen A 结构示意 图10 Soraphen A代谢通路 RDA是基于对应分析发展而来的一种排序方法,将对应分析与多元回归分析相结合,每一步计算均与环境因子进行回归,又称多元直接梯度分析. RDA分析是一种排序分析,即在一个可视化的低维空间(二维)重新排列样本,使得样本之间的距离最大程度地反映出平面散点图内样本之间的关系,直观的看出样本分布和环境因子间的关系[25]. 图11 SWIS差异性代谢产物与环境因子的RDA分析 图中蓝色箭头代表不同的环境因子,红色箭头代表不同的代谢产物.环境因子之间的夹角为锐角时,表示两个环境因子之间正向关,钝角为负相关.环境因子的射线越长,说明该影响因子的影响程度越大.图中代谢产物名字均采用英文缩写表示 从代谢产物与环境因子之间的夹角可以看出(图11),环境因子NH4+、NO2-与大部分代谢产物呈显著正相关关系,NO3-、ORP、COD、盐度与大部分代谢产物呈负相关关系.从环境因子之间的夹角可以看出,NO3-、ORP、COD、盐度两两之间呈正相关;NO2-与NH4+、COD都呈负相关.环境因子射线的长度表明,NO3-和COD对代谢产物影响较大.同时RDA排序图反映出不同微生物代谢产物对环境因子的适应性不同,即代谢产物在RDA排序图中的位置越接近,表示其对环境适应性越相似.RDA分析结果表明,在SWIS中,环境因子对微生物代谢产物存在影响,随着有机负荷波动和不同高度剖面的变化,代谢产物受到ORP与NO3-的影响较大,呈负相关. 3.1 本研究通过PLS-DA模型分析了SWIS微生物代谢组学在不同有机负荷、不同剖面的变化,建立了一套基于UPLC- MS技术的SWIS微生物代谢组学研究方法.通过筛选生物标志物,分析了差异性产物的代谢通路,利用RDA分析探索了该代谢产物与主要环境因子(COD,NH4+,ORP等)的相关性. 3.2 在有机负荷波动条件下(COD=250-500mg/L),微生物代谢产物因进水营养物质的不同存在差异性.其中,在中等负荷下(COD=400mg/L),微生物代谢产物与基质层高度显著相关, 共筛选到53个潜在生物标志物, 定性得到12种化合物,包括:葡萄糖醛酸苷(19-Norandrosterone glucuronide)、戊烯二酸(Glutaconic acid)、杯苋甾酮(cyasterone)等. 同时, ORP与NO3-对微生物代谢产物存在显著负相关关系. [1] Yang Z Z, Zhang S, Li C J, et al. Study on mechanism of nitrogen and phosphorus removal in matrix mixing subsurface wastewater infiltration system [J]. Water Treatment Technology, 2016,42(1):56- 61. [2] Pan J. Study on microbial diversity and enzyme activity of subsurface wastewater infiltration system [D]. Shenyang: Northeastern University, 2009. [3] Li Y H, Li H B, Yang C, et al. Characteristics of decontamination and release of N2O in subsurface wastewater infiltration system under nitrogen load fluctuation [J]. Journal of Safety and Environment, 2018, 18:270-273. [4] Badri D V, Chaparro J M, Zhang R F, et al. Application of natural blends of phytochemicals derived from the root exudates of arabidopsis to the soil reveal that phenolicrelated compounds predominantly modulate the soil microbiome [J]. Journal of Biological Chemistry, 2013,288:4502-4512. [5] Wang X, Sun T H, Li H B, et al. Nitrogen removal enhanced by shunt distributing wastewater in a subsurface wastewater infiltration system [J]. Ecological Engineering, 2010,36:1433-1438. [6] Pan J, Yuan F, Huang L L, et al. Nitrogen removal in subsurface wastewater infiltration system with and without intermittent aeration [J]. Ecological Engineering, 2016,94:471-477. [7] Tan Y F. Molecular mechanism of nitrogen-transformation microecological processes in multi-media ecological treatment systems [D]. Beijing: Peking University, 2011. [8] Guo Y Z, Zhao Y P, Zhu T T, et al. A metabolomic view of how low nitrogen strength favors anammox biomass yield and nitrogen removal capability [J]. Water Research, 2018,143:387-398. [9] Justyna A, Hubert B, Jacek N, et al. Main strategies, analytical trends and challenges in LC-MS and ambient mass spectrometry-based metabolomics [J]. Trends in Analytical Chemistry, 2018,108:278-295. [10] Ma J F. ORP value variation characteristics of underground infiltration system [D]. Shenyang: Northeastern University, 2014. [11] Duan Y L, Li Y H, Li H B, et al. Influence of hydraulic load on microbial distribution and N2O release in subsurface infiltration system [J]. Chinese Journal of Environmental Engineering, 2016, 10(11):6431-6438. [12] Wang M M, Cheng H T, Xue M, et al. Cell metabolomics analysis process and technology based on combined nuclear magnetic resonance and mass spectrometry techniques [J]. International Journal of Pharmaceutical Research, 2011,38(2):130-136. [13] Paola M, Giovanna F. Modeling of the microbial inactivation by high hydrostatic pressure freezing [J]. Food Control, 2017,73:8-17. [14] Wang B B, Teng Y, Xu Y F, et al. Effect of mixed soil microbiomes on pyrene removal and the response of the soil microorganisms [J]. Science of the Total Environment, 2018,640:9-17. [15] Shen G A, Duan L X, Qi X Q. Plant Metabolomics Data Analysis and Database [J]. Life Science, 2015,8:996-999. [16] David E G, Edward E B, Peter I B, et al. Separation and mass spectrometry in microbial metabolomics [J]. Science Direct, 2008,11: 233-239. [17] Malgorzata P, Julia J, Michal J M. Sample preparation procedures utilized in microbial metabolomics: An overview [J]. Chromatography B, 2017,1043:150-157. [18] Shao T T, Miki K, Yasummune N, et al. Random sample consensus combined with partial least squares regression (RANSAC-PLS) for microbial metabolomics data mining and phenotype improvement [J]. Bioscience and Bioengineering, 2016,122:168-175. [19] Cui C Y, Ma L M, Zhao J F. Study on Microbial Characteristics of Artificial Rapid Infiltration System [J]. China Population Resources and Environment, 2008,18:539-541. [20] Wang Y Q, Zhang W J, Nie C X, et al. Evaluation of sample extracting methods of FCSM by Lactobacillus acidophilus based on a UPLC-Q- TOF-MS global metabolomics analysis [J]. Biotechnology and Industrial Microbiology, 2018,49:392-400. [21] Xiao R, Ma Y, Zhang D J, et al. Discrimination of conventional and organic rice using untargeted LC-MS-based metabolomics [J]. Cereal Science, 2018,82:73-81. [22] Li Y H, Li H B, Wang X, et al. Denitrification of domestic sewage by biological packing underground infiltration system [J]. Chinese Journal of Environmental Engineering, 2013,7(9):3369-3374. [23] Yang X H, Gu J F. Advances in research on soil microbes and their metabolites in mangrove forests [J]. International Pharmaceutical Antibiotics, 2011,32(3):97-102. [24] Zhang P J, Li Y M, Zhang Y N, et al. Application and prospect of toxicity quality markers of Chinese materia medica based on metabolomics [J]. Chinese Herbal Medicines, 2018,10:108-116. [25] Tong L L. Discussion on the application of RDA analysis in CANOCO4.5 in water ecological assessment [J]. Environmental Science, 2018,1:45-47. Microbial metabolomics analysis of subsurface wastewater infiltration system. YANG Lei, LI Ying-hua*, LI Hai-bo, SU Fei (School of Resources and Civil Engineering, Northeastern University, Shenyang 110000, China)., 2019,39(11):4812~4822 Based on metabolomics method, ultra-performance liquid chromatography-mass spectrometry (UPLC-MS) was used to screen potential biomarkers of subsurface infiltration systems under different organic loads. The samples were typed by partial least squares (PLS-DA) and principal component analysis (PCA) pattern recognition methods, and potential biomarkers were screened according to the model's variable importance factor (VIP). Furthermore, biological information involved in metabolites and metabolic pathways were revealed. Finally, correlation between metabolites and environmental factors was explored by RDA analysis. The results of PLS-DA model showed that when the influent organic load gradually increased from 250mg/L to 400mg/Land 500mg/L, there was a significant difference between the metabolites in the same soil profile. A total of 230differential metabolites with a VIP value greater than 1.5 were screened out, which were mainly composed of organic acids, such as lactic acid, tartronic acid, dioleoylphosphatidic acid. The microbial metabolites had a significant correspondence with the soil profile. Under the condition of hydraulic load 0.14m3/(m2·d) and COD 400mg/L, 53metabolites with a VIP value greater than 1.5 were screened out from H2 (500mm), H4 (1000mm) and H6 (1500mm), respectively, which was mainly composed of acid and ketone substances, such as 6-Hydroxyondansetron sulfate, 19-Norandrosterone glucuronide and Methylcarbamyl PAF C-8. The results of RDA suggested that with the variation of pollution load and substrate profile, metabolites were negatively influenced by oxidation reduction potential (ORP) and nitrate (NO3-). subsurface wastewater infiltration system;metabolomics;microorganism;potential biomarke X523 A 1000-6923(2019)11-4812-11 杨 蕾(1995-),女,辽宁庄河人,东北大学硕士研究生,主要研究方向为污水生态处理技术.发表论文3篇. 2019-04-29 国家自然科学基金资助项目(41571455,51578115) * 责任作者,教授, liyinghua@mail.neu.edu.cn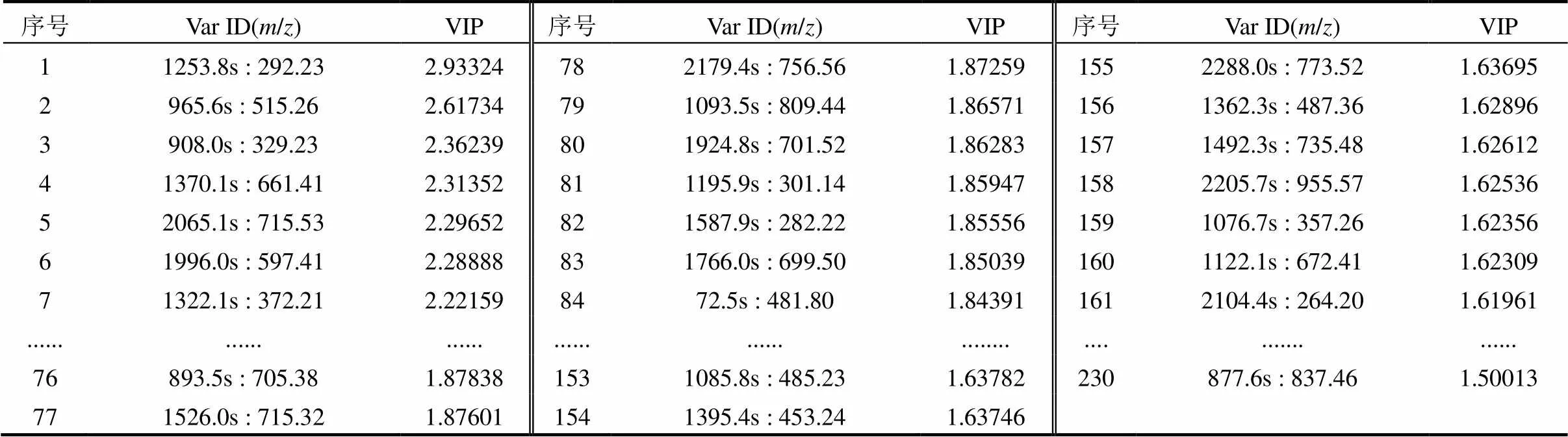

2.3 不同高度生物标志物的筛选

2.4 SWIS差异性代谢产物与环境因子的冗余分析(RDA)
3 结论
