19个香菇菌株基于ISSR、SRAP和TRAP分子标记的遗传多样性分析
2019-11-13汪昊牛玉蓉荣成博
汪昊 牛玉蓉 荣成博

摘要:利用简单重复序列间扩增(inter-simple sequence repeat,简称ISSR)、相关序列扩增多态性(sequence-related amplified polymorphism,简称SRAP)和目标区域扩增多态性(target region amplified polymorphism,简称TRAP)分子标记对19个香菇栽培菌株进行遗传多样性分析。结果显示,共筛选出34对(条)多态性丰富的引物,获得303条数据条带,其中多态性条带254条,多态性比例为83.83%,在遗传相似系数为0.75的水平上,将19个菌株分为6类。3种标记的多态性比例排序如下:TRAP(91.14%)>ISSR(83.15%)>SRAP(80.00%)。由结果可知,综合不同分子标记进行香菇的遗传多样性分析,可更加准确地反映菌株间的亲缘关系。
关键词:香菇;遗传多样性;ISSR;SRAP;TRAP
中图分类号: S646.1+20.32 文献标志码: A 文章编号:1002-1302(2019)17-0054-05
我国是世界上最早进行香菇人工栽培的国家,据今已有800多年的历史[1]。此外,我国也是香菇最大的生产国、消费国和出口国[2]。据中国食用菌协会统计,2016年全国香菇总产量为898万t,香菇已经是我国栽培量最大的食用菌品种。随着产业规模的快速发展,对香菇良种高效选育的要求越来越迫切,对种质资源的收集鉴定评价及进一步明确资源间的遗传关系,是新品种选育的重要前提。由于香菇表型较易受栽培基质、栽培模式、管理方法等因素影响,变异性较大,因此,从分子水平对香菇种质遗传多样性进行研究,可以更加高效准确地得出菌株间的亲缘关系。
分子标记技术自20世纪80年代发展以来,已被应用于多种作物的种质资源和育种研究中,特别是在构建分子遗传图谱、基因定位、分子辅助育种等方面取得了很大进展[3]。本研究利用基于PCR的简单重复序列间扩增(inter-simple sequence repeat,简称ISSR)、相关序列扩增多态性(sequence-related amplified polymorphism,简称SRAP)和目标区域扩增多态性(target region amplified polymorphism,简称TRAP)3种分子标记,对我国规模栽培的香菇品种进行遗传多样性分析,以期快速准确并多角度地明确香菇栽培菌株的亲缘关系及演化规律,从而更好地保护和利用香菇种质资源。
1 材料与方法
1.1 供试菌株
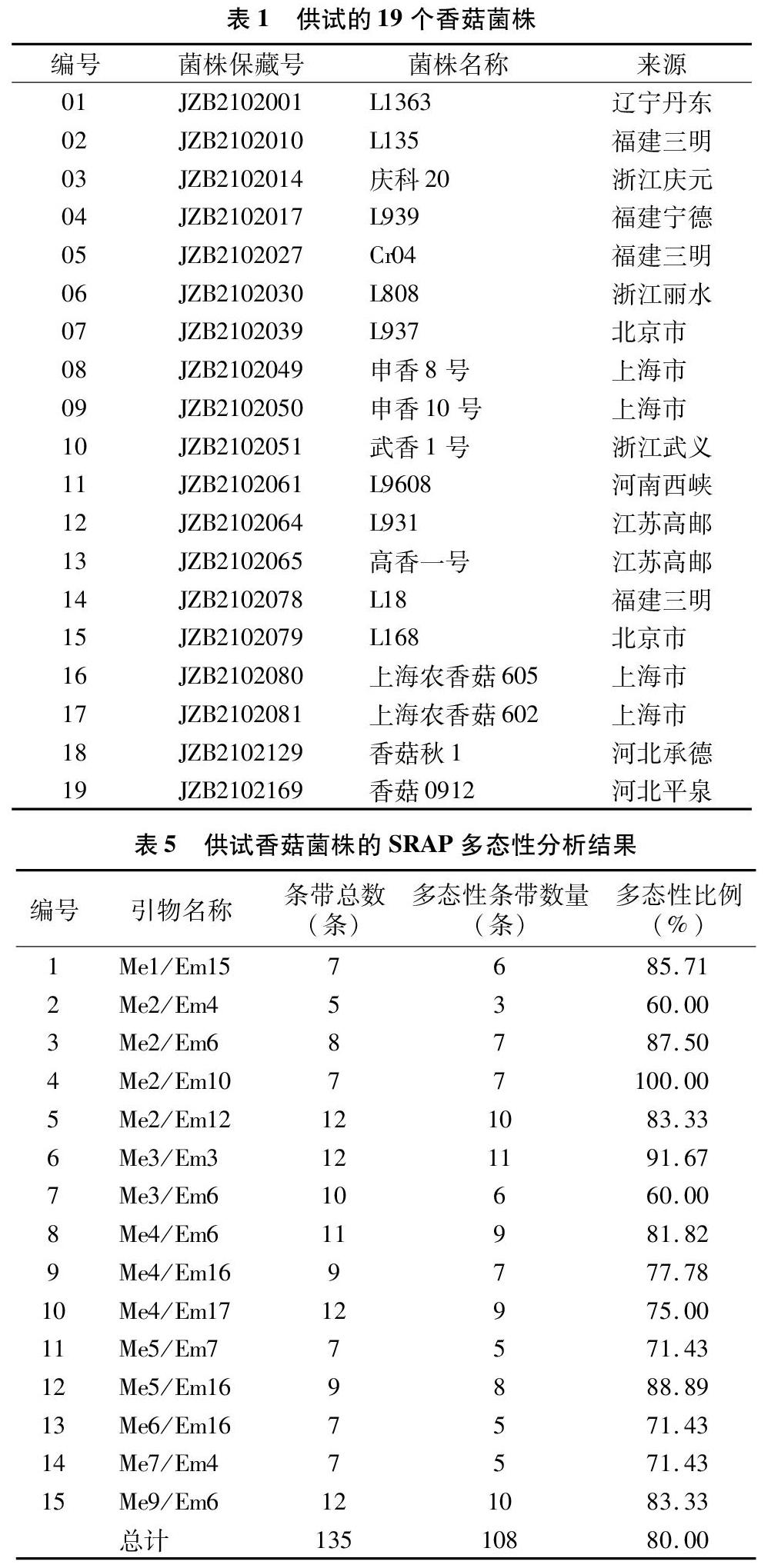
本研究开展的时间为2017年10月至2018年6月,供试的19个香菇菌株均由北京市农林科学院植物保护环境保护研究所保藏并提供,各供试菌株的名称、编号及来源见表1。
1.2 培养基
用于香菇菌丝培养的综合马铃薯葡萄糖琼脂(PDA)培养基配方(1 L):去皮马铃薯200 g熬汁,葡萄糖20 g,琼脂 20 g,KH2PO4 3 g,MgSO4 1.5 g,蛋白胨5 g,维生素B1 10 mg,加蒸馏水定容至1 000 mL,121 ℃灭菌20 min。
1.3 引物与试剂
试验所用引物均由生工生物工程(上海)股份有限公司合成;DNA marker、Taq PCR Mix,购自宝生物工程(大连)有限公司;基因组DNA提取试剂盒,购自德国QIAGEN公司。
1.4 香菇基因组的提取
用打孔器将活化的香菇各菌种定量接种在PDA培养基平皿中,于25 ℃恒温培养箱中培养10 d左右。分别刮取 100 mg 菌丝,用试剂盒提取香菇各菌株的基因组DNA,用紫外-可见分光光度计检测各DNA在260、280 nm下的吸光度,并将19种DNA的浓度调为一致。
1.5 PCR反应体系和条件
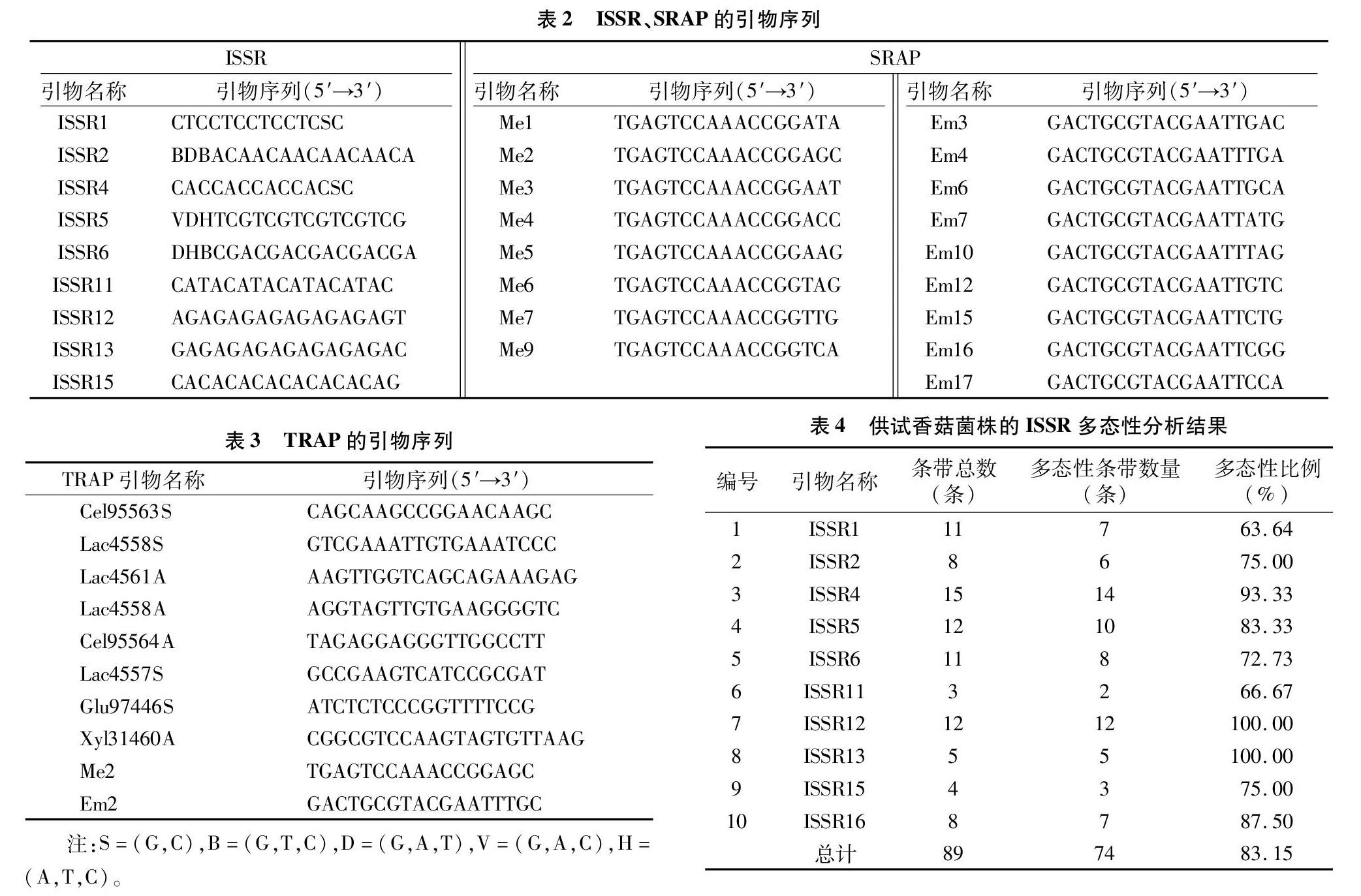
25 μL的ISSR-PCR反應体系:12.5 μL 2×Taq PCR Mix、1 μL ISSR引物(0.4 μmol/L)、1 μL DNA(30 ng)、10.5 μL 灭菌双蒸水。反应条件:95 ℃ 5 min;95 ℃ 30 s,48 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min,4 ℃保存。相关引物序列设计参考文献[7],详见表2。
25 μL的SRAP-PCR反应体系:12.5 μL 2×Taq PCR Mix、1 μL SRAP上游引物(0.4 μmol/L)、1 μL SRAP下游引物(0.4 μmol/L)、1 μL DNA(30 ng)、9.5 μL灭菌双蒸水。反应条件:95 ℃ 5 min;95 ℃ 30 s,48 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min,4 ℃保存。相关引物序列设计参考文献[7],详见表2。
25 μL的TRAP-PCR反应体系:12.5 μL 2×Taq PCR Mix、1 μL TRAP上游引物(0.4 μmol/L)、1 μL TRAP下游引物(0.4 μmol/L)、1 μL DNA(30 ng)、9.5 μL灭菌双蒸水。反应条件:95 ℃ 5 min;95 ℃ 30 s,35 ℃ 30 s,72 ℃ 1 min,10个循环;95 ℃ 30 s,45 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min,4 ℃保存。相关引物序列为笔者自己设计,详见表3。
1.6 数据统计与分析
将扩增产物进行1.5%琼脂糖凝胶电泳,用紫外成像系统观察并拍照。各泳道在同一位置上扩增出的强带或可分辨性好的弱带,视为扩增阳性,并赋值“1”,未扩增出条带,视为扩增阴性,赋值“0”。用NTSYSpc 2.10软件进行聚类分析,采用非加权组平均法(unweighted pair-group method with arithmetic means,简称UPGMA)构建系统树。
2 结果与分析
2.1 供试香菇菌株的ISSR分析结果
随机选取2个香菇菌株的DNA,用18条ISSR引物进行PCR扩增,从中筛选出10条扩增条带较多的引物(表4)。以19个供试香菇菌株的基因组DNA为模板,依次用筛选得到的10个ISSR引物进行PCR扩增,共扩增出89条DNA片段,其中多态性条带数为74条,多态性比例为83.15%,扩增片段长度多为200~5 000 bp(图1为以ISSR4为引物的扩增结果),其遗传多样性聚类分析结果见图2。
2.2 供试香菇菌株的SRAP分析结果
随机选取1个香菇菌株的DNA,用SRAP引物对(表2中2种SRAP引物的随机组合)进行PCR扩增,从中筛选出15对多样性较好、条带清晰的引物组合(表5)。以19个供试香菇菌株的基因组DNA为模板,共扩增出135条DNA片段,其中多态性条带为108条,多态性比例为80.00%,扩增片段长度在200~5 000 bp之间(图3为以Me3/Em6为引物对的扩增结果),其遗传多样性聚类分析结果见图4。
2.3 供试香菇菌株的TRAP分析结果
TRAP扩增引物包括锚定引物和随机引物,随机选取1个香菇菌株的DNA,使用TRAP引物对(8个锚定引物,2个随机引物)进行PCR扩增,从中筛选出9对多样性较好、条带清晰的引物组合(表6),以19个供试香菇菌株的基因组DNA为模板,共扩增出79条DNA片段,其中多态性条带为72条,多态性比例为91.14%(表6、图5为以Em2/Xyl31460A为引物对的扩增结果),其遗传多样性聚类分析结果见图6。
2.4 19个香菇菌株的聚类分析结果
由3种分子标记聚类分析结果可知,供试的19个香菇菌株相似性在0.59~0.99之间,菌株间具有一定的遗传差异。在遗传相似系数为0.75的水平上,ISSR将19个香菇菌株分为5大类,SRAP将19个香菇菌株分为4大类,TRAP将19个香菇菌株分为9大类。在所有供试菌株中,JZB212014和JZB2102081的遗传相似系数为0.575,亲缘关系最远;JZB212050和JZB2102051的遗传相似系数为0.97,亲缘关系最近。
3 讨论
由于历史上的引种利用等问题,我国香菇栽培菌株同种异名、同名异种的现象较为严重[4],研究者们曾经利用多种标记探究了香菇野生种质和栽培种质的遗传多样性[5-8]。由于采用单一标记往往较难获得理想的结果,本研究采用3种标记逐一筛选并综合分析构建了香菇的遗传聚类树(图7)。用10条ISSR引物、15对SRAP引物和9对TRAP引物扩增结果进行多态性分析,共得到303个标记数据,其中多态性条带254条,多态性比例为83.83%。在遗传相似系数为0.75的水平上,将19个菌株分为6类。3种标记的单独聚类分析结果与综合聚类结果略有差异,但总体趋势相同,某些菌株只能由1种分子标记区分, 有的可由2种或者3种标记方法同时区分。例如在0.99的水平上,ISSR、SRAP无法将JZB212050、JZB2102051这2个菌株区分,而利用TRAP标记得到2个菌株的遗传相似系数为0.906。由3种标记对多态性的贡献率可见,TRAP(91.14%)>ISSR(83.15%)>SRAP(80.00%),可能由于3种分子标记所检测的位点不同,所以单个标记对菌株分类是有局限性的,须充分利用不同分子标记的优点来进行综合分析。
TRAP是2003年由Hu等提出的一种新型分子标记[9],可利用已知cDNA序列或表达序列标签(EST)序列为固定引物,配合随机引进行遗传图谱构建、重要性状标记和遗传多样性分析等[3]。本研究以美国国立生物技术信息中心(NCBI)数据库中的香菇纤维素降解酶系、半纤维素降解酶系和木质素降解酶系的相关基因为靶标基因序列,利用TRAP标记分析,对19个香菇栽培菌株进行了遗传变异研究。结果表明,香菇各菌株之间显示了较高的纤維素、半纤维素和木质素降解酶基因的遗传多态性, 为从特定基因水平上研究香菇遗传亲缘关系提供了有用的信息。郭春芳等研究发现,TRAP标记比ISSR标记更适合茶树特异种质资源和重要农艺性状的筛选[10]。Miklas等研究发现,TRAP具有标记植物抗性基因的潜力[11],而此方面的研究在食用菌重要农艺性状基因标记方面还没有相关报道。今后,随着大规模测序工作的进展,TRAP标记技术将在食用菌品种改良中获得更多应用。
参考文献:
[1]Chang S T,Miles P G. Historical record of the early cultivation of Lentinus in China[J]. Mushroom J Tropics,1987,7:31-37.
[2]Chang S T. Past and present trends in the production of Lentinla edodes in Asia[J]. Mushroom Biology and Mushroom Products,2002,4:1-8.
[3]郭长奎,于 静,罗淑萍. 新型分子标记TRAP应用研究进展[J]. 生命科学研究,2009,13(4):366-369,376.
[4]边银丙. 我国香菇栽培种质资源与种质资源信息库建设[J]. 浙江食用菌,2008,16(1):12-15.
[5]郭金英,宋彦龙,李 超,等. 四十个野生香菇菌株遗传多样性分析[J]. 北方园艺,2018(10):157-160.
[6]孙 勇,林芳灿. 中国香菇自然种质遗传多样性的RAPD分析[J]. 菌物系统,2003,22(2):387-393.
[7]刘靖宇,宋秀高,叶 夏,等. 香菇菌株遗传多样性ISSR、RAPD和SRAP综合分析[J]. 食用菌学报,2011,18(3):1-8.
[8]卓 英,谭 琦,陈明杰,等. 香菇主要栽培菌株遗传多样性的AFLP分析[J]. 菌物学报,2006,25(2):203-210.
[9]Hu J G,Vick B A. Target region amplification polymorphism:a novel marker technique for plant genotyping[J]. Plant Molecular Biology Reporter,2003,21(3):289-294.
[10]郭春芳,唐玉海,孙 云,等. 11个茶树品种遗传多样性的ISSR和TRAP比较分析[J]. 中国农学通报,2008,24(1):340-346.
[11]Miklas P N,Hu J G,Grunwald N J,et al. Potential application of TRAP (targeted region amplified polymorphism) markers for mapping and tagging disease resistance traits in common bean[J]. Crop Science,2006,46(2):910-916.
