多距离聚类有效性指标研究
2019-11-11陈倩倩陈应霞
刘 丛,陈倩倩,陈应霞
1(上海理工大学 光电信息与计算机工程学院,上海 200093) 2(华东师范大学 计算机科学与软件工程学院,上海 200062)E-mail:Congl2014@usst.edu.cn
1 引 言
作为一种非监督学习方法,聚类已经广泛地应用于图像处理、数据挖掘、模式识别、计算机视觉等众多领域.该方法使用某种规则将数据集划分成一组互不重叠的子类,使得同一子类中的元素具有极大的相似性,不同子类中的元素具有极大的相异性[1].
近年来聚类技术受到研究者的广泛关注,大量的聚类算法被相继提出[1].可简单分为基于划分的聚类、基于密度的聚类[2]、基于层次的聚类、基于网格的聚类以及基于模型的聚类.在众多聚类算法中,基于划分的聚类[3,4]研究最为广泛.其使用类内紧凑度设计聚类目标函数,并优化该目标以获得最佳聚类结果.但该类算法含有以下几个问题:1)多数算法是基于欧氏距离而设计,该距离在对超球型数据聚类时效果比较好;但是对于非超球型数据,效果并不理想;2)多数算法需要提前指定聚类数目[5];3)通常使用基于梯度的优化算法求解,对初始值比较敏感,容易陷入局部最优.
针对问题1),使用不同的距离可以对非超球型数据聚类.基于密度的聚类方法使用密度信息度量数据间的相似性.基于层次的聚类使用类间最大、类间最小或平均距离作为数据的度量标准.近年来许多新的距离如Path距离[6]、流行距离[7]、马氏距离、切比雪夫距离、核距离[8]等被逐渐提出.但如何选择合适的距离或者将多种距离融合在一起是目前面临的一个挑战.将多种距离分别加权是一个很好的解决方法,但如何设置一个合适的权重非常困难.文献[3,4]将加权思想引入目标函数中,不同距离分配不同的权重,并根据不同数据进行动态变化,但该算法会导致数值比较小的距离分配较大的权重.针对问题2),有效性指标是常用的自动检测聚类数目的方法.该方法使用类内紧凑度和类间分离性来设计目标函数,通过寻找两者之间的平衡来自动获得最佳聚类数目.但该算法大都是基于欧氏距离而设计,所以对非超球型数据效果不理想.文献[8]使用核距离设计有效性指标自动检测非超球型数据的聚类数目,但核函数中的核参数需要手动调节,降低了算法的鲁棒性.针对问题3),进化算法在求解全局最优问题上有非常好的优势,该算法通过模拟自然界中的规律来获得最优解.近年来基于进化算法的聚类方法发展非常迅速[9,10].随着研究的深入,基于多目标进化算法的聚类方法也被相继提出.MSFCA[11]利用全局模糊紧凑性和分散性作为两个目标函数设计聚类算法并应用在图像分割中.MOCK[12]将类内紧凑度和类间连通性作为两个目标函数,并使用多目标精英进化策略算法PESA-II[13]对两目标同时优化.MOEASSC[14]利用类内偏差和最小化权重熵与类内分散性作为两个目标函数得到稳定性的聚类结果.虽然基于进化算法的聚类方法越来越多的被提出,但现有的多目标进化算法大都是基于单一的距离度量,其中以欧氏距离最为常见.该类算法对处理超球型数据效果较好,但对非超球型数据效果不理想.
针对上述问题,近期我们曾提出一种基于多目标多距离度量的聚类数目自动确定算法(MOEACDM)[15],该算法分别使用两个距离设计两个目标函数,并使用非支配排序遗传算法(NSGA-II)对其优化.但该算法由于使用基于标签的编码方式,所以导致算法时间复杂度比较大.基于此,本文提出基于多目标进化算法的多距离聚类有效性指标算法(Multiple distance validity index based on a multiobjective evolutionary algorithm)(MoMDVI).首先使用基于代表点的编码来设计染色体;其次分别基于两个距离设计两个聚类目标函数;并设计了基于差分进化和分布估计两种子代种群产生策略;最后使用RMMEDA算法[16]对其同时优化.实验证明,提出的算法获得了比较好的效果.
2 相关工作
本节主要介绍聚类有效性指标和多目标优化的两个相关工作.
2.1 有效性指标
有效性指标是常用的自动检测聚类数目方法.常用的有效性指标包括XB指标[17]、DB指标[18]等.其主要思想为通过寻找类内紧凑度和类间分离性之间的平衡来寻找最佳聚类数目.给定含有n个数据样本的待分类数据X={x1,…,xn}.使用XB指标将该数据集划分到k个类{C1,…,Ck}中.XB指标可描述为公式(1)所示.
(1)

算法1.XB指标确定聚类数目
输入:数据集X,聚类数目的搜索空间{kmin,…,kmax}
输出:最佳聚类数目k*
1.Fork=kmintokmax;
2.{C1,…,Ck}=FCM(X,k);//使用FCM聚类算法和当前类数目k对数据聚类,并获得聚类结果{C1,…,Ck}.
3.使用公式(1)计算当前聚类结果的XB值;
4.k=k+1;
5.End For
6.最佳聚类数目k*=min(XB{kmin,…,kmax}).
该算法的缺陷有两点:①模型基于欧氏距离设计,对超球型数据效果比较好,但对非超球型数据效果不理想.②使用不同的k值多次运行FCM算法,增加了出现局部解的概率.
2.2 多目标进化算法
在现实生活中,很多工程问题可以通过优化多个目标函数而获得最优决策,称之为多目标优化问题.该问题描述为公式(2)所示.
F(Θ)=(f1(Θ),…,fM(Θ))
(2)
其中Θ=(θ1,…,θh)表示一组h维的决策向量.F(Θ)表示含有M个子目标值的目标向量.在多目标优化问题中,各个目标往往是互相冲突的,因此不可能使所有目标同时达到最优,而是获得一组各个目标值所折衷的解集,称之为Pareto front解集.
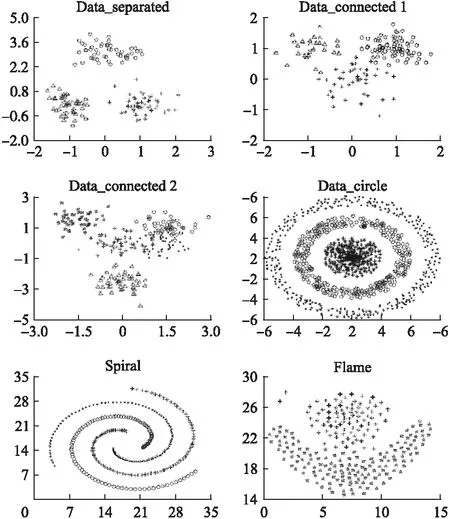
定义1.Pareto支配:如果Θ1支配Θ2,可记作Θ1Θ2,当且仅当对任意的l1∈{1,…,M},满足fl1(Θ1)≤fl1(Θ2)并且存在一个l2∈{1,…,M},满足fl2(Θ1) 定义2.Pareto front:Pareto最优解集在函数空间上对应的曲面,即为Pareto front,简称为PF.对于PF中的任意一个解Θ*,在当前解集中不能找到另一个解Θ′,满足Θ′Θ*. 本节主要介绍提出算法的主要细节,包括距离定义、目标函数设置、染色体编码与初始化,进化算子改进及聚类结果输出. 首先选择距离空间.可选的距离有很多,包括欧氏距离、Path距离[6]、流行距离[7]、马氏距离、切比雪夫距离、核距离等.理想状态下,选择的距离越多,其覆盖的数据结构也会越多.但过多的距离会导致过多的目标函数,也增加算法的运行时间.所以在此使用两种具有较大差异性的距离,分别为欧氏距离和Path距离.原因在于欧氏距离比较适合处理超球型数据集,而Path距离适合处理非超球型的数据集. X={x1,…,xn}表示含有n个数据点的待分类数据集.d(xj1,xj2)表示数据xj1和xj2的距离,满足j1,j2∈{1,…,n}.d1(xj1,xj2)表示两个数据的欧氏距离,d2(xj1,xj2)表示两个数据的Path距离. 据上所述,传统的有效性指标在处理非超球型数据时效果并不理想,基于此本文分别使用两个距离设计两个有效性指标.该指标考虑类内紧凑度和类间分离性之间的平衡.首先考虑类内紧凑度.传统有效性指标通常通过数据点到类中心之间的距离计算类内紧凑度.但在Path距离空间中,类中心已失去了其物理意义.因此本文通过类代表点来代替类中心点,类代表点是类中的一个实际样本.当分为k类时,类内紧凑度如公式(3)所示. (3) 其中ri是X中的一个数据,满足ri∈X.在公式(3)中ri表示第i个类的类代表点,并满足i∈{1,…,k}.该紧凑度Com(k)随着k值的增加呈减小趋势.其次考虑类间分离性,在此使用任意两个类的类代表点来设计类间分离性.可表示为如公式(4)所示. (4) i1,i2∈{1,…,k}表示任意两个类的类代表点的序号.Sep(k)随着k值的增加呈现增加的趋势.通过寻找Com(k)和Sep(k)之间的平衡来自动检测最佳聚类数目.由于Com(k)和Sep(k)具有不同的数量级,故分别对紧凑度和分离性进行归一化处理.首先归一化类内紧凑度Com(k),当k=1时,Com(1)可取最大值;当k=kmax时Com(kmax)可取最小值,其中kmax表示可接受的最大类数目上限.极端条件下,当k=n时,Com(n)=0.归一化后的类内紧凑度如公式(5)所示. (5) 其次归一化类间分离性Sep(k),当k=1时,Sep(1)可取最小值;当k=kmax时,Sep(kmax)可取最大值.归一化后的类间分离性如公式(6)所示. (6) 根据类内紧凑度和类间分离性之间的平衡,建立新的有效性指标如公式(7)所示: F(k,r)=Comnor(k)+Sepnor(k) (7) 其中r表示一组需要求解的类代表点r={r1,…,rk}. 将公式(7)中的距离分别改写成欧氏距离和Path距离,则基于欧氏距离和Path距离的有效性指标表示如公式(8)所示: (8) 其中Comd1(k)和Comd2(k)分别表示基于欧氏距离和Path距离的类内紧凑度.Sepd1(k)和Sepd2(k)分别表示基于欧氏距离和Path距离的类间分离性.Comd1(1),Comd2(1),Sepd1(kmax)和Sepd2(kmax)需要提前计算.对于Comd1(1)和Comd2(1),通过遍历寻找分成1类时的类内紧凑度.对于Sepd1(kmax)和Sepd2(kmax),可使用Kmedoids算法[19]将数据分为kmax类来获得. 接下来通过多目标算法来求解两个未知参数k和r={r1,…,rk}. 本节重点介绍染色体编码与初始种群生成.由于聚类数目也是需求解的一个参数,所以本节需要用到可变长编码方式.现有的编码方式有很多,如基于标签的编码、基于实数的编码等.由于基于标签的编码会产生较大的染色体长度,继而导致运行时间较长,在此使用基于实数的编码方式.如前所述,可接受的聚类数目上限为kmax,故本节的染色体长度为kmax.染色体可表示如公式(9)所示. γ={γ1,…,γkmax} (9) 其中γi表示第i个基因并满足γi∈(0,1].由于最佳类数目k*应满足k*≤kmax,所以对每个基因γi设置一个标签来判断该基因是否能被选中作为类代表点.γi≤0.5表示该基因可被选中作为有效代表点,并参与后续计算.γi>0.5表示该基因不能作为有效代表点,直接将其舍弃.将所有γi≤0.5的基因挑选出,可获得一组类代表点基因,如公式(10)所示. γv={γv1,…,γvk} (10) 其中k表示有效代表点的数目,满足k≤kmax.下一步将γvi映射成一组整数,该整数表示类代表点在整个数据中的序号.该映射方式为公式(11)所示. Iγvi=「γvi*n*2⎤ (11) 其中Iγvi∈{0,…,n}表示第i个类代表点所在整个数据集中的序号.再次解码,则获得k个代表点r={r1,…,rk},其中ri=xIγvi. 初始种群一般通过随机生成.为了提高算法的收敛速度,本文使用一种预聚类算法来产生该初始种群.据上所述,本文的染色体是一组表示类代表点序号的实数,该组实数解可通过Kmedoids算法获得.在预聚类中,需要将两个距离和不同的聚类数目加入其中.据此分析,初始种群由三种策略组成:(1)部分种群通过随机生成;(2)部分种群通过使用Kmedoids算法使用欧氏距离和不同的聚类数目生成;(3)部分种群使用Kmedoids算法使用Path距离和不同的聚类数目生成. 本文使用RMMEDA算法作为多目标算法优化器.据文献[20]所示,在迭代初期正则化信息并不明确,会导致建立的模型会略有偏差.基于此,本文使用差分进化算子来弥补RMMEDA算子产生的缺陷.α表示差分进化算子和RMMEDA算子产生的种群数目比例.popsize表示的种群规模.popsize*(1-α)个染色体由RMMEDA算子建模采样获得,popsize*α个染色体由差分进化算子获得.α随着迭代次数的增加而发生变化,由于迭代前期正则化信息不明显,过多的使用差分进化算法来产生新的种群,迭代后期随着正则化信息越来越明确,过多的使用RMMEDA算子产生新的种群.则α变化如公式(12)所示. (12) 其中maxg表示最大迭代次数,g表示当前代数.当产生子代种群后,使用非支配排序在父代种群和子代种群中挑选较优的popsize个染色体作为下一代种群.该进化算子如算法2所示. 算法2.进化算子 输入:第g代种群pop(g)={γ1(g),…,γpopsize(g)} 输出:第g+1代种群pop(g+1)={γ1(g+1),…,γpopsize(g+1)} 1.使用公式(12)计算参数α; 2.提取pop(g)正则化信息,并使用该信息进行建模; 3.对上述模型进行采样,获得popsize*(1-α)个子代染色体, offpop(g)={off_γ1(g),…,off_γpopsize*(1-α)(g)}; 4.使用差分进化算法产生popsize*α个子代染色体 offpop(g)={off_γpopsize*(1-α)+1(g),…,off_γpopsize(g)}; 5.使用非支配排序与拥挤距离从pop(g)∪offpop(g)中选择popsize个染色体作为第g+1代种群. 迭代结束后,可获得最终的popsize个染色体.使用非支配排序,从所有染色体中选出Pareto front解集,也称为非支配解集.每个非支配解都被认为是可行解,并且可使用公式(10)和(11)映射成一组类代表点.将数据集中的剩余数据划分到与其最近的类代表点所在的类中.由于在寻找最近类代表点时可使用欧氏距离和Path距离.所以针对每组类代表点,剩余数据都可获得两种归类方式.如果Pareto front中含有PFsize个非支配解集,则最终可获得2*PFsize聚类结果.而后使用人工选择的方式挑选出最佳聚类结果. 为了验证MoMDVI算法的有效性,本文使用8个测试数据展示本文算法的优势.该数据包括3个超球型数据、3个非超球型数据及2个真实数据.其中部分测试数据如图1所示. 图1 测试数据集Fig.1 Testing datasets Data_separated、Data_connected1和Data_connected2为3个超球型数据,每个数据含有若干超球型的类.这3个数据为人工生成数据.Data_circle、Spiral和Flame[21]为非超球型数据.其中第一个数据为人工生成数据,后两个为现有文献中常用的非超球型数据.Iris和Wine为两个UCI真实数据. 实验中,MoMDVI参数设置如下,为了降低运行时间,将kmax设置为10.种群规模popsize设置为200,最大迭代次数maxg设置为100.为了定量的评估提出算法的性能,本实验采用最常用的Rand指标[22]和F-measure指标[23]对聚类效果进行评价,计算指标时,每个数据的分类标记已知.在实验中两个指标简写为R和F,每个聚类结果对应的聚类数目用clusternumber(CN)表示. 本节使用Data_connected1和Data_circle两个数据集展示使用MoMDVI算法产生的Pareto front和对应的聚类结果.由于每个非支配集需使用欧氏距离(ED)和Path距离(PD)对剩余数据归类,所以每个非支配集对应两个聚类结果.在此使用ED和PD区分. 图2 Data_connected1的Pareto front集和对应聚类结果Fig.2 Pareto front and clustering results of Data_connected1 首先分析Data_connected1数据.如图2(a)所示,该数据得到5个Pareto非支配解,我们选择3个点进行聚类结果分析,图中已用椭圆标记.图2(b)、(c)和(d)分别对应图2(a)中被椭圆标记的非支配解.由该图可以看出使用欧氏距离对第2个标记的非支配解解码,可以获得最佳聚类效果. 其次分析Data_circle数据,如图3(a)所示.可得到9个Pareto点,仍然选择3个Pareto点分析.图3(d)中的第2个图为最佳的聚类结果,可以看出Path距离更适应于处理非超球型数据集. 图3 Data_circle的Pareto front集和对应聚类结果Fig.3 Pareto front and clustering results of Data_circle 为了验证MoMDVI算法的有效性,本节将其与现有的算法进行比较.首先选择两种常用的有效性指标XB和PBM[24]指标.由于本文算法是基于进化算法而设计,第三种对比算法为GCUK[25]算法,该算法使用遗传算法对DB指标进行优化.第四种对比方法为RAC-Kmeans算法[26].前三种对比算法都是基于欧氏距离设计的,第四种算法不需要考虑距离选择. 对比算法的参数设置如下.对于XB、PBM、GCUK和MoMDVI,kmax设置为10.对于RAC-Kmeans,该参数设置为n.对于GCUK和MoMDVI,种群规模popsize设置为200,最大迭代次数maxg设置为100. 在对比过程中,使用的指标为Rand和F-measure指标.每种算法分别运行10次,Mean_R、Mean_F、Max_R和Max_F分别表示两种指标在10次运行中获得的平均值和最大值.CN表示获得的最佳聚类数目其中括号中表示10次运行获得该聚类数目的次数. 表1展示了5种算法分别对8个测试数据聚类结果.通过该表可以得出,所有算法对于数据Data_separated都能获得比较好的聚类,原因在于该数据比较简单.对于数据Data_connected1和Data_connected2,所有的算法都可以获得相对较好的聚类效果,但MoMDVI获得的结果精度最高.通过这3个数据可知,MoMDVI处理超球型数据时,可以获得比较好的效果.对于非超球型数据Spiral、Data_circle和Flame,算法XB、PBM和GCUK都不能获得好的效果,这表明欧氏距离对非超球型数据效果不理想.使用算法RAC-Kmeans和MoMDVI对数据Spiral和Data_circle聚类,可以获得比较好的聚类效果;但是对于数据Flame,MoMDVI算法比RAC-Kmeans获得更好聚类效果.这表明MoMDVI对非超球型数据聚类也可以获得比较好的聚类效果.对于真实数据Iris和Wine数据,所有算法聚类结果相差不大,但MoMDVI算法在聚类精度上更有优势.因此通过该实验可以得出基于两种距离设计的MoMDVI算法对球型数据和非球型都表现出比较好的聚类效果. 表1 不同聚类算法的聚类数目(CN)及准确度(Mean_R/Max_R,Mean_F/Max_F)的比较Table 1 Comparison of the CN、Mean_R/Max_R and Mean_F/Max_F by different clustering algorithms 由于MoMDVI为解决MOEACDM[15]具有较高的时间复杂度而设计.本节对比两种算法的运行时间.两种算法使用相同的种群规模(popsize=200)和相同的迭代次数(maxg=200).使用的运行软件为Matlab2014b,使用的电脑配置为2.5G赫兹CPU,4G内存.表2为两种算法在4个数据集上运行使用时间. 通过表2可以得出,由于MoMDVI使用代表点编码,该算法通过缩小染色体的长度来降低算法的运行时间,所以比MOEACDM算法使用更少的运行时间. 表2 两种算法使用相同参数获得运行时间Table 2 Comparison computation time(s)by using two methods with the same(popsize,maxg) 针对传统有效性指标仅考虑欧氏距离的缺陷,本文提出了一种基于多距离的有效性指标(MoMDVI)来自动检测最佳聚类数目.该算法使用欧氏距离和Path距离分别设计有效性指标来自动检测聚类数目,并使用多目标进化算法对两种有效性指标进行并行优化.由于在Path距离中,类中心点已失去其意义,本文使用类代表点代替类中心来设计有效性指标.每个染色体由一系列实数组成,部分实数可转化为类代表点序号的形式.在设计进化算子时,将差分进化算子与RMMEDA进化算子融合,使其更快收敛.提出的算法既能自动检测超球型数据聚类数目,也可以自动检测非超球型数据聚类数目. 提出的算法还有两个问题需要改进.第一,本文算法只使用欧氏距离和Path距离作为两种距离进行分析,接下来可以考虑将更多的距离加入到算法模型中.第二,如何在最终Pareto非支配集中自动选择最佳解也是下一步需要考虑的工作.3 基于多目标进化算法的多距离有效性指标(MoMDVI)
3.1 距离定义
3.2 目标函数设置
3.3 染色体编码与初始化
3.4 进化算子
3.5 输出最佳聚类结果
4 实验结果与分析
4.1 聚类结果展示


4.2 精度对比

4.3 运行时间对比

5 结束语
