基于高密度连锁图谱定位玉米株高QTL
2019-10-25刘敬贤黄亚群陈景堂
刘敬贤 黄亚群 陈景堂
摘要:为了解析株高性状的遗传基础,以X178和NX531为亲本构建的124份RIL群体为研究材料,基于高密度SNP标记构建的包含7 278个bin的bin-map连锁图谱,对辛集、保定2个地点RIL群体的株高、穗位高、穗位系数3个性状进行QTL定位分析,共检测到16个QTL位点,有9个QTL的表型贡献率大于10.00%。其中辛集检测到7个,单个QTL表型贡献率范围4.67%~13.94%;保定检测到9个,单个QTL表型贡献率范围0.35%~25.56%。在2个环境下检测到qEHX3和qEHB3的置信区间存在重叠。在第1连锁群上289.16~296.77 Mb发现控制株高的qPHB1和穗位高的qEHB1-2定位区间相邻。在bin1.07定位到的qPHX1-1区间内存在br2(brachytic2)基因,bin1.09~1.1定位到的qPHX1-2区段内存在d8(dwarf8)基因,bin3.07定位到的qEHX3区段内存在ccd8基因,这3个基因影响节间的伸长,与株高、穗位高的发育相关。该研究结果为株高相关性状QTL精细定位、克隆提供理论依据。
关键词:玉米;株高;穗位高;穗位系数;高密度连锁图谱
中图分类号:S513.03 文献标志码: A 文章编号:1002-1302(2019)13-0038-04
随着我国农业的快速发展,全程机械化生产是解决“三农”问题的关键。玉米倒伏直接影响机械化生产,而品种自身抗倒能力的强弱直接决定了能否采用机械化生产。在玉米的诸多株型性状中,株高、穗位高与玉米抗倒能力密切相关。Horner等以F44和F6为材料进行7轮回交选择,发现穗位降低9%,倒伏率减少25%[1]。张泽民等研究表明,通过降低穗位系数,可以提高其抗倒性[2]。付志远等研究发现,穗上节间数与穗位高及穗位系数显著相关,可以通过增加穗上节间数来减小穗位系数,增强玉米的抗倒性能[3]。而选育抗倒、适合于机械化收获的玉米品种,必须了解株高、穗位高等性状的遗传机制。
一些研究者已发现株高、穗位高性状受主基因+多基因控制,且基因的加性、显性和上位性效应均起作用;在不同的遗传群体中这些基因作用的大小有差异,以基因的加性效应为主[4-6]。严建兵等利用简单序列重复(SSR)等分子标记对株高等性状进行定位研究,发现了一些与玉米株高、穗位高有关的数量性状基因座(QTL)[7-10]。这些相关QTL因其定位区间大,目前还没有应用于育种实践。虽然,杨梅利用所设计的SSR和Indel标记对玉米第3染色体上控制株高的主效QTL qPH3.2.1、qPH3.2.2、qPH3.3進行了精细定位,分别将定位区间缩小到7.6、7.2、11 Mb,仍无法精准地预测到候选基因[11]。
随着高通量、操作简便、成本低廉的第3代测序技术广泛应用,利用高密度的单点多态(SNP)标记检测控制数量性状的关键位点已成为众多学者的研究工具[12-13]。在高粱[14]、玉米[15]、水稻[16]、小麦[17]、棉花[18]等多种作物上均有利用SNP标记对株高等性状进行定位分析的报道。Wang等利用SNP标记对玉米株高进行定位,发现在控制株高的QTL区间内存在na1、td1和d3[19]。
虽然Sheridan[20]等学者,利用玉米突变体发现了与株高相关的基因,但对这些基因几乎未能实现克隆[20-21]。到目前为止,只有Teng等对ZmGA3ox2基因进行了克隆[22]。而利用高通量的SNP标记可将位点定位到较小的区段,实现目标性状位点的精细定位和候选基因的有效预测。本研究采用玉米自交系X178和NX531为亲本构建的重组自交系(RIL),对株高相关性状进行调查,并利用高密度的连锁图谱对其进行定位,挖掘株高相关性状紧密连锁的分子标记,检测主效QTL区域,为株高相关性状QTL克隆和分子标记辅助育种提供理论依据。
1 材料与方法
1.1 试验材料
以玉米农大108亲本之一X178和农单5亲本之一NX531杂交,以单粒传法连续自交构建的124份F9重组自交系(RIL)为试验材料。农大108和农单5均为国家审定品种,具有广泛种植面积。
1.2 试验设计
RIL群体及其2个亲本于2017年分别在国家玉米改良中心河北分中心试验基地(简称保定,BD,38°87′N,115°47′E)和河北农业大学辛集试验基地(简称辛集,XJ,37°94′N,115°22′E)进行春播(4月20日)和夏播(6月18日)。2个试验点均采用随机区组试验设计,单行区,2次重复,小区行长 3.0 m,行间距0.6 m,种植密度设置为 75 000株/hm2,并设置保护行。保定试验点,无前茬作物,播种前施农家肥作为基肥,在播种前和拔节期各浇灌1次水;辛集试验点,前茬作物为小麦,施用三元(N ∶ P2O5 ∶ K2O=18 ∶ 20 ∶ 5)复合肥做基肥,播种后进行浇水。其他田间管理同大田生产。
1.3 株高相关性状测定指标及方法
在玉米成株期,对辛集和保定2个试验点RIL群体每株系选取3株进行株高(plant height,简称PH)、穗位高(ear height,简称EH)的测定,并计算穗位系数(ear height coefficients,简称EHC)[18]。株高:地面至雄穗顶部的距离(cm);穗位高:地面至穗位节处的距离(cm);穗位系数:穗位高与株高的比值。
1.4 表型数据统计分析
利用SPSS 19.0对所调查株高、穗位高和穗位系数进行描述性统计分析、正态性Kolmogorov-Smirnov检验。
1.5 遗传连锁图谱的构建及QTL定位分析
采用7 278个bin标记构建的覆盖全基因组2 017.13 Mb bin-map遗传图谱[23],根据复合区间作图法(complex interval mapping,简称CIM),使用R/qtl软件包中的cim_scan命令,对株高、穗位高、穗位系数进行QTL定位,window设为10 cM。运行参数为默认值,LOD值设置为2.5,QTL置信区间用1.5个LOD值衰减方法进行判定,用R命令的1 m来计算每个QTL的加性效应及其对应的表型贡献率。
2 结果与分析
2.1 RIL群体及其亲本株高相关性状的表型统计分析
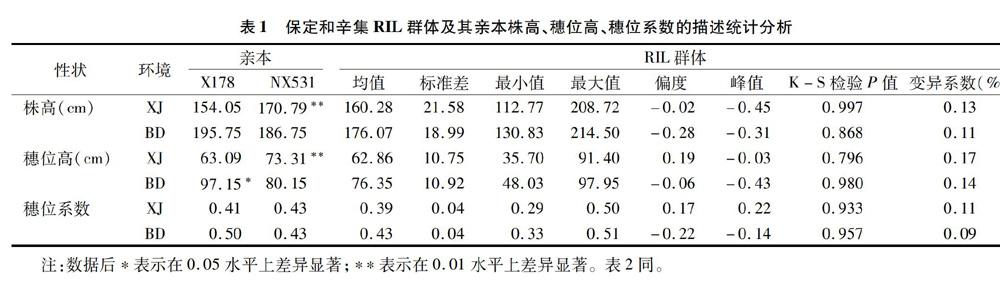
保定和辛集2个试验点RIL群体及其亲本株高、穗位高、穗位系数表型数据统计分析结果见表1。对亲本株高、穗位高、穗位系数3个性状进行差异显著性t检验发现,亲本X178的株高、穗位高在辛集环境下极显著低于亲本NX531(P<0.01),亲本X178的穗位高在保定环境下显著高于亲本NX531(P<0.05)。RIL群体株高、穗位高、穗位系数在2个环境下的最大值均高于相应的高值亲本,最小值均低于相应的低值亲本,表现为双向超亲分离,具有较大的变异范围。对RIL群体株高、穗位高、穗位系数的分布状况进行单样本K-S检验,P值为0.796~0.997,说明3个性状均服从正态分布。RIL群体株高、穗位高、穗位系数3个性状表现典型的数量性状特征,符合QTL定位的要求。
2.2 高密度遗传连锁图谱的构建
利用已获得的7 278个重组bin标记,构建的高密度bin标记遗传图覆盖全基因组2 017.13 Mb,相邻的2个bin之间的物理距离最大为3.28 Mb,最小为80.00 kb,平均为 277.00 kb;构建的遗传连锁图谱总长为2 569.00 cM,相邻的bin标记之间平均遗传距离为0.35 cM。
2.3 RIL群体各个株型相关性状的QTL分析
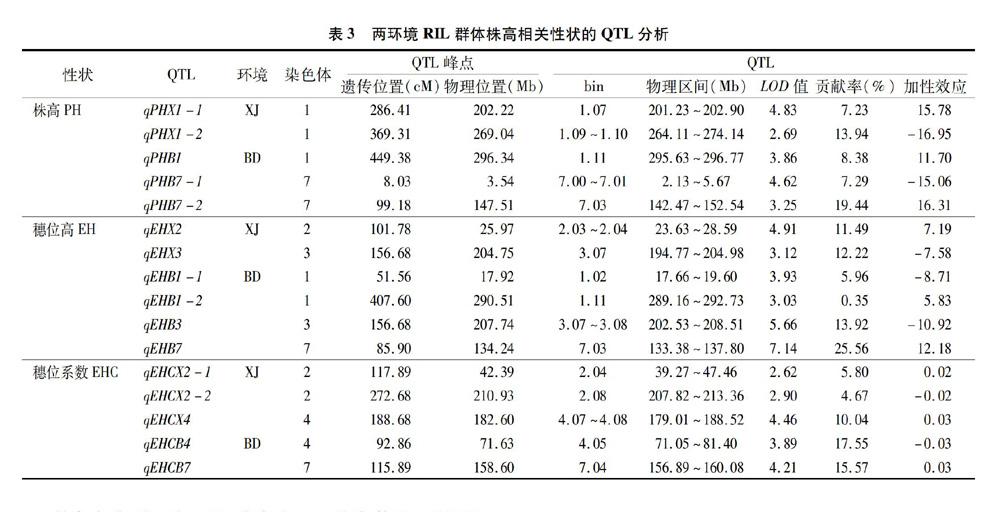
对玉米RIL群体株高、穗位高、穗位系数3个性状进行QTL定位分析,共定位到16个QTL(表3),其中在辛集检测到7个,分布在1、2、3、4号染色体上,单个QTL表型贡献率为 4.67%~13.94%,其中有4个QTL的表型贡献率大于10.00%,单个QTL的遗传图距为4.27~17.79 cM,物理距离为1.67~10.21 Mb,其中有2个QTL的物理距离在5.00 Mb以内;在保定检测到9个,分布在1、3、4、7号染色体上,单个QTL表型贡献率范围为0.35%~25.56%,其中有5个QTL的表型贡献率大于10.00%,单个QTL的遗传图距为2.07~18.83 cM,物理距离为1.14~10.35 Mb,其中有6个QTL的物理距离在 5.00 Mb 以内。
株高定位到5个QTL,分布在1、7染色体上,可解释 7.23%~19.44%的表型变异。在第1染色体269.04 Mb位置上的qPHX1-2可解释13.94%的表型变异,在第7染色体147.51 Mb位置上的qPHB7-2可解释19.44%的表型变异,并且qPHX1-2和qPHB7-2的增效等位基因均来自母本X178。穗位高定位到6个QTL,分布在第1、2、3、7染色体上,可解释0.35%~25.56%的表型变异,其中定位到4个QTL的表型贡献率大于10.00%,qEHX2和qEHB3的增效等位基因均來自父本NX531,qEHX3和qEHB7的增效等位基因均来自母本X178,其中bin3.07(204.75 Mb)位置上的qEHX3和bin3.07~3.08(207.74 Mb)位置上的qEHB3的峰值物理位置相距5.98 Mb,其置信区间存在重叠,说明该区段上控制穗位高的QTL具有较强的稳定性与可靠性。第1连锁群上在289.16 Mb~296.77 Mb之间发现控制穗位高和株高QTL定位区间临近,可能是存在紧密连锁的控制株高、穗位高的基因,也可能是一因多效。穗位系数检测到6个QTL,分布在2、4、7染色体上,可解释4.67%~17.55%的表型变异,3个QTL的表型贡献率大于10.00%,其中在第4染色体bin 4.05(71.63 Mb)位置上的qEHCB4可解释的表型贡献率最大。
3 结论与讨论
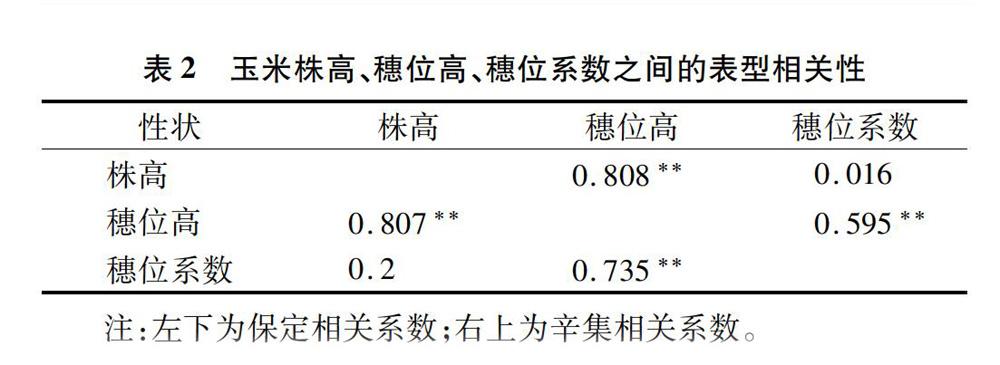
本研究发现辛集、保定2个环境下株高、穗位高和穗位系数表型值间相关关系与QTL定位相关性近似一致。如株高与穗位高表现出高度的相关关系,在第1连锁群上 289.16 Mb~296.77 Mb发现控制株高的qPHB1和穗位高的qEHB1-2定位区间临近。这一研究结果也证实了其他研究试验[24]。李清超等在多个区域同时检测到控制株高和穗位高的QTL,并且株高和穗位高具有较强的相关关系[24]。
株高、穗位高是重要的农艺性状,是抗倒、机械化收获的重要指标参数。本研究利用高密度连锁图谱,检测到16个与株高、穗位高、穗位系数相关的QTL。该定位结果与前人研究进行比较,发现本研究在第1染色体17.66~19.60 Mb区段检测到的控制穗位高的qEHB1-1,位于李浩川等定位到的穗位高QTL区间内[25];在第3染色体194.77~204.98 Mb区段检测到控制穗位高的qEHX3和202.53~208.51 Mb区段检测到控制穗位高的qEHB3,在第3染色体上检测到控制穗位高的QTL与杨晓军等[9]、Guan等[21]、Li等[26]的定位结果存在重叠,并且qEHX3和qEHB3的峰值物理位置相距 2.99 Mb;在第7染色体133.38~137.80 Mb区段检测到控制穗位高的qEHB7,该QTL位于杨晓军等在第7染色体上检测到控制穗位高的QTL定位区间内[9];在第2染色体39.27~47.46 Mb区段检测到控制穗位系数的qEHCX2-1,与李浩川等定位到的穗位高QTL区间存在重叠[25]。Guan等在qEHX3定位区间内发现ccd8基因,通过影响玉米节间的伸长,影响玉米穗位高[21]。依据这些位点在不同的群体、不同试验环境均被检测到这一结果,认为qEHB1-1、qEHX3、qEHB3、qEHB7、qEHCX2-1是真实存在的,为遗传稳效QTL,是控制株高的重要位点。
本研究在bin1.07(201.23~202.9 Mb)区段定位到株高的qPHX1-1,其定位区间仅有1.67 Mb,生物信息学研究发现在该区域内存在br2(brachytic2)基因。玉米中br2基因编码ABC转运体,参与生长素的极性运输,玉米br2的突变主要影响下部茎节间的生长[27]。在bin1.09-1.10(264.11~274.14 Mb)位置上定位到株高的qPHX1-2,在这一区段内存在d8(dwarf8)基因。拟南芥、小麦、玉米中的GAI,Rht-1和d8为直系同源基因,GAI、Rht-1和d8基因编码含有SH2结构域,类似于核转录因子的蛋白质,可能参与赤霉素信号转导[28]。在bin3.07(194.77~204.98 Mb)位置上定位到株高的qEHX3,在这一区段内存在ccd8(carotenoid cleavage dioxygenase8)基因。Guan等发现ccd8基因参与独角金内酯的信号转导途径,Zmccd8突变会显著降低茎直径,影响植株节间的伸长,使不定根发育迟缓[21]。
參考文献:
[1]Horner E S,Lutrick M C,Chapman W H,et al. Effect of recurrent selection for combining ability with a single-cross tester in maize[J]. Crop Science,1976,16(1):5-8.
[2]张泽民,贾长柱. 玉米株型对遗传增益的影响[J]. 遗传,1997,19(2):35-38.
[3]付志远,邵可可,陈德芝,等. 穗上节间数与玉米抗倒伏能力的相关性分析[J]. 河南农业大学学报,2011,42(2):149-154.
[4]兰进好,褚 栋. 玉米株高和穗位高遗传基础的QTL剖析[J]. 遗传,2005,27(6):925-934.
[5]王铁固,马 娟,张怀胜,等. 玉米穗位高的主基因+多基因的遗传模型分析[J]. 贵州农业科学,2012,40(4):10-13.
[6]郑德波,杨小红,李建生,等. 基于SNP标记的玉米株高及穗位高QTL定位[J]. 作物学报,2013,39(3):549-556.
[7]严建兵,汤 华,黄益勤,等. 不同发育时期玉米株高QTL的动态分析[J]. 科学通报,2003,48(18):1959-1964.
[8]汤 华,严建兵,黄益勤,等. 玉米5个农艺性状的QTL定位[J]. 遗传学报,2005,32(2):203-209.
[9]杨晓军,路 明,张世煌,等. 玉米株高和穗位高的QTL定位[J]. 遗传,2008,30(11):1477-1486.
[10]何坤辉,常立国,崔婷婷,等. 多环境下玉米株高和穗位高的QTL定位[J]. 中国农业科学,2016,49(8):1443-1452.
[11]杨 梅. 玉米株高QTL qPH3.2和qPH3.3的精细定位[D]. 武汉:华中农业大学, 2017:20-30.
[12]Zhou Z Q,Zhang C S,Zhou Y,et al. Genetic dissection of maize plant architecture with an ultra-high density bin map based on recombinant inbred lines[J]. BMC Genomics, 2016,17(1):178.
[13]Cui M,Jia B,Liu H H,et al. Genetic mapping of the leaf number above the primary ear and its relationship with plant height and flowering time in maize[J]. Frontiers in Plant Science,2017,8:1437.
[14]Zou G H,Zhai G W,Feng Q,et al. Identification of QTLs for eight agronomically important traits using an ultra-high-density map based on SNPs generated from high-throughput sequencing in sorghum under contrasting photoperiods[J]. Journal of Experimental Botany,2012,63(15):5451-5462.
[15]Zhang W Q,Zhang M C,Li Z H,et al. Dissection of the molecular genetic architecture of the ratio of ear to plant heights in response to ethylene by a RIL population with SNPs marker in maize[J]. Acta Physiologiae Plantarum,2017,39(6):142.
[16]Huang X E,Feng Q,Qian Q,et al. High-throughput genotyping by whole-genome resequencing[J]. Genome Research,2009,19(6):1068-1076.
[17]Zou J,Semagn K,Iqbal M,et al. Mapping QTLs controlling agronomic traits in the ‘AttilaבCDC go spring wheat population under organic management using 90K SNP array[J]. Crop Science,2017,57(1):365-377.
[18]Qi H K,Wang N,Qiao W Q,et al.Construction of a high-density genetic map using genotyping by sequencing (GBS) for quantitative trait loci (QTL) analysis of three plant morphological traits in upland cotton (Gossypium hirsutum L.)[J]. Euphytica,2017,213(4):83.
[19]Wang B B,Liu H,Liu Z P,et al.Identification of minor effect QTLs for plant architecture related traits using super high density genotyping and large recombinant inbred population in maize (Zea mays)[J]. BMC Plant Biology,2018,18(1):1-12.
[20]Sheridan W F. Maize developmental genetics:genes of morphogenesis[J]. Annu Rev Genet,1988,22(1):353-385.
[21]Guan J C,Koch K E,Suzuki M,et al. Diverse roles of strigolactone signaling in maize architecture and the uncoupling of a branching-specific subnetwork[J]. Physiologia Plantarum,2012,160(3):1303-1317.
[22]Teng F,Zhai L H,LiuR X,et al.ZmGA3ox2,a candidate gene for a major QTL,qPH3.1,for plant height in maize[J]. Plant Journal,2013,73(3):405-416.
[23]賴国荣,张 静,刘 函,等. 基于GBS构建玉米高密度遗传图谱及营养品质性状QTL定位[J]. 农业生物技术学报,2017,25(9):1400-1410.
[24]李清超,李永祥,杨钊钊,等. 基于多重相关RIL群体的玉米株高和穗位高QTL定位[J]. 作物学报,2013,39(9):1521-1529.
[25]李浩川,陈 琼,杨继伟,等. 基于双单倍体群体的玉米株高和穗位高QTL分析[J]. 河南农业大学学报,2016,50(2):161-166.
[26]Li X P,Zhou Z J,Ding J Q,et al. Combined linkage and association mapping reveals QTL and candidate genes for plant and ear height in maize[J]. Frontiers in Plant Science,2016,7(833):1-11.
[27]Multani D S,Briggs S P,Chamberlin M A,et al. Loss of an MDR transporter in compact stalks of maize br2 and sorghum dw3 mutants[J]. Science,2003,302(5642):81-84.
[28]Peng J,Richards D E,Hartley N M,et al. ‘Green revolution genes encode mutant gibberellin response modulators[J]. Nature,1999,400(6741):256-261.
