基于序列公共稀疏误差对称差异度分割的健壮脑肿瘤良恶性分类方法
2019-10-15公慧玲李致勋
公慧玲,李致勋
(南昌大学信息工程学院,南昌330031)
0 引言
在针对脑肿瘤图像的良恶性智能识别过程中,肿瘤区域的准确检测和分割是其中最为关键的一步。目前,业界有以下几类分割方法:①基于机器学习的方法,如马尔科夫随机场MRF 等[2];②基于统计学习的方法,如高斯混合模型GMM 等[3];③基于轮廓的技术,如主动轮廓、变形模型等[4];④基于深度学习的方法,如CNN、RCNN 等方法[5]。但是,当前研究多集中于单帧图像,由于肿瘤区域软组织相对较低的对比度,单帧图像中对边缘的判断是一个极大的挑战,漏分和错分情况极易出现。
为了更好地利用CT 或MR 图像中的三维序列特性,本文提出一种基于稀疏误差对称差异度的脑肿瘤显著性分割方法,通过对序列间对应的超像素区块进行提取并分析公共稀疏误差,再通过该稀疏误差对图像中每个像素进行有效性计算,以达到各层间信息互补的目的。最后利用左右脑半球间的对称差异度对脑肿瘤区域显著性进行计算和分割,最大程度降低肿瘤范围漏分或错分情况,并在此基础上提出了一系列健壮特征用于脑肿瘤的良恶性分类。
1 基于序列显著性帧间配准的脑肿瘤显著性映射
1.1 全图像健壮超像素区块的分割
此步骤中对整张图像进行的超分辨率分割会产生若干个超像素区块,这些区块将用于有意义区块的提取,同时减少噪声干扰并提高处理速度。这里首先利用Quickshift 算法进行粗超像素分割[6],然后再通过Kmeans 算法对各超像素区块中信号强度的熵、平均信号强度、信号方差3 个特征进行聚类,聚合成N 个类别由于此处每个类别是相邻相近信号区域的融合,所以聚类后的超像素区块划分更具合理性和健壮性。
1.2 序列超像素间公共稀疏重构误差的获取
为了用序列信号来弥补单帧图像中信号缺失的部分,此步利用上步所获取的N 个区块,在M 帧序列图像中对对应区块中的所有像素进行信号强度分布分析,分析公式为:

其中f 函数用于判别Cp中的超像素区块在相邻各帧图像It中出现的概率。如果某区块在某帧中出现,则f=1,否则f=0。Zp值越大代表着Cp中的超像素在序列中出现的概率越大,同时表明其为共同有效区块的可能性越大。当获取了每个类的分布情况后,则可以生成符合序列图像的公共重构基Fcomm:

其中α2为阈值,用于去除在序列中较少出现的类别。对图像It中所包含的超像素,进行稀疏变换后可得到稀疏编码为:

位于海南三亚的亚特兰蒂斯是一座海洋主题的度假胜地,囊括了惊险刺激的水世界冒险乐园、神秘奇幻的水族馆、互动性极强的海豚湾、不同风味的餐厅酒吧及餐饮点位、全海景客房及套房、高端雅致的水疗馆、时尚新潮的精品店铺以及大型会议中心与多功能厅。

1.3 左右脑半球间对比差异度及肿瘤显著性提取
由于人脑组织近似对称的结构特性,脑肿瘤区域可以通过两个脑半球的非对称区域获得。即当某半球脑组织中出现肿瘤后,该区域与另一半球对比将出现不一致性,从而体现出显著性。
这里采用Glocker 所提出的离散MRF 模型用于获取对比差异度[7]。设某脑半球中的像素标签集ℓ={1 ,… ,n} 和与之对应的变形离散偏移标记Δ={d1,… dn} ,对于每一个控制节点p,通过变形以确定左右脑半球标签间的对应关系。变形公式如下:

由于此处仅要分析对齐误差,所以脑肿瘤显著性值可以变形为下列公式:

该式函数值在两侧不具有对称性时更大,而具有对称性时更小,即两侧不均等时为肿瘤的可能性更大。
在获得显著性后,由于肿瘤信号和正常组织信号差异具有恒定的符号关系,例如CT 图像中肿瘤区域的信号强度通常都会高于正常组织,所以最终可以通过非对称符号正负来判别哪边存在肿瘤。
2 肿瘤区域健壮形状及纹理特征提取
在准确获取脑肿瘤轮廓之后,脑肿瘤形态的健壮表达是准确良恶性识别的关键步骤。本文根据脑肿瘤的特点,提出以下基于形状和纹理的健壮特征:
形状特征:①似圆度:通过计算最大拟合圆周长与实际轮廓周长之比,描述肿瘤的边缘是否平滑和规则;②平均归一化径向长度:通过计算肿瘤的中心到边缘的距离,来描述肿瘤的位置形态;③凸起的数量:因不同类型的肿瘤具有不同的凸起数目,所以可通过中值滤波器和多项式曲线拟合来计算径向距离,用于跟踪针状度;④轮廓椭圆归一化:因边界中含有较多的突起和凹陷,则椭圆轮廓具有更高的复杂度,因此它也可以用于合理地描述肿瘤轮廓的复杂度。
纹理特征:灰度共生矩阵描述的纹理信号间的强度关系,常用于图像的纹理特性分析[8]。在用于具有256 个灰度级的医学图像时,矩阵大小设为256×256。参数设置像素相对距离设置为1、2、3,相对角度设定为0°、45°、90°、135°。然后在每个距离和角度上分别提取四个特征:对比度、能量、同一性和相关性。最后,把相邻切片的特征进行融合。
3 算法实现
本文算法的具体实现流程如下:
第一步:预处理。为了减少设备间差异造成的影响,此步主要对图像使用2D 高斯低通滤波器来进行平滑处理,以及使用线性正则化方法来进行图像增强处理。
第二步:脑肿瘤显著区域提取及分割。此步首先利用本文所述的基于序列公共稀疏误差对称差异度进行显著性计算。获取显著性值后,采用图割的方法进行最终的肿瘤区域分割。值得一提的是,由于在显著性计算中,目标区域具有较大的显著性值,而背景区域具有较小的显著性值,因而可以在此处直接套用图割算法。
第三步:脑肿瘤显著性轮廓特征提取。似圆度、平均归一化径向长度、凸起的数量、轮廓椭圆归一化等形状特征,以及灰度共生矩阵的纹理特征可以为后续良恶性分类提供健壮的信息表达。
第四步:脑肿瘤良恶性分类。由于支持向量机SVM 方法具有高维空间下的非线性映射能力,以及不涉及概率演算复杂性的优势[9],所以此处采用SVM 方法对脑肿瘤良恶性进行分类识别。
4 实验结果及分析
为了验证算法的效果,本文在Intel 奔腾Xeon4 1.8G CPU,32G 内存配置的计算机及Ubuntu 64 位操作系统平台上进行实验。为与临床诊断实际需求一致,样本尽可能处于同一年龄段,病例图像全部选自30-60岁之间人群。并且选取良性脑膜瘤、恶性胶质瘤各30例,男女比例为1:1,在CT 和MR 设备上,每个病例产生74 张DICOM 图像序列图像以供分析。

图1 良性CT图像肿瘤分割结果
从实验视觉效果上来看,图1(b)和图2(b)显示出了公共稀疏重构误差的超像素区块,并且肿瘤处具有最亮的目标显著度。并且可以看出本文方法比仅利用单帧图像的方法能够更好地勾画出肿瘤区域。
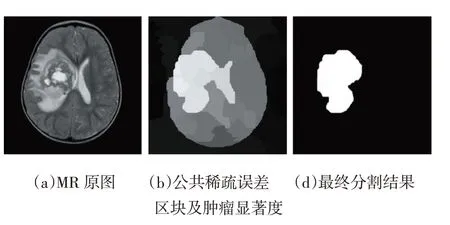
图2 恶性MR图像肿瘤分割结果
为了评估算法的良恶性分类整体性能,本文与基于统计学习的方法和基于主动轮廓的分割方法进行对比,然后在它们分割的基础上用SVM 分类器进行分类。此处采用5 个评价指标,准确率(ACC)、敏感性(SEN)、特异性(SPE)、正预测值(PPV)和负预测值(NPV)。其中正确分类和错误分类的恶性肿瘤数为TP和FN,正确分类和错误分类的良性肿瘤数为TN 和FP,计算公式如下:

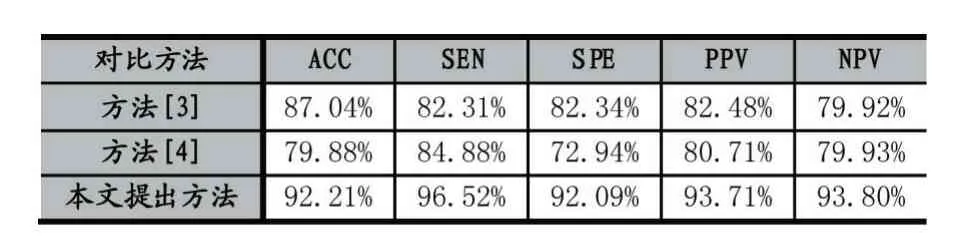
表1 算法性能对比结果
通过表1 所描述的量化对比数据可以看出,相对于基于统计方法分割的识别方法和基于主动轮廓模型分割的识别方法,本文提出的基于序列公共稀疏误差对称差异度的肿瘤精确分割方法具有更好的识别准确率。
5 结语
本文针对单帧脑肿瘤区域图像信号存在缺失的问题进行研究,提出了一种基于序列公共稀疏误差对比差异度的脑肿瘤显著性分割算法。实验表明,该方法能够获得更加准确的脑肿瘤区域,并且在此基础上利用似圆度等4 种无量纲健壮形状特征对脑肿瘤进行良恶性分类时,本文方法也具有更好的分类效果。
