利用基因芯片技术研究缺氧/复氧条件下抑制miR-132表达对人视网膜微血管内皮细胞中基因表达的影响
2019-10-08张枥心
张枥心
(湖南省儿童医院眼科 长沙 410006)
视网膜新生血管(retinal neovascularization, RNV)是一种发生在视网膜的病态血管增生,它的发生与多种疾病相关联,包括视网膜病变、视网膜静脉阻塞和年龄相关性黄斑变性[1-2]。有证据[3-4]表明,缺血、缺氧是导致RNV的主要原因,但潜在的机制还不明确。RNV是一个多步骤的复杂过程,涉及各种血管生成因素的复杂相互作用,包括血管生成因子、炎性细胞因子、趋化因子和生长因子等;另外,细胞外基质的变化也会影响RNV[5]。系统地研究缺血、缺氧导致RNV的分子机制,将有助于寻找有效治疗RNV的手段。
microRNA(miRNAs,miR)是一类长度为17~25个核苷酸的短链非编码RNA。miRNAs通过结合到mRNA的3′非翻译区(3′-UTR)来调控mRNA的稳定性和(或)翻译,从而调节靶基因的表达[6]。miRNAs与RNV的关联在近期的一些文献中可见报道,例如Yang等[7]报道miR-181a通过下调血管内皮生长因子(vascular endothelial growth factor, VEGF)的表达抑制了小鼠模型中RNV;在人视网膜微血管内皮细胞(human retinal microvascular endothelial cells, HRMEC)中,miR-200b/c通过下调VASH2抑制了HRMEC的增殖和迁移,因此推测miR-200b/c有可能抑制血管生成[8];Wang等[9]发现miR-370通过抑制KDR基因的表达而抑制视网膜毛细内皮细胞的生长和诱导凋亡,预示其可能在治疗RNV中发挥作用;Chen等[10]发现miR-29a通过影响小鼠模型中的血管紧张素原的表达,从而抑制了早产儿视网膜病变中的RNV。众多研究指出miR-132对血管形成发挥重要作用[11-13],并且缺氧能显著上调miR-132的表达水平[14],但是miR-132参与血管形成的机制,尤其是影响RNV形成的机制尚不完全清楚。
基因芯片检测主要是指用来检测基因组DNA和mRNA差异的一种生物技术装置,特点是可以快速同时检测大量的目标基因,检测通量远远大于传统Southern印迹、Northern印迹和定量聚合酶链反应(quantitative polymerase chain reaction, qPCR)等检测手段[15]。
基于mRNA基因芯片检测快速和超高通量的优势,本实验采用基因芯片技术研究缺氧/复氧(hypoxia/reoxygenation,H/R)条件下抑制miR-132表达对HRMEC中基因表达水平的影响。研究结果将有助于进一步了解miR-132调节RNV的机制。
1 材料与方法
1.1 细胞的培养 HRMEC购于Cell Systems公司(Kirkland, WA, 美国)。HRMEC培养于添加了10%胎牛血清(Life TechnoLogies, Carlsbad, CA, 美国)和内皮细胞生长添加剂(EGM SingleQuots; Lonza, Basel, 瑞士)的内皮基础培养液-2(EBM-2; Lonza, Walkersville, MD, 美国)中。细胞的培养环境为加湿的含5% CO2的细胞培养箱。培养液每3 d更换1次。对HRMEC做任何干预前的24 h把培养液换成不添加胎牛血清和生长添加剂的培养液。
1.2 细胞的处理 将HRMEC分成3个组,分别进行以下干预。①HRMEC细胞对照组:正常培养状态下的细胞;②HRMEC细胞+H/R处理组:将HRMEC暴露于一个产生缺氧的密封腔中(0.5% O2),时长为6 h,然后将细胞培养在常氧条件下(复氧),时长为6 h;③HRMEC细胞+H/R处理+miR-132抑制剂组:先使用miR-132抑制剂(终浓度为100 nmol/L)和转染试剂Lipofectamine 2000(Invitrogen Life TechnoLogies,美国)瞬时转染HRMEC,再对上述瞬时转染的HRMEC进行如“②HRMEC+H/R处理组”的干预。
1.3 RNA提取和质检 使用RNA提取试剂盒Rneasy MiNi Kit(Qiagen, CA, 美国)从HRMEC提取总RNA。RNA质控:先使用总RNA质检试剂盒Eukaryote Total RNA Nano(Agilent)处理总RNA,再在生物分析仪2100(Agilent,美国)上进行RNA质控检测。质控指数采用RNA完整性数值(RIN)。
1.4 基因芯片检测和分析 基因的差异表达分析使用mRNA微整列(HOA 7.1)实施。该芯片共包含29 204个基因。芯片参考国际公认的RefSeq 及 Ensembl 序列资料库,设计长链为60个碱基寡核酸的人类表达谱基因探针,内容以蛋白质编码基因为主。对RNA样本标记染料分子Cy5,然后将RNA分子杂交到mRNA阵列上。杂交后的阵列使用Agilent 0.1 XDR (Phalanx Biotech, 中国台湾地区)扫描,接着使用一种专门化的微阵列数据储存和分析的软件包Rosetta Resolver 7.2 (Merck & Co., Inc, NJ, 美国)进行功能富集分析(gene ontology,GO)。表达谱的聚类分析和可视化使用无监督的层次聚类分析法进行(Ernest Orlando Lawrence Berkeley National Laboratory, Berkeley, CA, 美国)。
1.5 定量反转录聚合酶链反应(qRT-PCR)检测 使用RNA提取试剂盒Rneasy MiNi Kit从HRMEC提取总RNA。采用反转录试剂盒(Qiagen,CA,美国)将RNA反转录为cDNA,然后采用PCR荧光定量试剂盒(Qiagen,CA,美国)检测cDNA中特定基因的表达水平。引物信息见表1。
1.6 数据分析 采用软件GraphPad Prism 5(Graphpad Software, Inc., La Jolla, CA, 美国)进行数据分析,数据用“均数±标准差”表示。用附带Bonferronit检验的单因素方差分析来分析数据,P<0.05为差异有统计学意义。对于GO分析的结果,仅列举BvsA的前10位集群数据,然后对GO分析中排行第1位集群数据中基因表达差异>64倍的基因进行局部深度数据挖掘分析。
2 结果
2.1 对基因探针在各分组中信号差异分布的检测结果
2.1.1 柱状图分析 图1显示所有检出的基因探针(不含控制探针及 flagged 探针)于分组比对中信号差异的分布。由图1可见:各组间比较的信号差异成正态分布,属于正常。
2.1.2 火山图分析 图2显示分组比对中差异基因探针的分布。由图2可见,分组比对中差异基因探针的分布正常。
2.1.3 差异表达基因的数量统计 对每组对比的差异基因数目进行了统计,每一项的变化基因数目都在2 000左右,提示需要在下一步分析中进行富集处理。
2.1.4 主成分分析 主成分分析用于评估生物重复及处理条件间的整体差异。图3表明:第1主成分是H/R处理,第2主成分是加入miR-132抑制剂,变量百分值分别为 53.11%和46.89%。

图1. 基因探针在各分组中信号差异分布,差异倍数柱状图
图2. 分组比对中差异基因探针的分布,差异表达基因的筛选阈值为差异倍数≥1且P<0.05,差异探针标示为蓝色点

图3. 主成分分析图
2.2 对基因探针在各分组中信号的聚类分析结果 本分析采用无监督层次聚类分析(unsupervised hierarchical clustering analysis)来分析实验样本间整体基因表达的相似性。由图4可见,B组和C组的相似性大于B组和A组的相似性以及C组和A组的相似性,符合预期。
2.3 对基因探针在各分组中信号的GO分析结果 BvsA的分子功能(MF)的总体分析结果显示,排行前3位的集群依次是:GO:0046983~protein dimerization activity、GO:0008092~cytoskeletal protein binding和GO:0042802~identical protein binding。表2是BvsA分子功能具体基因的分析结果,列举了排行第1的集群中,表达量差异>64倍的基因[Log2 (B/A)>6或者Log2 (B/A)< -6]。

图4. 聚类分析
BvsA生物学过程(BP)的总体分析结果显示,排行前3位的集群依次是:GO:0001944~vasculature development、GO:0001568~blood vessel development和GO:0042127~regulation of cell proliferation。表3是BvsA生物学过程具体基因的分析结果,列举排行第1的集群中,表达量差异>64倍的基因[Log2 (B/A)>6或者Log2 (B/A)<-6]。

表2 B vs A分子功能的具体基因分析结果

表3 B vs A生物学过程的具体基因分析结果
BvsA细胞定位(CC)的总体分析结果显示,排行前3位的集群依次是:GO:0031012~extracellular matrix、GO:0005578~proteinaceous extracellular matrix和GO:0005604~basement membrane。表4是BvsA细胞定位具体基因的分析结果,列举排行第1的集群中,表达量差异>64倍的基因[Log2 (B/A)>6或者Log2 (B/A)<-6]。

表4 B vs A细胞定位的具体基因分析结果
2.4 富集GO分析结果的深度分析 对BvsA的数据进行GO-MF、GO-BP、GO-CC分析,从分析结果中选取表达量差异>64倍的基因[Log2 (C/B)>6或者Log2 (C/B)<-6];对于选取的基因,考察它们在CvsA和CvsB中表达量的差异状况。
表5~7展示了上述分析的结果,分别涉及GO-MF、GO-BP和GO-CC。表5表明:H/R上调了CDH13的表达,下调了TFAP2C和HPGD的表达,而miR-132是上述表达调控中“必需程度”接近100%的中间执行者;同时,H/R上调了TCF4的表达,下调了CEBPA的表达,而miR-132是上述表达调控中的中间执行者之一,还存在其他中间执行者。表6表明:H/R下调了IL18的表达,而miR-132是上述表达调控中“必需程度”接近100%的中间执行者;同时,H/R上调了CXCL8、ANGPTL4、PLAT、PLAU和THY1的表达,而miR-132是上述表达调控中的中间执行者之一,还存在其他中间执行者。表7表明:H/R上调了COL17A1、TNC和MMP14的表达,而miR-132是上述表达调控中“必需程度”接近100%的中间执行者;H/R上调了GPC6、COL5A2和VCAN的表达,下调了DCN的表达,而miR-132完全不是上述表达调控中的中间执行者;同时,H/R上调了ANGPTL4的表达,下调了PI3的表达,而miR-132是上述表达调控中的中间执行者之一,还存在其他中间执行者。
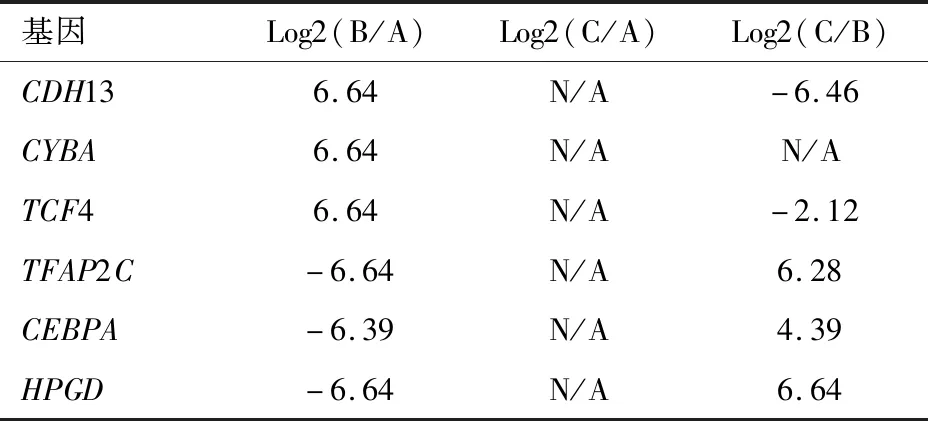
表5 对B vs A的GO-MF数据局部深度挖掘的分析结果
注:N/A示无

表6 对B vs A的GO-BP数据局部深度挖掘的分析结果
注:N/A示无
2.5 RT-qPCR对于基因芯片等检测结果的验证 图5示,H/R显著上调了miR-132的转录表达,而H/R+anti-miR-132逆转了上述作用;图6示,H/R显著下调了IL-18和PI3的转录表达,而H/R+Anti-miR-132逆转了上述作用;H/R显著上调了ANGPTL4、PI3、COL17A1、MMP14、CXCL8、PLAT、PLAU、THY1、GPC6、VCAN的转录表达,而H/R+anti-miR-132逆转了上述作用。

表7 对B vs A的GO-CC数据局部深度挖掘的分析结果
注:N/A示无

图5. H/R和H/R+anti-miR-132对miR-132转录表达的影响 与对照组比较,**示P<0.01;Con为对照组,H/R为H/R组,H/R+Inh为H/R+anti-miR-132组

图6. H/R和H/R+anti-miR-132对IL-18、PI3、ANGPTL4、PI3、COL17A1、MMP14、CXCL8、PLAT、PLAU、THY1、GPC6、VCAN转录表达的影响 与对照组比较,*示P<0.05,**示P<0.01,***示P<0.001; 与H/R组比较,#示P<0.05,##示P<0.01,###示P<0.001;Con为对照组,H/R为H/R组,H/R+Inh为H/R+anti-miR-132组
2.6 查找与RNV密切关联的基因 对于“2.4对富集GO分析结果的局部深度数据挖掘分析”圈定的基因,结合NCBI Pubmed数据库进行了深度挖掘,进一步在其中圈定了3个与RNV密切关联的基因,它们分别是IL18、ANGPTL4和PI3。
根据2.4和2.5两步局部深度数据挖掘的结果,推演出了在H/R条件下miR-132调节RNV的分子机制图(图7)。

图7. 在H/R条件下miR-132调控 RNV的分子机制图
3 讨论
基因芯片检测产生了大量数据。在B/A中,上调基因数目为2 207,下调基因数为2 482。这表明缺氧和后续的复氧导致大量基因表达水平的改变。在C/B中,上调基因数目为1 989,下调基因数为1 894。由此可见在H/R条件下抑制miR-132也可以影响众多基因的表达。通过对比B/A和C/B中的差异基因,发现很多相同的基因。这表明miR-132介导了H/R对部分基因的调节。我们对B/A的数据进行了GO-MF、GO-BP、GO-CC分析,并重点分析了结果中差异最显著的基因,即表达量差异>64倍的基因[Log2 (C/B)>6或者Log2 (C/B)<-6]。生物学过程分析结果中排行前3位的集群依次是:GO:0001944~vasculature development、GO:0001568~blood vessel development和GO:0042127~regulation of cell proliferation。这些生物学过程均与RNV有密切联系。由此可见,缺氧和后续的复氧主要影响了血管的生成。
我们进一步考察了B/A中差异最显著的基因在C/A和C/B中的变化状况。H/R上调了CDH13、COL17A1、TNC、MMP14的表达,下调了TFAP2C、HPGD、IL18的表达,miR-132是上述表达调控中“必需程度”接近100%的中间执行者。H/R上调了TCF4、CXCL8、ANGPTL4、PLAT、PLAU、THY1、GPC6、COL5A2和VCAN的表达,下调了CEBPA、DCN、PI3的表达,miR-132是上述表达调控中的中间执行者之一,还存在其他中间执行者。这些结果表明miR-132介导了H/R对这些基因的调节。
通过在NCBI Pubmed数据库进行查询,发现3个与RNV密切关联的基因,它们分别是IL18、ANGPTL4和PI3。对于IL18,Singh等[15]发现血管增生促进物同型半胱氨酸处理视网膜色素细胞(RPE)后,IL18的表达被下调,提示IL18可能是RNV的抑制物;Doyle等[16]报道玻璃体内注射IL18能治疗恒河猴的年龄相关性黄斑退化,提示IL18可以作为抗VEGF治疗的辅助物,在抑制RNV中发挥作用;Shen等[17]报道IL8具有抗渗透和抗血管生成的作用,与VEGF之间存在相互抑制。在本研究中,发现H/R能显著下调IL18的表达,与上述报道相符,而且我们进一步创新性地发现miR-132是这一过程的“中间执行者”,具有100%的“必需程度”。对于ANGPLT4,Jee等[18]报道了在增生性镰状视网膜病变(proliferative sickle retinopathy, PSR)中,ANGPTL4显著上调,提示其在RNV过程中的促进作用,并且是治疗PSR的潜在靶点;Babapoor-Farrokhran等[19]报道在增生型糖尿病视网膜病变(PDR)中,ANGPTL4是强烈促进血管生成的促进物;Yokouchi等[20]报道了ANGPTL4能在体外促进视网膜内皮细胞的血管生成活性。在本研究中,发现H/R能显著上调ANGPTL4的表达,与上述报道相符,而且进一步创新性地发现miR-132是这一过程的“中间执行者”,具有<100%的“必需程度”,与“其他中间执行者”共同上调ANGPTL4的表达。对于PI3,Alvarez等[21]报道抑制PI3可以抑制视网膜增生。在本研究中,发现H/R能显著下调PI3的表达,我们进一步创新性地发现miR-132是这一过程的“中间执行者”,具有<100%的“必需程度”,与“其他中间执行者”共同下调PI3的表达。
COL17A1、MMP14、CXCL8、PLAT、PLAU、THY1、GPC6、VCAN通过调节血管内皮细胞的活力、黏附能力和细胞外基质的重构对血管形成发挥了重要作用。miR-132通过调节这些基因有可能影响RNV。
综上所述,通过基因芯片检测发现miR-132介导了H/R对部分基因的调节,其中部分基因有可能参与RNV的发生、发展,为进一步阐明miR-132调节RNV的机制提供了研究基础。
