长江刀鲚选育群体转录组EST-SSR的分布特征分析*
2019-09-27于爱清施永海徐嘉波陆根海张海明谢永德刘永士
于爱清 施永海 徐嘉波 陆根海 张海明 谢永德 刘永士
长江刀鲚选育群体转录组EST-SSR的分布特征分析*
于爱清 施永海①徐嘉波 陆根海 张海明 谢永德 刘永士
(上海市水产研究所 上海市水产技术推广站 上海 200433)
本研究利用MISA软件挖掘长江刀鲚()肌肉和肝脏转录组中的微卫星标记,为刀鲚选育群体的种质资源评估和分子标记辅助育种奠定基础。结果显示,从71869条Unigenes中共获得33896条重复单元长度为1~6碱基的微卫星序列;刀鲚转录组中不同类型微卫星的重复基序具有不同的分布特征,其中,单核苷酸重复、二核苷酸重复和三核苷酸重复为主要的微卫星重复类型,分别占总微卫星数目的34.94%、49.47%和13.34%;不同微卫星重复类型的优势重复基序亦有所不同,其中,A/T为单核苷酸重复基序的优势重复基序占86.25%,AC/GT为二核苷酸重复基序的为优势重复基序占75.25%,AGG/CCT为三核苷酸重复基序的优势重复基序占28.57%;不同微卫星重复基序核苷酸的数量和重复次数亦有所不同,重复次数伴随着重复单元中核苷酸数量的增加而呈现降低的趋势;从100对四核苷酸重复的SSR引物中筛选获得了16对多态性微卫星标记,并以此为基础,对长江刀鲚选育群体(F3)的遗传学特征进行了初步评估,结果显示,长江刀鲚选育群体F3的平均有效等位基因数(N)、平均观测杂合度(H)、平均期望杂合度(H)和Shannon多样性指数分别为1.7580、0.3414、0.3977和0.6278。以上结果表明,基于刀鲚转录组数据批量开发微卫星是切实可行的,所开发的多态性微卫星标记能够应用于长江刀鲚选育群体的遗传背景评估和进一步的遗传育种研究。
刀鲚;转录组;微卫星;分子标记
刀鲚()隶属于鲱形目(Clupeiformes)、鳀科(Engraulidae)、鲚属(),又名长颌鲚,俗称刀鱼、毛刀鱼,主要分布于我国黄渤海、东海海域及通海的江河,其中,又以长江下游的产量最高,因其鱼体丰腴肥满、肉质细嫩鲜美而享有“长江三鲜”之首的美誉(施永海等, 2014、2015; 邓平平等, 2014)。历史上,我国刀鲚的种质资源极为丰富。然而,近年来,由于酷捕滥渔、水域环境污染、水文条件改变、生态环境恶化及海岸工程建设等诸多因素,长江刀鲚自然种质资源急剧衰退,产量波动很大,且呈现出逐年降低且个体日趋小型化的趋势,甚至不能形成优势种群(施永海等, 2014、2015; 魏广莲等, 2013; 袁传宓, 1988),亟需对刀鲚的种质资源进行保护和恢复。而刀鲚基因组信息匮乏、遗传背景不清晰、繁殖周期较长等问题亦在一定程度上制约了其种质资源的开发和恢复。刀鲚种质资源的有效保护和恢复,除了政府相关部门采取相关行政干预(建立禁渔期、加强捕捞监管、适时增殖放流等)之外,还需相关部门适时地监测洄游型刀鲚的遗传多样性,从整体上把握刀鲚野生群体的遗传多样性水平和遗传背景,同时加快刀鲚良种的选育进程,特别是分子标记辅助良种选育的研究。
分子标记由于能够对动物整个基因组的遗传变异进行探究,且相对稳定,而被应用于水产动物的遗传育种研究,其中,微卫星标记(Microsatellite)的应用最为广泛。微卫星标记,又称为简单重复序列(Simple sequence repeat, SSR),是指由1~6个核苷酸组成的简单串联重复序列,这些序列的重复单位和重复次数的差异使其具有高度变异性和多态性,被广泛应用于水产动物种质资源评估和鉴定、遗传图谱的构建及分子标记辅助育种等方面(杨铭等, 2017; 管奥等, 2018; 白翠翠等, 2016)。近年来,学者利用线粒体基因D-loop(张燕萍等, 2017; Cheng, 2011)、线粒体Cytb (魏广莲等, 2012; Ma, 2010)、RAPD (马春艳等, 2004)、AFLP(葛家春等, 2008)等方法研究了刀鲚不同群体的遗传结构和遗传差异,而对刀鲚微卫星标记开发的报道相对较少。Rong等(2013)利用FIASO法构建了微卫星富集文库方法,从89对刀鲚微卫星引物中获得了20对多态性微卫星标记。马春燕等(2011)利用FIASO法构建微卫星富集文库的方法,从70对刀鲚微卫星引物中获得了12对多态性微卫星标记。陈芳等(2012)利用磁珠富集法,从198对刀鲚微卫星引物中获得了34对多态性微卫星标记。邓平平等(2014)利用磁珠富集法,从59对刀鲚微卫星引物中获得了9对多态性微卫星标记。利用磁珠富集法从水产动物基因组中开发的微卫星标记,多态性相对较高,但相对费时费力,效率相对较低,且标记大规模的开发成本相对较高,远远不能满足刀鲚高密度遗传图谱的构建、QTL精细定位和分子标记辅助育种等对分子标记需求量较大的研究,亟需大规模开发分子标记。而高通量转录组测序技术所开发的微卫星,又被称为表达序列标签SSR (Expressed sequence tag SSR, EST-SSR),其多态性虽然相对低于基因组所开发的微卫星(G-SSR),但由于数量多、通量大、周期短、成本低,且直接与某些功能基因相关联(李东明等, 2017),而被广泛应用于水产动物分子标记大规模的开发研究。
本研究基于刀鲚肌肉和肝脏的高通量转录组数据,对长江刀鲚转录组中的微卫星位点进行筛选,分析刀鲚EST-SSR的序列特征、分布和组成等信息,并利用所筛选的多态性微卫星标记对长江刀鲚选育群体(F3)的遗传学特征进行初步评估,以期能够为刀鲚遗传多样性的分析、种质资源的保护和利用、高密度遗传连锁图谱的构建及分子标记辅助良种选育奠定基础。
1 材料与方法
1.1 实验材料与数据来源
实验所用长江刀鲚来自于上海市水产研究所核心选育群体(F3)的2龄刀鲚,提取刀鲚肌肉和肝脏的RNA后,构建高通量转录组测序文库,利用Illumina HiSeq4000进行高通量转录组测序。刀鲚肌肉和肝脏的转录组测序总共获得28.20 Gb的数据量,原始测序数据经Trinity软件(http://trinityrnaseq.sourceforge. net/)组装并去冗余后,获得平均长度为932.49 bp的Unigenes 71869条,N50达到1364 bp,质量不低于20的碱基比例(Q20)为97.31%,表明测序结果良好(未发表)。
1.2 EST-SSR的筛查
以刀鲚转录组中的Unigenes作为参考序列,利用MISA(MIcroSAtellite identification tool)软件(http: //pgrc.ipk-gatersleben.de/misa/misa.html)搜索刀鲚肝脏和肌肉转录组中的SSR位点,其中,单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸的最少重复次数分别设置10、6、5、5、5和5。复合SSR两个位点间最大间隔碱基数设置为100。
1.3 EST-SSR多态性引物的设计、筛选和初步验证
利用Primer Premier 3.0软件对筛选出的微卫星序列批量设计引物,主要设置参数为:引物长度和PCR产物分别为18~25 bp和100~500 bp,55℃≤退火温度(T)≤65℃,40%≤GC含量≤60%,上下游引物的T≤2℃。考虑到四碱基重复的微卫星标记相对二碱基重复和三碱基重复具有较长的演化历史和更加稳定的突变积累,本研究随机选取100对基于四核苷酸重复微卫星序列所设计的PCR扩增引物交由生工生物工程(上海)股份有限公司合成,并以刀鲚核心选育群体(F3)为实验材料筛选具有多态性的微卫星引物。PCR扩增产物经10%非变性聚丙烯酰胺凝胶电泳检测后,利用银染法显色定影,条带显色清晰后用数码相机拍照保存,进行微卫星引物后续的多态性分析。利用GenAlEx 6.0软件(Peakall, 2006)计算长江刀鲚选育群体的有效等位基因数(N)、观测杂合度(H)、期望杂合度(H)、Shannon多样性指数()等遗传多样性参数。参照Bostein等(1980)的计算公式计算多态信息含量(Polymorphism information content, PIC):
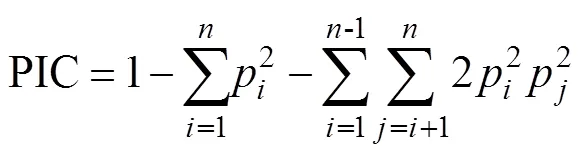
式中,P、P分别为群体中第、个等位基因频率,为等位基因数。
2 结果
2.1 刀鲚转录组中EST-SSR位点的数量与分布
利用MISA软件挖掘刀鲚转录组中的EST-SSR标记(搜索结果见表1)。从刀鲚71869条Unigenes中共获得33896个SSR位点,占所评估序列总数目的47.16%;22463条Unigenes包含SSR位点,占总Unigenes的31.25%;含有1个以上的SSR位点的Unigenes有7701个,约占总Unigenes的10.71%;具有复合型SSR位点的Unigenes有2989个,约占总Unigenes的4.16%(表1)。
2.2 刀鲚EST-SSR的基序类型和频率特征
基于微卫星重复单元的类型和重复次数,将不同的SSR位点进行分类(表2)。结果显示,从单核苷酸重复SSR到六核苷酸重复SSR在刀鲚转录组中均有分布,但数量分布上具有不同程度的差异。其中,二核苷酸重复SSR数量最多,占SSR位点总数的49.47%;单核苷酸重复SSR数量次之,占SSR位点总数的34.94%;三核苷酸重复SSR的数量亦较为丰富,占SSR总位点数的13.34%;四核苷酸重复SSR的数量相对较少,占SSR总位点数的2.11%;五核苷酸和六核苷酸重复SSR的数量最少,分别占SSR总位点数的0.09%和0.06%。
表1 刀鲚转录组中EST-SSR位点的搜索结果
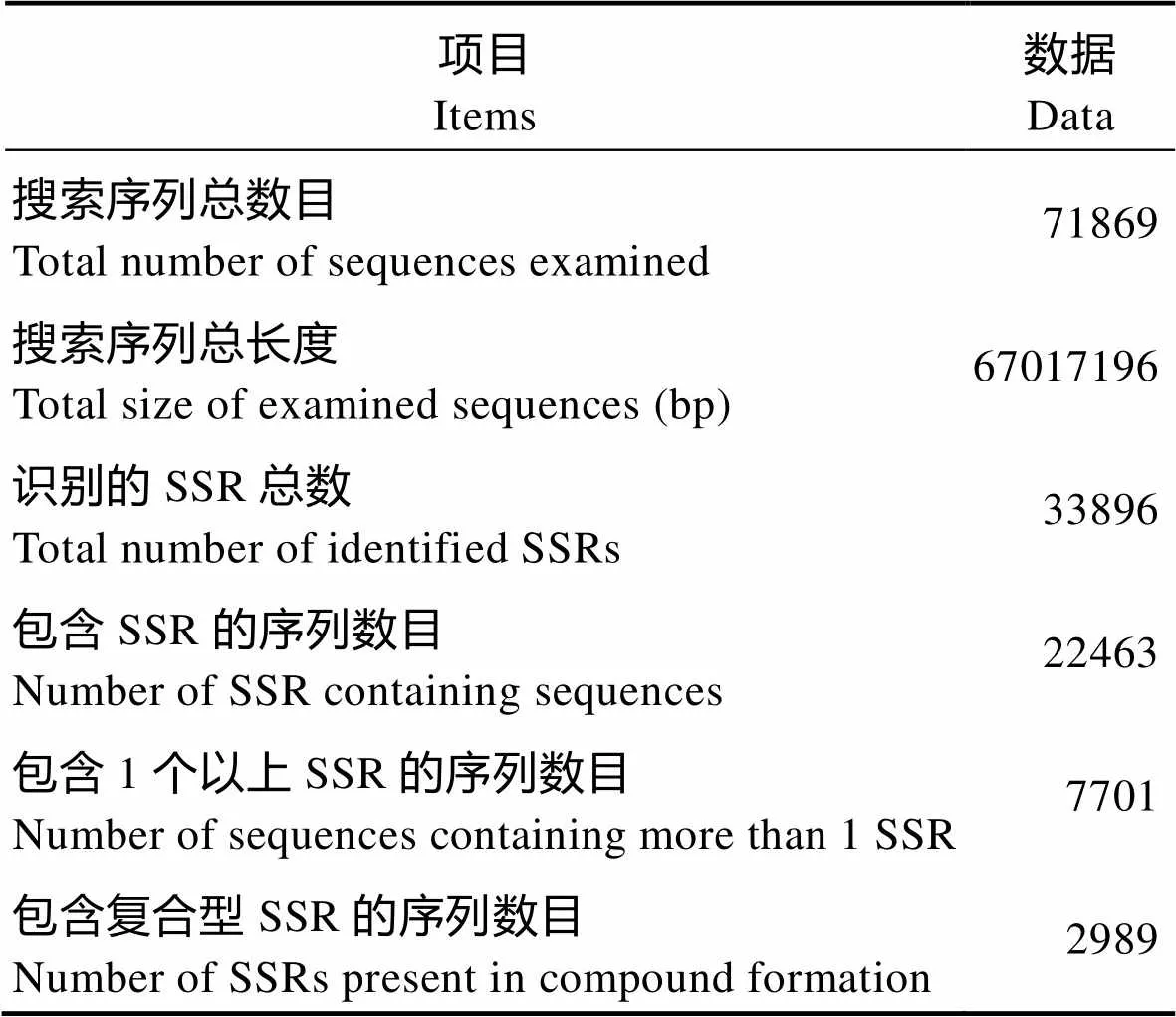
Tab.1 EST-SSR search results in the transcriptome of C. ectenes
此外,刀鲚转录组EST-SSR中共有94种核苷酸重复基序,主要核苷酸重复基序亦有不同类型的优势重复基序。其中,单核苷酸重复SSR的优势重复基序为A/T,占86.25%,这可能与Poly A被纳入计算结果有关;二核苷酸重复SSR的优势重复基序为AC/GT,占75.25%;三核苷酸重复SSR的优势重复基序为AGC/CTG和AGG/CCT,分别占27.16%和28.57%;四核苷酸重复SSR的优势重复基序为AAAC/GTTT和ACAG/CTGT,分别占17.51%和16.81%。
2.3 刀鲚EST-SSR不同基序重复次数分布
刀鲚SSR主要重复基序的重复次数的分布情况见表3。刀鲚SSR基序的重复次数大部分为5~24次,其他重复次数相对较少,仅ATC/ATG出现了45次重复。低重复次数(5~9次重复)的SSR位点最多,约占61.21%;中等重复次数(10~11次重复)的SSR位点次之,约占22.84%;而>11次重复的SSR位点仅占15.95%。其中,单核苷酸基序重复次数均≥10,且重复11次以上的最多,占45.60%;二核苷酸基序以低重复次数为主,占92.26%,重复11次以上的较少,仅占0.02%;三核苷酸至六核苷酸基序均以重复次数为5的最多,其余重复次数的重复基序相对较少。
2.4 刀鲚EST-SSR多态性引物的初步筛选和分析
以本单位核心选育群体F3随机选择的30尾鱼苗的基因组DNA为模板,针对100对四核苷酸重复SSR位点的引物进行多态性检验,最终筛选到16对具有多态性的微卫星引物(表4)。
基于16对经筛选获得的多态性微卫星标记,对长江刀鲚核心选育群体(F3)进行遗传多样性评估 (表5)。结果显示,长江刀鲚核心选育群体(F3)的平均有效等位基因数(N)、平均观测杂合度(H)、平均期望杂合度(H)和Shannon信息指数()分别为1.7580、0.3414、0.3977和0.6278。基于等位基因频率计算不同位点的多态信息含量,结果显示,16对多态性微卫星标记中,有2对属于高度多态,10对属于中度多态,4对属于低度多态。可见,基于刀鲚转录组开发的多态性EST-SSR能够应用于刀鲚选育群体的种质资源评估,具有较高实用性。
表2 刀鲚转录组中EST-SSR的主要重复基序的类型和数量
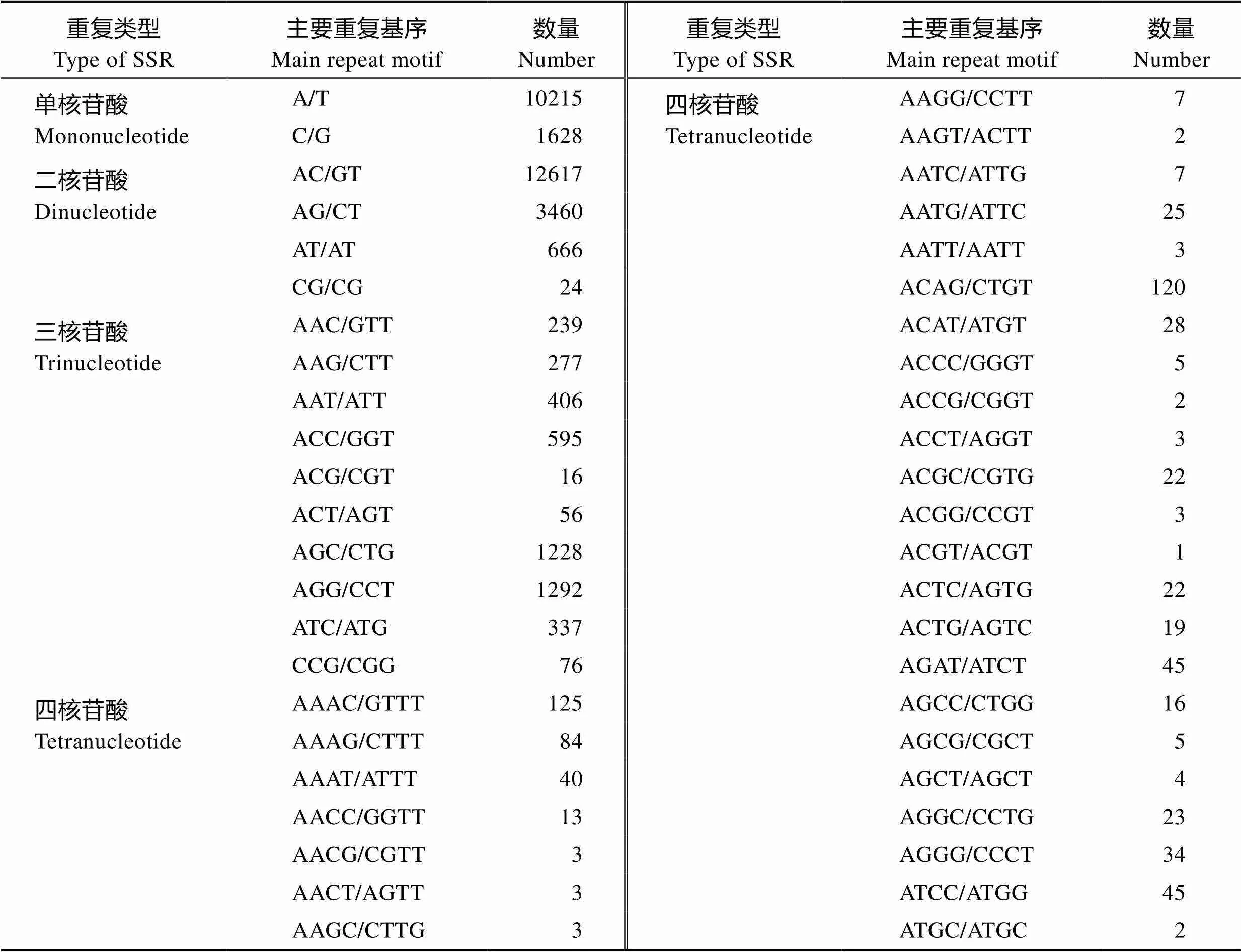
Tab.2 The type and number of main repeat motif of EST-SSR in the transcriptome of C. ectenes
表3 刀鲚转录组主要EST-SSR重复基序的重复次数
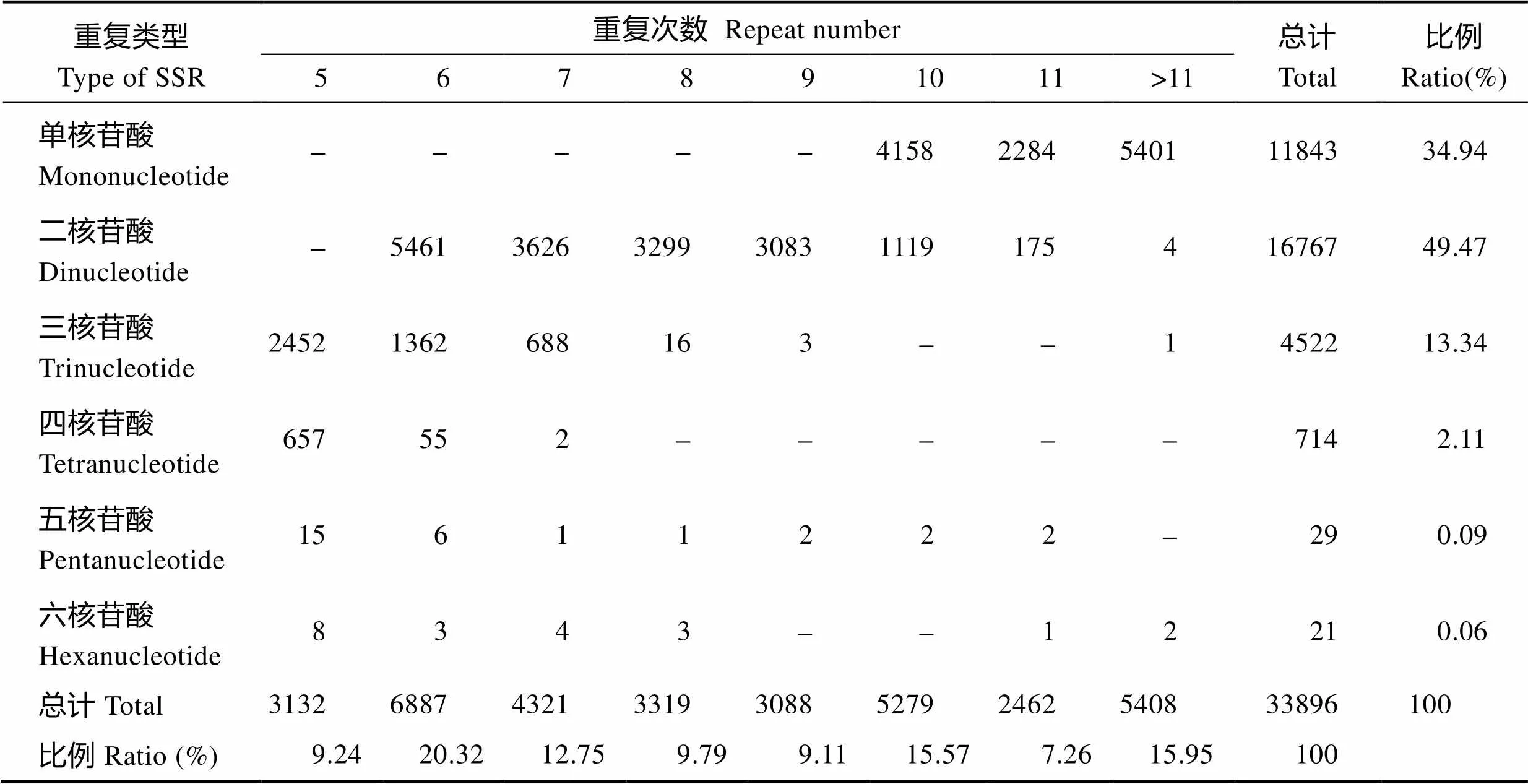
Tab.3 The repeat number of main repeat motif of EST-SSR in the transcriptome of C. ectenes
表4 刀鲚多态性微卫星引物
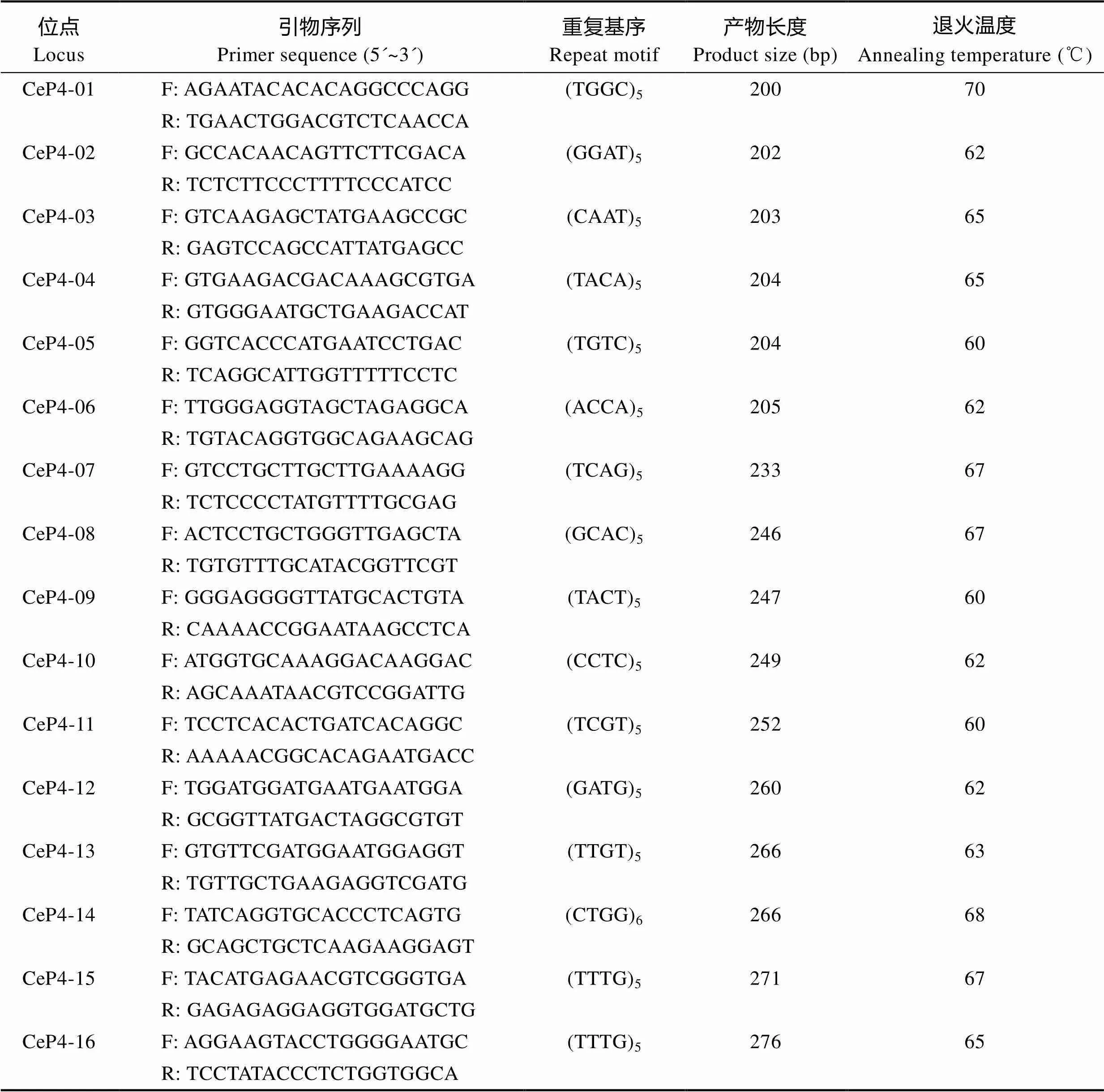
Tab.4 Polymorphic SSR primers of C. ectenes
3 讨论
微卫星分子标记在动物进化过程中,由于DNA分子的复制、缺失、滑移或错配及姐妹染色单体的不均交换等原因而具有高度变异性,被广泛应用于水产动物群体遗传结构分析(滕爽爽等, 2018)、亲子鉴定(张丹等, 2018)、遗传图谱构建(郭香等, 2013)、数量性状定位(顾颖等, 2016)和分子标记辅助育种(孙际佳等, 2017; 顾颖等, 2016)等方面的研究。对于具有基因组信息的物种而言,微卫星标记的大规模开发相对方便;而对于目前还没有基因组信息的物种,则可利用高通量转录组测序技术大规模开发微卫星标记。刀鲚作为我国具有较高经济、营养和养殖价值的江海洄游性鱼类,其基因组信息、转录组信息及可用分子标记资源均极为匮乏,有鉴于全基因组测序的成本相对较高,因此,亟需开展刀鲚的转录学研究以获得大量的可用分子标记。此外,我国长江流域的刀鲚产量最为丰富,相应的野生群体遗传多样性较高和资源分布较广,是开展刀鲚遗传育种研究的理想种质资源库。而本研究的刀鲚最初的繁育亲本为长江流域的野生群体,历经3代高强度的人工选育,亟需适时地利用合适的分子标记检测其遗传变异情况,以制定科学有效的育种策略。
表5 16个微卫星标记的遗传参数
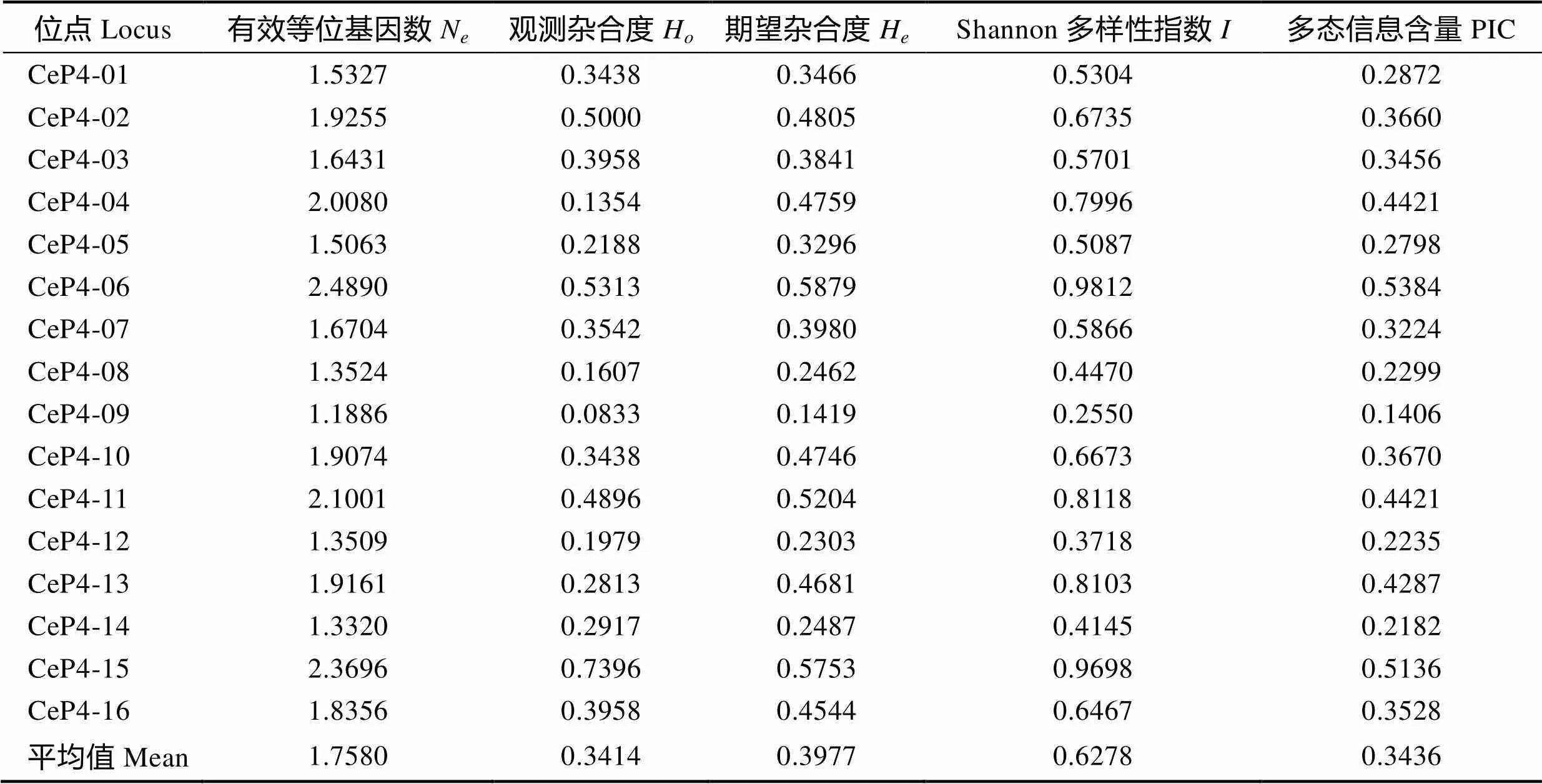
Tab.5 Genetic parameters at 16 microsatellite markers
本研究基于刀鲚转录组数据,利用生物信息学方法挖掘刀鲚EST-SSR,获得了33896条刀鲚EST-SSR,这些EST-SSR的种类较为丰富,且1~6核苷酸重复基序均有不同程度的分布。其中,以出现频率为49.47%的二核苷酸重复基序为主,其次是单核苷酸重复基序(34.94%),三核苷酸重复基序亦较为丰富,占13.34%,其他类型的核苷酸重复基序相对较少。蔡磊等(2015)研究表明,多数物种EST-SSR中的重复基序均以二核苷酸重复基序为主,这与本研究结果一致;而其余类型的优势重复基序则有较大的差异,这可能与不同物种转录组文库构建的组织来源、不同物种的种间特异性、位点突变频率及选择性进化机制有关。在刀鲚所有EST-SSR中,不同的核苷酸重复基序具有不同的重复次数,其中,单核苷酸重复基序大部分属于中高等重复次数,其余类型重复基序的大部分重复次数在5~9之间,相对低于其他鱼类基因组中的微卫星重复次数,可能与编码区和非编码区所受到的选择压力差异有关。不同重复基序的碱基优势重复类型表现出不同程度的偏倚性,单核苷酸重复基序以A/T类型为主,可能是由于富含A/T的微卫星退火温度相对较低,利于双链DNA的解链,通过DNA复制的重组和滑动机制而增加A/T重复基序的机率(倪守胜等, 2018)。而二核苷酸重复基序以AC/GT为主,这与鱼类基因组微卫星中以AC/GT重复为主相符;其余重复基序的优势重复碱基类型在不同物种间差异极大,可能与不同物种的种间差异性有关。此外,本研究发现,刀鲚EST-SSR重复单元的拷贝次数随着重复单元长度的增加而下降,微卫星的数量亦呈现出逐步减少的趋势。
近年来,相关学者在鱼类中利用高通量转录组测序技术陆续开发出了多态性较高的微卫星标记。蔡磊等(2015)利用高通量转录组测序技术从诸氏鲻虾虎鱼()肝脏转录组中获得6225个微卫星位点,并从76对引物中筛选获得32对具有多态性的微卫星标记。龚诗琦等(2016)利用高通量转录组测序技术从黄姑鱼()转录组获得12254个微卫星标记,并从80对引物中筛选获得18个具有多态性的微卫星标记。岳华梅等(2016)利用高通量转录组测序技术从兴国红鲤()的垂体和性腺转录组中获得13652个微卫星标记,并从30对引物中筛选获得了9个具有多态性的微卫星标记。本研究从100对四碱基重复的微卫星引物中筛选获得了16对具有多态性的微卫星标记,引物多态率相对低于其他鱼类的研究结果。造成多态性较低的原因,一方面是由于本研究选取的四碱基重复的微卫星相对于二碱基和三碱基重复的微卫星比较保守,且变异率低;另一方面是由于基于转录所获得的EST-SSR扩增产物可能跨越内含子或者PCR引物位于内含子和外显子的结合处,而造成扩增失败导致多态率较低。此外,筛选多态性引物所用样本的种类、数量、遗传差异程度以及不同物种的DNA序列的保守性对于微卫星位点多态性的筛选亦有不同程度的影响。
基于Botstein等(1980)对于多态信息含量(PIC)的评估标准,当PIC>0.5、0.25
本研究利用生物信息学方法筛选刀鲚高通量转录组Unigenes中的微卫星标记,分析刀鲚EST-SSR分布和组成特征,并利用所开发的多态性微卫星标记初步分析了刀鲚核心选育群体(F3)的遗传特征。研究结果不仅丰富了刀鲚分子标记的数量,而且对刀鲚后续选育群体的育种效果评估及分子标记辅助育种实践具有重要的指导意义。
Bai CC, Liu SF, Zhuang ZM. Characteristic analysis of microsatellite DNA in the genome of Gobiidae. Progress in Fishery Sciences, 2016, 37(5): 9–15 [白翠翠, 柳淑芳, 庄志猛. 虾虎鱼科(Gobiidae)基因组微卫星DNA的分布特征. 渔业科学进展, 2016, 37(5): 9–15]
Botstein D, White RL, Skolnick M,. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314–331
Cai L, Yu LJ, Chen XQ,. A preliminary screening and characteristic analysis of microsatellite markers from transcriptome sequences in. Biotechnology Bulletin, 2015(9): 146–151 [蔡磊, 余露军, 陈小曲, 等. 诸氏鲻虾虎鱼转录组序列中微卫星标记的初步筛选及特征分析. 生物技术通报, 2015(9): 146–151]
Chen F, Zeng LY, Cheng QQ. Development of thirty-four novel polymorphic microsatellite markers in(Clupeiformes: Engraulidae) and cross-species amplification in two closely related taxa. Journal of Genetics, 2012, 91(1): 1–7
Cheng QQ, Zhang QY, Ma CY,. Genetic structure and differentiation of four lake populations of(Clupeiformes: Engraulidae) based on mtDNA control region sequences. Biochemical Systematics and Ecology, 2011, 39(4–6): 544–552
Deng PP, Shi YH, Zhang GY,. Isolation of microsatellite inby magnetic beads. Biotechnology Bulletin, 2014(6): 145–149 [邓平平, 施永海, 张根玉, 等. 磁珠富集法分离刀鲚微卫星标记. 生物技术通报, 2014(6): 145–149]
Ge GC, Cao T, Chen CJ,. Analysis of genetic diversity based on amplified fragment length polymorphism fingerprint offrom Yangtze River. Journal of Nanjing University (Natural Sciences), 2008, 44(3): 332–338 [葛家春, 曹廷, 陈婵娟, 等. 利用扩增片断长度多态性技术分析长江刀鲚的遗传多样性. 南京大学学报(自然科学版), 2008, 44(3): 332–338]
Gong SQ, Wang ZY, Xiao SJ,. Development and verification of SSR based on transcriptome of yellow drum,. Journal of Jimei University (Natural Science), 2016, 21(4): 241–246 [龚诗琦, 王志勇, 肖世俊, 等. 黄姑鱼转录组SSR的开发与验证. 集美大学学报(自然科学版), 2016, 21(4): 241–246]
Gu Y, Lu CY, Zhang Q,. Analysis of genetic diversity and growth traits inusing microsatellite QTL markers. Freshwater Fisheries, 2016, 46(4): 9–18 [顾颖, 鲁翠云, 张芹, 等. 微卫星QTL标记分析豫选黄河鲤群体遗传结构及生长性状相关性. 淡水渔业, 2016, 46(4): 9–18]
Guan A, Wu YT, Chen Y,. Deep sequence-based transcriptome analysis of microsatellites in the cuttlefish (). Progress in Fishery Sciences, 2018, 39(3): 144–151 [管奥, 毋玉婷, 陈宇, 等. 曼氏无针乌贼转录组微卫星特征分析. 渔业科学进展, 2018, 39(3): 144–151]
Guo X, Li Q, Kong LF,. Microsatellite-based consensus map of the Pacific oyster (). Journal of Fisheries of China, 2013, 37(6): 823–829 [郭香, 李琪, 孔令锋, 等. 基于微卫星标记整合长牡蛎遗传图谱. 水产学报, 2013, 37(6): 823–829]
Li DM, Yang AG, Wu B,. Development and application of the EST-SSR markers in. Progress in Fishery Sciences, 2017, 38(2): 137–142 [李东明, 杨爱国, 吴彪, 等. 栉江珧()EST-SSR标记的开发与应用. 渔业科学进展, 2017, 38(2): 137–142]
Ma CY, Cheng QQ, Zhang QY. Genetic variation of(Clupeiformes: Engraulidae) revealed by the complete cytochrome b sequences of mitochondrial DNA. Journal of Experimental Marine Biology and Ecology, 2010, 385(1): 14–19
Ma CY, Cheng QQ, Zhang QY. Development of 12 polymorphic microsatellite markers inJordan and Seale, 1905 (Clupeiformes: Engraulidae) and cross-species amplification inLinnaeus, 1758. Environmental Biology of Fishes, 2011, 91(2): 243–249
Ma CY, Liu M, Ma LB,. Genetic diversity inby RAPD analysis. Marine Fisheries Research, 2004, 25(5): 19–24 [马春艳, 刘敏, 马凌波, 等. 长江口刀鲚遗传多样性的随机扩增多态DNA(RAPD)分析. 海洋水产研究, 2004, 25(5): 19–24]
Ni SS, Yang Y, Liu SF,. Microsatellite analysis ofusing next-generation sequencing method. Progress in Fishery Sciences, 2018, 39(1): 107–113 [倪守胜, 杨钰, 柳淑芳, 等. 基于高通量测序的虾夷扇贝基因组微卫星特征分析. 渔业科学进展, 2018, 39(1): 107–113]
Peakall R, Smouse PE. GENALEX 6: Genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Resources, 2006, 6(1): 288–295
Rong XJ, Xu YJ, Wang QY,. Isolation and characterization of polymorphic microsatellite markers from. Genetics and Molecular Research, 2013, 12(4): 6011–6017
Shi YH, Zhang GY, Zhang HM,. Analysis and comparison of nutritional quality of muscle fromFED formulated feed and live feed. Chinese Journal of Animal Nutrition, 2014, 26(2): 427–436 [施永海, 张根玉, 张海明, 等. 配合饲料和活饵料喂养刀鲚肌肉营养品质分析与比较. 动物营养学报, 2014, 26(2): 427–436]
Shi YH, Zhang GY, Zhang HM,. Complete artificial propagation and embryonic development of. Journal of Shanghai Ocean University, 2015, 24(1): 36–43 [施永海, 张根玉, 张海明, 等. 刀鲚的全人工繁殖及胚胎发育. 上海海洋大学学报, 2015, 24(1): 36–43]
Sun JJ, Li GF, Liu L,. Analysis of microsatellite DNA markers emphasis on correlation with some economically important traits in Mandarin fish. Chinese Journal of Fisheries, 2017, 30(1): 11–18 [孙际佳, 李桂峰, 刘丽, 等. 翘嘴鳜微卫星标记及其与主要经济性状的相关分析. 水产学杂志, 2017, 30(1): 11–18]
Teng SS, Fang J, Shao YQ,. Microsatellite analysis of genetic variation in the fast growth families of the second generation ofand correlation with growth traits. Acta Hydrobiologica Sinica, 2018, 42(4): 681–689 [滕爽爽, 方军, 邵艳卿, 等. 泥蚶G2代快速生长家系遗传结构的微卫星分析及其与生长性状的关联. 水生生物学报, 2018, 42(4): 681–689]
Wei GL, Xu GC, Gu RB,. Studies on the genetic diversity of farmed and wild populations ofby analysing mitochondrial DNA Cytb genes. Acta Agricultural Universitatis Jiangxiensis, 2012, 34(6): 1216–1221 [魏广莲, 徐钢春, 顾若波, 等. 基于mtDNA Cytb序列分析养殖与野生刀鲚群体的遗传多样性. 江西农业大学学报, 2012, 34(6): 1216–1221]
Wei GL, Xu GC, Gu RB,. Biology and artificial breeding progress in. Journal of Yangtze University (Natural Science), 2013, 9(7): 31–35 [魏广莲, 徐钢春, 顾若波, 等. 刀鲚的生物学及人工养殖研究进展. 长江大学学报(自然科学版), 2013, 9(7): 31–35]
Yang M, Yu Y, Zhang XJ,. 2017. Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp (). Marine Sciences, 41(2): 96–102 [杨铭, 于洋, 张晓军, 等. 基于转录组数据的凡纳滨对虾微卫星标记开发. 海洋科学, 2017, 41(2): 96–102]
Yuan CM. The resources ofin middle and lower Yangtze River, and the status and the causation of the population formation changes. Chinese Journal of Zoology, 1988, 23(3): 12–15 [袁传宓. 长江中下游刀鲚资源和种群组成变动状况及其原因. 动物学杂志, 1988, 23(3): 12–15]
Yue HM, Zhai Q, Song MY,. Development of microsatellite markers inusing next- generation sequencing. Freshwater Fisheries, 2016, 46(1): 24–28 [岳华梅, 翟晴, 宋明月, 等. 基于转录组测序的兴国红鲤微卫星标记筛选. 淡水渔业, 2016, 46(1): 24–28]
Zhang D, Fu JJ, Zhang LD,. The parentage analysis of bighead carp () based on ten microsatellite markers. Genomics and Applied Biology, 2018, 1–11 [张丹, 傅建军, 张利德, 等. 鳙基于10个微卫星标记的亲子鉴定分析. 基因组学与应用生物学, 2018, 1–11]
Zhang YP, He G, Wang S,. Analysis of genetic diversity ofandbased on mitochondrial DNA D-loop sequence. Jiangsu Agricultural Sciences, 2017, 45(20): 59–64 [张燕萍, 贺刚, 王生, 等. 基于线粒体DNA D-loop序列分析刀鲚与短颌鲚的遗传多样性. 江苏农业科学, 2017, 45(20): 59–64]
Characteristic Analysis of Microsatellites in SelectedUsing a Transcriptome Dataset
YU Aiqing, SHI Yonghai①, XU Jiabo, LU Genhai, ZHANG Haiming, XIE Yongde, LIU Yongshi
(Shanghai Fisheries Research Institute, Shanghai Fisheries Technology Extension Station, Shanghai 200433)
The objective of this study was to identify microsatellites from the transcriptome sequences ofusing MISA software. These microsatellites could create very useful resources in the evaluation of germplasm resource and marker-assisted breeding of. The results showed that a total of 33,896 microsatellites were identified with repeating units, at lengths of 1~6 bases, from 71,869 unigenes. Different types of repeat SSRs had considerably different distribution characteristics. The majority of the microsatellite loci consisted of mono-, di-, and tri-nucleotide motifs (34.94%, 49.47%, and 13.34%, respectively). Dinucleotide microsatellite repeating units were the most abundant in thetranscriptome, and the AC/GT repeating units were the most ascendant repeating unit (75.25%). The dominant repeating units for the mononucleotide and trinucleotide motifs were A/T (86.25%) and AGG/CCT (28.57%), respectively. Different nucleotide repeat motifs had different repetitions that had a reducing trend, with the increase in the number of nucleotides in repeat motifs. Among the 100 designed primer pairs, 16 pairs proved to be polymorphic microsatellite markers. In the present study, 16 polymorphic microsatellite loci were characterized to evaluate the genetic diversity of the selected(F3). The results showed that the average number of effective alleles (N), the average observed heterozygosity (H), the average expected heterozygosity (H),and the average Shannon´s information index () of the selected(F3) were 1.7580, 0.3414, 0.3977, and 0.6278, respectively. These results indicate that it is feasible to develop microsatellite markers based on thetranscriptome and polymorphic microsatellite loci obtained in this study will facilitate further studies on the population genetic management and conservation of.
; Transcriptome; Microsatellites; Molecular markers
SHI Yonghai, E-mail: yonghais@163.com
* 上海市科技兴农重点攻关项目[沪农科攻字(2016)第6-2-2号]、上海市科学技术委员会重点科技攻关项目(17391900300)和上海市科学技术委员会重点科技攻关项目(11391901300)共同资助 [This work was supported by the Shanghai Key Scientific and Technological Project on Agriculture from Shanghai Municipal Agricultural Commission (2016-2-2), Shanghai Key Scientific Technological Project from Shanghai Science and Technology Commission (17391900300), and Shanghai Key Scientific Technological Project from Shanghai Science and Technology Commission (11391901300)]. 于爱清,E-mail: aiqingyu0125@163.com
施永海,教授级高级工程师,E-mail: yonghais@163.com
2018-08-02,
2018-08-29
S937.3
A
2095-9869(2019)05-0101-09
10.19663/j.issn2095-9869.20180802001
http://www.yykxjz.cn/
于爱清, 施永海, 徐嘉波, 陆根海, 张海明, 谢永德. 刘永士. 长江刀鲚选育群体转录组EST-SSR的分布特征分析. 渔业科学进展, 2019, 40(5): 101–109
Yu AQ, Shi YG, Xu JB, Lu GH, Zhang HM, Xie YD, Liu YS. Characteristic analysis of microsatellites in selectedusing a transcriptome dataset. Progress in Fishery Sciences, 2019, 40(5): 101–109
(编辑 冯小花)
