模拟产生具有相关关系的终点指标数据及R软件实现*
2019-09-17张玲云张晓敏卓仁杰刘丽亚
张玲云 张晓敏 卓仁杰 刘丽亚△
肿瘤药物疗效的金标准评价指标为总生存期(overall survival,OS),但是应用该指标作为"最终"临床研究终点会使整个研发周期延长、研发成本提高。实际工作中,常常考虑应用有效的短期替代终点代替总生存期作为肿瘤药物疗效评价的"最终"临床研究终点。常见肿瘤临床试验替代终点包括肿瘤大小,无进展生存期(progression-free survival,PFS),至疾病进展生存期(time-to progression,TTP)及无病生存期(disease-free survival,DFS)等[1-2]。目前,关于如何验证替代终点是否能够有效替代真实终点仍存在许多争议,但是,毋庸置疑的是替代终点与总生存期具有强相关关系是替代终点有效的必要条件[3-4]。一项研究表明,2005年到2007美国食品药品监督管理局(the U.S.Food and Drug administration,FDA)批准通过的53种肿瘤新药中有9种(17%)肿瘤临床试验的研究终点指标为PFS[5]。由此可知肿瘤药物临床试验研究当中不同结局变量间普遍存在相关关系[6]。因此,关于如何模拟产生具有相关关系的生存时间数据是肿瘤临床试验模拟研究亟待解决的问题之一。
本文介绍并讨论了三种模拟产生具有相关关系生存时间数据的方法,并通过实例介绍其R软件实现[7]。
方法与原理
1.基于Copula函数法
目前,快速发展的Copula理论是构建具有相关非正态变量联合分布函数的有效手段之一,Copula函数实际上是一类将联合分布函数与它们各自的边缘分布函数连接在一起的函数,因此也有人将它称为连接函数。Copula函数的提出最早可以追溯到1959年,SKlar通过定理形式将多元分布与Copula函数联系起来[8]。
Copula函数种类很多,如Gaussian,t,Clayton,Frank,Gumbel,Plackett Copula等,其中由于Gaussian Copula函数可根据变量边缘分布及相关系数矩阵唯一确定变量的联合分布函数,故采用Gaussian Copula函数产生具有相关关系的生存时间数据可以根据实际情况设定相关系数ρ[9]。
以相关的二元分布为例介绍如何应用Gaussian Copula函数构建具有相关关系的二元非正态变量x1和x2的联合分布函数。
(1)模拟产生样本量为N相关系数为ρ的二元正态分布(Z1,Z2),
(1)
(2)分别计算两变量Z1和Z2的累积分布用U1和U2表示,
U1=Φ(Z1)
U2=Φ(Z2)
(2)
(U1,U2)表示具有相关关系的二元均匀分布,范围分别为(0,1)。
(3)假定待估计具有相关关系的二元非正态变量X1和X2的边际分布函数分别为,F1(·),F2(·)则
(3)
假定生存时间服从指数分布,同时考虑截尾时间变量,则根据Gaussian Copula函数模拟产生具有相关关系的生存时间变量的具体步骤如下,
(1)模拟产生样本量为N相关系数为ρ的二元正态分布(Z1,Z2);
(2)依次计算二元正态分布(Z1,Z2)每个变量的累积分布,用(U1,U2)表示,即二元均匀分布,范围是(0,1);
(3)生存时间服从指数分布,则生存时间X的分布函数及其反函数分别为F(t)=1-e-θt,F-1(t)=-ln(1-F1(t))/θ1。将U1看作是1-F1(t)代入变量分布函数的反函数估计生存时间X。同理,估计生存时间Y;
(4)模拟产生截尾时间变量C,C~Uniform[0,a];
(5)根据上述步骤模拟产生的(X,Y,C)及公式Zx=min(X,Y,C),Zy=min(Y,C),δx=I(X≤min(Y,C))、δy=I(Y≤C)产生TTP(即Zx)与OS(即Zy)变量以及相应截尾变量,I表示指示函数。
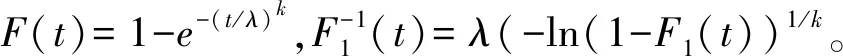
例1 生成1000个相关关系为0.5的TTP与OS,要求TTP与OS的中位生存时间分别为2个月、4.5个月且截尾时间变量服从均匀分布[0,36]。
说明:题目设定的相关系数是Copula函数指定的二元正态分布相关系数,而非TTP与OS相关系数。
程序如下:
install.packages("mvtnorm")
library(mvtnorm)
n=1000 #样本量为1000
corr=0.5 #相关关系为0.5
sigma=matrix(c(1,corr,corr,1),ncol=2) #相关系数矩阵
x=rmvnorm(n=n,mean=c(0,0),sigma=sigma) #模拟产生二元正态分布随机变量
p1=pnorm(x[,1],mean=0,sd=1,lower.tail=TRUE,log.p=FALSE) #变量Z1的累积分布函数U1
p2=pnorm(x[,2],mean=0,sd=1,lower.tail=TRUE,log.p=FALSE) #变量Z2的累积分布函数U2,
TTP0=qgamma(p1,1,rate=log(2)/2,lower.tail=TRUE,log.p=FALSE) #TTP服从中位生存时间为2的指数分布
OS0=qgamma(p2,1,rate=log(2)/4.5,lower.tail=TRUE,log.p=FALSE) #OS服从中位生存时间为4.5的指数分布
censor=runif(n=n,min=0,max=36) #截尾变量censor服从均匀分布[0,36]
OS=pmin(OS0,censor) #实际的OS为OS0与censor的最小值
TTP=pmin(TTP0,OS0,censor) #实际的TTP为TTP0,OS0和censor的最小值
TTP_cen=(TTP0<=OS) #如TTP0小于等于实际的OS则认为疾病进展事件发生,表示疾病进展在死亡前、观察截止前被观测到
OS_cen=(OS0<=censor) #如OS0小于等于截尾时间则认为终点事件发生,表示死亡事件在观察截止前被观测到
Data_EX1=cbind(TTP,OS,TTP_cen,OS_cen) #最终生成的数据集
另上述程序假定生存时间均服从指数分布,如生存时间服从Weibull分布的,上述生成TTP0、OS0的步骤可修改为,
TTP0=qweibull(p1,1,2/log(2),lower.tail=TRUE,log.p=FALSE) #TTP服从中位生存时间为2的Weibull分布
OS0=qweibull(p2,1,4.5/log(2),lower.tail=TRUE,log.p=FALSE) #OS服从中位生存时间为4.5的Weibull分布。
2.混合指数模型法
肿瘤病人在死亡前常经历数次疾病进展,如图1所示,然而疾病进展后的死亡风险一般高于疾病进展前,因此构建产生生存时间数据模型时应充分考虑疾病发生发展机制。采用混合指数模型法分别模拟产生疾病进展前、后生存时间,不同阶段的死亡风险可依据实际情况设定不同参数[10]。

图1 肿瘤患者疾病进展示意

(4)
如果k≠i则λi≠λk。
假定受试者只经历一次疾病进展后死亡,即n=2时,t的概率密度函数可表示为:
(5)
则根据混合指数模型模拟产生具有相关关系生存时间数据的具体步骤如下:
(1)模拟产生样本量为N,参数为λ1的指数分布,TTP;
(2)模拟产生样本量为N,参数为λ2的指数分布,PPS;
(3)OS=TTP+PPS,则将步骤(1)与步骤(2)产生的模拟数据相加得到OS。
基于该法不能考虑受试者在疾病进展前发生死亡事件,Fleischer等人于 2009年对该法进行了改进[11]。首先,假定TTP与OS存在相关关系,TTP服从参数为λ1的指数分布。其次,引入X变量,表示未经历疾病进展的患者从随机化分组至死亡的时间,服从参数为λ2的指数分布,X~Exp(λ2)。由于TTP与X变量相互独立,设定PFS=min(TTP,X),则PFS仍服从指数分布,PFS~Exp(λ1+λ2)。最后,假定疾病进展后生存期(post-progression survival,PPS)仍服从参数为λ3指数分布,PPS~Exp(λ3)。因此,OS可表示为,
(6)
此时OS风险函数不再是一个常量,随着疾病进展而改变。一般来讲,疾病进展后的风险率高于进展前(λ3>λ2,即疾病进展前死亡风险为λ2,疾病进展后死亡风险为λ3)。基于上述假定,PFS与OS的相关关系为:
(7)
在其他参数不变前提下,随着λ1或λ2的增加PFS与OS的相关关系下降,随着λ3的增加PFS与OS的相关关系增加,见图2。
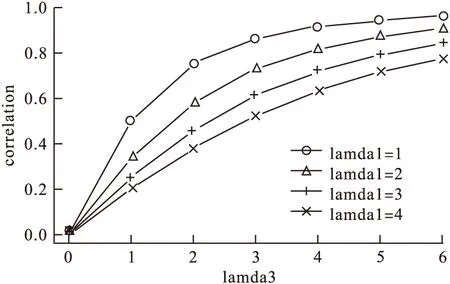
图2 PFS与OS在不同参数组合下相关关系情况(lamda2=1)
*:lamda1,表示TTP服从参数为lamda1的指数分布;lamda2,表示未经历疾病进展患者从随机化分组至死亡的时间服从参数为lamda2的指数分布;lamda3,表示PPS服从参数为lamda3的指数分布;correlation表示PFS与OS相关系数。
例2 模拟产生1000个具有相关关系的PFS与OS数据,要求TTP、PPS的中位时间分别为5个月及15个月,未经历疾病进展患者的中位生存时间为7.5个月。
程序如下:
n=1000 #样本量为1000
rate1=log(2)/7.5 #未经历疾病进展患者的死亡风险函数
rate2=log(2)/5 #TTP的风险函数
rate3=log(2)/15 #PPS的风险函数
x=rexp(n,rate1) #模拟产生未经历疾病进展患者的死亡时间
ttp=rexp(n,rate2) #模拟产生TTP
pfs=pmin(ttp,x) #模拟产生PFS
pps=rexp(n,rate3) #模拟产生PPS
os=pps+pfs #模拟产生OS
cor(pfs,os) #估计PFS与OS相关关系
3.混合Bayes模型法
Michael等人于2002年提出一种能够精确模拟产生具有相关关系的多元分布新方法,称为混合Bayes模型法[12]。该法应用Bayes思想,易于推广,其本质是根据后验分布模拟产生与先验具有相同边际分布的样本,两个边际分布的相关关系由似然函数决定。
假定随机变量X1的先验分布为
g(x1;θ)
(8)
参数θ表示多维变量;在X1=x条件下,根据抽样分布K|(X1=x)获得样本,构建似然函数可表示为
h(k|x1;η)
(9)
将公式(8)与公式(9)相乘,得变量X1和K联合分布
j(x1,k;θ,η)=g(x1;θ)h(k|x1;η)
(10)
对X1积分得K的边际分布

(11)
给定K=k条件下,估计X2的后验分布
p(x2|k;θ,η)=j(x2,k;θ,η)/m(k;θ,η)
(12)
变量X1与X2有相同的边际分布,X2分布为不同后验分布的加权平均,准确地再现了先验分布。假定生存时间满足Gamma分布,则变量X1与X2的边际分布均为Gamma分布。
应用混合Bayes模型法模拟产生具有相关关系的生存时间变量的具体步骤如下:
(1)确定先验分布X1~Γ(a,b),根据先验分布模拟产生X1,样本量为N;
(2)由于Poisson分布的共轭先验分布是Gamma分布,因此条件抽样分布为K|(X1=x)~Poisson(τx),据此分布抽样获得样本量为N的K;
(3)根据先验以及抽样获得的K估计后验分布,X2~Γ(a+k,b+τ);
(4)根据每个后验分布依次模拟产生X2,据此获得的二维随机变量(X1,X2)具有相关关系且边际分布均为Gamma分布,相关系数为
(13)
(5)模拟产生截尾时间变量C,C~Uniform[0,a];
(6)根据上述步骤模拟产生的(X1,X2,C)及公式Zx1=min(X1,X2,C),Zx2=min(X2,C),δx1=I(X1≤min(X2,C)),δx2=I(X2≤C)产生TTP(即Zx1)与OS(即Zx2)变量以及相应截尾变量,I表示指示函数。
例3 模拟产生1000个相关系数为0.78的TTP与OS,要求TTP服从指数分布中位时间为5个月。
说明:依据实际情况需要模拟产生生存时间数据需考虑截尾时间。本文介绍混合Bayes模型法模拟产生截尾时间变量的步骤同例1一致,故R程序一致,同时由于考虑截尾时间将会影响TTP与OS的相关关系,故本例未考虑截尾时间变量。本例模拟数据产生后可评估TTP与OS的相关系数与设定参数是否一致,从而检验模拟过程是否正确。
程序如下:
n=1000 #样本量为1000
shape1=1 #形态参数为1的Gamma分布是指数分布
rate1=log(2)/5 #尺度参数
prior=rgamma(n,shape1,scale=1/rate1) #先验分布为指数分布,即TTP
tau=0.5 #Poisson分布的τ设定为0.5
lamda=data.frame(tau*prior) #估计条件分布K|(X1=x)~Poisson(τx)参数
likelihood=rpois(n,lamda[,1]) #据条件分布抽样获得K,样本量为1000
shape2=data.frame(shape1+likelihood) #根据先验形态参数以及抽样获得的K估计1000个后验分布的形态参数
rate2=1/(1/rate1+tau) #依据相关关系公式估计后验分布的尺度参数
posterior=rgamma(n,shape2[,1],scale=1/rate2) #据1000个后验分布抽样获得1000个后验样本,即OS
cor(prior,posterior) #估计TTP与OS相关系数

表1 三种模拟产生具有相关关系生存时间数据方法的优缺点
*:PFS,progression-free survival,无进展生存期;TTP,time-to progression,至疾病进展生存期;PPS,post-progression survival,疾病进展后生存期。
讨 论
关于如何模拟产生具有相关关系的生存时间数据广泛应用于研究与实践领域,主要用于评估统计分析方法的统计学性能。本文介绍了三种模拟产生具有相关系数生存时间数据的方法,其相关系数参数均能设定,但是由于截尾数据的存在,模拟设定的相关系数将会受到影响,且随截尾变量均匀分布上限的缩短而降低。
在实际工作中应用三种方法产生模拟数据时需注意:(1)Copula函数法设定的相关系数是二元正态分布相关系数而非生存时间相关;(2)混合指数模型法可以控制PPS的长短,如果模拟试验研究考虑该变量作为研究的变化参数时,则只能采用该法产生模拟数据;(3)混合Bayes模型法不能设定中位生存时间;(4)Copula函数法和混合Bayes模型法除能模拟产生具有相关关系的生存时间变量外,也可推广应用至其他分布类型数据的模拟产生,但是应用混合Bayes模型法要求具有相关关系的先验与后验分布类型一致。
本研究为建立一套科学、可行、又可操作的产生具有相关关系的模拟数据的方式提供依据,进而解决科研工作者在生存资料模拟研究中关于产生模拟数据的困惑。本研究的局限性在于产生截尾时间数据的方式均基于均匀分布且截尾类型仅考虑右侧截尾情况,因此在实际工作中产生模拟数据的方式需依据实际情况而定。
