旱柳转录组测序及生物学分析
2019-09-10李敏郭聪李玉娟冯新民王莹张健谈峰
李敏 郭聪 李玉娟 冯新民 王莹 张健 谈峰









摘要:对盐敏感旱柳沿江柳和耐盐旱柳9901的杂交F代根部进行了RNA-Seq测序和分析,共获取了107950条Unigene,平均长度为1076.96bp。通过与COG、GO等8个数据库比对,60848个基因获得注释信息,其中38182条Unigene在GO数据库中获得注释,24101条Unigene在KEGG数据库中获得注释。GO和KEGG富集分析结果表明,差异表达基因主要调节核糖体代谢、植物激素信号转导等生物学功能。
关键词:旱柳;转录组分析;盐胁迫;差异表达基因
中图分类号:S718.43
文献标识码:A
文章编号:1000-4440(2019)02-0271-11
土壤盐渍化是阻碍植物生长并严重降低植物生产力的主要环境胁迫因素之一。根据粮农组织和教科文组织的不完全统计资料,全世界约有4.0x108hm鹽碱地,而中国约有3.6x107hm。由于自然环境变化和人为因素,导致土壤退化,土壤盐碱化面积逐渐增加。盐胁迫对光合作用、能量生产和营养获取等植物基本过程具有很大的负面影响,它破环细胞膜的完整性和各种酶的活性,这可能进一步导致许多有害作用,例如缺水、渗透胁迫、二次氧化胁迫以及动态平衡和离子毒性的破坏。许多研究者借助分子和基因组学的方法阐明耐盐植物的耐盐机制。通过转录组分析,许多耐盐基因已经被发现,涉及盐胁迫信号转导的几种重要途径,例如盐敏感性蛋白(SOS)、钙依赖性蛋白激酶(CDPK)和有丝分裂原激活蛋白(MAP)。
过去几十年,不同植物耐盐胁迫的转录组分析已被报道,比如棉花、大豆、冰叶日中花、草地早熟禾、欧洲油菜、沙漠杨树和小麦。这些研究结果表明,与氧化还原过程和脯氨酸代谢有关的差异表达基因以及许多转录因子都在植物耐盐中发挥重要作用。目前,一些模式植物,比如水稻、玉米和拟南芥等已经进行了转录组测序。这些模式植物的DNA序列信息和转录组序列信息都已经很完善,可用于测序、绘图和组装基因等研究。然而,非模式木本盐生植物的转录组研究很少。非模式木本盐生植物基因组和转录组数据库的缺乏很大程度上限制了其相关的研究。
柳树(Salix L.)属于杨柳科物种,其种类繁多,在中国就存在256种以上,其生长潜力大,顶端优势比较明显,腋芽萌发力比较强,具有抗性强、适应性广等特性。柳树根部是应对盐胁迫的首要部位,根部对Na*的选择性吸收外排和区域化对柳树响应盐胁迫具有重要作用。本研究利用llumina HiSeq高通量测序平台对旱柳沿江柳和耐盐旱柳9901的杂交F,代根部进行转录组测序分析,比较耐盐柳树和盐敏感柳树中基因表达的差异,以期通过RNA-Seq技术获得与耐盐性相关的基因序列,为揭示柳树耐盐分子机制和培育柳树耐盐新品系提供参考。
1 材料与方法
1.1 材料及处理
试验材料为盐敏感旱柳沿江柳(采自江苏沿江地区农业科学研究所)和耐盐旱柳9901(采自山东省林业科学研究院)的杂交F代。2017年6月,将F,代进行耐盐分离群体试验,选择15个耐盐材料和15个盐敏感材料,每5个材料混合为1个样品,作为1个重复。经0mmol/L和150mmol/LNaCl溶液处理4h后,取柳树的新鲜根,立即放入液氮中保存备用。
1.2 总RNA的提取及检测
使用TIANCEN公司的多糖多酚植物试剂盒提取柳树根的总RNA,样品的纯度、浓度和完整度分别用Nanodrop、Qubit2.0、Aglient2100分析。柳树总RNA的提取、质量检测及无参考基因组序列的转录组高通量测序分析均由北京百迈客生物科技有限公司进行。
1.3 cDNA文库构建和转录组测序
用检测合格的柳树总RNA样品构建eDNA文库。以mRNA为模板,利用AMPure XP beads试剂盒依次进行纯化、末端修复和片段大小选择,通过PCR富集得到柳树eDNA文库。eDNA文库质量用Agilent2100检测,再通过llumina Hi-Seq平台完成RNA-Seq测序分析。
1.4 De novo组装及质量控制
获得的原始数据(Rawreads)经过滤得到Cleanreads。将其用Trinity拼接,得到contigs。利用DeBruijin图方法和测序Read信息,在片段集合中读取转录本序列,最长contig即为最终转录本的Unigene。每个样品测序产出不少于6Gb Clean data,Q30碱基百分比达到85%。
1.5 Unigene功能注释
使用BLAST软件将差异表达基因(DEG)序列与NR、Swiss-Prot、GOCOG、KOG、eggNOG4.5、KEGG数据库分别比对,获得相关注释信息。
2 结果与分析
2.1 旱柳转录组测序与de novo组装
利用lluminaHi-Seq对12个旱柳根样品进行转录组测序,共获得83.81Gb Clean data,各样品Clean data均达到6.31Gb以上(表1)。Q30碱基百分比均不小于89.21%,GC含量百分比在44%以上,可以用于下一步的de novo组装。利用Trinity软件将所有短序列拼接组装得到107950条Unigene,总长度116257763bp,平均长度为1076.96bp。其中长度在500~1000bp的Unigenes所占比例最高,为29.39%,Unigene的N50为1690bp,组装完整性较高(图1),获得的Mapped reads可用于后续分析。
2.2 旱柳差异基因表达量分析
在差异表达分析过程中采用了公认有效的Benjamini-Hochberg方法对原有假设检验得到的显著性p值进行校正,并最终采用校正后的p值,即FDR(False discovery rate)作为差异表达基因筛选的关键指标,以降低对大量基因的表达值进行独立统计假设检验带来的假阳性。
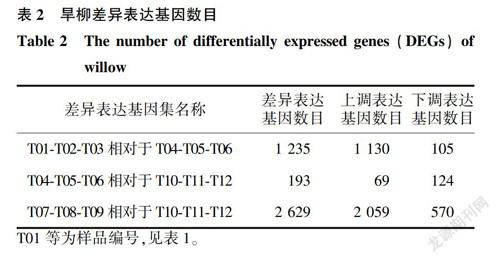
在筛选过程中,将FDR小于0.01且差异倍数FC[两样品(组)间表达量的比值]≥2作为筛选标准。在盐敏感柳树中,以0mmol/LNaCl溶液处理为对照,在150mmol/L盐胁迫下,共有1235个DEG表达量发生了改变,其中1130个DEG上调,105个DEG下调;在耐盐柳树中,2629个DEG表达量发生了改变,其中2059个DEG上调,570个DEG下调。在150mmol/L盐胁迫下,盐敏感柳树与耐盐柳树相对比,共有193个DEG表达量发生了改变,其中69个DEG上调,124个DEG下调(表2)。将不同组的差异基因进行对比并绘制维恩图,展示对比后各组特有的差异基因数量,以及共有的差异基因数量。其中盐敏感柳树在不同盐浓度处理下与耐盐柳树在不同盐浓度处理下一共有853个重复差异基因;盐敏感柳树和耐盐柳树在150mmol/L盐胁迫下与盐敏感柳树在不同盐处理下有59个差异基因,而与耐盐柳树在不同盐处理下有99个差异基因,三者有5个共同的差异基因(图2)。
2.3 旱柳差异表达基因功能注释
通过选择BLAST参数(E值≤x10~')和HMMER参数(E值≤x10),将全部Unigene序列与COG、GO等8个数据库进行比对(表3、表4),最后得到60848个有注释信息的基因。其中长度在1000bp以上的基因注释效率为52.39%,而未得到注释的Unigene为47102条(43.63%)。
2.4 旱柳差异表达基因GO分类和功能富集
将旱柳差异表达基因在GO数据库中进行比对,有38182条Unigene在GO数据库中得到相关注释,其中每2个样品相比得到的差异基因在GO数据库中注释率最多达69.28%,注释的基因参与生物学过程、细胞组分和分子功能调控(表5),功能组涉及到54个(图3、图4、图5)。生物学过程的注释显示,Unigene表达差异显著顺序依次为代谢过程、细胞过程和单组织过程;在细胞成分分类信息中,细胞部分和细胞显著富集;在分子功能分类中,富集程度最高的是生物合成和催化活性。
2.5 旱柳差异表达基因KEGG注释和富集分析
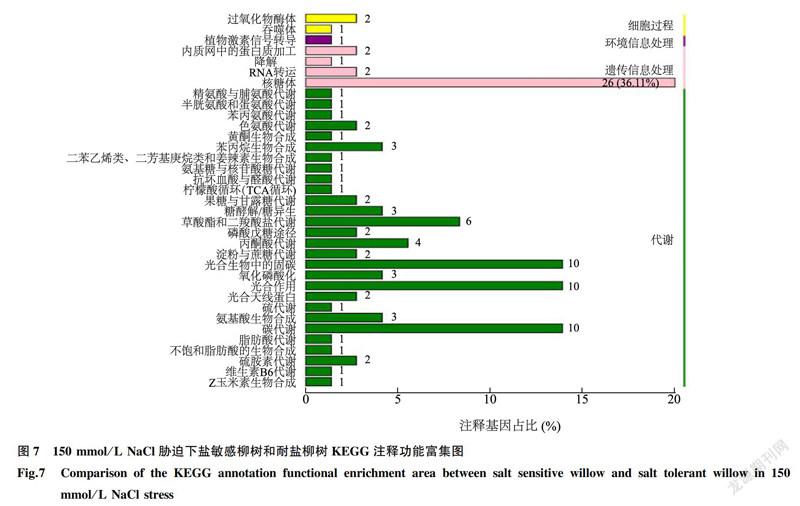
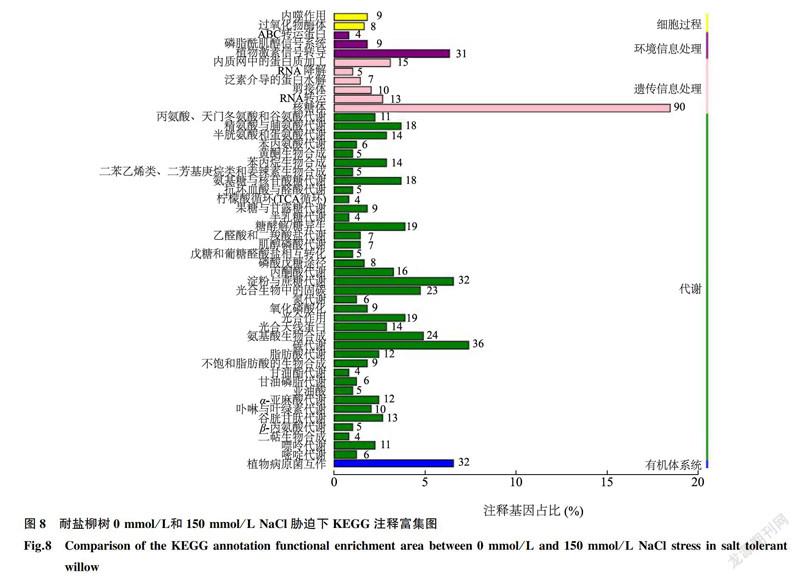
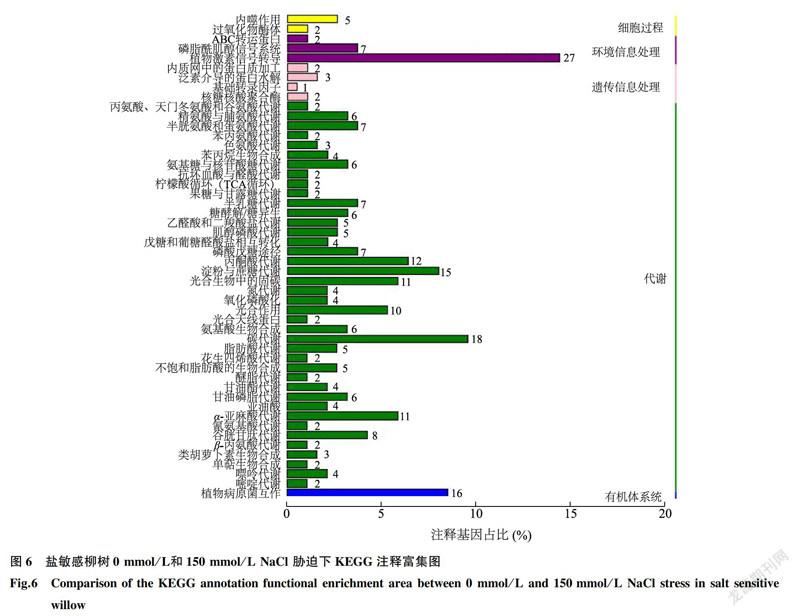
有24101条(39.61%)Unigene在KEGG数据库得到注释,涉及209条KEGG代谢通路。其中盐敏感柳树组,以0mmol/LNaCl溶液处理为对照,在150mmol/L盐胁迫下,获得注释的189条差异表达基因(DEG)可以被分配到77个KEGG通路(图6);150mmol/L盐胁迫下,盐敏感柳树组与耐盐柳树组对比,获得注释的72条差异表达基因可以被分配到35个KEGG通路(图7)。耐盐柳树组,以0mmol/LNaCl溶液处理为对照,在150mmol/L盐胁迫下,获得注释的487条差异表达基因可以被分配到97个KEGG通路(图8),其中在同一个KEGG通路中10个以上差异表达基因主要涉及细胞途径、遗传信号响应、环境信号响应、新陈代谢和生物系统5个方面。这5个方面中新陈代谢类最大,包括碳水化合物、氨基酸、多糖生物合成、能量、次生物质等;生物系统类最小。同时,KEGG注释结果也显示,差异基因主要参与核糖体、植物激素信号转导、淀粉与蔗糖代谢、碳代谢以及植物病原菌的相互作用等通路(表6)。
2.6 旱柳差異表达基因注释结果分析
在盐胁迫下,差异表达基因,上调2倍以上才有意义,现筛选高水平差异表达(log2FC达7倍以上)的基因(表7)。高水平差异表达的注释基因中,在150mmol/L盐胁迫下,盐敏感柳树相对于耐盐柳树类似叶绿体的基因BMK_Unigene_064460表达下调水平显著。与对照相比,盐胁迫下盐敏感柳树中高水平差异基因表达都是上调的,主要包括稳定蛋白、细胞色素p450家族蛋白、细胞周期蛋白等。与对照相比,盐胁迫下耐盐柳树中高水平差异基因表达都是上调的,主要包括水通道蛋白、TPR结构蛋白、磷酸烯醇式丙酮酸羧激酶、谷胱甘肽s转移酶等。
3 讨论
当植物受到干旱、高盐、高温、低温等非生物胁迫时,植物细胞表达相关抗逆基因来适应外界不利刺激。这些抗逆基因与各种代谢途径、抗氧化过程等密切相关。本试验中,将盐胁迫下的F,代和正常生长的F,代旱柳植株转录组进行比较,发现两者之间的基因表达具有较大差异,包括差异表达基因数量、表达量、发生上下调的基因数量。植株对盐胁迫的抵御能力往往需要这些差异表达的基因来调节。我们对正常生长和盐胁迫下的F,代旱柳植株.的根进行了转录组测序,并以FDR小于0.01且差异倍数大于等于2的标准进行筛选,分别在0-盐敏(清水处理的盐敏感柳树)F代柳树相对于150-盐敏(150mmol/L处理的盐敏感柳树)F,代柳树,150-盐敏F,代柳树相对于150-耐盐(150mmol/L处理的耐盐柳树))F,代柳树以及0-耐盐(清水处理的耐盐柳树)F,代柳树相对于150-耐盐F,代柳树中得到符合该标准的差异表达基因。0-盐敏F,代柳树相对于150-盐敏F,代柳树和0-耐盐F,代柳树相对于150-耐盐F代柳树的差异表达基因数量分别为1235和2629,说明耐盐F代柳树的耐盐机制中参与调控的基因较多,可能有更好的抵御能力。150-盐敏F代柳树相对于150-耐盐F代柳树的差异表达基因数目为193,进一步说明两种F,代柳树对盐胁迫的抵御能力是有差异的。
不司组织器官、发育阶段以及不同处理下样本间基因表达的比较能够有效地揭示分子机制。本研究中,我们对正常生长和盐胁迫下的柳树F,植株进行转录组测序分析,结果显示60848个Unigene通过COG、GO等8个数据库比对得到注释信息,其中有38182个Unigene在GO数据库中得到相关注释,24101个Unigene在KEGG数据库中得到注释。通过进一步GO富集分析发现,大量差异表达基因在代谢过程、细胞过程、单组织过程、细胞部分生物合成以及催化活性等GO条目中显著富集。通过KEGG显著性富集分析发现差异表达基因主要在核糖体、植物激素信号传导、淀粉与蔗糖代谢、碳代谢以及植物病原菌的相互作用等通路较为活跃,说明它们在柳树响应盐胁迫中发挥重要作用。
另外,我們对log2FC达到7倍以上的差异表达基因进行了注释分析。结果表明,盐敏感柳树中高水平差异基因都是上调表达,主要包括蛋白质、细胞色素p450家族蛋白以及细胞周期蛋白等。耐盐柳树中高水平差异基因也都是.上调表达的,主要包括水通道蛋白、TPR蛋白、磷酸烯醇式丙酮酸羧激酶以及谷胱甘肽s转移酶等。因此,这些差异表达基因可能是柳树耐盐的关键因子。
总之,通过转录组分析,我们比较全面地了解了柳树响应盐胁迫的转录水平变化,为进一步研究柳树耐盐分子机制,培育柳树耐盐品系提供了重要的信息。
参考文献:
[1] SHABALA S,CUIN T A. Potassium transport and plant salt tolerance :[ J]. Physiol Plant,2008,133(4):651-669.
[2] NAN G,ZHANG Y,LI S,et al. NaCl stress-induced transeriptomics analysis of Salix linearistipularis (Salix mongolica)[J]. J Biol Res (Thessalon),2016,23:1.
[3] CHINNUSAMY V,ZHU J,ZHU J K. Salt stress signaling and mechanisms of plant salt tolerance[ J]. Genet Eng,2006,27:141-177.
[4] MUNNS R,TESTER M. Mechanisms of salinity tolerance[J]. An-nu Rev Plant Biol,2008,59:651-681.
[5] CHEN S,POLLE A. Salinity tolerance of Populus[J]. Plant Biol(Stuttg),2010,12(2):317-333.
[6] NISHIYAMA R,LE D T,WATANABE Y,et al. Transcriptome analyses of a salt-tolerant cytokinin-deficient mutant reveal differ-ential regulation of salt stress response by cytokinin deficiency[J].PLoS ONE,2012,7(2):e32124.
[7] WARD J M,HIRSCHI K D,SZE H. Plants pass the salt[J].Trends Plant Sci,2003,8(5):200-201.
[8] NISHIYAMA R,WATANABE Y,FUJITA Y,et al. Analysis of cytokinin mutants and regulation of cytokinin metabolic genes re-veals important regulatory roles of cytokinins in drought,salt andabscisic acid responses ,and abscisic acid biosynthesis[J]. PlantCell,2011 ,23(6):2169-2183.
[9] KRASENSKY J,JONAK C. Drought,salt,and temperature stress-induced metabolic rearrangements and regulatory networks[ J ]. JExp Bot,2012,63(4):1593- 1608.
[ 10] SANCHEZ-BARRENA MJ,MARTINEZ-RIPOLL M,ZHU J K,et al. The structure of the Arabidopsis thaliana SOS3 :molecularmechanism of sensing calcium for salt stress response[ J]. J MolBiol,2005,345(5):1253- 1264.
[ 11] NAKAGAMI H,PITZSCHKE A,HIRT H. Emerging MAP kinase pathways in plant stress sigalling[ J]. Trends Plant Sci,2005,10(7):339-346.
[12] XUP,LIU z,FAN X,et al. De novo transcriptome sequencing and comparative analysis of differentially expressed genes in Gos-sypium aridum under salt stress[J]. Gene,2013,525(1):26-34.
[13] BUYUK I,INAL B,ILHAN E,et al. Genome-wide identification of salinity responsive HSP70s in common bean[J]. Mol Biol Rep,2016,43(11):1251-1266.
[ 14] TSUKAGOSHI H,SUZUKI T,NISHIKAWA K,et al. RNA-seq analysis of the response of the halophyte,Mesembryanthemum crys-tallinum (ice plant)to high salinity[J]. PLoS ONE,2015,10(2):e0118339.
[ 15] BUSHMAN B S,AMUNDSEN K L,WARNKES E,et al. Tran-scriptome profiling of Kentucky bluegrass (Poa pratensis L.)ac-cessions in response to salt stress[ J]. BMC Genomics,2016,17:48.
[16] LONG W,ZOU X,ZHANG X. Transcriptome analysis of canola (Brassica napus)under salt stress at the germination stage [ J ].PLoS ONE,2015,10(2):e0116217.
[17] ZHANGJ,JIANG D,LIU B,et al. Transcriptome dynamics of a desert poplar (Populus pruinosa)in response to continuous salinitystress[J]. Plant Cell Rep,2014 ,33(9):1565-1579.
[18] GOYALE,AMITSK,SINGH RS,et al. Transcriptome profiling of the salt-stress response in Triticum aestivum cv. Kharchia Local[J]. Sci Rep,2016,6:27752.
[19] KUMARIS,SABHARWAL V P,KUSHWAHA H R,et al. Tran-scriptome map for seedling stage specific salinity stress response in-dicates a specific set of genes as candidate for saline tolerance inOryza sativa L.[J]. Funct Integr Genomics,2009,9(1):109-123.
[ 20] OHTSU K,SMITH M B,EMRICHSJ,et al. Global gene expression analysis of the shoot apical meristem of maize (Zea mays L. )[J]. Plant J,2007,52(3):391-404.
[21] WEBER A P,WEBER K L,CARR K,et al. Sampling the Arabi-dopsis transcriptome with massively parallel pyrosequencing[J]. Plant Physiol,2007,144(1):32-42.
[22]李敏,王瑩,李玉娟,等.2个柳树亲本耐盐性比较试验[J].浙江农业科学,2017(7):1220- 1222.
[23] WICKER T,SCHLAGENHAUF E,GRANER A,et al. 454 sequencing put to the test using the complex genome of barley[J].BMC Genomics,2006,7:275.
[ 24] MOROZOVA O,HIRST M,MARRA M A. Applications of new sequencing technologies for transcriptome analysis[J]. Annu RevGenomics Hum Genet,2009,10:135-151.
