SMBBR处理焦化废水性能及菌群结构响应关系
2019-08-28李卫平郝梦影敬双怡于玲红孙岩柏杨文焕
李卫平,郝梦影,敬双怡,于玲红,孙岩柏,杨文焕
SMBBR处理焦化废水性能及菌群结构响应关系
李卫平,郝梦影,敬双怡,于玲红,孙岩柏,杨文焕*
(内蒙古科技大学能源与环境学院,内蒙古 包头 014010)
采用特异性移动床生物膜反应器(SMBBR)处理焦化废水,连续监测生物反应器处理性能.通过HS-GC/MS和Illumina高通量测序探究污染物降解与生物膜菌群结构的响应关系;利用CCA分析废水变量对微生物菌群结构的影响关系.结果表明,系统稳定运行50d时总酚去除率达96.62%,100d时硫氰化物和氰化物完全降解,其中酚、硫氰化物和氰化物对NH4+-N的降解具有毒性抑制作用.HS-GC/MS结果显示,经过好氧处理后,80%以上的有机物被完全去除,其中包括全部酚、部分含N、O杂环化合物和长链烷烃等.测序结果表明,反应时间的不同,生物膜菌群丰度和多样性存在差异.反应期间Proteobacteria(变形菌门)相对丰度最高(20.57%~34.55%),促进了苯酚的降解;优势菌属为、、;此外,(陶厄氏菌属)、、(根瘤菌属)和(硫杆菌属)为系统中苯酚、SCN-和CN-的降解优势菌.CCA分析表明,pH值与(硝化菌属)正相关性最大,有效控制pH值可有助于硝化反应的稳定运行.本文研究结论可为生物膜法处理焦化废水提供理论依据.
SMBBR;焦化废水;好氧降解;菌群结构;废水变量
焦化废水是一种高污染难降解的有机工业废水,具有较高的毒性和致癌作用[1-4].生物处理焦化废水一直是研究领域的热门课题.目前,焦化废水生物处理工艺大多采用A/O、A2/O、A/H/O或A/O2等传统活性污泥法[5-6],Sharma and Philip[7]验证了好氧活性污泥法可有效去除苯酚及其衍生物、含N杂环化合物、多环芳烃等有机污染物;刘国新[8]和蒙小俊[9]等分析了好氧处理焦化废水过程中微生物群落的组成及多样性,结果表明:微生物在不同的培养阶段群落结构和丰度具有显著差异;另外, Ma[10]和Zhu[11]等采用焦磷酸测序分析表明微生物属如,,,,and可能在焦化废水中苯酚、硫氰化物和氰化物的降解中起重要作用.然而,当前国内对于生物膜法处理焦化废水中污染物降解和与污染物降解相关的微生物菌群结构之间的响应关系研究尚少;此外,环境变量对污染物的降解和微生物菌群结构组成的影响知之甚少.
本研究采用连续好氧处理装置——特异性移动床生物膜反应器(SMBBR)来处理厌氧预处理后的焦化废水,挂膜启动完成后,连续180d监测膜生物反应器的处理性能,并通过顶空气相色谱/质谱联用(HS-GC/MS)评估反应装置中主要污染物的降解情况.同时,采用Illumina Miseq测序平台对试验接种污泥和系统稳定运行后不同时间点的生物膜样品进行微生物菌群结构分析,考察生物膜上的生物量变化与污染物降解的响应关系.最后,通过典范对应分析(CCA)探究废水变量和微生物菌群之间的相关性,分析废水变量对微生物菌群结构的影响关系.本研究可以为SMBBR应用于焦化废水处理的可行性提供理论依据.
1 材料与方法
1.1 试验材料与装置
试验用水取自包钢焦化厂调节池,试验原水首先经过厌氧水解酸化作用使得有机大分子难降解污染物质转化为小分子物质[12].厌氧预处理采用AMBBR装置,控制HRT为3d.预处理后采用好氧曝气装置SMBBR连续运行180d.SMBBR是基于MBBR基础上的一种改进装置,其投加具有发明专利的SDC-03型特异性填料[13-14].试验期间控制SMBBR中HRT为5d,进水负荷60mL/min,DO 3~ 6mg/L,温度20~35℃,pH值 7.5~8.5,MLSS控制在500~1000mg/L,控制C:N:P=100:5:1.调节池中原水水质为:COD浓度1500~3000mg/L,NH4+-N 60~ 100mg/L,TN 180~220mg/L,TP 1~3mg/L.试验接种污泥取自本焦化厂好氧池新鲜污泥.试验装置流程如图1所示.
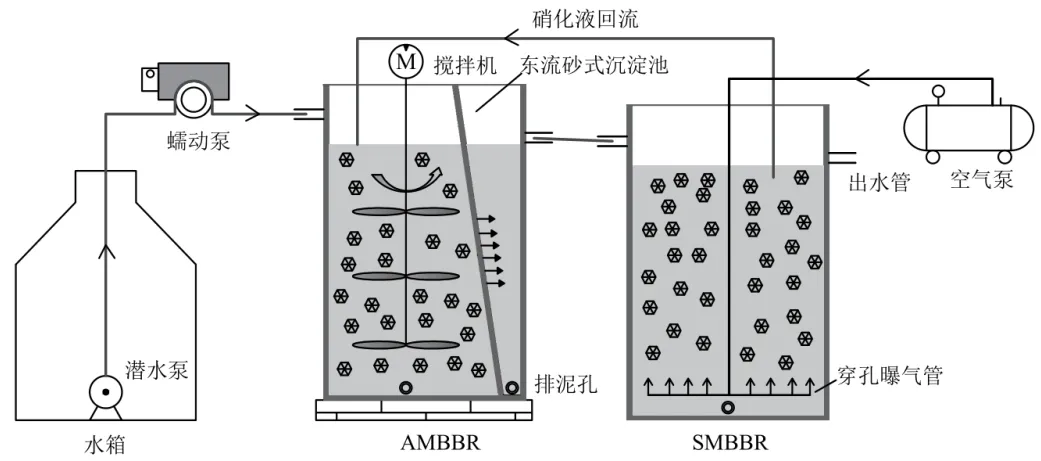
图1 试验装置流程图
1.2 挂膜启动与样品收集
采用闷曝排泥法挂膜,首先向装置中投加填充率为45%的填料,然后进水将填料浸泡24h后加入新鲜污泥闷曝24h,排掉1/3的上清液,再进水闷曝12h;之后连续进水,控制进水流量为60mL/ min,挂膜启动期间每2d排掉1/3底泥并投加新鲜污泥,试验30d时填料开始出现黄褐色菌斑,挂膜启动完成.
系统稳定运行后,每3~5d取好氧装置进出水样用以监测生物反应器的处理性能,为了测定废水中污染物组成,分别取试验原水、SMBBR进出水水样,置于4℃冰箱保存,每个样品分别由3个平行样混合而成.对于微生物分析,将接种的原始污泥和挂膜启动后6个不同时间点采集填料上生物膜样品分别记为d0、d30、d60、d90、d120、d150、d180,并在-20℃保存至DNA提取.每个样品由同一天的不同时间(8:00、13:00、17:00)收集的3个重复生物膜样品混合制备.
1.3 常规分析方法
在化学分析之前,将水样以3000r/min离心10min,然后通过0.45mm膜过滤,测定COD(重铬酸钾-微波消解法)、总酚(4-氨基安替比林试剂法)、总氮(过硫酸钾-紫外分光光度法)、氨氮(纳氏试剂法)、氰化物(氯胺T试剂法)、硫氰化物(铁离子显色分光光度法)[15].采用便携式多参数水质分析仪测量T、DO和pH值.
1.4 HS-GC/MS分析
取SMBBR进出水水样分别在碱性、中性和酸性条件下使用二氯甲烷进行有机萃取,然后将3份萃取液合并,加少量无水硫酸钠干燥,最后用氮气吹脱将萃取液浓缩至1mL待测[16].气相色谱型号: Aglient 7890B,质谱型号:5977B;气相色谱条件为:采用HP-5MS石英毛细柱(30m×0.25mm×0.25μm),柱温45~200℃,45℃维持3min,20℃/min升温至200℃,维持10min;柱流量为1.2mL/min,进样口温度为250℃,柱前压65.2729kpa,进样量1uL,不分流,载气为高纯氦气.质谱条件:电离方式EI,电子能量70eV,传输线温度250℃;离子源温度230℃;四级杆温度150℃;质量范围50~550.
1.5 Illumina Miseq测序
取SMBBR中填料放置于烧杯中,采用超声震荡仪在温度25℃条件下震荡10min使填料上生物膜脱落,放置-20℃的条件下保存.将样品送至上海美吉生物科技有限公司进行高通量测序检测,将检测后的数据采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析,同时运用mothur软件对物种多样性指数进行运算.
1.6 统计分析
将每个样品值与所有样品的平均值之差除以标准偏差来标准化废水水质和生物反应器性能变量,通过Mantel试验来检验废水变量(包括环境变量和生物反应器性能变量)和生物膜菌群OTU之间的关系.同时,通过Canoco 4.5软件对微生物群落和废水变量之间的相关性进行CCA分析,由蒙特卡洛分析结果<0.05被认为是显著相关[17].
2 结果与讨论
2.1 生物反应器处理性能
如图2所示,SMBBR在进水水质波动较大的情况下仍具有较好的处理效果.试验初期,由于微生物处于挂膜启动阶段,接种污泥需要一定的适应时间,所以反应30d之前,COD、总酚、TN、NH4+-N、硫氰化物和氰化物的去除率相较中后期较低.挂膜启动后,COD去除率逐渐升高并趋于稳定,由之前平均去除率67.6%逐渐达到83.19%.随着微生物逐渐适应反应环境,填料上生物膜逐渐增厚,反应50d后,总酚开始快速降解,70d后,去除率达到最大并趋于稳定,可初步推测酚降解菌的适应期为50d.反应器运行80d后,硫氰化物和氰化物去除率开始明显上升,此时COD和总酚去除率已接近最大分别为82.14%和96.62%,表明酚浓度对硫氰化物和氰化物的降解具有抑制作用,此结论与潘霞霞等[18]研究结果一致.NH4+-N去除率在反应30d后逐渐上升,而在100~140d之间有较大下降,随后趋于稳定,而此时硫氰化物和氰化物的去除率达到最大,几乎完全降解,表明随着SCN-和CN-的迅速降解释放NH4+,导致反应中NH4+-N浓度上升,同时,SCN-和CN-对NH4+-N的去除具有较强的抑制作用,Staib[19]和Vazquez[20]等均证实了此观点.而整个试验期间总氮去除率都较低,平均去除率为56.67%,然而相较于Joshi等[21]研究的活性污泥法处理焦化废水整个试验期间总氮几乎无变化的情况具有较大改善,由此证明SMBBR具有较强的脱氮效果[22-23].
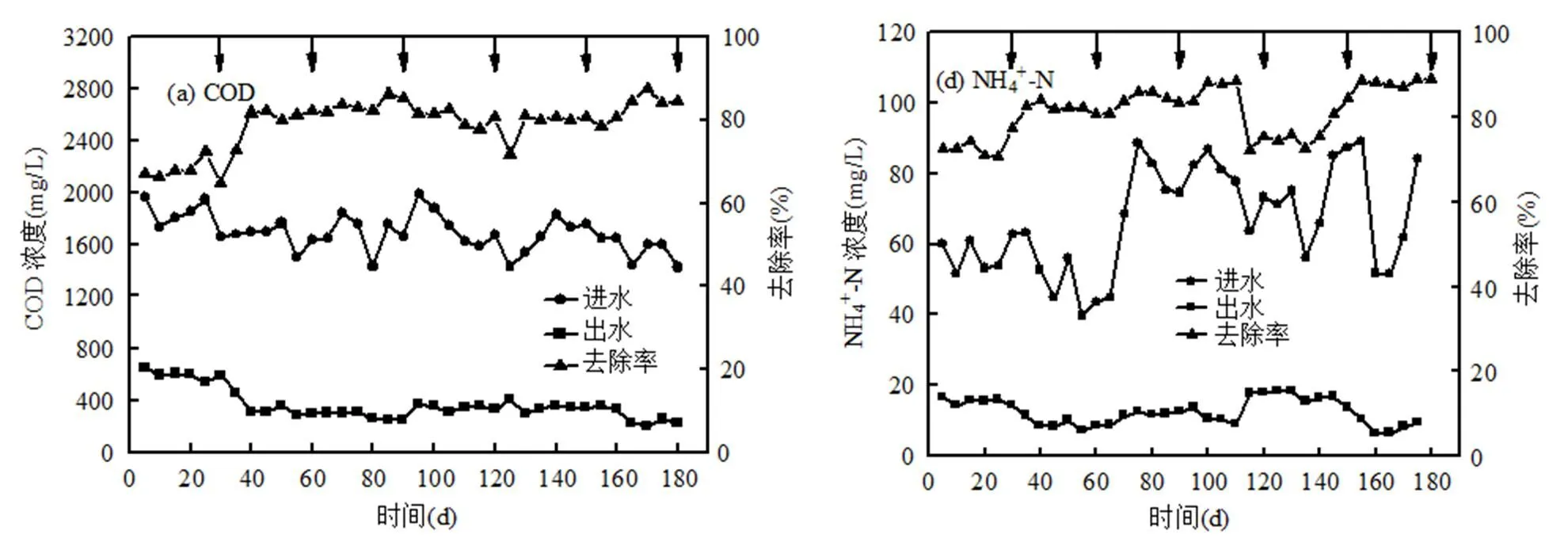
垂直向下的箭头表示Illumina Miseq测序的生物膜样品采集时间
为了更进一步探究焦化废水中有机污染物的组成和反应过程中主要污染物的降解转化,采用HS-GC/MS进行监测分析,表1为反应过程中主要有机污染物降解的分析结果.从表1中可以看出,SMBBR进水中污染物组成成分相比于原水有所降低,说明好氧前的厌氧预处理去除了大约1/3的有机污染物,2-甲基苯酚、对甲基苯酚、喹啉和吲哚在此阶段已被降解,大大减轻了后续好氧装置SMBBR处理负荷,由此证明,厌氧预处理在焦化废水生物处理中发挥重要作用,该研究结果与Wang等[24]研究结果一致.同时,经过好氧处理后,80%以上的有机物被完全去除,其中包括全部酚、部分含N、O杂环化合物和长链烷烃等,该结果与先前实验室所测结果吻合.

表1 焦化废水中有机物的去除效果
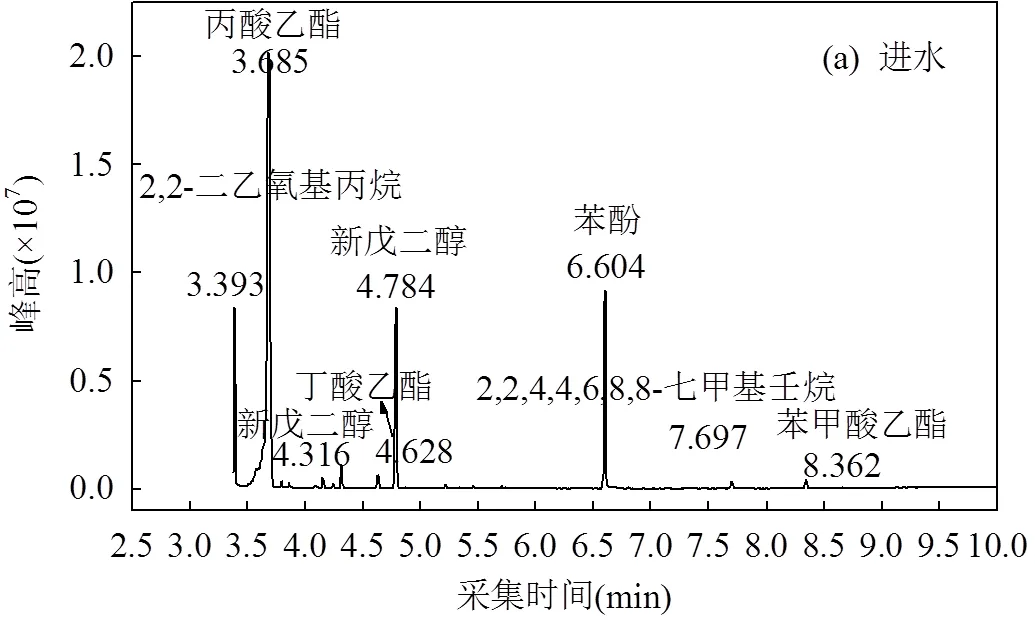
如图3所示,进水中有机污染物主要为:丙酸乙酯、苯酚、新戊二醇、2,2-二乙氧基丙烷、丁酸乙酯、2,2,4,4,6,8,8-七甲基壬烷和苯甲酸乙酯.经过好氧处理后,废水中仍残留丙酸乙酯、新戊二醇和2,2-二乙氧基丙烷等,残留量分别为58.09%、15.55%、7.07%,说明此类化合物难于生化降解,还需结合其它物理化学方法进行进一步处理.
2.2 微生物群落组成和结构变化
在反应过程中的不同时间段,随着废水中污染物的降解,填料上生物膜菌群结构发生相应变化,通过Illumina高通量测序分别对(d0、d30、d60、d90、d120、d150、d180)6个时间点的生物膜菌群结构进行检测,其中d0为接种的原始污泥.测序结果揭示了总共506个细菌OTU的665876个有效序列读数,质控后的序列长度分布在421~460之间,平均长度为434.6bp.测序结果如表2所示.
从表2中可以看出,所有样品覆盖率均在99%以上,表明废水中几乎所有细菌均被检出.从样品的序列数、OTUs和Chao1指数可知,反应运行30d的生物膜菌群丰度相比于原始污泥已有明显增加,从Shannon指数和Simpson指数可知,生物膜菌群多样性也逐渐丰富,说明此时接种的污泥已适应新的环境并开始生长繁殖,挂膜启动完成.30d后,从OTUs、Shannon指数和Simpson指数看出菌群丰度和多样性大幅增加,此时废水中COD去除率逐渐上升.50d后苯酚迅速降解,推测此时系统中苯酚降解菌快速生长繁殖,从而导致菌群丰度的迅增.而当反应至120和150d时,菌群丰度和多样性却有所下降,由生物反应器处理性能可知,此阶段硫氰化物和氰化物降解速率达到最大值,而由其降解释放大量NH4+-N,抑制了微生物生长,导致菌群丰度和多样性降低,此结论与Zhang等[25]研究的高浓度氨和游离氨可以抑制生物增长结论一致.反应一段时间后,菌群丰度和多样性又恢复到之前水平,说明生物膜上的微生物具有较强的适应性.
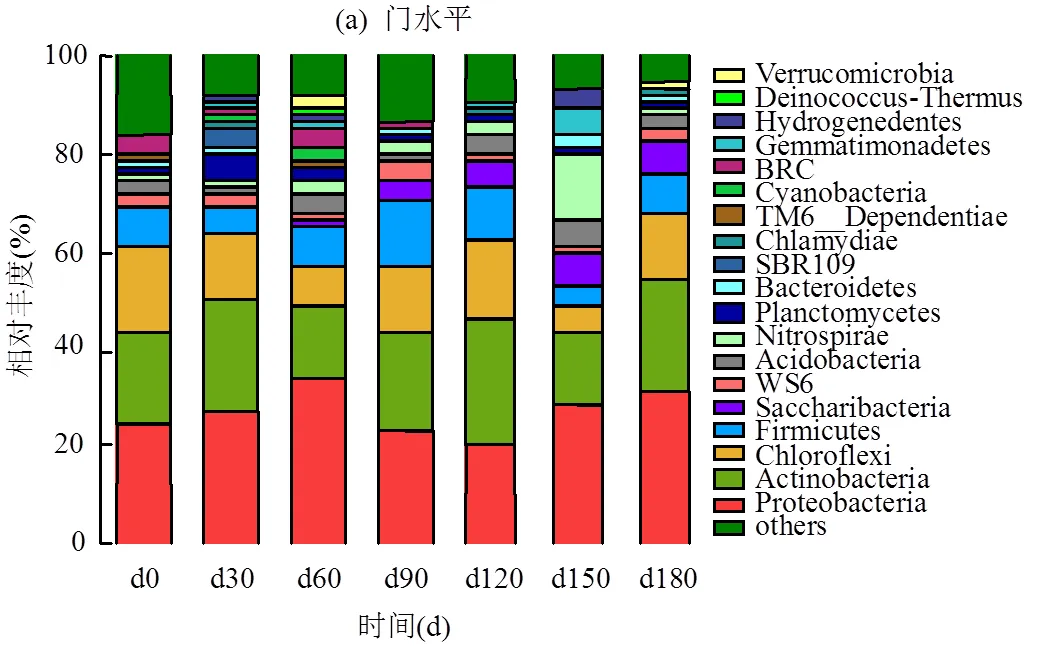
为了进一步解析反应过程中污染物降解与生物膜菌群结构变化的响应关系,图4为采用R语言工具制作的细菌群落结构在门和属水平上的组成和丰度.生物膜上微生物菌群结构的优势门主要有Proteobacteria(变形菌门)、Actinobacteria (放线菌门)、Chloroflexi (绿弯菌门)、Firmicutes (厚壁菌门)、Saccharibacteria (螺旋菌门)、WS6、Acidobacteria (酸杆菌门)、Nitrospirae (硝化螺旋菌门)、Planctomycetes (浮霉菌门)和Bacteroidetes (拟杆菌门),其中Proteobacteria相对丰度最高为20.57%~ 34.55%.Manefield[26]和Ibarbalz[27]已研究过Proteobacteria在不同废水和不同类型生物反应器的苯酚降解系统中经常被观察到,这突出了Proteobacteria在去除酚类化合物中的快速适应和代谢显著性.由图4(a)可看出,Proteobacteria在60d时丰度达到最大值34.55%,此时系统中苯酚降解速率最大.Kim等[28]等研究好氧污泥中可能由于毒性化合物的抑制作用,没有检测到硝化细菌的存在.而从本研究中可知Nitrospirae存在整个反应期间,相对丰度为1.32%~13.34%,反应150d时Nitrospirae丰度最大.这是由于随着系统中SCN-和CN-的完全降解,其对硝化过程的抑制解除,此时Nitrospirae快速生长繁殖,NH4+-N去除率逐渐恢复稳定水平.由此证明SMBBR为硝化细菌提供了理想的生存环境,从而具有较强的脱氮能力.
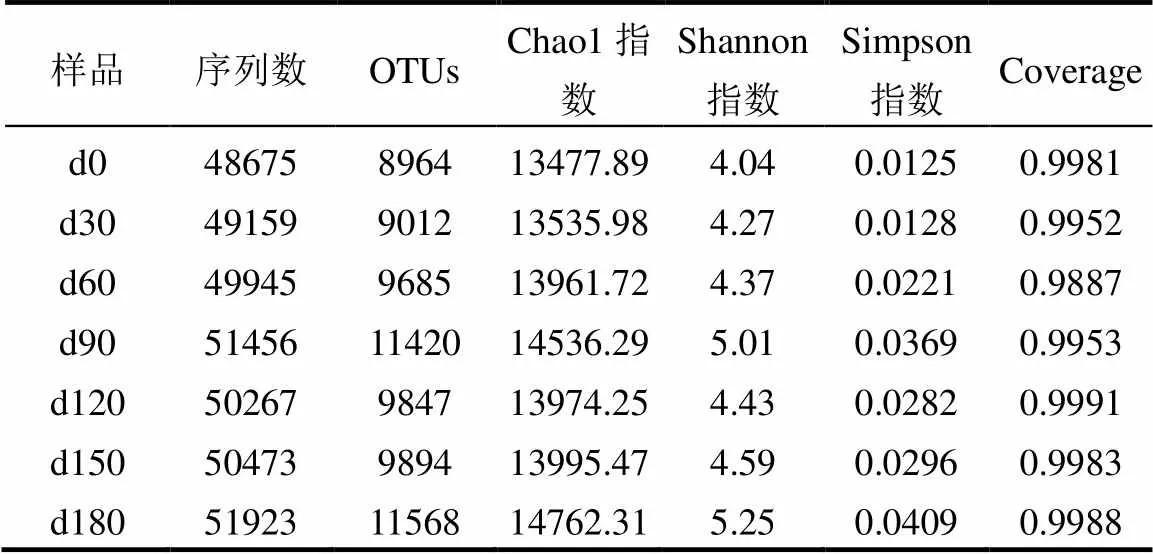
表2 生物膜菌群丰度和多样性指数
从图4(b)属水平热图可知,生物膜菌群结构优势属为norank_f_ODP1230B8.23 (9.06%~18.45%)、unclassified_o_Micrococcales (6.10%~12.31%)、norank_f_Anaerolineaceae (3.61%~8.24%)、norank_ p_Saccharibacteria (3.18%~6.85%)和Luteococcus (3.07%~5.83%).研究表明Thauera (陶厄氏菌属)、Ottowia、unclassified_o_Rhizobiales (根瘤菌属)和Thiobacillus (硫杆菌属)为几种常见的苯酚、SCN-和CN-降解菌[29-30].从图4(b)可以看出,Thauera、Ottowia和unclassified_o_Rhizobiales在反应60和90d时丰度较大,分别为2.11%和2.16%、1.63%和1.61%、2.40%和3.02%,由此可以说明此阶段苯酚迅速降解.Thiobacillus在反应120和150d时丰度较大,为1.70%和2.13%,此阶段SCN-和CN-去除率达到最大.而Nitrospira (硝化螺旋菌属)整个反应期间均被检出,丰度为1.08%~3.43%,总氮去除率相比于活性污泥法具有较大改善,氨氮去除受到抑制后仍能恢复到较高水平,再次证明了SMBBR具有较好的脱氮效果.
2.3 废水变量与微生物群落结构的关系
Yuste等[31]研究表明微生物的繁殖代谢与所处的环境变量息息相关.由废水中污染物的降解转化所引起的环境改变可直接影响微生物菌群间的竞争关系,从而导致优势菌群丰度的改变.表3为通过Mantel测试的相关性分析来评估废水变量与生物膜菌群结构的可能联系.
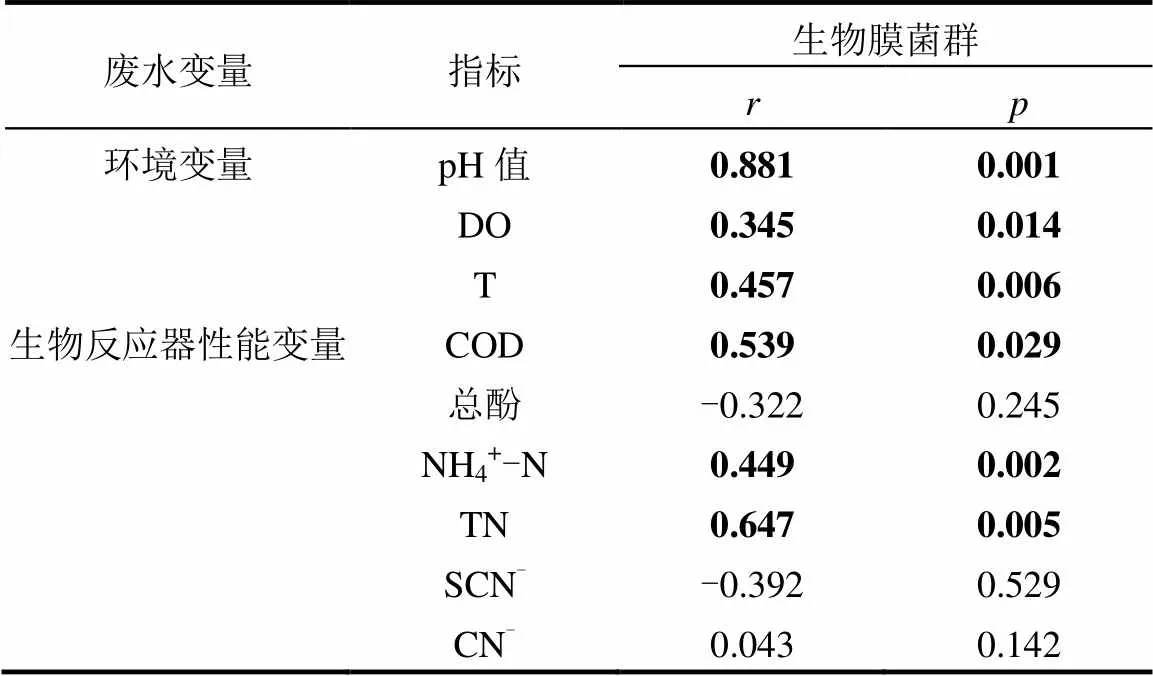
表3 由Mantel试验所得废水变量与菌群结构相关性
注:代表统计相关系数,代表值,粗体数字表示显著相关性(<0.05).
从表3可以看出,生物膜菌群结构与环境变量pH值(=0.881,=0.001)、DO (=0.345,=0.014)和温度(T) (=0.457,=0.006)均显著相关.为了辨别细菌群落的可能关键功能,还分析了生物反应器性能与系统发育群落结构之间的关系.结果显示,生物膜菌群结构与生物反应器性能变量COD (=0.539,= 0.029)、NH4+-N (=0.449,=0.002)和TN (=0.647,= 0.005)均显著相关.本文发现NH4+-N浓度在反应过程中对生物膜菌群结构及其功能产生毒性作用,而pH值可以控制游离氨和铵离子之间的平衡来维持系统的稳定性[32-33].因此,本文发现pH值与生物膜菌群结构显著相关.
采用CCA分析进一步研究废水变量对微生物菌群结构的影响作用大小,基于999个蒙特卡罗排列的自动正向选择和方差膨胀因子,在CCA中包含了3个环境变量(pH值、DO、T)和3个生物反应器性能变量(COD、NH4+-N、TN)如图5所示.图中废水变量用带有箭头的线段表示,线段的长短代表影响的大小;三角符号代表属水平下丰度前10的菌种,由菌种向废水变量引垂线,垂点距离变量箭头的相对位置越近,表示该菌种与该变量的正相关性越大;废水变量之间的夹角代表其相关性,为锐角时说明两废水变量之间是正相关.

图5 废水变量与菌群结构的CCA分析
S1: norank_f_ODP1230B8.23; S2: unclassified_o_Micrococcales; S3: norank_f_Anaerolineaceae; S4: norank_p_Saccharibacteria; S5: Luteococcus; S6: norank_f_OM1_clade; S7: norank_o_JG30-KF-CM45; S8: Woodsholea; S9: norank_p_WS6; S10: Nitrospira
从图5微生物菌群结构属水平上的CCA分析结果可知,第一轴与pH值、COD、NH4+-N和TN呈正相关,与DO、T呈负相关;第二轴与DO、pH值、COD和NH4+-N呈正相关,与T和TN呈负相关.与T、TN呈正相关,与pH值呈负相关;与pH值、DO、T呈显著正相关,与TN呈负相关;与pH值、COD、NH4+-N呈显著正相关,与T呈负相关.废水变量间COD和NH4+-N呈显著正相关.
整体来看,pH值、T、COD和NH4+-N均为影响生物膜菌群结构的主要环境因子.其中,pH值对菌群结构影响作用最大.由菌种向pH值变量引垂线可知,pH值与S10:(硝化菌属)正相关性最大,Klankeo等[34]研究表明硝化菌对环境条件pH值的变化异常敏感,pH值能够影响硝化菌的增殖速率和酶的活性.因此,有效控制pH值可有助于实现焦化废水好氧处理过程中最佳的生物脱氮性能和系统稳定性.
3 结论
3.1 SMBBR中的生物膜在克服焦化废水毒性抑制并适应环境后,能够有效降解焦化废水中的污染物质,尤其具有较强的脱氮能力.
3.2 生物膜菌群结构组成中优势门为Proteobacteria,核心属为norank_f_ODP1230B8.23、unclassified_o_Micrococcales和norank_f_ Anaerolineaceae.此外,Thauera、Ottowia、unclassified_ o_Rhizobiales和Thiobacillus为系统中苯酚、SCN-和CN-的降解优势菌.
3.3 pH、T、COD和NH4+-N均为影响生物膜菌群结构的主要环境因子,其中pH值与正相关性最大,有效控制pH值可有助于实现废水处理最佳的生物反应器性能和系统稳定性.
[1] Sharma N K, Philip L. Combined Biological and Photocatalytic Treatment of Real Coke Oven Wastewater [J]. Chemical Engineering Journal, 2016,295:20-28.
[2] 蒙小俊,李海波,盛宇星,等.焦化废水活性污泥PAH双加氧酶基因多样性分析 [J]. 中国环境科学, 2017,37(1):367-372. Meng X J, Li H B, Sheng Y X, et al. Diversity of PAH dioxygenase genes of activated sludge from coking wastewater [J]. China Environmental Science, 2017,37(1):367-372.
[3] Liu Y, Liu J, Zhang A, et al. Treatment effects and genotoxicity relevance of the toxic organic pollutants in semi-coking wastewater by combined treatment process [J]. Environmental Pollution, 2016, 220(A):13-19.
[4] Zhang M, Tay J H, Qian Y, et al. Coke plant wastewater treatment by fixed biofilm system for COD and NH3-N removal [J]. Water Research, 1998,32(2):519-527.
[5] Li H, Cao H, Li Y, et al. Innovative Biological Process for Treatment of Coking Wastewater [J]. Environmental Engineering Science, 2010, 27(4):313-322.
[6] Wang Y Q, Lv Y L, Shan M J, et al. Engineering Application Research of Enhanced Microbiological Treatment for Coking Wastewater [J]. Applied Mechanics and Materials, 2011,108:257-261.
[7] Sharma N K, Philip L. Treatment of Phenolics, Aromatic Hydrocarbons, and Cyanide-Bearing Wastewater in Individual and Combined Anaerobic, Aerobic, and Anoxic Bioreactors [J]. Applied Biochemistry and Biotechnology, 2014,175(1):300-322.
[8] 刘国新,吴海珍,孙胜利,等.市政污泥接种焦化废水好氧降解能力及微生物群落演替的响应分析[J]. 环境科学, 2017,38(9):3807-3816.Liu G X, Wu H Z, Sun S L, et al. Aerobic degradation and microbial community succession of coking wastewater with municipal sludge [J]. Environmental Science, 2017,38(9):3807-3816.
[9] 蒙小俊,李海波,曹宏斌,等.焦化废水活性污泥细菌菌群结构分析[J]. 环境科学, 2016,37(10):3923-3930. Meng X J, Li H B, Cao H B, et al. Bacterial community composition of activated sludge from coking wastewater [J]. Environmental Science, 2016,37(10):3923-3930.
[10] Ma Q, Qu Y, Shen W,et al. Bacterial community compositions of coking wastewater treatment plants in steel industry revealed by Illumina high-throughput sequencing [J]. Bioresource Technology, 2015,179:436-443.
[11] Zhu S, Wu H, Wei C, et al. Erratum to:Contrasting microbial community composition and function perspective in sections of a full- scale coking wastewater treatment system [J]. Applied Microbiology and Biotechnology, 2016,100(2):949-960.
[12] Zhao J L, Jiang Y X, Yan B, et al. Multispecies acute toxicity evaluation of wastewaters from different treatment stages in a coking wastewater-treatment plant [J]. Environmental Toxicology and Chemistry, 2014,33(9):1967-1975.
[13] 李卫平,李 杰,朱浩君,等. SMBBR工艺不同填料处理生活污水[J]. 环境工程学报, 2015,9(9):4427-4433. Li W P, Li J, Zhu H J, et al. Treatment of domestic sewage by SMBBR process with different fillers [J]. Chinese Journal of Environmental Engineering, 2015,9(9):4427-4433.
[14] Ma D H, Liu C, Zhu X B, et al. Acute toxicity and chemical evaluation of coking wastewater under biological and advanced physicochemical treatment processes [J]. Environmental Science and Pollution Research, 2016,23(18):18343-18352.
[15] 国家环境保护总局《水和废水监测分析方法》编委会.水和废水监测分析方法[M]. 4版.北京:中国环境科学出版社, 2002:131-284. Editorial board of the State Environmental Protection Administration. Water and wastewater monitoring and analysis methods. The fouith edition [M]. Beijing: China Environmental Science Press, 2002: 131-284.
[16] 张 琲,解立平,秦梓雅,等.污泥水蒸气气化焦油化学组分的分布特征 [J]. 中国环境科学, 2018,38(12):193-200. Zhang B, Xie L P, Qin Z Y, et al. Distribution characteristics of chemical components of the tar from sewage sludge steam gasification [J]. China Envirocnmental Sciene, 2018,38(12):193-200.
[17] Joshi D R, Zhang Y, Gao Y, et al. Biotransformation of nitrogen- and sulfur-containing pollutants during coking wastewater treatment: Correspondence of performance to microbial community functional structure [J]. Water Research, 2017,121:338-348.
[18] 潘霞霞,李媛媛,黄会静,等.焦化废水中硫氰化物的生物降解及其与苯酚、氨氮的交互影响[J]. 化工学报, 2009,60(12):3089-3096. Pan X X, Li Y Y, Huang H J, et al. Biodegradation of thiocyanate and inhibitory interaction with phenol , ammonia in coking wastewater [J]. CIESC Journal, 2009,60(12):3089-3096.
[19] Staib C, Lant P. Thiocyanate degradation during activated sludge treatment of coke-ovens wastewater [J]. Biochemical Engineering Journal, 2007,34(2):122-130.
[20] Vazquez I, Rodriguez J, Maranon E, et al. Simultaneous removal of phenol, ammonium and thiocyanate from coke wastewater by aerobic biodegradation [J]. Journal of Hazardous Materials, 2006,137(3): 1773-1780.
[21] Joshi D R, Zhang Y, Tian Z, et al. Performance and microbial community composition in a long-term sequential anaerobic-aerobic bioreactor operation treating coking wastewater [J]. Applied Microbiology and Biotechnology, 2016,100(18):8191-8202.
[22] 莘明亮,隋秀斌,朱浩君,等. A/SMBBR工艺处理高氨氮戊二胺废水[J]. 水处理技术, 2017,43(6):114-117. Xin M L, Sui X B, Zhu H J, et al. Pilot-scale study on A/SMBBR process for high ammonia nitrogen DN5wastewater treatment [J]. Technology of Water Treatment, 2017,43(6):114-117.
[23] 梁东博,卞 伟,王文啸,等.低温条件下好氧颗粒污泥培养及其脱氮性能研究 [J]. 中国环境科学, 2019,39(2):188-194. Liang D B, Bian W, Wang W X, et al. Aerobic granular sludge formation and nutrients removal characteristics under low temperature [J]. China Envirocnmental Sciene, 2019,39(2):188-194.
[24] Wang W, Ma W, Han H, et al. Thermophilic anaerobic digestion of Lurgi coal gasification wastewater in a UASB reactor [J]. Bioresource Technology, 2011,102(3):2441-2447.
[25] Zhang T, Ding L, Ren H, et al. Ammonium nitrogen removal from coking wastewater by chemical precipitation recycle technology [J]. Water Research, 2009,43(20):5209-5215.
[26] Manefield M, Griffiths R I, Leigh M B, et al. Functional and compositional comparison of two activated sludge communities remediating coking effluent [J]. Environmental microbiology, 2005, 7(5):715-722.
[27] Ibarbalz F M, Figuerola E L, Erijman L. Industrial activated sludge exhibit unique bacterial community composition at high taxonomic ranks [J]. Water Research, 2013,47(11):3854-3864.
[28] Kim Y M, Park H, Cho K H, et al. Long term assessment of factors affecting nitrifying bacteria communities and N-removal in a full- scale biological process treating high strength hazardous wastewater [J]. Bioresource Technology, 2013,134:180-189.
[29] 葛启隆,岳秀萍,王国英.一株苯酚降解菌的分离鉴定及响应面法优化其固定化 [J]. 中国环境科学, 2014,34(2):518-525. Ge Q L, Yue X P, Wang G Y. Isolation and identification of a phenol-degrading strain and optimization for its immobilization using response surface methodology [J]. China Envirocnmental Sciene, 2014, 34(2):518-525.
[30] Felfoldi T, Szekely A, Goral R, et al. Polyphasic bacterial community analysis of an aerobic activated sludge removing phenols and thiocyanate from coke plant effluent [J]. Bioresource Technology, 2010,101(10):3406-3414.
[31] Yuste L, Corbella M E, Turiegano M J, et al. Characterization of bacterial strains able to grow on high molecular mass residues from crude oil processing [J]. Fems Microbiology Ecology, 2000,32(1): 69-75.
[32] Lay-Son M, Drakides C. New approach to optimize operational conditions for the biological treatment of a high-strength thiocyanate and ammonium waste: pH as key factor [J]. Water Research, 2008, 42(3):774-780.
[33] 甄建园,于德爽,王晓霞,等.低C/N(<3)条件下SNEDPR系统启动及其脱氮除磷特性研究 [J]. 中国环境科学, 2018,38(8):2960-2967. Zhen J Y, Yu D S, Wang X X, et al. The nutrient removal characteristic of SNEDPR system during start-up and steady operation phases treating low C/N (<3)sewage [J]. China Envirocnmental Sciene, 2018, 38(8):2960-2967.
[34] Klankeo P, Nopcharoenkul W, Pinyakong O. Two novel pyrene-degradingsp. andsp. isolated from soil [J]. Journal of Bioscience & Bioengineering, 2009, 108(6):488-495.
Performance and flora structure response relation in the SMBBR treatment of the coking wastewater.
LI Wei-ping, HAO Meng-ying, JING Shuang-yi, YU Ling-hong, SUN Yan-bai, YANG Wen-huan*
(School of Energy and Environment, Inner Mongolia University of Science and Technology, Baotou 014010, China)., 2019,39(8):3332~3339
In this paper, a specific moving bed biofilm reactor (SMBBR), was engaged to treat the coking wastewater with its treatment performance being monitored continuously. The response relation between pollutants degradation and biofilm flora structure was illustrated by HS-GC/MS and Illumina high-throughput sequencing, and the influence of wastewater variables on microbial flora structure was analyzed by CCA. Results showed that the total phenol removal rate was 96.62percent when the system was operated stably for 50days, the thiocyanide and cyanide were completely degraded at 100days, phenol, thiocyanide and cyanide had toxic inhibition on NH4+-N degradation. HS-GC/MS results exhibited that after the aerobic treatment more than 80percent of the organic pollutants, including all phenols, partially the N, O heterocyclic compounds and long-chain alkanes were removed. The sequencing results displayed that there existed difference in the biofilm flora abundance and diversity based on the change of the reaction time. Proteobacteria, which promotes the degradation of phenol, had the highest relative abundance during the reaction. The dominant genera were,, and. In addition,,,andproved to be the dominant degrading bacterium of phenol, SCN-and CN-in the system. CCA analysis exhibited that the pH value is the most positively correlated with, and effective pH control can contribute to the stable operation of the nitrification reaction. All these provide a theoretical basis for the biofilm treatment of the coking wastewater.
SMBBR;coking wastewater;aerobic degradation;microbial structure;wastewater variable
X703.1
A
1000-6923(2019)08-3332-08
李卫平(1973-),男,陕西神木人,教授,博士,主要从事水处理与水环境控制研究.发表论文40余篇.
2019-02-18
内蒙古自然科学基金资助项目(2018LH003);内蒙古自治区高校项目(NJZY16164);包头科技计划重点领域技术攻关项目(2017Z1009-1)
* 责任作者, 副教授, yangwenhuan80@163.com
