小麦冬前最大分蘖期根数的QTL定位
2019-08-23尹国红成东梅于金林赵伟峰田纪春邓志英
彭 涛,尹国红,成东梅,于金林,赵伟峰,高 燕,田纪春,邓志英
(1.山东农业大学/作物生物学国家重点实验室,山东 泰安 271018; 2.济源市农业科学院,河南 济源 459002)
根系是植物吸收水分和养分的重要器官,其生长发育状况直接影响植株地上部的发育和产量[1-4]。小麦是须根系作物,其根系由初生根和次生根组成[5-6]。初生根由种子胚根发育而成,一般于小麦种子萌发后7~10 d集中发生,功能期主要集中在出苗到拔节期。次生根的发生,一般从小麦植株分蘖期开始,到抽穗开花时停止发育[6],其发生有2个高峰,一个是冬前最大分蘖期,另一个是拔节到抽穗期。薛丽华等[7]研究发现,冬前最大分蘖期的次生根数与小麦的有效穗数密切相关。李鸿斐等[8]研究表明,强筋小麦冬前最大分蘖期的次生根数显著高于中筋和弱筋小麦。综上,小麦冬前最大分蘖期的初生根数、次生根数及总根数等根系性状对小麦的产量和品质具有重要的影响。
CARADUS[9]分析作物不同根系形态性状的遗传力发现,根数、根长、根体积、根表面积、根直径、根质量、根冠比、根毛长度等衡量根系形态的指标具有较高的遗传力,表现出典型的数量性状遗传。由于根系深埋在土壤中,存在工作量大、难以获得完整准确的根系数据等问题,所以大多数根系研究主要利用水培或凝胶培养的方式在室内模拟进行根数、最大根长、根直径、根表面积、根体积、根鲜质量、根干质量等的遗传分析[10-15],而在大田土壤环境条件下开展小麦根系性状的QTL定位分析的研究较少[16]。迄今,仅见王升星等[16]在大田土壤环境条件下以196份小麦自然群体为材料,于小麦越冬前、拔节期进行次生根数测定,利用185对SSR标记进行基因型关联分析,共关联到32个显著标记位点,分布于14条染色体上,其中,Barc81(1BL)、Wmc617(4DS)和Gwm190(5DS) 在3个环境下与次生根数稳定关联。但其并未对总根数、初生根数进行QTL定位分析。为此,本研究利用在根系性状方面差异显著的花培3号和豫麦57及其构建的含有168个株系的加倍单倍体(DH)群体为材料,在3种土壤栽培环境条件下,对小麦冬前最大分蘖期根数(初生根数、次生根数和总根数)进行测定及QTL定位分析,旨在揭示小麦冬前最大分蘖期根数的分子遗传基础,以期找到有助于培育健壮根系的新的QTL,为小麦根系重要功能基因的克隆及分子标记辅助选择育种奠定基础。
1 材料和方法
1.1 试验材料
试验材料为168个DH群体,其由高产多抗的花培3号(母本),优质、高产、综合性状优良的豫麦57(父本)杂交产生的F1通过花药培养,并经染色体加倍获得。
DH群体的遗传图谱由本实验室于2008年构建[17],含有定位于小麦21条染色体的323个位点(包括284个SSR、37个EST-SSR、1个ISSR和1个HMW-GS标记),使用MapChart 2.1[18]软件作图。图谱全长2 485.7 cM,平均2个标记间的遗传距离是7.67 cM,形成24个连锁群。
1.2 试验设计
2013—2014年进行盆栽试验,于山东农业大学网室内进行,简称泰安市盆栽试验(E3)。选取DH群体168个家系及亲本大小均匀一致、饱满完整的种子各20粒,浸泡于10% H2O2中30 min,然后用蒸馏水冲洗干净,播种于带纱网孔的发芽板上,置入光照培养箱中。设置白天温度为(22±2)℃,光照16 h,夜间温度为(14±2)℃,黑暗8 h。种子萌发后,从每个家系中挑取12株生长健壮、长势一致的幼苗,分别移栽到3个塑料盆中,重复3次。塑料盆底部有孔,盆深33 cm,上口直径为32 cm,下口直径为26 cm,盆栽土壤由75%农田土壤和25%细砂土混合而成,其中,农田土壤过筛去除石子、根茎等杂质。每盆装土15 kg,装土前拌入复合肥(含总养分45%,N∶P2O5∶K2O=15∶15∶15) 4 g。将土壤和肥料充分混匀后装盆并浇水沉实,次日移栽。移栽日期为10月12日。在小麦幼苗生长期,及时防治杂草和病虫害,进行正常的水肥管理。
2014—2015年进行田间栽培试验,试验材料种植在河南省济源市农业科学院试验田和山东农业大学试验农场,简称济源市田间试验(E1)和泰安市田间试验(E2),完全随机区组设计,2次重复,3行区种植,行长2 m,行距0.20 m,播种日期分别为10月5日和10月10日,基本苗150万株/hm2。按当地小麦产量比较试验方案进行田间管理。
1.3 测定项目及方法
对于盆栽试验,于12月22日取样,取样前先用水管浇透各塑料盆土壤,再将塑料盆浸泡在水中,然后仔细清洗各单株根系。每个家系随机选6株,统计小麦冬前最大分蘖期初生根数、次生根数和总根数。
对于田间试验,山东省泰安市试验田和河南省济源市试验田的小麦材料分别于12月20日和12月25日取样,用铁铲小心挖出带土幼苗,再把带土幼苗浸泡在水中,然后仔细清洗各单株根系,统计小麦冬前最大分蘖期初生根数、次生根数和总根数。
1.4 数据处理和QTL分析
利用Excel 2003对数据进行汇总和整理,使用SPSS 19.0对数据进行正态分布检验、方差分析和相关性分析。
利用基于混合线性模型[19]的复合区间作图软件QTLNetwork 2.1[20]对小麦冬前最大分蘖期初生根数、次生根数和总根数等进行QTL分析。QTLNetwork 2.1软件的扫描窗口设定为10 cM,步长设定为1 cM,以单个环境中3个重复的平均值输入数据,以P=0.005为统计检测阈值,即当某个根数性状分子标记的P值小于统计检测阈值时,认为该标记处存在1个与根数性状有关的QTL。最后将检测到的所有QTL以及它们之间的上位性互作整合到一个全QTL模型中,用基于Gibbs抽样的贝叶斯方法估计遗传效应。按照 MCINTOSH等[21]的方法命名QTL。
2 结果与分析
2.1 小麦冬前最大分蘖期根数分析
亲本豫麦57的初生根数、次生根数和总根数均高于花培3号,尤其是次生根数和总根数。在DH群体中,冬前最大分蘖期初生根数、次生根数和总根数变异范围较大。与2个田间试验相比,盆栽条件下根数增多,变异范围更大(表1)。不同环境条件下初生根数、次生根数和总根数3个性状的偏度和峰度均较小,呈正态分布(表1,图1—3),说明该群体适合对根数进行QTL定位。
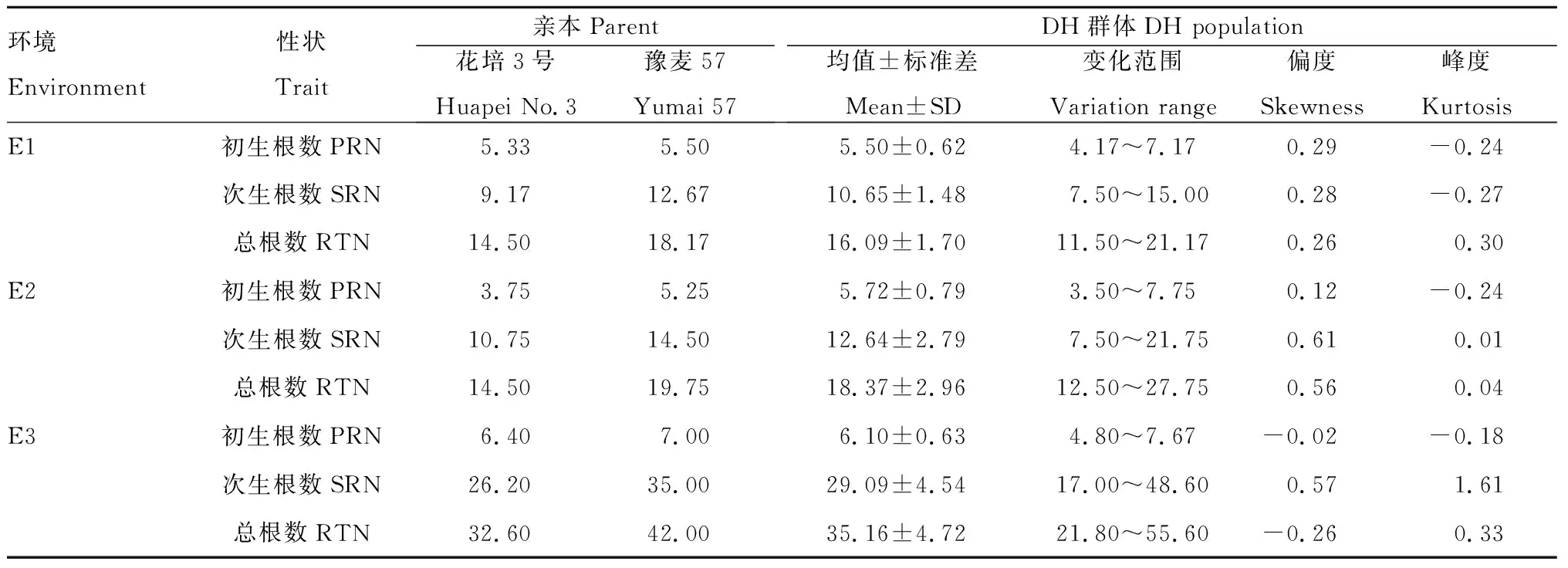
表1 小麦冬前最大分蘖期DH群体根数的分析Tab.1 Analysis of root number of wheat DH population at maximum tillering stage before winter
注:E1:济源市田间试验,E2:泰安市田间试验,E3:泰安市盆栽试验;PRN:初生根数,SRN:次生根数,RTN:总根数。下同。
Note:E1 represents field test in Jiyuan City,E2 represents field test in Tai’an City,and E3 represents pot culture test in Tai’an City;PRN represents primary root number,SRN represents secondary root number,and RTN represents total root number.The same as below.
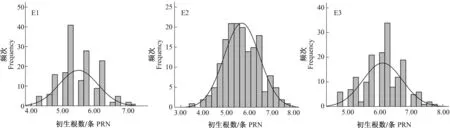
图1 3个环境下小麦DH群体初生根数分布Fig.1 Distribution of primary root number of wheat DH population under three environments
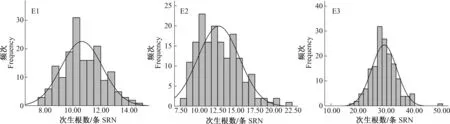
图2 3个环境下DH群体次生根数分布
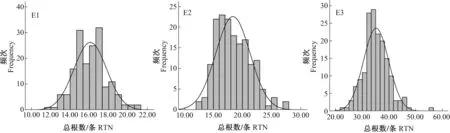
图3 3个环境下DH群体总根数分布
2.2 小麦冬前最大分蘖期根数性状间的相关性分析
由表2可知,小麦冬前最大分蘖期总根数与初生根数、次生根数均呈极显著正相关。与初生根数相比,次生根数与总根数相关性更高,在3个环境下相关系数分别为0.900、0.964、0.732,说明总根数主要由次生根数决定。次生根数与初生根数均呈正相关,但均未达到显著水平。

表2 小麦冬前最大分蘖期DH群体根数性状间的相关性分析(E1/E2/E3)Tab.2 Correlation analysis among root number traits of wheat DH population at maximum tillering stage before winter
注:**表示在0.01水平相关性极显著。
Note:** represents significant correlation at 0.01 level.
2.3 小麦冬前最大分蘖期根数的QTL定位及效应分析
3个栽培环境下,共定位了7个加性效应QTL (表3和图4) 和7对上位性互作QTL (表4),分布在1B、2B、2D、3B、4A、4D、5D、6B、6D、7D染色体上。
对于初生根数,仅在E2环境下检测到1个控制初生根数的加性效应QTL,位于2B染色体上,表型贡献率为7.03%,增效等位基因来源于母本花培3号。E2环境下未检测到上位性互作QTL。
对于次生根数,在3个环境下均检测到控制次生根数的加性效应QTL,共4个,分别位于1B、4A、4D染色体上,可解释表型变异的4.67%~16.56%,其中2个为主效QTL,位于1B染色体上的qSrn1B具有最大的表型贡献率,可解释16.56%的表型变异,其增效基因来源于父本豫麦57;位于4D染色体上的qSrn4D可解释10.70%的表型变异,其增效基因来源于母本花培3号。在E1和E3环境下,检测到3对控制次生根数的上位性互作QTL,分别位于染色体1B-2B、2B-4A、3B-5D上,可分别解释6.90%、8.24%、5.24%的表型变异。
对于总根数,在E2和E3环境下,共检测到影响总根数的2个加性效应QTL,分别位于1B和6D染色体上,可分别解释表型变异的12.80%和9.17%,其中,位于1B染色体上的qRtn1B是主效QTL,其增效基因来源于父本豫麦57。在E1和E3环境下,检测到4对控制总根数的上位性互作QTL,分别位于染色体6B-7D、2B-4A、2D-6D、3B-5D上,可分别解释14.45%、9.78%、7.57%、5.65%的表型变异,其中1对位于染色体6B-7D上的上位性互作 QTL(qRtn6B和qRtn7D)的表型贡献率达14.45%,表明上位性效应对该群体冬前最大分蘖期总根数的影响较大。

表3 小麦冬前最大分蘖期DH群体根数的加性效应QTL Tab.3 Additive QTLs for root number of wheat DH population at maximum tillering stage before winter
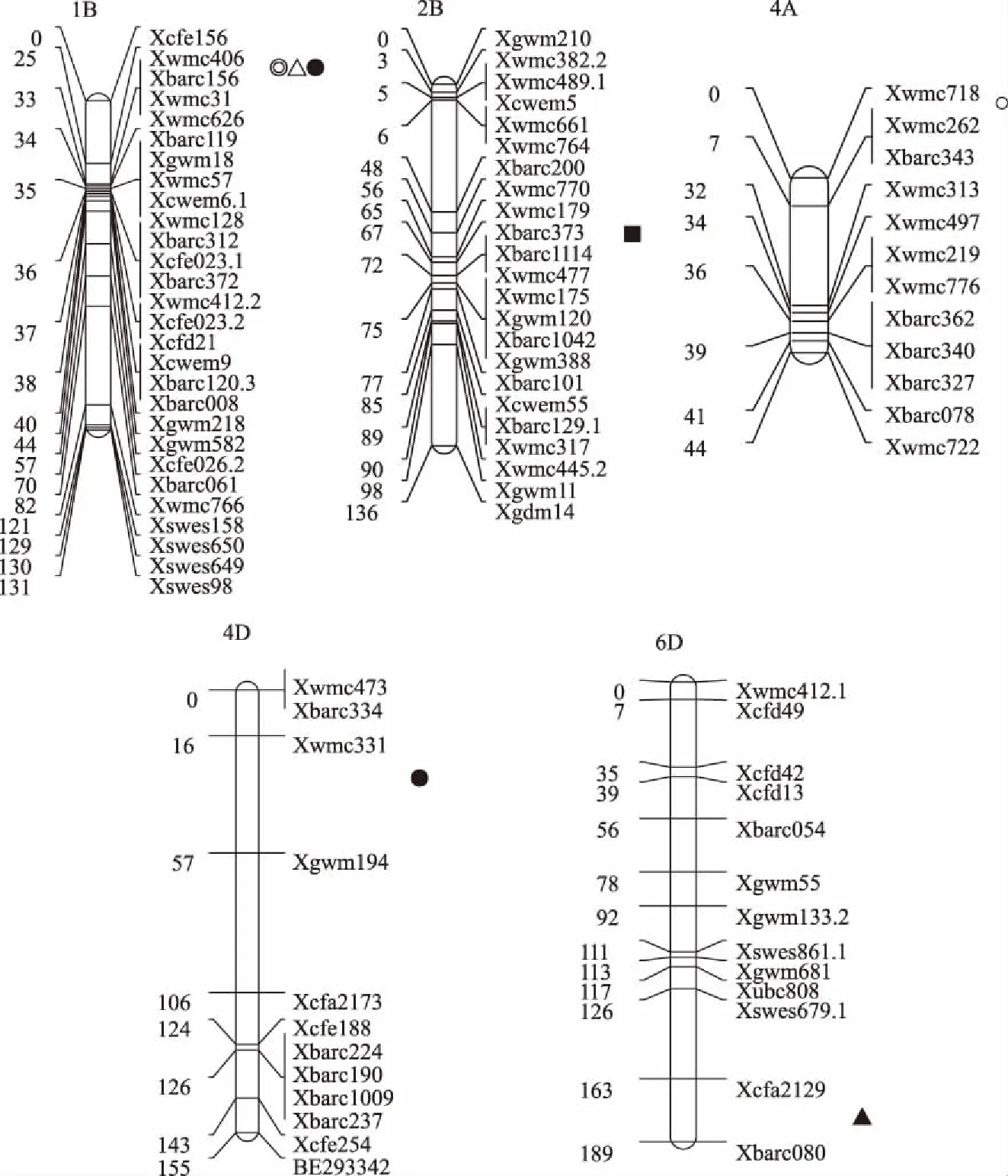
■代表E2环境下的初生根数,○、◎ and ●分别代表E1、E2、E3环境下的次生根数,△、▲分别代表E2、E3环境下的总根数
总体来看,位于1B染色体上影响次生根数的QTL在2个泰安市环境下均被检测到,该位点对次生根数能解释4.67%~16.56%的表型变异,且该位点来源于父本豫麦57,为1个典型的主效QTL位点。但该位点在E1环境下未检测到,需增加检测年份或地点,以确定该位点在不同环境下的表达特性。在E3环境下,分别在2B-4A、3B-5D染色体上检测到对次生根数、总根数有共同效应的上位性互作QTL,可解释表型变异的5.24%~9.78%。

表4 小麦冬前最大分蘖期DH群体根数的上位性互作QTL位点Tab.4 Epistatic QTLs for root number of wheat DH population at maximum tillering stage before winter
3 结论与讨论
因根系深埋在土壤中,目前国内外对小麦根系性状的遗传定位研究主要基于水培条件,而本研究在土壤条件下测定小麦DH群体的根数,其结果更具有实际参考价值。
本研究在田间环境和盆栽环境条件下,以花培3号和豫麦57杂交获得的168个株系的DH群体为试验材料,在土壤栽培条件下对冬前最大分蘖期的根系性状进行了QTL定位分析,共定位了小麦冬前最大分蘖期初生根数、次生根数、总根数3个根系性状的7个加性效应QTL和7对上位性互作QTL,分布在1B、2B、2D、3B、4A、4D、5D、6B、6D、7D染色体上,单个QTL可以解释4.67%~16.56%的遗传变异。根数的加性效应QTL分别定位在1B、2B、4A、4D、6D染色体上,除1B染色体上的位点来自于父本豫麦57外,其余位点均来自于母本花培3号。其中,单个QTL对根数的表型贡献率大于10%的主效QTL有3个,包括2个次生根数QTLqSrn1B(16.56%)、qSrn4D(10.70%)和1个总根数QTLqRtn1B(12.80%)。在1B染色体的标记区间Xwmc406—Xbarc156同时存在控制次生根数、总根数的QTLqSrn1B、qRtn1B,分别解释次生根数、总根数表型变异的16.56%、12.80%,可为小麦根系的分子标记辅助选择奠定基础。经对比分析发现,在1B染色体上Xwmc406—Xbarc156区间控制次生根数和总根数的QTL,与利用同一DH群体定位的小麦穗部可育小穗数[22]、小麦籽粒清蛋白和球蛋白含量[23]QTL位于相同区间位置,表明控制次生根数和总根数的基因可能同时控制小麦穗部可育小穗数、籽粒清蛋白和球蛋白含量,其对小麦产量和品质均能产生影响;在4A染色体上控制次生根数的QTL与利用同一DH群体定位的籽粒产量、总小穗数、可育小穗数[22]、幼苗根长[24]QTL位于相同区间位置,表明控制次生根数的基因可能同时控制小麦籽粒产量、总小穗数、可育小穗数和幼苗根长。
本研究在1B、4A、4D染色体上检测到在大田、盆栽环境下影响越冬前最大分蘖期次生根数的4个加性效应QTL,而王升星等[16]在1A、1B、2D、3A、3B、4A、4D、5A、5B、5D、6B、6D、7A、7B等14条染色体上关联到大田环境下控制越冬前、拔节期次生根数的32个显著标记位点,其中越冬前次生根数显著关联标记有11个,分布在1B、2D、3A、4D、5A、5D染色体上,表明1B、4A、4D等染色体上存在控制次生根数的重要位点。本研究在2B染色体上检测到1个控制初生根数的QTL,其所在标记区间与先前定位到的控制根数的QTL标记区间一致[13-15,25-28]。
通过比较发现,一些染色体区间是控制根系性状的热点区间,这与在不同作物中发现相关性状QTL常常定位于相同或相近染色体区段的研究结论一致[22,29-31]。本研究中,在染色体1B的 Xwmc406—Xbarc156标记区间同时存在控制次生根数和总根数的3个QTL,其中,2个主效位点qSrn1B和qRtn1B分别解释次生根数和总根数表型变异的16.56%和12.80%。王升星等[16]在该区间内关联到了2个控制次生根数的QTL;屈春艳[32]在该区间关联到了4个控制小麦根数的QTL,其中,QRn.sdau-1B.1的表型贡献率达13.54%;李振兴等[26]也在该区间定位了2个与根数相关的磷胁迫响应QTL,其中,1个QTL的表型贡献率达16.77%。染色体1B上具有控制次生根数和总根数的重要位点,该位点可能存在一因多效基因或紧密连锁基因,本研究为进一步在该区段内发掘控制根系相关性状的重要基因以及分子标记育种奠定了基础。
