新疆准东油田石油污染土壤萘双加氧酶基因的多态性
2019-08-20薄明森吕杰李兴海马媛
薄明森 吕杰 李兴海 马媛
摘要:为了分析新疆准东油田石油污染土壤中萘双加氧酶功能基因的多态性,采用无机盐培养基,以萘为唯一的碳源物质,对准东油田石油污染土壤中的萘降解菌进行液体富集培养,用高效液相色谱法(high performance liquid chromatography,简称HPLC)对降解产物进行检测,同时采用萘双加氧酶基因通用引物对萘双加氧酶基因片段进行扩增、测序及分析。高效液相色谱检测结果显示,萘被降解成小分子产物,证明液体培养基均富集培养获得了萘降解菌群。从准东油田共分离得到了49条萘双加氧酶基因片段,共编码35条氨基酸序列,GenBank登录号为KY304781~KY304829。获得的萘双加氧酶基因系统发育树结果显示,研究获得的萘双加氧酶基因分为4个类群。通过高效液相色谱分析及萘双加氧酶基因的分子检测,证明准东油田石油污染土壤中含有萘降解菌株,这些萘降解菌株是可以通过液体富集培养获得的,并且可以通过固体平板获得降解萘的单菌株。
关键词:萘降解菌;高效液相色谱;萘双加氧酶基因;克隆文库
中图分类号: S151.9+5;S182;X53文献标志码: A
文章编号:1002-1302(2019)09-0110-05
芳香族烃类化合物是煤炭、石油等化石燃料的天然组成成分,植物体内含有多种酚类产物,它们不完全燃烧亦可产生芳香烃类化合物。另外,在应对石油短缺问题时,“煤变油”过程中会产生大量结构复杂的芳香烃化合物[1]。极低含量的多环芳烃(polycyclic aromatic hydrocarbons,简称PAHs)便会对人体造成严重危害,是芳香烃化合物中对环境危害最大的一类[2]。PAHs家族包括150多种成员,其中16种被美国环境保护署列为优先考虑的污染物[3]。原油中含有2%~4%的芳香烃,1~5个不等的缩合芳烃[4],因此,从石油开采区原油污染土壤中可以检测到高含量的PAHs[5]。该类物质化学结构稳定,难溶于水,在自然环境中很难降解,因而如何消除此类有机污染物,在很长时间内一直困扰着人们。微生物降解被认为是去除PAHs的有效手段,随着微生物降解菌中双加氧酶的分离及研究的深入,这一难题正被安全有效地解决[6]。
双加氧酶基因是芳烃开环降解的第1步,是关键酶及限速酶基因。将自然界中的双加氧酶基因按照α亚基序列信息的聚类分析,可以分为4类:甲苯/联苯双加氧酶,萘或多环芳烃双加氧酶,苯甲酸双加氧酶,邻苯二甲酸双加氧酶。因为多环芳烃双加氧酶具有其独特的特征,所以能够利用芳香烃双加氧酶α亚基的保守域设计通用引物,用于研究环境中PAHs降解的功能菌群,是行之有效的手段[7-10]。
研究微生物降解PAHs的途径以及对降解过程中涉及的酶的研究可以为了解微生物对PAHs的转化能力及代谢方式提供基础,是将微生物降解应用于污染修复的前提。双加氧酶基因作用于多环芳烃氧化开环的初始步骤,其多态性及含量决定了土壤的自我修复能力。对于萘双加氧酶而言,因其活性中心具有相对较小的结构上的保守性,其底物范围相对比较广泛[1],因此,萘双加氧酶的基因也常被作为原油污染土壤中微生物菌群降解多环芳烃的指标物进行分子检测[5]。萘双加氧酶的催化能力差异较大,因此需要对萘双加氧酶基因资源进行多态性研究,并以此为指导依据,筛选高效降解菌进行污染物的原位修复仍是未来工作的热点[11]。
本研究采集新疆准东油田原油污染不同年限的沙质土壤,用以分析不同年限原油污染土壤中nah基因的多态性变化,同时以萘为唯一碳源,液体富集培养萘降解菌群,对可培养萘降解菌群中nah基因的多态性进行研究。通过对准东油田石油污染土壤中非培养、可培养萘降解菌群双加氧酶基因多态性的研究,揭示其原位基因的组成特征,并通过对可培养萘降解菌群双加氧酶基因的分析,研究降解菌群的培养规律。
1 材料与方法
1.1 材料
1.1.1 试验材料 准东油田采油区位于新疆准噶尔盆地东部,地理位置为88°40′~89°22′E,43°40′~44°20′N。试验采集准东油田同一个采油区3个不同石油污染年限的土壤,土壤样本编号为ZD1、ZD2、ZD3。于2015年6月采集样本,采集样本时与采油区工作人员了解具体情况,ZD1为废弃单罐井,石油污染时间为5年以上;ZD2为闸阀井,污染时间为1年(持续污染);ZD3位于采油井邊,污染时间为3年。采样时去除表面土壤,采集深度为2~20 cm,每个样点采集5份土样。将采集的土样装入无菌密封聚丙烯袋中,在低温下运送至实验室,于4 ℃保存备用。
1.1.2 试验试剂 PowerSoilTM DNA Isolation Kit土壤基因组DNA抽提试剂盒,购自MO BIO公司;细菌基因组DNA提取试剂盒,购自生工生物工程(上海)股份有限公司;LA Taq DNA聚合酶和DNA marker,购自大连TaKaRa公司;DNA凝胶回收试剂盒,购自杭州Axygen公司;pCR2.1-T vector,购自美国Invitrogen公司;DH5α大肠杆菌感受态细胞,购自北京全式金生物技术有限公司;Hha Ⅰ、Rsa Ⅰ、Hinf Ⅰ,购自美国NEB公司;其余试剂均为国产分析纯。
1.1.3 试验仪器 PCR仪(TC-412),购自TECHNE公司;凝胶成像仪,购自SYNGENE公司;离心机(SIGMA 3-18K),购自Sartorius AG公司;琼脂糖水平电泳仪(DYCP-31DN),购自北京六一生物科技有限公司。
1.1.4 培养基 液体富集培养基为无机盐基础培养基,由大量元素(99 mL)和微量元素(1 mL)2个部分组成。大量元素成分:1.0 g/L (NH4)2SO4,0.8 g/L K2HPO4,0.2 g/L KH2PO4,0.2 g/L MgSO4·7H2O,0.1 g/L CaCl2·2H2O,0.005 g/L FeSO4·7H2O。
微量元素成分:0.039 9 g/L MnSO4·H2O,0.042 8 g/L ZnSO4·H2O,0.034 7 g/L (NH4)6Mo7O24·4H2O[12]。pH值为7.0±0.2,于121 ℃灭菌30 min。在固体培养基中添加 1.5% 琼脂粉。
1.2 方法
1.2.1 液体富集培养萘降解菌群 等量混合5份石油污染土壤,称取10 g,加入100 mL液体富集培养基中,再加入 0.05 g 萘作为唯一碳源及能源物质,于180 r/min、32 ℃避光培养7 d。培养结束后吸取10 mL富集培养液,离心收集菌体,将无菌培养基在洗脱后加入新的液体培养基中,此过程重复5次。
1.2.2 高效液相色谱法(high performance liquid chromatography,简称HPLC)检测萘降解产物 高效液相色谱法检测的样本为液体培养96 h后的样本(外源添加的萘肉眼不可见),用15 mL乙酸乙酯萃取,重复3次。将含有降解产物的乙酸乙酯于12 000 r/min离心3 min,弃沉淀,干燥后加 1 mL 甲醇溶液溶解。测试条件:以甲醇和水(体积比为 82 ∶18)为流动相,流动速度为1 mL/min,进样量为10 μL,检测波长为 270 nm,采用C18柱,整个过程的温度为40 ℃[13]。
1.2.3 土壤基因组DNA的提取与萘降解菌基因组DNA的提取 等量混合每个样点采集的土样,分别称取0.4 g土样,采用PowerSoilTM DNA Isolation Kit土壤基因组DNA抽提试剂盒对土壤基因组DNA进行提取,操作过程按照说明书相关步骤进行。液体富集萘降解菌群采用生工生物工程(上海)股份有限公司生产的细菌基因组DNA提取试剂盒进行提取,提取时离心收集5 mL富集培养菌液中的菌体。3个土壤所对应的样品编号为ZD1、ZD2、ZD3,液体摇瓶对应的编号为Y。
1.2.4 萘双加氧酶基因片段的PCR扩增 采用萘双加氧酶基因(nah基因)的通用引物(NAH-F:5′-CAAAARCACCTGATTYATGG-3′;NAH-R:5′-AYRCGRGSGACTTCTTTCAA-3′),以不同石油污染年限的土壤總DNA和液体富集培养萘降解菌群总DNA为模板进行PCR扩增。PCR反应条件参照已报道的条件进行[14]。每个样本平行扩增3个重复,扩增结束后,电泳检测。
1.2.5 萘双加氧酶基因克隆文库的构建与限制性片段长度多态性(restriction fragment length polymorphism,简称RFLP)分型筛选阳性克隆子 为了避免单次扩增的偏向性,混合3个PCR扩增平行样本产物进行凝胶回收。将回收产物与 pUC18-T 载体连接,转化大肠杆菌(Escherichia coli) DH5α感受态细胞,每个样本随机挑取96个白斑,从而构建准东油田不同年限石油污染土壤以及液体富集培养萘降解菌群中萘双加氧酶基因的克隆文库。
用碱裂解法提取克隆文库质粒DNA,用M13-F、M13-R通用引物对进行载体PCR扩增,反应条件:94 ℃ 4 min;94 ℃ 30 s,51 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 5 min。对PCR产物进行琼脂糖凝胶电泳检测,对阳性克隆子进行筛选,之后将阳性克隆PCR产物用限制性内切酶HhaⅠ、RsaⅠ、Hinf Ⅰ进行3轮酶切,酶切产物用2.5%琼脂糖凝胶进行电泳检测,将不同RFLP条带类型对应的克隆子送至生工生物工程(上海)股份有限公司进行测序,将测序与比对正确的序列信息提交至GenBank数据库。
1.2.6 系统发育分析 将测序获得的萘双加氧酶基因序列在GenBank中进行BLAST同源性比对,下载相似性最高的萘双加氧酶基因作为标准序列,用MEGA 7软件,采用邻接法进行聚类分析,并构建系统进化树,并用该软件对萘双加氧酶的氨基酸序列进行聚类分析[15]。
2 结果与分析
2.1 萘降解菌群降解产物的检测结果
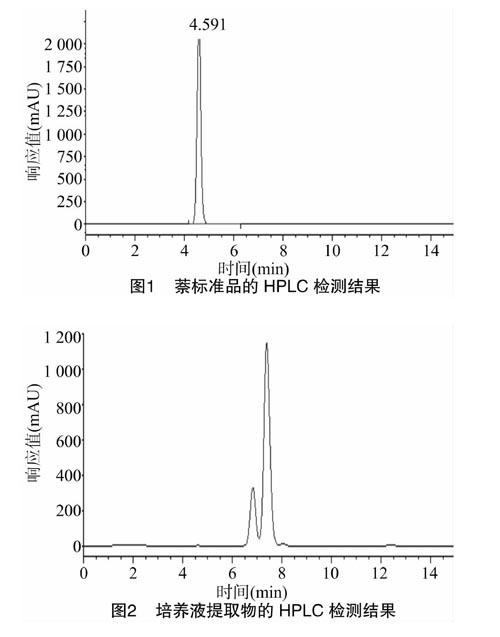
由图1和图2可以看出,萘标准品的保留时间为 4.591 min,而对萘降解培养液提取物进行检测时发现,在 7.5 min 时出现1个大的吸收峰,在6.7 min时出现1个小峰。HPLC试验结果表明,在液体培养基中添加的萘,在96 h后已被降解成2个分子量小于萘的降解产物,证明通过液体培养获得了准东油田萘降解菌的可培养菌群,而对于2个组分的降解产物,有待进一步研究其结构。
2.2 nah基因片段的PCR扩增结果
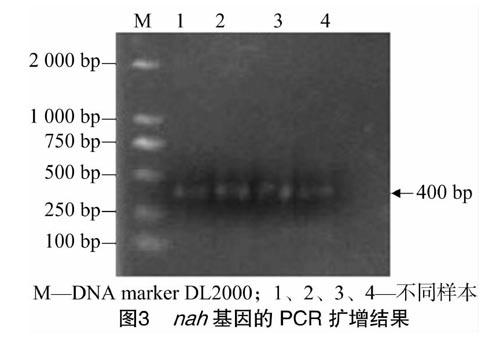
以不同石油污染年限的土壤总DNA和液体富集培养萘降解菌群的总DNA为模板,进行PCR扩增。如图3所示,通过PCR扩增,得到了清晰的特异性条带,条带大小为400 bp左右,目标条带大小与预期扩增的nah基因片段大小相符。
2.3 nah基因片段的RFLP分析结果
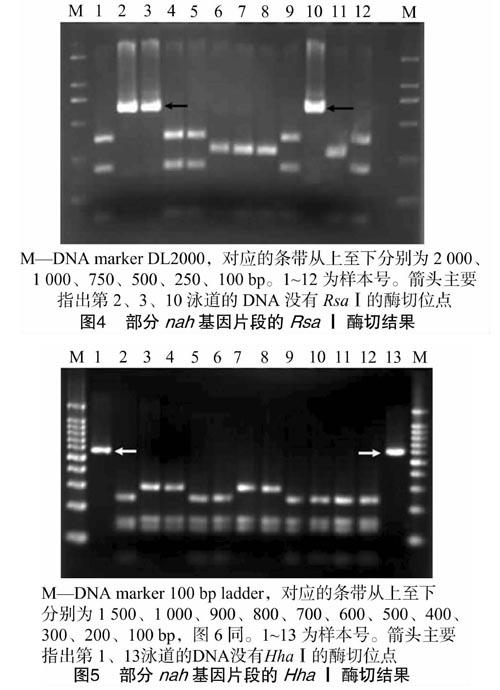
对克隆文库中的阳性克隆子进行PCR筛选,将筛选获得的阳性克隆子PCR产物用限制性内切酶Hha Ⅰ、Rsa Ⅰ和Hinf Ⅰ进行3轮酶切。原本预期采用2种识别4个碱基的限制性内切酶,但是nah基因的部分PCR条带不能被Hha Ⅰ、Rsa Ⅰ酶切,因此最终采用2种限制性内切酶。用3种限制性内切酶进行RFLP分析的部分结果如图4、图5和图6所示,进一步分析可知,ZD1共有18个带型,ZD2有13个带型,ZD3有13个带型,Y有12个带型。
2.4 萘双加氧酶基因的系统发育分析
将测序结果经过BLAST同源比对,去除错误结果,最终获得的nah基因片段数如下:ZD1有16条,ZD2有11条,ZD3有11条,Y有11条。将序列信息提交至GenBank数据库,GenBank登录号为KY304781~KY304829。
将获得的萘双加氧酶基因序列在GenBank中进行BLAST相似性比对,将相似性較高的序列作为标准序列,用MEGA 7软件,采用邻接法聚类分析构建的系统发育树。由图7的系统发育树可以看出,从准东油田石油污染土壤中获得的萘双加氧酶基因可以分为Cluster a(Cluster表示簇)、Cluster b、Cluster c、Cluster d 4个类群,其中Cluster b含有2个不同的小类群,Cluster c含有2个不同的小类群,Cluster d含有3个不同的小类群,每个小类群内的相似性在98%以内。Cluster a、Cluster b和Cluster c两两间的相似性均在95.5%以上,其中Cluster d与其他类群的相似性均低于85%。
萘双加氧酶BLAST同源比对得分最高的序列多来自石油污染土壤中的免培养细菌,仅2条序列来自Pseudomonas(假单胞菌属)菌株的萘双加氧酶基因,且这2条序列均存在于Cluster b类群中。此外,比对获得的来源于免培养细菌nah基因的标准序列,除登录号为KC878841的序列外,其他标准序列均来自大庆油田石油污染土壤[5],相似性均高于98%,而摇瓶培养获得的nah基因与Yang等研究得到的来源于克拉玛依油田土壤细菌的nah基因序列信息相比,相似性反而均低于97%[16]。
将萘双加氧酶基因序列翻译成氨基酸序列,用MEGA 7进行聚类分析,由图8可以看出,本试验获得的萘双加氧酶α亚基的核酸序列为49条,共编码35条氨基酸序列,与核酸序列聚类结果相同,共分为4个类群,其中Cluster b占整个文库的70%,Cluster a占整个文库的22%,为最大的2个类群。从聚类结果可以看出,来自3个不同石油污染年限土壤中的nah基因并没有很好地聚类,但是从摇瓶培养获得的nah基因却聚类成独自的类群。
3 结论
新疆地区的石油储量丰富,但是在几十年的开采过程中,难免会有原油污染物随着开采加工过程进入土壤中。准东油田采油区大部与古尔班通古特沙漠重合,其土壤微生态环境脆弱,原油渗漏极易破坏微生物菌群结构。原油渗漏后所含的烃类能给微生物提供足够的碳源,因此沙质土壤中的原核微生物多样性往往大于背景土壤,认为有机污染物降解主要由原核微生物完成[17]。目前未见针对新疆准东油田开展的原油污染土壤中芳香烃降解酶基因多态性以及石油污染土壤中不同污染物降解菌株分离的报道。
萘双加氧酶的底物范围相对比较广泛,因此萘双加氧酶基因也常被作为原油污染土壤中微生物菌群降解多环芳烃的指标物进行分子检测。本研究选择萘作为限制性培养因子,液体培养萘降解菌群,通过HPLC检测证明,通过试验富集培养获得了萘降解菌群。如果采用该菌群进行污染物的原位修复,会极大地避免采用外来菌株受到土著微生物的竞争捕食作用。此外本研究针对新疆准东油田原油污染土壤中来源的非培养和可培养萘降解菌的功能基因进行多态性研究。结果表明,从3个不同年限石油污染土壤内获得的nah基因并没有很好地聚类,但是从摇瓶中获得的nah基因却聚类形成独自的类群,证明萘降解菌群结构与环境中的营养物质成分相关,但是与萘含量并不相关。摇瓶获得的萘降解菌群的nah基因并未在环境样本中获得,说明该菌群在土壤中的含量极低。
通过对准东油田石油污染土壤中非培养和可培养萘降解菌群双加氧酶基因的多态性研究,揭示其原位功能基因的结构特征,并通过对可培养萘降解菌群双加氧酶的基因分析,研究降解菌群的培养规律,可以为进一步从准东油田石油污染土壤中获得萘降解单菌株及其他稠环芳香烃降解菌株提供借鉴。
参考文献:
[1]章 俭,夏春谷. 芳香烃双加氧酶的结构与功能研究[J]. 化学进展,2004,16(1):116-122.
[2]Ravelet C,Grosset C,Montuelle B,et al. Liquid chromatography study of pyrene degradation by two micromycetes in a freshwater sediment[J]. Chemosphere,2001,44(7):1541-1546.
[3]罗洪君,王绪远,赵 骞,等. 石油污染土壤生物修复技术的研究进展[J]. 四川环境,2007,26(3):104-109.
[4]Liang Y T,Zhang X,Wang J,et al. Spatial variations of hydrocarbon contamination and soil properties in oil exploring fields across China[J]. Journal of Hazardous Materials,2012,241(4):371-378.
[5]Yang Y Y,Wang J,Liao J Q,et al. Distribution of naphthalene dioxygenase genes in crude oil-contaminated soils[J]. Microbial Ecology,2014,68(4):785-793.
[6]宋兴良,王江涛,张 哲. 多环芳烃蒽高效降解菌的筛选及其降解中间产物分析[J]. 海洋环境科学,2010,29(6):815-818.
[7]Gibson D T,Parales R E. Aromatic hydrocarbon dioxygenases in environmental biotechnology[J]. Current Opinion in Biotechnology,2000,11(3):236-243.
[8]Abu-Omar M M,Loaiza A,Hontzeas N. Reaction mechanisms of mononuclear non-heme iron oxygenases[J]. Chemical Reviews,2005,105(6):2227-2252.
[9]Visser S P,Kumar D. Iron-containing enzymes versatile catalysts of hydroxylation reactions in nature[M]. London:Royal Society of Chemistry,2011:42-63.
[10]Iwai S,Johnson T A,Chai B L,et al. Comparison of the specificities and efficacies of primers for aromatic dioxygenase gene analysis of environmental samples[J]. Applied and Environmental Microbiology,2011,77(11):3551-3557.
[11]王 涛,蓝 慧,田 云,等. 多环芳烃的微生物降解机制研究进展[J]. 化学与生物工程,2016,33(2):8-14.
[12]Hesham A,Wang Z Y,Zhang Y,et al. Isolationand identification of a yeast strain capable of degrading four and five ring aromatic hydrocarbons[J]. Annals of Microbiology,2006,56(2):109-112.
[13]Qi J,Wang B,Li J,et al. Genetic determinants involved in the biodegradation of naphthalene and phenanthrene in Pseudomonas aeruginosa PAO1[J]. Environmental Science and Pollution Research International,2015,22(9):6743-6755.
[14]Baldwin B R,Nakatsu C H,Nies L. Detection and enumeration of aromatic oxygenase genes by multiplex and real-time PCR[J]. Applied and Environmental Microbiology,2003,69(6):3350-3358.
[15]Kumar S,Stecher G,Tamura K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution,2016,33(7):1870-1874.
[16]Yang Y Y,Wang J,Liao J Q,et al. Abundance and diversity of soil petroleum hydrocarbon-degrading microbial communities in oil exploring areas[J]. Applied Microbiology and Biotechnology,2015,99(4):1935-1946.
[17]譚银萍,马 媛,吕 杰. 石油污染对土壤细菌结构特征的影响[J]. 生物技术,2016,26(2):193-198.
