2016-2017 年广东地区猪繁殖与呼吸综合征病毒的分子检测及NSP2、ORF5 基因变异分析
2019-08-20周吉培申翰钦刘相聪于林洋董建国张乐宜刘燕玲梁鹏帅宋长绪
徐 铮,周吉培,申翰钦,刘相聪,于林洋,董建国,2,张乐宜,刘燕玲,王 磊,梁鹏帅,宋长绪
(1.华南农业大学动物科学学院 国家生猪种业工程技术研究中心,广东 广州510642;2.信阳农林学院牧医工程学院,河南 信阳464000)
猪繁殖与呼吸综合征(PRRS)系由猪繁殖与呼吸综合征病毒(PRRSV)引起的母猪繁殖障碍和各阶段猪呼吸道症状的高度接触性传染性疾病,现已成为严重威胁全球养猪业的主要疫病之一。该病毒基因组为单股正链RNA,长约15 kb,包含10 个开放阅读框(ORF),其中ORF5 和NSP2 基因变异最大,是作为分析PRRSV 遗传变异的理想靶基因[1]。该病毒可分为2 个基因型:以Lelystad-virus(LV)株为代表株的欧洲型和以VR-2332 株为代表株的美洲型病毒株[2],分别是Wensvort 等[3]在1991 年和Benfield 等[4]于1992 年分离得到。郭宝清等[5]1996 年首次在国内分离到一株病毒,并命名为CH-1a,成为我国经典毒株的代表。2001 年在美国分离到具有NSP2 蛋白的(111+1+19)共131 个氨基酸不连续缺失为特征的NADC30 株[6]。2006 年我国分离到PRRSV 的变异株—高致病性猪繁殖与呼吸综合征病毒(HP-PRRSV)[7],其代表性毒株为JXA1和HUN4,此类毒株的NSP2 蛋白存在(1+29)共30个不连续氨基酸缺失的特征[8]。
本研究对2016-2017 年采自广东省各地疑似PRRSV 感染病料进行检测,并对其中的9 份阳性样品的NSP2 和ORF5 基因进行测序和比对分析,以揭示广东地区猪群PRRSV 最新流行动态和遗传变异特点,希望为该地区PRRSV 的科学防控提供一定的有效参考依据。
1 材料与方法
1.1 病料样品 采集和送检PRRSV 的906 份病猪淋巴结、脾脏、肺脏等组织病料,样品来自广东省各地市的疑似发病猪场。
1.2 菌株、细胞及载体 大肠杆菌DH5α 感受态细胞、Marc-145 细胞均由华南农业大学国家生猪种业工程技术研究中心保存;pMD-19T 克隆载体,购自宝生物工程(大连)有限公司。
1.3 主要试剂 病毒DNA/RNA 提取试剂盒,购自广州美基(欣研)生物科技有限公司;PrimeScipt®One Step RT-PCR kit、DNA Marker DL-2 000,均购自宝日医生物技术(北京)有限公司;胶回收试剂盒为Promega 公司产品;EB 替代染料,购自广州华奇盛生物科技有限公司。
1.4 引物设计与合成 参照GenBank 中收录的JXA1 和VR-2332 等毒株基因序列,应用Primer Premer5.0 分别设计2 对NSP2 和ORF5 基因的特异性引 物。 NSP2-F:5′-CCTCCGTGGTGCAACAAATCTTG-3′; NSP2-R: 5′-CGATGATGGCTTGAGCTGAGTAT-3′,扩增片段大小为1 064 bp。ORF5-F:5′-GTGTCAGGCATTGTGGCTGTG-3′;ORF5-R:5′-CATTATTGGCGTGTAGGTGATTAGAAAA-3′,扩增片段大小为821 bp。引物由苏州金唯智生物科技有限公司合成。
1.5 病料总RNA 的提取及RT-PCR 扩增 取0.5 g左右无菌研磨好肺、脾等病料组织,置于1.5 mL 离心管内,以1 mL 无菌PBS 进行重悬,反复3 次冻融后,8 000 r/min 离心10 min,取上清液按照病毒RNA提取试剂盒说明书提取病料组织病毒RNA。以提取的RNA 为模板,利用PrimeScipt®One Step RTPCR kit 对目的片段进行扩增。扩增体系为50 μL,PrimeScript 1 step Enzyme Mix 2 μL,2×1 Step Buffer 25 μL,上游引物2 μL,下游引物2 μL,RNA 模板4 μL,RNase Free ddH20 15 μL。PCR 反应循环参数为:50 ℃30 min;94 ℃2 min;94 ℃30 s,60 ℃30 s,72 ℃90 s,35 个循环;72 ℃延伸10 min。取5 μL 反应产物进行琼脂糖凝胶电泳观察。
1.6 NSP2 和ORF5 基因的克隆及测序 PCR 产物凝胶回收后克隆于pMD-19T 载体上,转化DH5α 感受态细胞,阳性重组质粒送苏州金维智生物科技有限公司测序。
1.7 NSP2 和ORF5 基因的遗传变异分析 参考GenBank 收录的国内外的PRRSV 参考毒株序列,利用Lasergene 软件中MegAlign 程序对不同毒株的Nsp2 和ORF5 基因序列和推导氨基酸进行比对和同源性分析;运用MEGA5.0 软件的Maximum Likelihood 方法绘制发育遗传进化树。
2 结果
2.1 RT-PCR 检测PRRSV 结果 2016-2017 年共采集检测来自广东各地PRRSV 病料906 份,PRRSV检测为阳性的样本有399 份,阳性率为44%,其中2月份与9 月份PRRSV 核酸阳性率最低,约10%,8月份阳性率最高,达到了65%。提示PRRS 在广东流行的情况比较严重,不同月份之间检测差异应该与广东的气候和猪场免疫程序有很大关联。从阳性样品中挑选9 份,编号分别为:GDGZ、GDFS、GDSH、GDHY、GDHZ-1、GDHZ-2、GDMZ、GDYJ、GDSG进行NSP2、ORF5 基因变异分析。
2.2 NSP2 基因的变异特征
2.2.1 NSP2 基因核苷酸及其推导氨基酸序列比对分析 9 个NSP2 基因核苷酸序列之间同源性为88.9% ~100%,推导的氨基酸序列同源性为87.0%~100%;与欧洲型代表株LV 核苷酸同源性仅为40.1%~43.7%,推导氨基酸序列同源性为16.1%~18.0%;与经典美洲株NADC30 核苷酸同源性为56.3%~60.0%,推导氨基酸序列同源性为49.5%~52.4%;与美洲型代表株VR-2332 的核苷酸同源性为74.4%~77.1%,推导氨基酸序列同源性为66.8%~68.4%;与近年一度在南方流行低致病性代表株GM2 核苷酸同源性为75.7%~79.5%,推导氨基酸序列同源性为67.0%~70.7%;与中国经典株CH-1a 核苷酸同源性为85.9%~90.8%,推导氨基酸序列同源性为82.7%~88.2%;与中国高致病性代表株JXA1 和HUN4 核苷酸同源性分别为92.3%~98.3%和92.2%~98.2%,推导氨基酸序列同源性分别为91.5% ~98.1%和91.5% ~97.8%。结果表明,9 个NSP2 基因之间高度同源,与欧洲型之间同源性最低,与经典美洲株NADC30、美洲型代表株VR-2332 和低致病性代表株GM2 之间同源性不高,与中国经典株CH-1a 之间同源性较高,与中国高致病性代表株JXA1 和HUN4 同源性最高。
9 个NSP2 基因推导氨基酸序列与HP-PRRSV代表株高度相似,但与其他代表株相比,出现了多处多个氨基酸位点的突变(图1)。其中GDFS、GDYJ、GDSH、GDSG、GDMZ、GDHZ-1、GDHZ-2 共7 个毒株与JXA1 和HUN4 相比,具有相同的氨基酸缺失特征,即有1+29 个氨基酸的缺失,如图1 实线方框所示,也与文献报道的HP-PRRVS的NSP2 氨基酸缺失特征一致。 GDHY 和GDGZ株在相应位置发现缺失8+1+0 的特征,与CH-1a 的缺失特征相似,如图1 椭圆线框所示。从图中我们也看到NADC30 株出现了与文献报道一致的(111+1+19)131 个不连续氨基酸的缺失特征,但本次调查并没有发现与之缺失情况相似的毒株,即目前国内外报道较多的NADC30 或NADC30-like 株。2.2.2 NSP2 基因核苷酸序列的遗传进化分析 由9 个毒株与参考毒株的NSP2 基因序列构建的进化树显示,美洲型PRRSV 可分为5 个亚群(图2),即以HP-PRRSV JXA1、TJ 和HUN4 等为代表的亚群1,以中国经典株CH-1a 为代表的亚群2,以国内近年在华南和港台地区流行的低致病性毒株GM2 为代表的亚群3,以VR-2332 为代表的亚群4,以及以经典毒株NADC30 为代表的亚群5[9-10]。而9 株广东地区PRRSV 毒株均属于亚群1,表明近两年广东省流行的PRRSV 仍以高致病性毒株为主。
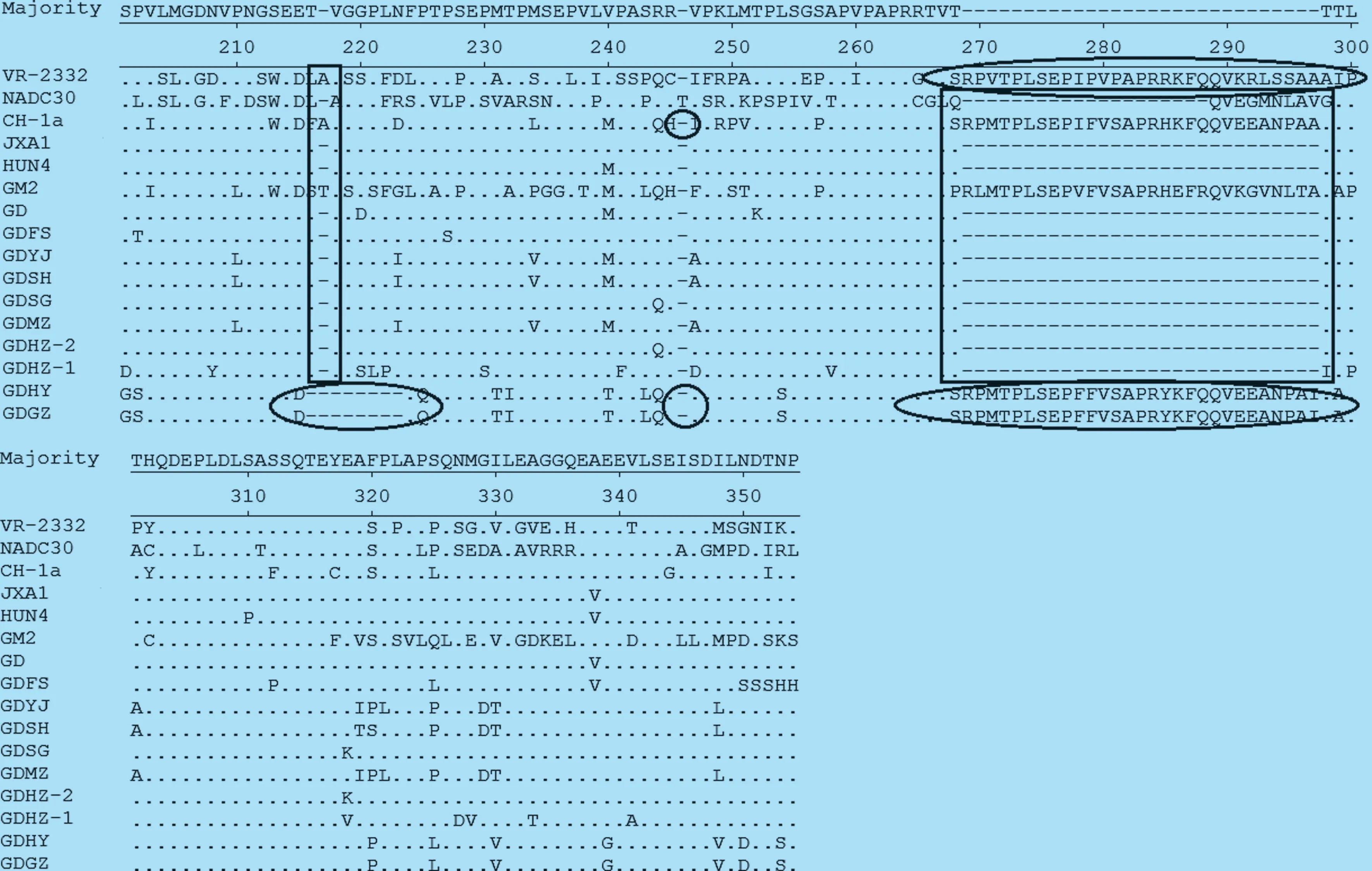
图1 NSP2 蛋白推导氨基酸序列比对分析
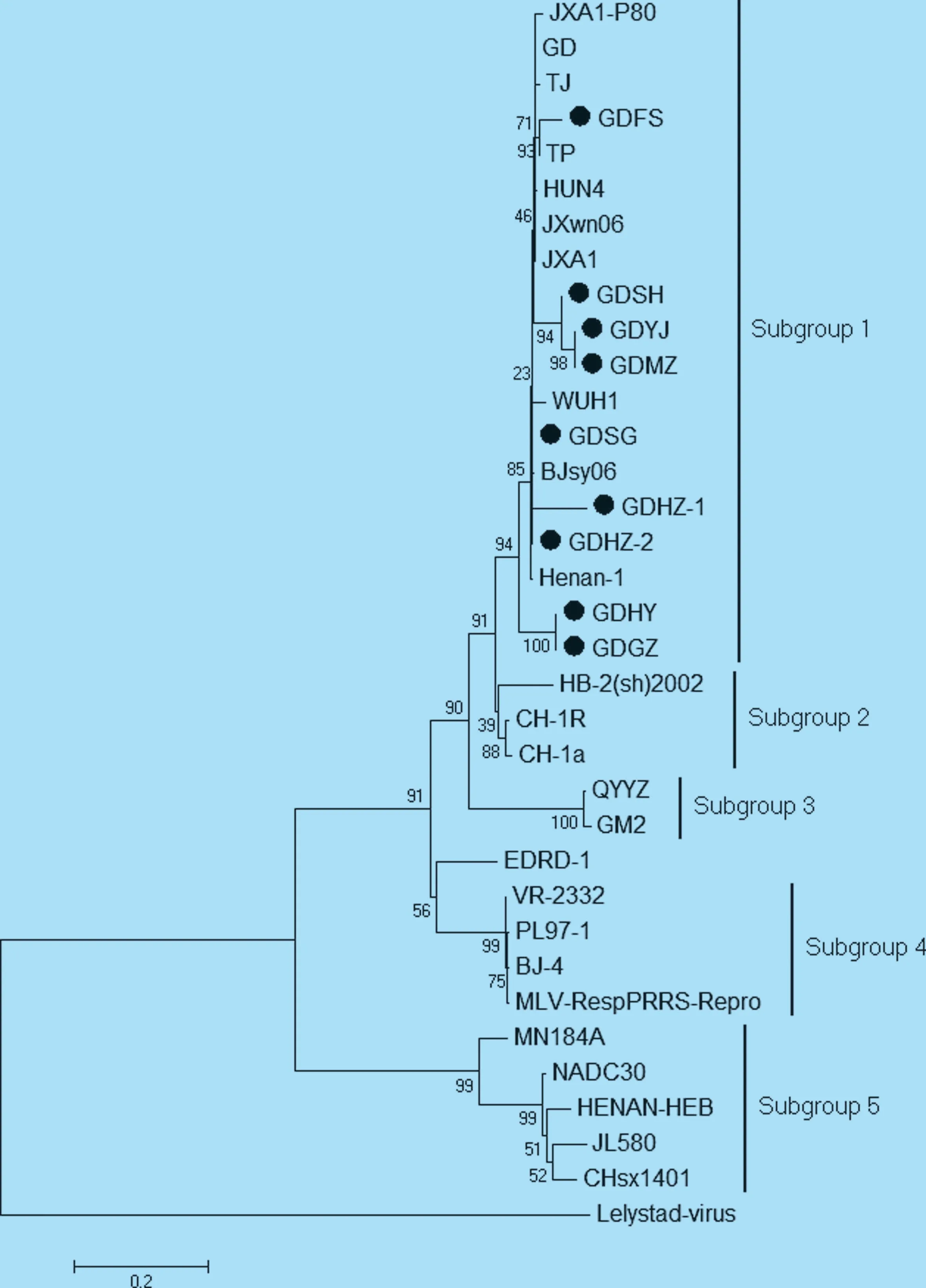
图2 NSP2 基因核苷酸序列遗传进化分析
2.3 ORF5 基因的变异特征
2.3.1 ORF5 基因核苷酸及其推导氨基酸序列比对分析 9 个ORF5 基因的核苷酸序列同源性为92.0% ~100%,其推导的氨基酸序列同源性为91.5%~100%;与欧洲型代表株LV 核苷酸同源性为62.6% ~64.3%,推导氨基酸序列同源性为56.6%~59.2%;与经典美洲株NADC30 核苷酸同源性为84.2%~86.6%,推导氨基酸序列同源性为83.1%~87.6%;与美洲型代表株VR-2332 的核苷酸同源性为87.2%~90.0%,推导氨基酸序列同源性为84.6%~90.0%;与近年在南方流行的低致病性代表株GM2 核苷酸同源性为81.6%~84.2%,推导氨基酸序列同源性为79.1%~83.6%;与中国经典株CH-1a 核苷酸同源性为91.7%~95.5%,推导氨基酸序列同源性为89.1%~93.5%;与中国高致病性代表株JXA1 和HUN4 核苷酸同源性分别为94.2%~99.7%和94.4%~99.8%,推导氨基酸序列同源性为95.0%~99.5%和95.0%~100%。结果表明,9 个NSP2 基因之间高度同源,与欧洲型之间同源性最低,与经典美洲株NADC30、美洲型代表株VR-2332 和低致病性代表株GM2 之间同源性略高,与中国经典株CH-1a 之间同源性较高,与中国高致病性代表株JXA1 和HUN4 同源性最高。
当前已确定美洲型的PRRSV GP5 蛋白有3 个表位,其中非中和表位2 个(aa27 ~aa30 和aa180 ~aa197),中和表位1 个(aa37 ~aa45),如图3 黑色框所示。如图3 所示,本试验的9 个毒株与参考株比较,在非中和表位aa27 ~aa30,主要是第29 位氨基酸发生突变,与美洲型代表株VR-2332 相比,GDSG、GDHY、GDGZ、GDFS 与其一致,GDYJ、GDSH、GDMZ、GDHZ-1 和GDHZ-2 的A29V,与CH-1A、JXA1、HUN4N 和ADC30 一致。在aa180 ~aa197 中,在185 位和189 位,9 个毒株与JXA1 和HUN4 一致,但与VR-2332 相比发生V185A 和I189L;在196 位,GDSH、GDSG、GDMZ、GDHY、GDGZ、GDFS 株与VR-2332、CH-1a 和HUN4 一致,GDYJ、GDHZ-1、GDHZ-2三个毒株发生Q196R。在aa37 ~aa45,主要突变发生在第39 位氨基酸,9 个毒株基本与国内高致病毒株JXA1 和HUN4 一致,与VR-2332 和NADC30 相比发生L39I。总体来说,国内高致病性毒株(含本试验9 株)与美洲型代表株VR-2332 相比,GP5 蛋白的2 个重要抗原相关区域的4 个氨基酸位点(29、39、185 和189)的变异较为明显:A29V、L39I、V185和AI189L。提示这种变化或与两者之间毒力差异相关,但要做出定论还需进一步研究。

图3 GP5 蛋白推导氨基酸序列比对分析
2.3.2 ORF5 基因核苷酸序列的遗传进化分析由9 个毒株与参考毒株的ORF5 基因序列构建的进化树显示,与NSP2 进化树相似,美洲型PRRSV 也可分为5 个亚群(图4)[9-10]。9 株广东地区PRRSV毒株均属于亚群1,表明近两年广东省流行的PRRSV 仍以高致病性毒株为主。
3 讨论
HP-PRRSV 近年来在我国大部分地区流行甚至暴发,使我国养猪业损失惨重,大量的政府主导强制免疫的HP-PRRSV 活疫苗涌入市场,使得弱毒疫苗有可能在选择压作用下发生毒力返强[11]。本试验结果显示,PRRSV 核酸阳性率达到44%,抽检的9 个毒株均与高致病毒株高度同源且处于同一亚群,在如此高强度免疫的状态下存在这种情况,提示可能为疫苗株返强。另外,弱毒疫苗株和野毒株同时在猪群中存在,也使得毒株之间发生重组的风险增加[12]。

图4 ORF5 基因核苷酸序列遗传进化分析
本研究通过对2016-2017 年间采集自广东各地的906 份疑似病料进行了RT-PCR 检验,测得阳性率为44%,为了进一步研究PRRSV 在广东分子流行病学情况和毒株基因变异的发展,随机挑选9个阳性样本进行NSP2 和ORF5 基因序列及推导氨基酸比对,并对遗传序列进行分析。结果显示,9 个样品均属于HP-PRRSV,表明广东流行的PRRSV 仍以高致病性毒株为主,这也与于林洋等[13]2016-2017 年在广东、李海琴等[14]2013-2014 年在江西和李彬等[15]2013-2014 年在华中地区调查的结果一致,表明HP-PRRSV 仍是南方地区的优势毒株。但周峰等[16]、郭天准等[17]和董建国等[18]2012-2015 年间在河南调查的结果发现除HP-PRRSV 之外,还出现了与NADC30 高度同源的新变异株-NADC30-like 株的流行,该变异株与国内HP-PRRSV株有重组现象,且致病力有所增强[19],李真等[20]2014-2016 年在华北的地区的调查也发现了相同的情况,而且还发现新的变异毒株GM2-like 株的存在。以上结果似乎表明中国南北方流行的毒株有所差异,提示近区域引种或精液交易可能是造成这种差异的原因,但具体原因有待进一步研究。总之,本研究调查发现猪场在普遍使用了PRRSV 弱毒活疫苗后,检出阳性率仍然较高,而且不断有新的变异株出现,说明目前市场的疫苗不能很好地控制该病的发生,一方面说明目前中国PRRSV 毒株呈多样化趋势,另一方面提示PRRSV 在不断的发生变异,究竟是毒力返强还是不同毒株之间的重组,或是病毒抗原表位突变导致免疫逃逸,以及这些变异与其毒力和致病力之间的相关性还需进行深入研究。
