耐酸与酸敏酒酒球菌环丙烷脂肪酸合酶基因的差异
2019-08-07陈其玲刘树文
田 雨 陈其玲 刘树文,2,3* 何 玲
(1 西北农林科技大学葡萄酒学院 陕西杨凌712100 2 陕西省葡萄与葡萄酒工程技术研究中心 陕西杨凌712100 3 西北农林科技大学合阳葡萄试验示范站 陕西渭南715300 4 西北农林科技大学园艺学院 陕西杨凌712100)
苹果酸-乳酸发酵 (Malolactic fermentation,MLF)是提升葡萄酒品质的重要工艺环节[1-2]。作为葡萄酒生产中启动并完成苹果酸-乳酸发酵的主要菌种,酒酒球菌应对葡萄酒酒精发酵结束后高酸、高乙醇含量、高SO2的能力对葡萄酒苹果酸-乳酸发酵尤为重要[3-4]。
为了在葡萄酒环境中生存,膜成分调整引起的细胞膜流动性调整是乳酸菌应对胁迫环境采取的一种机制,此过程中主要是影响膜内磷脂和脂肪酸成分[5-7]。环丙烷脂肪酸(Cyclopropane fatty acid,CFA)是抗胁迫机制中一种重要的膜脂肪酸,CFA 的存在可以降低质子通透性,增强膜的刚性[8-9]。乳酸菌在酸胁迫环境中会出现不饱和脂肪酸的环化现象[10-12]。而调控合成CFA 的环丙烷脂肪酸合酶(Cyclopropane fatty acid synthesis,CFAS)由环丙烷脂肪酸合酶基因 (Cyclopropane fatty acid synthesis gene,cfa)编码合成。
目前研究发现酒酒球菌在酸环境下生长的细胞会增加其CFA 含量以应对环境的胁迫作用[13-15]。To 和Grandvalet 等[16]在乳酸乳球菌中表达酒酒球菌ATCC BAA 1163 的cfa 基因,其中重组乳酸乳球菌cfa 的基因表达量及CFA 含量在酸性环境中升高。目前酒酒球菌CFA 相关的研究主要着眼于CFA 在的抗酸机制中的作用,尚无文献从耐酸性不同的酒酒球菌株间CFAS 的差异这方面探索基因与抗酸性的相关性。
本研究首先通过对野生酒酒球菌进行耐酸性筛选,筛选出耐酸能力很强的野生型酒酒球菌。利用筛选的菌株扩增出cfa 基因进行测序,同时与耐酸、酸敏突变菌进行比对。通过将野生耐酸菌株和酸敏菌株cfa 基因在植物乳杆菌中的表达,对不同耐酸表型菌株株间cfa 基因的差异进行研究,探索环丙烷脂肪酸基因与菌株抗酸性的相关性及菌株间该基因稳定差异造成的影响。
1 材料与方法
1.1 菌株与质粒
试验中所用的酒酒球菌菌株保藏于西北农林科技大学葡萄酒学院,包括筛选自内蒙古、山东、陕西、宁夏和河北5 个葡萄酒产区的34 株酒酒球菌和商业酒酒球菌31-DH,见表1。耐酸、酸敏突变菌株a3[17]和b2[17]由实验室离子注入诱变获得,植物乳杆菌ATCC33222 菌株来源于西北农林科技大学食品学院。质粒pMG36e 由江南大学惠赠。
表1 试验中34 株初筛酒酒球菌的产区来源Table 1 The regions of thirty four screening strains in the study
1.2 试验材料与设备
ATB 培养基参照参考文献[18],MRS 培养基参照参考文献[19],用HCl 调节培养基pH,试剂均为国产分析纯。
PrimeSTAR HS 酶,日本TaKaRa 公司;Ezup柱式细菌基因组DNA 抽提试剂盒、SacI/XbaI 内切酶,生工生物工程(上海)股份有限公司;DNA 胶回收试剂盒,天根科技(北京)有限公司;EZNA 质粒DNA 小量抽提试剂盒,美国OMEGA 公司。
AIR TECH 超净台,苏州安泰空气技术有限公司;MJ-250B 培养箱、HH-4 数显恒温水浴锅,上海森信实验仪器有限公司;TG16-WS 台式高速离心机,湖南湘仪离心机仪器有限公司;Thermal Cycler C1000 基因扩增仪,美国Bio-Rad 公司;Agilent Technologies 紫外分光光度计,美国安捷伦科技有限公司;BioTek ELx800 酶标仪,美国博腾仪器有限公司。
1.3 试验方法
1.3.1 酒酒球菌的筛选 将酒酒球菌菌株在pH 4.8 的ATB 培养基中活化2 次至对数生长期,以4%的接种量接种至pH 2.8 的ATB 培养基中培养,每隔24 h 取1.5 mL 菌液于24 孔板中测定其在600 nm 下的吸光度。根据吸光度的变化情况初步筛选抗酸和酸敏表型差异菌株。将筛选获得的较耐酸酒酒球菌菌株活化至对数生长期,以4%接种量接种至pH 2.4,2.6,2.8 的ATB 培养基中,利用紫外分光光度计检测其在600 nm 下的吸光度,绘制生长曲线。根据吸光度的变化情况筛选出抗酸能力强的菌株。
1.3.2 目的基因序列的PCR 扩增及序列比对根据基因序列设计引物并由生工生物工程(上海)股份有限公司合成,引物序列为cfa1(TTGAAATGGCAATCGTTTCAGCA ) 和 cfa2 ( CGGTTGTACCGTCGCCTTTA),进行PCR 扩增。PCR 产物用1%琼脂糖凝胶电泳进行检测,在全自动凝胶成像分析系统中拍照、分析。将经过检测的PCR 产物送样测序。将本试验筛选获得的耐酸野生菌、酸敏野生菌与本实验室诱变筛选的耐酸突变菌a3、酸敏突变菌b2[17]的cfa 基因进行序列比对。
1.3.3 酒酒球菌cfa 基因的外源表达 对酒酒球菌cfa 基因的PCR 产物进行切胶回收,具体步骤参照切胶回收试剂盒说明书;利用质粒提取试剂盒进行pMG36e 表达质粒的提取,操作步骤参照说明书。对回收的cfa 基因片段和质粒pMG36e 进行SacI/XbaI 双酶切,酶切产物进行1%的琼脂糖凝胶电泳并分别进行切胶回收,获得目的基因和质粒的酶切产物。将酶切产物连接,反应体系(10 μL):载体pMG36e(SacI/XbaI)1 μL,目的DNA 片段4 μL,ligation mix 5 μL,16 ℃连接过夜。
植物乳杆菌感受态细胞制备及电转方法参照[20]略做调整。取30 ng 质粒与200 mL 植物乳杆菌感受态细胞混合后,移入间距为0.2 cm 的电转杯中,冰上静置5 min,使用高压脉冲电转仪进行电击转化,电转参数为电压2.0 kV,电击常数4 ms。电击完毕后,迅速向电转杯中加入1.8 mL 含0.3 mol/L 蔗糖的MRS 培养基,37 ℃静置培养2~3 h,使细胞复苏。取200 μL 复苏的菌液,涂布在含200 μg/mL 红霉素的MRS 平板上,37 ℃静置培养48 h。将转化的菌体进行阳性克隆,菌落PCR 鉴定,提取重组质粒后进行双酶切鉴定。
1.3.4 植物乳杆菌的耐酸性测定 为测定重组植物的耐酸性,耐酸与酸敏野生菌的cfa 基因连接载体并成功导入植物乳杆菌后,将重组的植物乳杆菌接入pH 3.0~4.0 梯度MRS 培养基中测定生长曲线,以含有空载体的植物乳杆菌L0 作为对照。将重组植物乳杆菌菌株在pH 6.5 的MRS 培养基中活化2 次至对数生长期,以2%的接种量接种至pH 3.0~4.0 梯度MRS 培养基中培养,利用紫外分光光度计检测其在600 nm 下的吸光度,绘制生长曲线。
2 结果与分析
2.1 酒酒球菌的筛选生长曲线
2.1.1 酒酒球菌菌株初筛 图1为35 株酒酒球菌在pH 2.8 的ATB 培养基中稳定期时利用酶标仪检测的OD600值。

图1 酒酒球菌在pH 2.8 环境下的初筛结果Fig1 The results of the screening Oenococcus oeni in pH 2.8
由图1可以看出,菌株CS-7b 的生长状况最好 (OD600值为1.039),SD-2a、ME-5b、ME-2c 和31-DH 菌株的生长状况较其它菌株存在一定优势(OD600值在0.8~1.0 之间)。其余菌株生长状况比较相近 (OD600值在0.6 左右)。菌株SD-2d、SX-1b、SD-1b 和ME-2a 的生长情况较差(OD600值低于0.5)。由于试验中所用的酸敏突变株b2 是由SX-1b 诱变得来,因此确定SX-1b 为野生酸敏酒酒球菌的代表菌株。同时对生长情况较好的CS-7b、SD-2a、ME-5b、ME-2c 和31-DH 这5 株菌进行进一步耐酸能力强的野生菌株的筛选。
2.1.2 酒酒球菌菌株的复筛 以pH 2.8 培养基筛选出的CS-7b、SD-2a、ME-5b、ME-2c 和31-DH 这5 株酒酒球菌利用紫外分光光度计测定其在pH 2.4,2.6,2.8 的ATB 培养基中的生长曲线。图2为5 株酒酒球菌在pH 2.8 的培养基中的生长曲线。图3为5 株酒酒球菌在pH 2.6 的培养基中的生长曲线。
由图2可以看出,CS-7b、SD-2a、ME-5b、ME-2c 和31-DH 这5 株在pH 2.8 培养基中均能存活和生长,CS-7b 和ME-5b 稳定期的状态(OD600值达到1.8 左右)与酒酒球菌在正常pH 条件下稳定期的状态基本一致,长势明显优于SD-2a、ME-2c和31-DH。5 株菌在pH 2.8 培养基中对数生长期均明显延长,SD-2a、ME-2c 和31-DH 大约在第5天进入稳定期,而CS-7b 和ME-5b 在第7 天进入稳定期。结果表明,酒酒球菌菌株在酸性环境中生长时会出现对数生长期延长,稳定期滞后的现象,符合酒酒球菌适应胁迫环境的生长趋势。

图2 酒酒球菌野生株在pH 2.8 培养基中的生长曲线Fig.2 Growth curve of wild O.oeni cultured in ATB medium (pH 2.8)
由图3看出,CS-7b 和ME-5b 在pH 2.6 培养基中存活能力明显高于其余3 株菌 (稳定期OD600值0.3~0.35 之间),生长曲线趋势也比较符合细菌生长周期。其余3 株菌生长曲线基本没有变化,确定菌体没有生长。CS-7b 和ME-5b 两株菌有更为明显的耐酸优势。在pH 2.4 环境下继续培养、筛选和确认时发现5 株菌均没有生长。
CS-7b 和ME-5b 的耐酸能力相似,菌株的耐酸极限是pH 2.6,结果确定了CS-7b 和ME-5b为研究中所筛选的36 株野生酒酒球菌中最耐酸的菌株。经过对36 株野生酒酒球菌的初筛、复筛及综合比较发现,最终确定用于研究的耐酸和酸敏菌株均为陕西省筛选的野生酒酒球菌及其诱变株,一定程度上保证了遗传背景的相似性。
2.2 环丙烷脂肪酸合酶基因的PCR 和序列分析
以提取的酒酒球菌全基因组为模板,以1.3.2节中的两对特异性引物进行PCR 扩增,产物用1%琼脂糖凝胶电泳进行检测并送样测序。
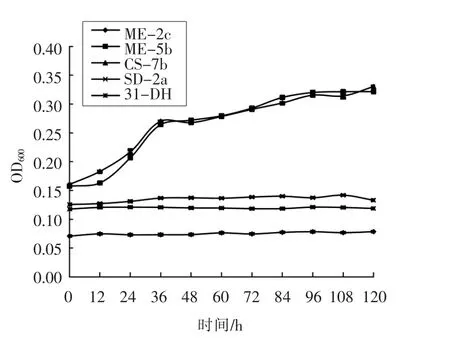
图3 酒酒球菌野生株在pH 2.6 培养基中的生长曲线Fig.3 Growth curve of wild O.oeni cultured in ATB medium (pH 2.6)
测序结果提交Genebank 核酸数据库,菌株ME-5b、CS-7b、a3、b2 的cfa 基因访问号依次为KX467881、KX467882、KX467883、KX467884。对扩增cfa 基因的测序结果显示,该基因全长1 188 bp,含有完整的开放阅读框,编码395 个氨基酸,测序结果显示酸敏野生菌和突变菌与PSU-1 菌株该基因的碱基序列完全一致,而耐酸野生株和突变菌均在684(C-T)、898(A-C)和924(A-G)处发生突变。将所得核苷酸序列推导的氨基酸序列与PSU-1 标准菌株中对应氨基酸序列进行比对,结果如图4所示。耐酸菌株该基因编码的氨基酸序列与标准菌株同源性达99%以上,3 个碱基突变中有一个为非同义突变,第300 个氨基酸由I(异亮氨酸)突变为L(亮氨酸)。
2.3 重组植物乳杆菌表达载体的构建
在耐酸野生菌CS-7b 和ME-5b 中,选择筛菌过程中生长较稳定的CS-7b 作为耐酸菌中cfa 基因的来源。酸敏野生菌SX-1b 作为酸敏菌cfa 基因的来源。将SX-1b 和CS-7b 中扩增的cfa 基因和载体进行酶切、连接和转化。菌落PCR 验证后,分别获得重组菌L1 和L2,提取重组质粒进行双酶切验证。验证结果见图5。重组质粒经过SacI/XbaI 双酶切后,释放约3 610 bp 和1 188 bp 大小的片段,分别对应pMG36e 和cfa 基因的大小,与图5的验证结果一致,说明重组质粒构建成功,获得研究所需要的重组植物乳杆菌。
2.4 重组植物乳杆菌的耐酸性变化

图4 耐酸、酸敏菌株与对照菌株PSU-1 中OEOE_RS05660 基因编码的氨基酸序列比对结果Fig.4 Alignment of the deduced amino acid sequence coded by gene OEOE_RS05660 from acid-resistant/acid-sensitive strains and Oenococcus oeni PSU-1

图5 pMG36e-cfa1/2 的酶切结果Fig.5 Identification of pMG36e-cfa1/2 by enzyme digestion
将对照重组植物乳杆菌L0 分别在pH 3.0,4.0,5.0,6.0,7.0 培养基中培养,结果表明L0 在pH 3.0 下没有生长,在pH 4.0,5.0,6.0,7.0 中生长良好,因此确定在pH 3.0~4.0 梯度培养基中培养重组植物乳杆菌及对照菌。图6为在pH 3.6 及pH 3.4 培养基中3 株菌的生长曲线图,pH 3.2 培养基中3 株菌均没有生长。L2 和L1 的生长极限在pH 3.4,相较于L0 的极限值出现突破,cfa 基因的导入使植物乳杆菌耐酸性明显升高。同时,在pH 3.6 培养基中,L2 和L1 的稳定期OD600值相较于L0 分别增加了约42%和33%,菌体生长的涨幅L2>L1;在pH 3.4 培养基中,L2 的生长情况也优于L1,说明一定程度上L2 在适应酸胁迫环境方面有一定的优势。
综合两株重组菌和对照菌生长情况的测定,结果表明,酒酒球菌cfa 基因的外源插入对于植物乳杆菌在酸性环境中的表现有明显提升,并突破对照菌的生长极限。同时提升程度与cfa 来源的酒酒球菌的耐酸性一致;酒酒球菌耐酸性越好,对应cfa基因的重组植物乳杆菌在酸性环境中生长的越好。
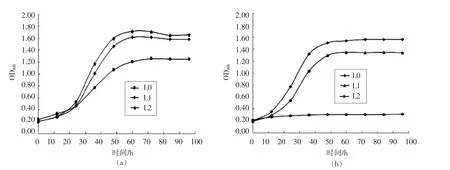
图6 重组植物乳杆菌在pH 3.6,3.4 MRS 培养基中的生长曲线Fig.6 Growth curve of recombinant L.plan cultured in MRS medium (pH 3.6,3.4)
3 结论
为研究不同耐酸表型的酒酒球菌cfa 基因的株间差异,同时进一步研究酒酒球菌CFA 在抗酸机制中的作用,本研究利用筛选自国内5 个产区的35 株野生酒酒球菌进行耐酸菌株的筛选,最终筛选出2 株可以在pH 2.6 培养基中生长的耐酸野生菌株CS-7b 和ME-5b。在对cfa 基因的测序比对中,将实验室诱变筛选获得的耐酸突变株和酸敏突变株加入比对,证明cfa 碱基突变与菌株耐酸能力具有相关性。将野生酸敏、耐酸菌株SX-1b和CS-7b 的cfa 基因在植物乳杆菌中表达,研究重组植物乳杆菌的耐酸能力变化。结果发现重组菌突破了原菌的生长极限,进一步证明cfa 与菌株耐酸能力具有相关性,且cfa 基因存在的碱基突变使重组菌株在耐酸能力方面存在差异。
研究中筛选的耐酸野生酒酒球菌可以作为研究酒酒球菌耐酸能力和作用机制的菌种资源,同时耐酸菌株具有较低的生长极限pH 以及在酸性培养基中稳定生长的特性为葡萄酒生产中实现高酸原料启动和完成苹果酸-乳酸发酵提供了可能。然而耐酸菌株是在培养基中进行筛选,pH 值为唯一的胁迫条件。由于酒精发酵结束葡萄酒中复杂的生存环境和多种胁迫条件,菌株能否存活并顺利启动,并完成苹果酸-乳酸发酵,以及对葡萄酒风味的具体影响还有待进一步研究。
研究中成功构建重组菌可以进一步研究其发酵苹果酸的能力和在其它胁迫因素下的耐受性,为植物乳杆菌工程菌在葡萄酒生产中作为发酵启动剂提供一定基础。由于酒酒球菌的耐酸性由多基因共同作用[21-22],cfa 基因及其株间出现的碱基突变在抗酸机制中发挥作用的机理需要进一步研究。
酒酒球菌很难导入外源质粒实现高效表达和基因敲除[3],因此本试验中所用的方法和思路也可以为酒酒球菌中其它可能与耐酸能力甚至抗胁迫能力相关的基因的研究提供参考。同时也证明在研究酒酒球菌抗胁迫基因过程中,基因在株间的差异是不容忽略的因素。
