马氏珠母贝TRADD基因克隆与组织表达分析
2019-07-17何军军梁海鹰房晓宸黄雪敏
何军军,梁海鹰,陈 崧,房晓宸,黄雪敏
马氏珠母贝基因克隆与组织表达分析
何军军,梁海鹰,陈 崧,房晓宸,黄雪敏
(广东海洋大学水产学院,广东 湛江 524088)
【目的】克隆马氏珠母贝()肿瘤坏死因子受体相关死亡域蛋白(TRADD)基因,并分析其在各组织中的表达。【方法】利用cDNA末端快速扩增技术(RACE)克隆获得马氏珠母贝基因的cDNA全长序列,利用实时荧光定量PCR(qPCR)方法分析基因在马氏珠母贝不同组织中的表达模式。【结果与结论】包含5′非编码区101 bp,3′非编码区144 bp和开放阅读框(ORF)591 bp,编码196个氨基酸。序列分析表明,PmTRADD没有信号肽和跨膜结构域,C端含有一个死亡结构域(DEATH)。将PmTRADD死亡结构域的氨基酸序列与其他物种的TRADD死亡结构域序列进行比对,发现不同物种的TRADD死亡结构域序列同源性较低。在马氏珠母贝各组织中均有不同程度表达,在鳃组织中表达最高,肝胰腺次之,闭壳肌中基本无表达。
马氏珠母贝; 肿瘤坏死因子受体相关死亡域蛋白; 基因克隆; 实时荧光定量
马氏珠母贝()最早发现于广西合浦县,其肉质鲜美、所产珍珠具有非常高的观赏价值和药用价值[1]。随着珍珠养殖业发展, 养殖规模不断扩大, 种质退化和病害问题日趋严重, 海水珍珠产量和质量显著下降, 严重威胁我国海水珍珠产业的持续健康发展[2]。因此, 研究贝类免疫相关基因, 了解贝类免疫防御机制至关重要。
肿瘤坏死因子受体相关死亡域蛋白 (tumor necrosis factor receptor associated death domain protein, TRADD) 是一种含死亡域 (death domain, DD) 的接头蛋白, 是TNF受体-1(TNFR1)介导的TNF信号传导的重要介质,在TNFα介导的程序性细胞凋亡中至关重要[3]。TRADD蛋白含有功能未知的N末端区域和C末端死亡结构域,与TNFR1的死亡结构域有23% 的相同之处[4]。TRADD的过表达可激活TNFR1样信号通路,用于细胞凋亡和转录因子NF-κB的激活,在TNFR1应答中发挥作用[5]。细胞凋亡在发育和免疫反应中也起重要作用。在必要的情况下,生物体利用死亡信号消除威胁其存活的个体细胞,细胞因子如Fas配体和TNFα,可作为死亡信号与靶细胞表面的死亡受体结合,从而传递凋亡信号[6],随后TRADD与受体死亡结构域结合并主要负责其它效应蛋白的募集,如RIP1(受体相互作用蛋白1)、TRAF2(TNF受体-相关因子2)与TNFR1信号复合物、FADD(Fas相关蛋白与死亡域)对TNFR1诱导的二级复合物进行招募[4-5,7-9]。这些衔接蛋白的募集导致MAP激酶和NF-κB的激活,也触发其他信号通路,包括细胞死亡,促炎反应等[8,10]。在日本脑炎病毒(JEV)实验中已经表明,神经元中TNFR-1的表达模式在JEV感染后发生变化,并且是通过肿瘤坏死因子受体相关死亡结构域(TRADD)启动死亡级联的[11-12]。此外,TRADD与TNFR1、TRAF的相互作用是通过其死亡结构域与二者结合发生效应,因此,TRADD可能同时结合TNFR1和TRAF,从而将TRAF募集到TNFR1复合物中[4]。
近年来,TRADD蛋白的很多研究主要集中在脊椎动物的免疫功能中,例如在小鼠模型中TRADD的缺失对TAK1 / P38 MAPK信号传导的影响机制,以及TRADD作为中枢衔接子,介导细胞死亡和炎症信号[13-14]。尽管TRADD通常被认为是细胞质蛋白,但它也可能在细胞核中起作用[13]。TRADD还被证明是各种心脏损伤的重要保护因素,包括内质网(ER)应激,缺血再灌注(I/R)损伤[15-16]。在软体动物中,细胞凋亡构成一种重要免疫反应,可以通过各种刺激引发,包括细胞因子,激素,毒性损伤,寄生虫或病原体[17-19]。但目前为止,纯粹的基因在软体类研究未见报道,这也为马氏珠母贝TRADD的研究增加了很大难度。本实验通过RACE技术获得马氏珠母贝基因的cDNA序列全长并进行了生物信息学分析,qRT-PCR技术研究其在马氏珠母贝6个组织中的表达情况,为进一步了解在马氏珠母贝中的免疫学功能奠定基础。
1 材料与方法
1.1 材料
养殖于广东省湛江市徐闻县的2龄马氏珠母贝为实验用贝。取马氏珠母贝闭壳肌、鳃、外套膜、肝胰腺、性腺和血淋巴6个组织作为本次实验材料,冻存于-80℃冰箱备用。
1.2 试剂
PrimeSTAR Max DNA Polymerase、rTaq DNA聚合酶、Reverse Transcriptase M-MLV(RNase H)、反转录试剂盒、DNA Marker、Amp、克隆载体pMD-18T等购自TAKARA公司,DEPC购自生工生物工程(上海)股份有限公司,RACE试剂盒购自Clontech公司;Trizol和SYBR®Select Master Mix购自赛默飞世尔科技公司,Trans1-T1 Phage Resistant感受态细胞购自全式金公司。
1.3 方法
1.3.1 cDNA的合成
1.3.1.1 mRNA的提取 运用Trizol法提取马氏珠母贝闭壳肌等6个组织的总RNA,通过琼脂糖凝胶电泳验证其完整性,Nano Drop ND1000紫外分光光度计测各组织RNA浓度并分析纯度。5′RACE和3′RACE模板的制备参照SMARTRACE cDNA Amplification Kit(Clontech公司)的说明书,实时荧光定量cDNA模板则参照Reverse Transcriptase M-MLV(RNase H)说明书操作合成。
1.3.1.2 cDNA全长序列的获得 从本实验室构建的马氏珠母贝血细胞转录组文库中获得注释为TRADD的unigene序列[20],并设计特异性引物(表1)。采用巢式PCR扩增法进行5和3末端的RACE扩增,获得5和3末端序列,多聚酶链式反应扩增进行中间片段验证。先通过琼脂糖凝胶电泳检测产物,后根据纯化回收试剂盒说明书纯化目的基因片段;回收目的基因片段导入到pMD-18T载体中,再转化到Trans1-T1 Phage Resistant感受态细胞中,挑取阳性单克隆菌落进行菌落PCR检测,目的菌送生工生物工程(上海)股份有限公司分部广州测序部测序。

表1 PmTRADD基因克隆及荧光定量所用的引物序列
1.3.2 生物信息学分析 使用DNAMAN软件对已知的unigene序列和测序序列进行重叠拼接,获得基因的cDNA全长序列。使用NCBI数据库的ORF Finder (https://www.ncbi.nlm.nih.gov/orffinder/)软件预测该基因的开放阅读框(ORF)并推导其氨基酸序列。使用Ex PASy-Prot Scale(http://web. expasy. org/protscale/)预测其疏水性,用Motif Scan(http://myhits.isb-sib.ch/cgi-bin/motif_scan)搜索检测序列功能位点,采用Signal P 4.1(http://www.cbs.dtu.dk/services/Signal P/)预测其信号肽,通过Ex PASy-Prot Param(http://web. expasy. org/prot-scale/)分析其理化性质,SMART (http://smart.embl-heidelberg.de/smart/set_mode.cgi? NORMAL=1)预测其结构域。采用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_auto-mat.pl?page=/NPSA/npsa_sopma.html)在线网站预测二级结构,通过Clustalw在线软件进行多序列比对;使用Mega 6软件构建生物系统进化树。
1.3.3 荧光定量检测组织差异表达 通过实时荧光定量PCR检测基因在马氏珠母贝闭壳肌、鳃、外套膜、肝胰腺、性腺、血淋巴6个组织中表达情况,作为内参,每组样品进行3次技术重复,每个组织进行4次生物学重复。各组织相对表达量用2-△△CT法计算。运用SPSS 18.0对实验数据进行单因素方差分析,并用Duncan’s多重比较对均值进行差异显著性检验,显著性水平= 0.05。
2 结果分析
2.1 PmTRADD基因的cDNA克隆和序列分析
运用RACE技术扩增获得基因cDNA全长为836 bp,其中5UTR为101 bp, 3UTR为144 bp,包含20 bp的poly A尾巴(图1),ORF为591 bp,编码196个氨基酸,预测其分子量约为21.89 ku理论等电点为8.42。
2.2 PmTRADD蛋白理化性质分析
PmTRADD蛋白的脂溶性系数(Aliphatic index)为54.29,不稳定指数为63.04,属于不稳定蛋白。总平均亲水性(Grand average of hydropathicity)为-0.721,属于亲水性蛋白(图2),其中在第125位氨基酸达到最高亲水性,亲水指数为-2.322,在第180位氨基酸达到最高疏水性,疏水指数为1.556。PmTRADD不存在信号肽与跨膜结构域,表明该蛋白可能为非分泌蛋白。对PmTRADD序列进行功能位点检测可知,该蛋白有2个N-糖基化位点,1个酰胺化位点,7个蛋白激酶C磷酸化位点,6个酪蛋白激酶II磷酸化位点,1个N-肉豆蔻酰化位点,3个微体C端靶向信号。二级结构预测结果显示,该蛋白中α螺旋占41.84%,β转角占3.06%,无规卷曲占45.41%,延伸链占9.69%。同时进行结构域预测分析,结果显示PmTRADD氨基酸序列在第99~196位有一个DEATH结构域(图3)。最后预测马氏珠母贝PmTRADD蛋白分子的三维结构(图4)。

起始密码子ATG和终止密码子TAA用方框标注; 阴影部分为DEATH结构域
The initial codon ATG and the termination codon TAA are marked with the box; Shades of gray showed DEATH domain
图1基因的核酸序列及编码的氨基酸序列
Fig. 1 The nucleotide and amino acids sequences ofgene from
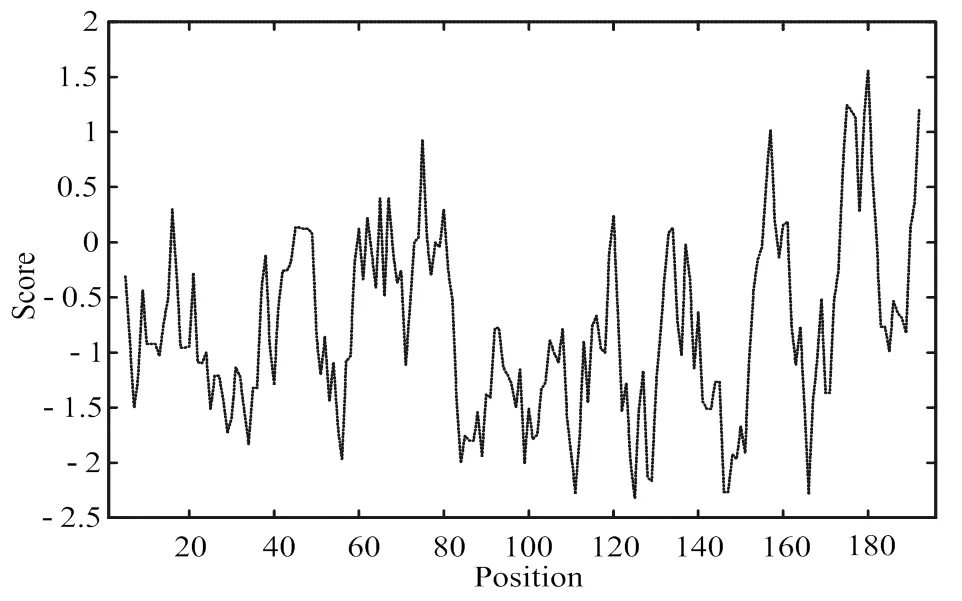
图2 PmTRADD蛋白疏水性

图3 SMART 软件预测的PmTRADD蛋白质结构

N: N末端; C: C末端(A: N-terminal; C: C-terminal)
2.3 多序列比对与进化树建立
运用NCBI在线Blastx比对,结果显示PmTRADD氨基酸序列与其它物种的相似性都较低。运用CLUSTALW软件将PmTRADD的死亡域(DD)氨基酸序列与智利鼠(, XP_004628516.1)、佛罗里达海牛(, XP_004387623.1)、海獭(, XP_022373460.1)、剑鱼(, XP_023191591.1)、九带犰狳(, XP_012376990.1)和文昌鱼(, AEO79023.1)的TRADD死亡域(DD)氨基酸序列进行多序列比对,结果表明物种间TRADD的同源性不高,其中与文昌鱼的同源性最高,同源性值为37.04%,与剑鱼的同源性最低,同源性值为29.90%(图5)。运用MEGA 6软件的Neighbor-Joining法构建马氏珠母贝氨基酸序列与原牛(, NP_001039361.1)、小鼠(, NP_001028333.1)、人类(, NP_001310481.1)、佛罗里达海牛(XP_004387623.1)、海獭(XP_022373460.1)、珍珠鸡(, XP_021269726.1)、虹鳟鱼(, NP_001118111.1)、北极红点鲑(, XP_023858136.1)和银鲑(, XP_020334958.1)的TRADD氨基酸序列的系统进化树,显示人类、原牛和小鼠这3类哺乳动物聚为一簇,虹鳟鱼、北极红点鲑和银鲑聚为一簇,并与哺乳类聚为一大支;佛罗里达海牛和海獭聚为一簇;而马氏珠母贝单独为一支,但是相对更靠近哺乳类。结果与传统分类相吻合(图6)。

“*”表示保守的氨基酸;“:”表示强相似的氨基酸;“.”表示弱相似的氨基酸
“*”indicate the conserved amino aicd;“:”indicate strong similar amino acid;“.”indicate weak similar amino acid
图5 PmTRADD的死亡结构域氨基酸序列多序列比对
Fig. 5 Multi-alignment of the DEATH domain of PmTRADD amino acid sequence
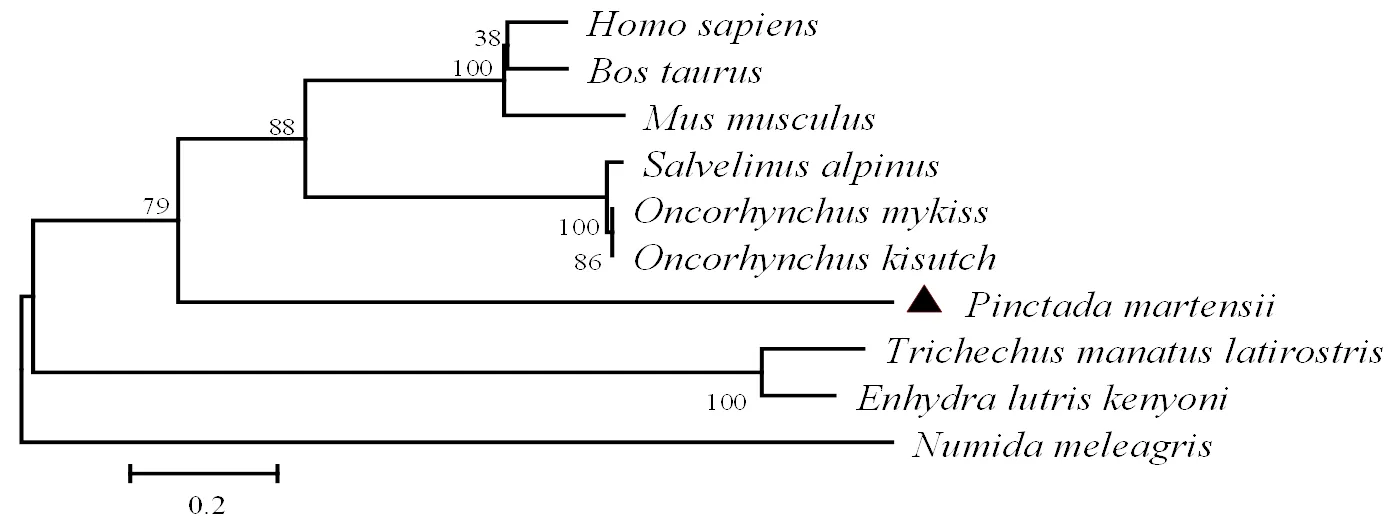
图6 马氏珠母贝TRADD蛋白质序列聚类分析(Neighbor-Joining法)
2.4 PmTRADD组织差异表达分析
实时荧光定量PCR检测在马氏珠母贝闭壳肌等6个组织中的表达情况,2-△△CT法计算相对表达量。发现基因在各组织中均有表达,其中鳃组织表达量最高,其次是肝胰腺,在闭壳肌中基本不表达(图7)。

A:闭壳肌; Gi:鳃; M:外套膜; He:肝胰腺; GO:性腺; B:血细胞;
相同小写字母表示差异不具统计学意义(> 0.05)
A: Adductor muscle; Gi: Gill; M: Mantle; He: Hepatopancreas; Go: Gonads; B: Hemocytes;Same lowercase letters indicate no statistically significant difference (> 0.05)
图7组织表达分布
Fig. 7expression distribution in different tissues
3 讨论
基因编码的蛋白质在C端存在一个死亡结构域。含有这种结构域的基因参与多种死亡受体诱导的细胞凋亡信号转导通路,在细胞正常凋亡中起重要作用。当机体发出凋亡信号或其它刺激时,TRADD被肿瘤坏死因子招募并结合,参与细胞凋亡。TRADD的过度表达导致2种主要TNF诱导的反应,细胞凋亡和NF-κB的激活,它们分别依赖于其与FADD 和TRAF2的特异性相互作用[21]。本实验利用RACE技术成功克隆出基因cDNA全长。经生物信息学分析发现PmTRADD无信号肽和跨膜结构域,说明该蛋白可能为胞内蛋白;二级结构预测中发现TRADD主要以α螺旋和无规则卷曲为主,三级结构中也发现其含有5个α螺旋和1个无规则卷曲,由此可推测两亲性的α螺旋在TRADD蛋白的信号传导和募集其它效应分子的过程中扮演着主要功能。对其结构域预测发现,该蛋白第99~196位氨基酸形成特征性死亡结构域。对死亡结构域部分进行多序列比对,结果表明物种间TRADD保守性较低,与其它6物种间的一致性为32.80%,其中与文昌鱼相似性最高,为37.04%。系统进化树结果显示马氏珠母贝单独聚为一支,与其它物种相离较远。软体动物中,基因的研究未见报道,但与其同源类基因相比较,在结构上均含有DEATH结构域,并且大多数DEATH结构域都由100个左右氨基酸形成,例如在香港牡蛎()中克隆分析的4种肿瘤坏死因子死亡受体基因都含有这些特征[22]。本研究从基因角度探究贝类TRADD蛋白在进化上是相对一致性,发现从软体动物到高等脊椎动物,TRADD蛋白发生较大变异性。
为进一步探讨功能,检测在马氏珠母贝不同组织中表达量情况,发现其在所检测组织中均有不同程度表达,在闭壳肌中基本无表达,在鳃中表达量最高,其次是肝胰腺。虽然在软体动物中未见TRADD更多报道,但在太平洋牡蛎()中发现的与TRADD紧密相关的TRAF2在鳃和肝胰腺中都有很高表达[23];TRAF2作为TRADD蛋白的主要招募对象之一[4],两者在鳃和肝胰腺中共同的高表达现象,可猜测在细胞凋亡和其它免疫反应中,TRADD能迅速招募TRAF2,并完成机能反应。鳃与肝胰腺被认为是贝类参与宿主对病原体的免疫防御反应的主要免疫器官, 是机体受外界刺激,炎症反应和损伤后修复等的主要参与者[24-25]。在马氏珠母贝鳃和肝胰腺中的高表达,说明其可能在马氏珠母贝细胞凋亡等免疫防御反应中担任着重要角色。本研究以马氏珠母贝中基因为研究对象,通过对其序列、理化性质、结构及表达进行研究,为进一步研究其在马氏珠母贝免疫防御中的功能奠定基础。
[1] 陆晓娟, 杨帅, 罗少杰,等. 马氏珠母贝前列腺素E合酶2基因的克隆与表达分析[J]. 基因组学与应用生物学,2019, 39(4):1-14.
[2] 吴羽媛, 郭志颖, 梁海鹰, 等. 马氏珠母贝基因的克隆与表达分析[J]. 广东海洋大学学报, 2017, 37(4):1-7.
[3] KIM J Y, LEE J Y, KIM D G, et al. TRADD is critical for resistance to TRAIL-induced cell death through NF-κB activation[J]. FEBS Letters, 2011, 585(14): 2144-2150.
[4] HSU H, SHU H B, PAN M G, et al. TRADD-TRAF2 and TRADD-FADD interactions define two distinct TNF receptor 1 signal transduction pathways[J]. Cell,1996, 84(2):299-308.
[5] HSU H, XIONG J, GOEDDEL D V. The TNF receptor 1-associated protein TRADD signals cell death and NF-κB activation[J]. Cell,1995,81(4): 495.
[6] ASHKENAZI, A. Death receptors: signaling and modulation[J]. Science,1998,281(5381): 1305-0308.
[7] POBEZINSKAYA Y L, KIM Y S, CHOKSI S, et al. The function of TRADD in signaling through tumor necrosis factor receptor 1 and TRIF-dependent Toll-like receptors[J]. Nature Immunology, 2008,9(9):1047-1054.
[8] CHEN, G. TNF-R1 signaling: a beautiful pathway[J]. Science, 2002, 296(5573): 1634-1635.
[9] MICHEAU O, JÜRG TSCHOPP. Induction of TNF receptor I-mediated apoptosis via two sequential signaling complexes[J]. Cell, 2003, 114(2): 181-190.
[10] WAJANT H, PFIZENMAIER K, SCHEURICH P. Tumor necrosis factor signaling[J]. Cell Death & Differentiation, 2003,10(1): 45-65.
[11] SWARUP V, GHOSH J, DAS S, et al. Tumor necrosis factor receptor-associated death domain mediated neuronal death contributes to the glial activation and subsequent neuroinflammation in Japanese encephalitis[J]. Neurochemistry International, 2008, 52(7): 1310-1321.
[12] SWARUP V, DAS S, GHOSH S, et al. Tumor necrosis factor receptor-1-induced neuronal death by TRADD contributes to the pathogenesis of Japanese encephalitis[J]. Journal of Neurochemistry, 2007, 103(2): 771-783.
[13] CHIO I I C, SASAKI M, GHAZARIAN D, et al. TRADD contributes to tumour suppression by regulating ULF-dependent p19Arf ubiquitylation[J]. Nature Cell Biology, 2012, 14(6): 625-633.
[14] XIE P. TRAF molecules in cell signaling and in human diseases[J]. Journal of Molecular Signaling, 2013, 8(1): 7.
[15] WANG F, WENG H, QUON M J, et al. FADD dominant negative dissipates the proapoptotic signalosome of the unfolded protein response in diabetic embryopathy[J]. American Journal of Physiology - Endocrinology And Metabolism, 2015, 309(10): 861-873.
[16] KRENZ M. Cell biology of ischemia/reperfusion injury[J]. International Review of Cell & Molecular Biology, 2012, 298: 229.
[17] KAZUTAKA T, TAKAHASHI K G. Mechanisms and immunological roles of apoptosis in molluscs[J]. Current Pharmaceutical Design, 2008, 14(2): 131-137.
[18]SOKOLOVA I M. Apoptosis in molluscan immune defense[J]. Invertebrate Survival Journal, 2009, 6(1): 49-58.
[19] ROMERO A, NOVOA B, FIGUERAS A. The complexity of apoptotic cell death in mollusks: An update[J]. Fish & Shellfish Immunology, 2015, 46(1): 79-87.
[20] WANG W, WU Y, LEI Q. Deep transcriptome profiling sheds light on key players in nucleus implantation induced immune response in the pearl oyster[J]. Fish & Shellfish Immunology, 2017, 118(2): 489-495.
[21]MARIE-CÉCILE MICHALLET, MEYLAN E, ERMOLAEVA M A, et al. TRADD protein is an essential component of the RIG-like helicase antiviral pathway[J]. Immunity, 2008, 28(5): 651-661.
[22] XIANG Z , XIAO S , WANG F , et al. Cloning, characterization and comparative analysis of four death receptorTNFRs from the oyster[J]. Fish & Shellfish Immunology, 2016, 59: 288-297.
[23] QU F, XIANG Z, ZHOU Y, et al. A molluscan TNF receptor-associated factor 2 (TRAF2) was involved in host defense against immune challenges[J]. Fish & Shellfish Immunology, 2017, 71: 105-115.
[24] 吴羽媛,梁海鹰,阮尧,等. 马氏珠母贝基因的克隆与组织表达分析[J]. 基因组学与应用生物学, 2018, 37(1): 238-246.
[25] 雷超,贝伟烈,郑哲, 等. 马氏珠母贝B型清道夫受体基因克隆与表达性分析[J]. 广东海洋大学学报, 2017, 37(1): 7-14.
Gene Cloning and Tissue Expression Analysis offrom
HE Jun-jun, LIANG Hai-ying, CHEN Song, FANG Xiao-chen, HUANG Xue-min
(,,524088,)
【Objective】To clone the tumor necrosis factor receptor associated death domain protein (TRADD) gene and analyze its expression patterns in tissues of.【Method】The full-length cDNA sequence ofwas cloned by rapid amplification of cDNA ends (RACE) and the expression of this gene was analyzed by RT-PCR.【Result and Conclusion】Thefull-length cDNA contained 5′ UTR (101 bp) , the 3′ UTR (144 bp) and open reading frame (591 bp) which encoded 196 amino acids. Analysis of deduced amino acids showed that it has no signal peptide and transmembrane domain, and the C-terminus contains a death domain (DEATH). Results from multi-sequence comparisons showed that the DEATH domain of PmTRADD has low homology compared with other species.gene was with higher expression level in the gill, followed by the hepatopancreas, and almost no expression in the adductor muscle.
;; Gene cloning; Real-time PCR
Q78;Q959.215
A
1673-9159(2019)04-0013-07
10.3969/j.issn.1673-9159.2019.04.003
2019-04-12
国家自然科学基金(31472306); 广东省海港建设与渔业产业发展专项(A201608B15)
何军军(1993-),男,硕士研究生,主要从事水产无脊椎动物增养殖和珍珠培育方向的研究。Email: 304458416@qq.com
梁海鹰(1971-),博士,教授。Email: zjlianghy@126.com
何军军,梁海鹰,陈崧,等. 马氏珠母贝基因克隆与组织表达分析[J].广东海洋大学学报,2019,39(4):13-19.
(责任编辑:刘岭)
