基于转录组的曼氏无针乌贼SSR与SNP位点信息分析
2019-07-08郭宝英祁鹏志唐祖蓉刘硕博
孙 扬,郭宝英,祁鹏志,陈 宇,唐祖蓉,刘硕博
(浙江海洋大学海洋科学与技术学院,国家海洋设施养殖工程技术研究中心,浙江舟山 316022)
曼氏无针乌贼Sepiella japonica,俗称墨鱼、目鱼,隶属于软体动物门Mollusca、头足纲Cephalopoda、十腕目Decapoda、乌贼科Sepiidae、无针乌贼属Sepiella[1],主要分布于我国浙江、福建沿海,具有很高的食用、药用及经济价值。浙江省历史上最高年产量达6万t,占当时全省海洋捕捞量的9.3%[2],然而自1980年以后,由于人类活动的愈发频繁造成沿海海域污染,捕捞力度过大,超过其种群自我修复的范围,捕捞产量急剧下降。近年来,为了恢复曼氏无针乌贼的资源,我国开启了一系列人工养殖和增殖放流行动。人工养殖的曼氏无针乌贼1 a可以繁殖2次,半年时间就能长至200 g[3],是我国海水养殖中极具潜力的养殖品种之一。在增殖放流方面,浙江海洋大学研究团队已经成功开展了一系列放流活动,据统计,于2009-2012年累计放流乌贼幼体(卵)3 921.44 万只(颗)[4],于 2013 年至 2017 年累计放流乌贼幼体(卵)4 900.67 万只(颗)。目前对增殖放流成果的主要评估方式是对标记个体的回捕率进行分析。近年来新兴的标记方法有,对鱼鳍鱼尾注入编码微型金属标记[5]、使用四环素处理幼鱼耳石标记[6]和分子标记法。其中编码微型金属标记法仪器费用昂贵,耳石标记法对鱼仔生理发育有一定影响,分子标记法操作简单,无需任何物理标记,仅通过放流个体的基因组DNA获取的遗传信息便可以对其增殖放流效果、种质资源、遗传组成进行科学合理有效的评估。如2012年,我国学者许凌雪[7]使用微卫星标记,对呼玛河哲罗鲑Hucho taimen放流效果进行了评估,近年来在草鱼Ctenopharyngodon idellus[8]、三疣梭子蟹Portunus trituberculatus[9]、许氏平鮋Sebastes schlegelii[10]、魁蚶Scapharca broughtonii[11]和胭脂鱼Myxocyprinus asiaticus[12]等诸多物种放流效果评估中得到了广泛的应用。
在各种分子标记中,简单重复序列(simple sequence repeat,SSR)又称作微卫星和单核苷酸多态性(single nucleotide polymorphisms,SNP),是当今分子育种、遗传多样性分析中应用最广的两种分子标记[13]。SSR普遍存在于真核生物基因组,是由若干个碱基组成的简单串联重复序列。然而传统手段开发SSR标记方法繁琐且成功率低,如利用富集技术筛选SSR标记,需要构建筛选基因文库,实验量大且耗费颇高[14]。而由转录组来源的SSR(EST-SSR)具有数据量大,适用性广等优势,目前已在动植物中广泛应用。在海洋生物中,已有达氏鲟Acipenser dabryanus[15]、凡纳滨对虾Litopenaeus vannamei[16]、白梭吻鲈Sander lucioperca[17]、青石斑鱼Epinephelus awoara[18]等基于转录本数据或EST数据库成功进行SSR标记开发的报道。SNP是由于单个核苷酸的变异而导致基因组层面DNA序列的多态性,通常表现为单个碱基的转换或颠换、缺失或插入[19]。SNP以其在基因组上多态性丰富著称,绝大多数SNP位点分布于基因组的非编码区,少部分分布于基因的编码区的位点通常称为编码SNP,这一类SNP在遗传学研究中具有重要意义[20]。SNP作为新兴的第三代分子标记,相比较于之前的分子标记,具有位点数量多、基因组内分布广泛、遗传稳定性高、检测手段简单且准确率高、某些SNP与生物特定的性状直接相关、某些SNP在不同群体中具有显著差异等优点[21]。目前SNP位点在水生生物中已被广泛应用,如对中国对虾Fenneropenaeus chinensis[22]进行放流效果评估,对青海湖裸鲤Gymnocypris przewalskii[23]、大口黑鲈Micropterus salmoides[24]进行分子育种辅助等。
本文拟通过对曼氏无针乌贼转录组序列分析,分析其SSR位点和SNP位点的组成和特征,完善曼氏无针乌贼分子标记,为今后曼氏无针乌贼的种质资源评估、遗传图谱构建和增殖放流效果评估工作提供有力的研究工具。
1 材料与方法
1.1 材料
用于转录组高通量测序分析的曼氏无针乌贼组织样品于2016年5月采自福建省宁德市苍南县苗种养殖示范基地,该海区水体污染少,繁育养殖技术成熟。选取健康乌贼活体解剖后取其视腺组织,使用液氮速冻保存备用。
1.2 方法
1.2.1 转录组EST微卫星序列获取
曼氏无针乌贼视腺样品总RNA的提取,cDNA文库的构建由华大基因公司完成。构建好的文库质检合格后使用IlluminaHiSeq4000平台进行测序[25]。测序所得的raw reads经过如下步骤筛选:去除包含接头的reads;去除未知碱基N含量大于5%的reads;去除低质量的reads。对过滤后得到的clean reads进行De novo组装。组装后得到的转录本使用MISA[26]工具进行微卫星的搜索。SSR搜索参数设置以不同数量级碱基的重复次数区分,从单碱基到六碱基所需最少重复次数依次为:12、6、5、5、4、4。
1.2.2 SNP位点挖掘
我们使用HISAT[27]将clean reads对比到Unigene,然后使用GATK[28]检测SNP并过滤低质量SNP。
1.2.3 SSR引物设计
使用Primer premier 5对检测到的SSR进行引物设计,筛选引物长度介于18~28 bp,引物退火温度介于 55~65 ℃产物长度筛选范围依次为 80~160 bp,80~240 bp,80~300 bp。
2 结果与分析
2.1 曼氏无针乌贼EST-SSR位点的数量与分布
转录组数据量一共测得44.61 Gb数据,组装并去冗余后得到127 575个Unigene,总长度为103 104 058 bp。共搜索到分布于50 626条序列中的各种SSR位点108 685个,平均每949 bp就含有1个SSR位点,SSR发生率39.68%(含有SSR的Unigene数目与总Unigene的数目之比),出现频率(检出SSR个数与总Unigene数目之比)[29]为85.19%。在50 626含有SSR的Unigene序列中,有25 548条unigene中,含有SSR位点数目在2个及以上。从SSR重复类型分析发现,单碱基重复46 561(42.84%),二碱基重复 31 227(28.73%),三碱基重复 16 230(14.93%),四碱基重复 13 899(12.79%),五碱基重复 603(0.55%),六碱基重复 165(0.15%)(图 1)。
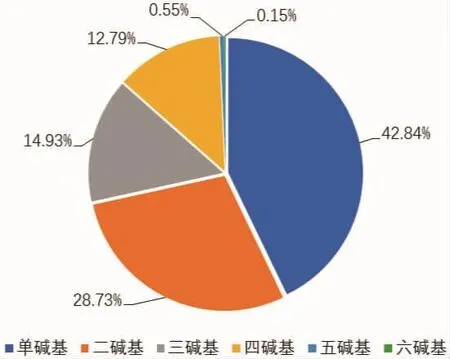
图1 曼氏无针乌贼转录组EST中1-6碱基类型微卫星的组成各类型分布情况图表Fig.1 The percentages of mono-,di-,tri-,quad-,penta-,and hexa-nucleotide repeats in SSR motif sequences in S.japonica transcriptome EST
2.2 曼氏无针乌贼EST-SSR基元类型和比例
从图1可以看出,曼氏无针乌贼EST-SSR重复基元类型丰富,共观察到124种重复基元,从单核苷酸至六核苷酸种类分别有2、4、10、28、45、35。在各碱基类型的微卫星中,碱基重复基元的数量分布具有差异。在这124种重复基元中,各类型重复基元分布如下:单碱基重复基元中以A/T为主,占总SSR的42.23%,C/G占总SSR的0.61%;二碱基重复基元中,AT/AT占比13.33%,AC/GT占比9.32%,AG/CT占比6.06%,剩余合计占比0.03%;三碱基重复基元中,占主体的有:AAT/ATT占比3.97%,AAG/CTT占比3.69%;在单碱基至三碱基中,重复基元 CG/CG(18),ACG/CGT(180),CCG/CGG(152),数量较少。四碱基重复基元中,AAAG/CTTT占比10.00%,AGAT/ATCT占比1.16%,其余26种类型总计占比1.63%。五碱基和六碱基SSR重复基元类型较多,但数目占总数的极少部分,出现频率极低。
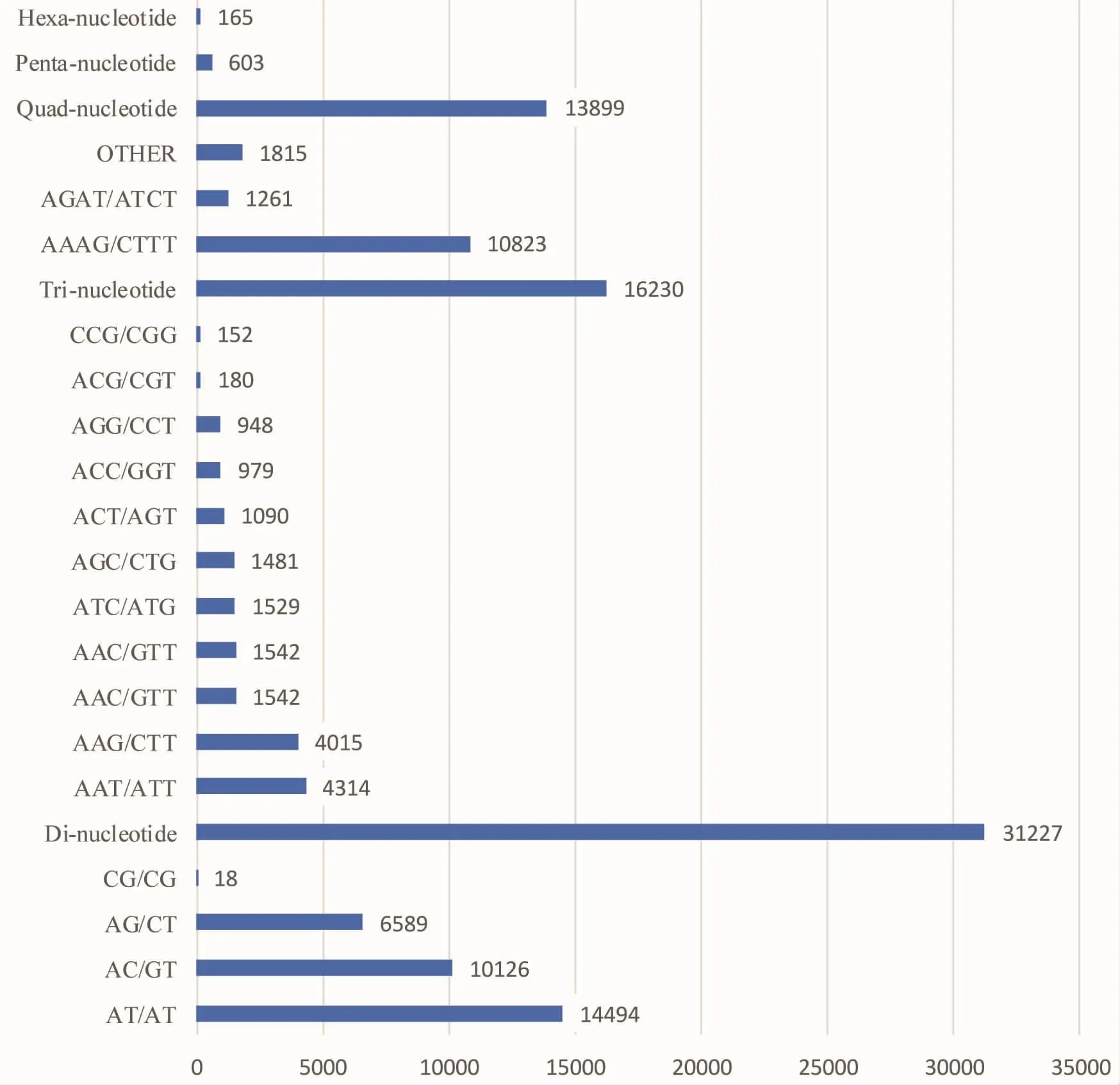
图2 曼氏无针乌贼转录组EST中SSR重复基元类型与数目统计图Fig.2 Observed counts of identified microsatellite loci for different repeat sequence motifs of di-,tri-,quad-,penta-and hexanucleotide repeats in S.japonica transcriptome EST
各数量级碱基的微卫星重复次数的变异范围也存在区别[30]。由表1可见,在二碱基中AC/GT,AG/CT,AT/AT重复次数变异范围为6~62次,集中在6~12次,而CG/CG重复次数变异为6~8次。二碱基中6次重复的微卫星最多,占总二碱基类型的22.03%。三碱基中数目占比较多的AAT/ATT,AAG/CTT重复次数变异范围为5~49次,集中在5~8次,其余重复基元重复次数变异范围为5~27次,集中于5~8次,三碱基中5次重复的微卫星最多,占总三碱基类型的38.43%。四碱基中占比最多的AAAG/CTTT重复次数变异范围为5~34次,集中于5~6次,其余重复基元重复次数变异范围为5~33次,集中于5~6次,四碱基5次重复的微卫星最多,占总四碱基类型的41.31%。五碱基中重复次数变异范围为4~11次,集中于4~5次,重复次数为4的微卫星最多,占总五碱基类型的79.27%。六碱基中重复次数变异范围为4~11次,只有少数微卫星重复次数不为4,重复次数为4的微卫星占总六次重复微卫星比87.27%。由此可见,曼氏无针乌贼中各碱基类型的微卫星位点随着碱基数目的增加,其中重复基元的变异逐渐减少;各碱基类型的微卫星序列均以满足最低重复次数筛选要求的序列最多;随着重复次数要求的增加,相对应的微卫星的数量逐级减少;随着微卫星碱基数的增加,其最高重复次数占比也逐渐增多。重复次数的范围也随着碱基数量增加而减小,KELKAR,et al[31]研究认为重复次数越高的微卫星其多态性也越高,这说明在曼氏无针乌贼中,二碱基微卫星多态性可能较高,随着碱基数量增加多态性也随之降低。

表1 曼氏无针乌贼各碱基重复次数分布表Tab.1 The repeating number of di-,tri-,quad-,penta-,and hexa-nucleotide repeats in SSR motif sequences in S.japonica transcriptome EST
2.3 曼氏无针乌贼SNP位点的数量与分析
SNP是基因组水平上单个碱基的变异,包括置换、颠换、缺失和插入[19]。SNP可在DNA、RNA和蛋白质不同水平影响基因的功能,尤其是位于编码区域内的编码SNP(cSNP)与基因的表达相关,直接影响着功能基因的作用途径[20],cSNP是对基因功能与表型性状进行分析研究的重要途径,通过其分布情况以及功能分析可以得到许多与物种生长发育性状相关的重要资料。
在曼氏无针乌贼转录组中共有12 323条Unigene中,检测到64 732个SNP位点,每条Unigene平均含 5.25 个 SNP 位点(表 2)。其中转换 Transition 45 975 个(71.02%),颠换 Transversion 18 757 个(28.98%)。6种单核苷酸变异中,A/G和C/T发生的频率最高,分别达到36.86%和34.16%。其他4种单核苷酸变异中A/C、A/T、C/G和G/T的频率分别为7.85%、8.78%、4.46%和7.89%。该研究结果极大丰富了曼氏无针乌贼SNP位点信息,为其遗传关联分析、种质资源评估奠定了基础。

表2 曼氏无针乌贼SNP位点分布表Tab.2 Distribution table of SNP locus in S.japonica
2.4 部分EST-SSR引物
将搜索到的SSR位点信息导入至Primer premier 5软件设计引物,从结果中筛选引物长度介于18~28 bp、引物退火温度介于55~65℃、产物长度筛选范围依次为80~160 bp的引物,挑选部分引物如表3所示。

表3 曼氏无针乌贼EST-SSR引物表Tab.3 Primer of EST-SSR in S.japonica
3 讨论
在各种DNA分子标记中,SSR与SNP标记技术已被广泛应用于分子育种,遗传分析,亲缘鉴定等科学研究活动中[32]。随着二代高通量测序技术的发展,通过转录组数据库来进行分子标记的发掘逐渐成为一种高速、有效的方法。在这之前通过DNA建库或探针富集法所开发出的曼氏无针乌贼SSR引物约25对[33-34],而吕振明等[30]使用De novo高通量测序法获得并验证了65对可用的SSR引物,说明该方法可靠、高效。本文对曼氏无针乌贼视腺组织转录组测序获得的127 575条Unigene序列,共得到分布于50 626条序列中的各种SSR位点108 685个,SSR发生率39.68%,出现频率为85.19%。平均每949 bp就含有1个SSR位点。利用Primer premier 5软件设计生成引物346 520对。本研究在搜索到的微卫星位点中,以一、二、三、四碱基占微卫星碱基类型的主体,而五、六碱基微卫星数量极少。微卫星数量随着碱基数量增加而减少,随着重复次数的增加而减少。本次研究搜索所得的微卫星数量多、类型丰富,可为今后进行曼氏无针乌贼微卫星引物的开发丰富材料,为其增殖放流效果评估提供有力的研究工具。
SNP位点标记作为第三代遗传标记技术具有位点出现频次高、基因组分布广、易于检测等优点,在海洋动物中可被应用于遗传图谱构建、QTL定位、关联分析等方面研究[35]。本研究在12 323条Unigene中共检索到64 732个SNP位点,SNP位点数量丰富,每条Unigene平均含5.25个SNP位点。其中转换Transition45 975个(71.02%),颠换Transversion18 757个(28.98%)。6种单核苷酸变异中,A/G和 C/T发生的频率最高,分别达到36.86%和34.16%。其他4种单核苷酸变异中 A/C、A/T、G/T和 C/G的频率分别为7.85%、8.78%、4.46%和7.89%。本研究首次报道了曼氏无针乌贼SNP位点,为后续曼氏无针乌贼与其它乌贼的进化关系、对其生长发育性状的分型研究等提供了重要的研究工具。
SSR标记作为一种高效的分子标记手段,其缺点是SSR标记具有特异性,须使用PCR进行验证[29]。本研究在后期还将会对已搜索得到的SSR位点的相应引物进行验证,以探索其多态性和稳定性。SNP作为分子标记常用检测方法有Taqman法、芯片法、酶切法、质谱法、测序法等[36],本研究后期拟选用可靠性高的限制性内切酶酶切法来对SNP进行检测。本研究结果表明通过高通量测序技术联合生物信息学方法开发SSR、SNP位点是一种高效、快速的方法。目前在曼氏无针乌贼中尚未有对SNP进行相关研究的报道,本研究有望对曼氏无针乌贼SNP的开发提供可靠的指导,同时对曼氏无针乌贼资源的种质评估、遗传分析、增殖放流效果评估等提供有力的研究工具。
