基于高通量测序的黄姑鱼转录组从头组装和基因注释分析
2019-06-17王余菊娄方瑞水柏年
王余菊,娄方瑞,2,水柏年
(1.浙江海洋大学水产学院,浙江舟山 316022;2.中国海洋大学水产学院,山东青岛 266003)
黄姑鱼Nibea albiflora,隶属于鲈形目Perciformes、石首鱼科Sciaenidea、黄姑鱼属Nibea。黄姑鱼较为广泛的分布在中国近海及日本和朝鲜半岛海域,其作为东海较为重要的捕捞和养殖种类,具有重要的经济价值[1-2]。然而,近年来随着黄姑鱼捕捞量的增加,其自然资源量逐步下降,黄姑鱼养殖业迅速发展,目前全国黄姑鱼养殖年产量已超过3 万t[3-4]。国内外学者在黄姑鱼的基础生物学、人工繁育、养殖生态和分子生物学等各方面已开展了较为广泛研究[4-6]。值得注意的是,关于黄姑鱼基因组学方面的数据信息较少,其基因组组装结果尚未发表,限制了黄姑鱼分子育种工作的开展。
转录组通常是指在生物的某一状态下,由特定的组织或细胞所转录出的全部RNA 的集合,反应了当时基因表达的情况[7]。转录组测序能够在缺少基因组信息的前提下,揭示基因与生物学特性和外界环境之间的关系,能够有效地开发大量基因资源[8]。转录组测定是对于生物基因功能及其结构进行分析的首要环节,且其相比较于全基因组测序,具备更为迅速的测序速度和更低的测序成本。目前转录组测序并已在大黄鱼Larimichthys crocea[9]、鮸鱼Miichthys miiuy[10]、口虾蛄Oratosquilla oratoria[11]、大竹蛏Solen grandis[12]和曼氏无针乌贼Sepiella maindroni[13]等多个海洋生物中得到广泛应用,ZHAN Wei,et al[14]报道了黄姑鱼转录组测序组装结果,Unigenes 的N50 为1 279 bp,组装后转录本序列较短,这可能影响后续应用。为了进一步开发黄姑鱼的基因资源,本研究中以采自舟山近海的黄姑鱼自然个体为材料,利用Illumina Hiseq XTen平台获得黄姑鱼转录组序列遗传变异等信息,从中挖掘黄姑鱼的基因数据和和微卫星分子标记,以期为后续黄姑鱼及其他黄姑鱼属鱼类的分子学探究提供数据基础。
1 材料与方法
1.1 样品采集
转录组测序所采用的黄姑鱼样品于2018 年10 月采自舟山附近海域,在20 ℃的实验室海水环境内暂养6 d,暂养期间不投喂,采集活体黄姑鱼的多种组织(肝、卵巢、鰓、眼和肌肉)分别用液氮速冻并保存在-80 ℃的超低温环境中。
1.2 RNA 提取、文库构建和上机测序
液氮研磨并等量混匀黄姑鱼组织,而后加入标准Trizol Reagent Kit 试剂获取混匀后组织的总RNA。基于琼脂糖凝胶电泳、Nanodrop 分光光度计、Qubit 荧光定量仪和Agilent 2100 生物分析仪对总RNA 的降解程度、纯度、浓度和完整性分别进行检测。检测合格后,基于磁珠法富集混匀后组织总RNA 中的mRNA,随后将其打断成特定短片段并以此为模板进行文库构建。委托北京诺禾致源公司使用Illumina Hiseq XTen 平台对库检合格的测序文库进行双末端150 bp 测序。
1.3 原始测序数据过滤和质量评估
使用Trimmomatic 软件对于测序平台中获取的原始测序序列(Raw Reads) 进行过滤以获取高质量的Clean Reads,其中,需要对带有测序接头的Raw Reads、Qphred<=20 的碱基达到所有碱基数量50%以上的Raw Reads 以及无法确定的碱基信息比例高于10%的Raw Reads 进行剔除;剩余的高质量Clean Reads 经FastQC 软件进行质量评估后用于后续分析。
1.4 转录本拼接和基因功能注释
使用Trinity 2.4.0 软件[15]对Clean Reads 进行从头组装获取转录本序列(Transcripts),所使用的参数为:--min_kmer_cov:3。基于map 到转录本序列上的reads 数及表达模式,Corset 1.05 软件[16]进一步对获取的转录本(Transcripts)进行层次聚类,从而获取最长的聚类序列(Unigenes),软件参数使用默认参数。Unigenes序列用于后续分析。而后,利用Diamond 和Blast 等比对软件基于七大基因注释数据库Nr (Non-Redundant Protein Sequence Database)、Nt (Nucleotide Sequence Database)、Pfam (Pfam protein families database)、KOG(Clusters of Orthologous Groups of proteins)、Swiss-prot,KEGG(Kyoto Encyclopedia of Genes and Genomes)和GO (Gene Ontology)对所有的Unigene 进行比对和注释,从而获得黄姑鱼的表达基因功能,基因功能注释软件及参数设置如表1。Diamond 是一个用于比对数据库蛋白,其蛋白质比对性能为blast+的500~20 000 倍,因此比对Nr、KOG 和Swiss-prot 等3 个蛋白数据库采用Diamond 软件。

表1 基因功能注释软件及参数Tab.1 The software and parameters associated with gene functional annotation.
1.5 编码序列(CDS)预测与简单重复序列标记(SSR)位点搜索
编码序列(cording sequences,CDS)预测分析步骤如下[11]:首先,将聚类获取的Unigenes 依次比对到Nr和Swissprot 蛋白库并获取比对成功的Unigenes 中的编码框核苷酸序列;第二,应用estscan3.0.3 软件预测未比对成功的Unigenes 中的编码序列核苷酸信息;最后,按照5'->3'的顺序,将编码阅读框碱基序列翻译为氨基酸序列。转录中的微卫星序列(simple sequence repeats,SSR)则主要依靠MISA 1.0 软件进行检测[13],软件设置使用默认参数,其中,重复单位及其最少重复次数分别设定为:单核苷酸重复10 次、双核苷酸重复6 次、三核苷酸重复5 次、四核苷酸重复5 次、五核苷酸重复5 次和六核苷酸重复5 次。
2 结果
2.1 黄姑鱼转录组测序质量评价和序列组装
转录组测序共获取43 385 432 条Raw Reads,经过去除低品质的原始序列,得到42 809 266 条高质量的Clean Reads。质量评估结果表明,Clean Reads 的碱基错误率、Q20、Q30 和GC 含量值分别为0.02%、97.15%、92.28%和49.27%。Clean Reads 拼接为57 654 条Transcripts,Transcripts 的平均长度和N50 值分别为1 950 bp 和3 255 bp;所有的transcripts 进一步聚类后获得32 623 条Unigenes,Unigenes 的平均长度和N50 值分别为1 646 bp 和2 777 bp。Trianscripts 和Unigenes 在不同长度区间内的频数统计结果如图1 所示。

图1 黄姑鱼转录组Transcripts 和Unigenes 长度分布Fig.1 Distribution of the length of transcripts and unigene in the N.albiflora transcriptome.
2.2 基因功能注释
2.2.1 基因功能注释成功率统计
基于7 大数据库对黄姑鱼转录组的Unigenes 序列进行基因功能分析,注释成功率如表2 所示。结果表明,28 645 条Unigenes(87.81%)匹配到至少一个数据库中,占总Unigene 的;7 023 条Unigenes(21.52%)可以匹配到所有数据库中。

表2 注释结果统计Tab.2 The annotation results.
2.2.2 Unigene 序列相似性分析
为了获取黄姑鱼基因序列及其功能信息,并进一步确定黄姑鱼与其近缘种基因序列的相似度,将转录组中的Unigenes 与NCBI 中的Nr 数据库进行比对。结果表明,黄姑鱼转录组中16 291、1 106、665、370 和346 条Unigenes 分别与大黄鱼、尖吻鲈Lates calcarifer、高体鰤Seriola dumerili、贝氏隆头鱼Labrus bergylta 和眼斑双锯鱼Amphiprion ocellaris的基因序列具有较高的相似性,剩余的3 085 条Unigenes 与132 种其他物种的基因序列具有相似性(图2)。
2.2.3 Unigene 功能分类
9 605 条在KOG 数据库中注释成功黄姑鱼转录组的Unigene 被归属于26 个KOG 功能大类中(图3)。其中,数量最多的功能类为信号转导机制(1 843 条),而后依次为一般功能预测(1 507 条)、转录后修饰、蛋白折叠和分子伴侣(946 条)等。

图2 Nr 数据库中Unigenes 注释信息Fig.2 The annotation information of all unigenes blast in Nr database
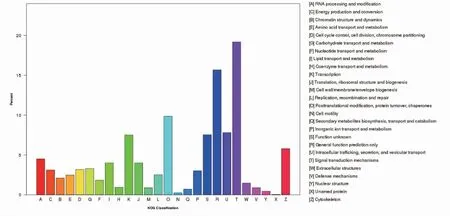
图3 黄姑鱼转录组Unigenes 的KOG 分类Fig.3 KOG classification of unigenes in the N.albiflora transcriptome
此外,与GO 数据库进行比对结果表明,17 812 条匹配成功的Unigenes 被富集在1 590 个GO 功能词条中,如图4。其中,在3 个GO 大类生物过程(Biological process)、细胞成分(Cellular component)和分子功能(Molecular Function)中分别有54 612、33 293 和22 539 条Unigenes 获得注释信息。在生物过程大类中,富集在细胞过程(GO:0008152)最多Unigenes 数量较多,达10 595 条,其次是代谢过程(GO:0009987)词条为8 527 条;在细胞成分大类中,富集在细胞(GO:0005623)和细胞组分(GO:0044464)词条中的Unigenes 数量较多,均为6 042 条;在分子功能大类中,富集在结合(GO:0005488)最多为11 082 条,其次是催化活性(GO:0003824)中词条为6 928 条。

图4 黄姑鱼转录组Unigenes 的GO 分类Fig.4 GO classification of unigenes in the N.albiflora transcriptome.
2.2.4 Unigene 代谢途径分析
参与同一代谢通路的黄姑鱼的Unigene 可以通过KEGG 分析富集在一起。黄姑鱼转录组数据的代谢途径分析结果表明,所有的Unigenes 与KEGG 数据后进行比对后,14 754 条Unigene 显著富集在231 种代谢通路中。其中,各大类中富集Unigene 数量较多的通路如图5 所示,涉及信号转导、内分泌系统、免疫系统等。

图5 黄姑鱼转录组Unigenes 的KEGG 分类Fig.5 The KEGG pathways of unigenes in the N.albiflora transcriptome (A,Cellular Processes; B,Environmental Information Processing; C,Genetic Information Processing; D,Metabolism; E,Organismal Systems).
2.3 CDS 预测和SSR 位点分析
通过Nr 和Swissprot 数据库,16 803 个CDS 从比对成功的Unigenes 中被提取;使用estscan 对未比对成功的Unigene 进一步分析后获取13 579 个预测CDS。同时,我们共获取了29 022个SSR 标记,这些标记位于13 508 条Unigenes 中,如图6 所示。结果表明,SSR 标记的数量与SSR 标记的重复单位数量成负相关,单碱基重复类型SSR 标记数量最多,为14 702 个;六碱基重复类型SSR 标记数量最少,仅为40 个。从SSR 重复基序角度来说,AC 重复是黄姑鱼最普遍的SSR 标记,此外,最普遍的三碱基重复是AGG 重复,AAAC 重复则是最普遍的四碱基重复。
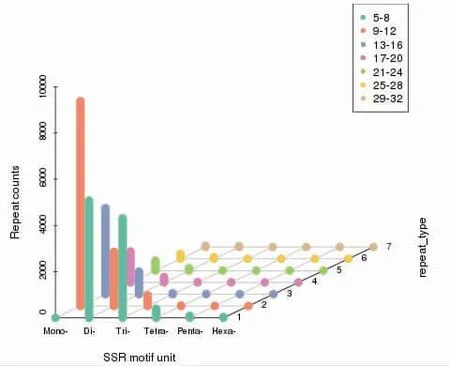
图6 黄姑鱼转录组中SSR 分析统计结果Fig.6 Number of SSRs in the N.albiflora transcriptome
3 讨论
3.1 黄姑鱼转录组测序与组装
近年来,转录组测序的优势性已经促使其在许多缺乏基因组信息的海洋生物中得到广泛应用。本次研究对黄姑鱼的多种组织进行混匀后首次测定了其转录组信息,测序结果丰富和完善了黄姑鱼的基因数据库资源。对原始序列中的低质量序列经过剔除后,约有98.67%的高质量序列。此外,对获取的高质量序列进行质控发现,序列的碱基错误率较低且具有相对较高的质量值。这进一步证明了本次黄姑鱼的转录组测序信息的准确性相对较高[22]。黄姑鱼转录组拼接出的Transcripts 和Unigenes 的N50 值分别为3 255 bp 和2 777 bp,同之前的黄姑鱼转录组组装结果相比有明显提高(UnigenesN50 值为1 279 bp)[14],表明黄姑鱼的转录本组装质量较好,适用于后续的基因功能分析。
3.2 功能注释
在转录组数据挖掘中,基因功能注释是极为关键的环节之一。本次研究中,约有87.81%的Unigenes 在至少一个数据库中成功获得功能注释,较高的注释率再次验证了本次转录组测序的大数据量和转录本拼接的有效性。此外,蛋白数据库中石首鱼科蛋白质信息较为丰富也是黄姑鱼转录组高注释率的原因。Nr 注释结果表明,大量的Unigenes 与大黄鱼的基因序列得到了匹配,这是因为大黄鱼的基因组测序早已完成,且其与黄姑鱼均属于石首鱼科[2]。另外,功能聚类和通路富集的分析也为我们快速挖掘与黄姑鱼生物学特性相关的基因奠定了基础。
3.3 分子标记筛选
在物种鉴定、群体遗传学及遗传图谱构建等研究中,分子标记开发可以提供较为有价值的基础信息。有研究认为,基因编码区域中可能存在着广泛的分子标记,而转录组测序获取的大的信息量实际上可能涵盖了成千上万的表达基因,因此,转录组测序技术可能在分子标记开发方面具有较大的优势性[7]。本研究中,MISA 软件共搜索到29 022 个SSR 位点,从而进一步丰富了黄姑鱼的分子标记资源,但所有的SSR 的有效性仍需进一步验证。
4 结论
本研究初次对黄姑鱼的转录组信息进行了测序,捕获的转录组数据有效性和可信度较高,从而丰富了黄姑鱼的基因数据库资源。但约有13%的Unigenes 没有得到注释,这些可能是黄姑鱼特有的基因转录本或是由于组装错误形成的转录本,需要后续三代测序数据进行进一步验证。此外,转录本的注释结果也为黄姑鱼生物学调控机制的基因功能、分子事件和信号通路奠定了基础。大量的SSR 位点的获取可为后续黄姑鱼分子标记的开发提供了参考。黄姑鱼转录本数据获得同时也为石首鱼科物种分化研究、种群适应进化研究、遗传多样性研究和系统发育学研究提供了数据保障,为黄姑鱼属种质资源的开发利用奠定了基础。
