南麦号系列小麦品种的染色体结构演变
2019-06-14唐宗祥王淑荣
葸 玮,蒋 进,唐宗祥,王淑荣
(1.四川农业大学植物遗传育种省级重点实验室/四川农业大学农学院,四川成都 611130;2.四川省南充市农业科学院,四川南充 637000)
评价亲本对其后代衍生品种的遗传贡献有利于小麦育种过程中的亲本选择。在这一类遗传分析中,分子标记是常用的工具。比如,DArT、SSR和Illumina 90KiSelect SNP等标记被广泛用来分析小麦骨干亲本对其衍生品种的遗传贡献[1-8]。这些分子标记能较好地追踪骨干亲本传递给后代的重要染色体区段,有利于这些区段的分子标记辅助选择。但分子标记不能反映染色体区段的具体结构变异,尤其是对于小麦背景中的外源染色体的存在状况,以上提到的分子标记方法不能很好地鉴定。因此,利用荧光原位杂交(FISH)技术,从染色体结构变异方面研究小麦亲本及其衍生系的基因组变异,会对分子标记分析结果起到很好的补充作用。近年来发展起来的非变性荧光原位杂交(nondenaturing fluorescenceinsituhybridization,ND-FISH)技术对这方面的研究具有促进作用。依赖于寡核苷酸探针的ND-FISH技术,具有方便、快捷和经济的优点[9-11]。利用寡核苷酸探针和ND-FISH 技术,可以反映不同小麦品种(系)的染色体结构差异[12-15],同时还可以体现同一串联重复序列的不同区段在不同染色体上的分布差异[12-13]。因此,利用基于寡核苷酸探针的ND-FISH技术,可以快速、方便地分析小麦亲本及其衍生系的染色体结构差异,从而跟踪亲本材料染色体在衍生系中的变异情况,这有助于小麦育种理论的完善和加强。本研究拟利用南麦号系列品种及其亲本,研究小麦亲本与其后代衍生品种的染色体结构变异情况,以期为小麦育种提供更直观的遗传变异参考。
1 材料与方法
1.1 供试材料
小麦亲本材料攀早抗及其衍生品种南麦302、南麦618、特研麦南88和荣春南麦1号用于本研究,南麦302、南麦618、特研麦南88的系谱均为16-1/攀早抗,荣春南麦1号的系谱为2001-43-1/攀早抗。由于16-1和2001-43-1的种子遗失,没有用于本研究。
1.2 方 法
1.2.1 根尖细胞有丝分裂中期染色体制片
供试小麦材料的根尖细胞有丝分裂中期染色体制备参照Han等描述的方法[16]。每份材料至少取5粒种子。
1.2.2 非变性荧光原位杂交(ND-FISH)
寡核苷酸探针Oligo-275.1、Oligo-275.2、Oligo-pSc119.2-1和Oligo-pTa535-1用于ND-FISH分析。其中Oligo-275.1和 Oligo-275.2根据已公布的串联重复序列pTa-275设计而得[12,17],Oligo-pSc119.2-1根据已公布的pSc119.2家族串联重复序列设计[10],Oligo-pTa535-1根据已公布的串联重复序列pTa-535设计而得[10,17]。ND-FISH分析参照Fu等[11]和Tang等[12]的方法。对于每一粒种子的根尖,至少观察5个细胞。
2 结果与分析
2.1 2A、6A、7A、5B、1D和2D染色体结构变异
从Oligo-pSc119.2-1的FISH带型看,小麦亲本攀早抗含1对1RS/1BL易位染色体(图1、图2),其衍生品种南麦302、特研麦南88和南麦618都含1RS/1BL易位染色体(图1、图2),仅荣春麦南麦1号是非1RS/1BL易位系品种(图1、图2)。寡核苷酸探针Oligo-275.1、 Oligo-275.2、 Oligo-pSc119.2-1和Oligo-pTa535-1反映出了2A、4A、5A、6A、7A、3B、5B、6B、1D和2D染色体在攀早抗及其衍生品种中的结构差异(图1、图2、图3)。
对于2A、6A、7A、5B、1D和2D染色体,品种间的结构差异主要由探针Oligo-275.1和 Oligo-275.2体现。攀早抗和南麦302的2A染色体无Oligo-275.1信号,其余品种的2A染色体都有该探针信号(图1、图3)。仅特研南麦88和荣春南麦1号的6A染色体带有Oligo-275.1的信号(图1、图3)。攀早抗的7A染色体不含Oligo-275.1和 Oligo-275.2信号,Oligo-275.1和Oligo-275.2信号在南麦302的7AL臂出现,但在特研麦南88、荣春南麦1号和南麦618的7AS和7AL臂都出现;Oligo-275.2的信号出现在特研麦南88的7AS和7AL臂以及南麦618的7AL臂,而荣春南麦1号7A染色体不含Oligo-275.2信号(图1、图2、图3)。除南麦618的5B染色体外,其余品种的5B染色体都带有Oligo-275.2信号(图2、图3)。攀早抗和南麦618的1D染色体无Oligo-275.1信号,其余品种的1D染色体都有该探针信号(图1、图3)。除特研麦南88的2D染色体外,其余品种的2D染色体都不带有Oligo-275.2信号(图2、图3)。
2.2 4A、5A、3B和6B染色体结构变异
攀早抗、南麦302、特研南麦88和荣春南麦1号的4AL臂都带有Oligo-pSc119.2-1和Oligo-pTa535-1的信号,以及较弱的Oligo-275.1信号,而南麦618的4AL臂不含Oligo-pSc119.2-1和Oligo-275.1信号(图1、图3)。攀早抗的5AS端部含Oligo-pSc119.2-1信号,5AL含Oligo-pTa535-1、Oligo-275.1和 Oligo-275.2信号;南麦302、特研南麦88和荣春南麦1号的5AS臂都不含Oligo-pSc119.2-1信号,而这3个品种的5AL臂都含Oligo-pSc119.2-1、Oligo-pTa535-1、Oligo-275.1和 Oligo-275.2信号;南麦618的5A染色体除了在5AS端部含Oligo-pSc119.2-1信号外,其余探针信号都不存在(图1、图2、图3)。几个品种的3B和6B染色体的结构差异主要由Oligo-pSc119.2-1信号体现。Oligo-pSc119.2-1在5个品种的3BS端部和亚端部都产生3个信号位点,攀早抗和南麦618的3BS端部Oligo-pSc119.2-1信号弱,而其余3个品种的该位置信号强(图1、图2、图3)。南麦618的6BS端部无Oligo-pSc119.2-1信号,其余4个品种的6BS端部都含有该信号(图1、图2、图3)。

A、C、E、G和I用寡核苷酸探针Oligo-pSc119.2-1(绿)和Oligo-pTa535-1(红)进行分析;B、D、F、H和J 用寡核苷酸探针Oligo-275.1(绿)进行分析。A和 B:攀早抗同一细胞;C和D:南麦302同一细胞;E和F:特研麦南88同一细胞;G和 H:荣春南麦1号同一细胞;I和 J:南麦618同一细胞。Oligo-pSc119.2-1主要在染色体的端部、亚端部和臂间产生信号(A、C、E、G和I),Oligo-275.1主要在着丝粒、近着丝粒和亚端部产生信号(B、D、F、H和J)。
A,C,E,G and I:Oligo probes Oligo-pSc119.2-1(green) and Oligo-pTa535-1(red) were used for ND-FISH; B,D,F,H and J:Oligo probe Oligo-275.1(green) was used for ND-FISH. A and B:The same cell of Panzaokang; C and D:The same cell of Nanmai302; E and F:The same cell of Teyanmai Nan 88; G and H:The same cell of Rongchun Nanmai 1; I and J:The same cell of Nanmai 618. Signals of Oligo-pSc119.2-1 mainly appeared on the telomeric,subtelomeric and intercalary regions of chromosomes(A,C,E,G and I). Oligo-275.1 mainly produced signals on the centromeric,pericentromeric and subtelomeric regions(B D,F,H and J).
图1 攀早抗及其衍生品种的ND-FISH分析结果
Fig.1 ND-FISH analysis of Panzaokang and its derived wheat varieties.

A、C、E、G和I用寡核苷酸探针Oligo-pSc119.2-1(绿)和Oligo-pTa535-1(红)进行分析;B、D、F、H和J 用寡核苷酸探针Oligo-275.2(绿)进行分析。A和B:攀早抗同一细胞;C和 D:南麦302同一细胞;E和 F:特研麦南88同一细胞;G和 H:荣春南麦1号同一细胞;I和J:南麦618同一细胞。Oligo-pSc119.2-1主要在染色体的端部、亚端部和臂间产生信号(A、C、E、G和I),Oligo-275.2主要在着丝粒、近着丝粒和亚端部产生信号(B、D、F、H和J)。
A,C,E,G and I:Oligo probes Oligo-pSc119.2-1(green) and Oligo-pTa535-1(red) were used for ND-FISH;B,D,F,H and J:Oligo probe Oligo-275.2(green) was used for ND-FISH. A and B:The same cell of Panzaokang; C and D:The same cell of Nanmai 302; E and F:The same cell of Teyanmai Nan 88; G and H:The same cell of Rongchun Nanmai 1; I and J:The same cell of Nanmai 618. Signals of Oligo-pSc119.2-1 mainly appeared on the telomeric,subtelomeric and intercalary regions of chromosomes(A,C,E,G and I). Oligo-275.2 mainly produced signals on the centromeric,pericentromeric and subtelomeric regions(B,D,F,H and J).
图2 攀早抗及其衍生品种的另一ND-FISH分析结果
Fig.2 Another ND-FISH analysis of Panzaokang and its derived wheat varieties
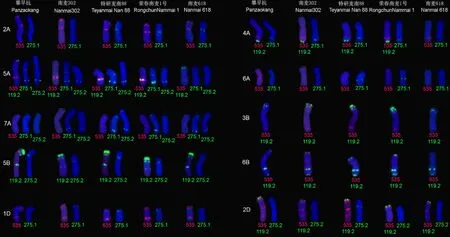
535 表示寡核苷酸探针Oligo-pTa535-1(红);119.2表示寡核苷酸探针Oligo-pSc119.2-1(绿);275.1表示寡核苷酸探针Oligo-275.1(绿);275.2表示寡核苷酸探针Oligo-275.2(绿)。
535 represents oligo probe Oligo-pTa535-1(red); 119.2 represents oligo probe Oligo-pSc119.2-1(green); 275.1 represents oligo probe oligo-275.1(green); 275.2 represents oligo probe oligo-275.2(green).
图3 攀早抗及其衍生品种中有结构变异染色体的截图对比
Fig.3The chromosomes for comparison among the structural alteration chromosomes of Panzaokang and its derived varieties
3 讨 论
3.1 FISH核型分析能够跟踪小麦品种的系谱
寡核苷酸探针Oligo-275.1和 Oligo-275.2能够反映出小麦A组染色体和部分B、D组染色体的结构差异[12]。本研究中,所用材料的2A、6A、7A、5B、1D和2D染色体的结构差异主要由这两个探针体现。结合Oligo-pSc119.2-1和Oligo-pTa535-1的信号模式,4A、5A、3B和6B染色体的结构差异也被检测到。尽管与攀早抗组合的另两个亲本16-1和2001-43-1没有用于本实验,但可以推测:特研麦南88、荣春南麦1号和南麦618的2A,南麦618的4A、5B和6B,4个衍生品种的5A和7A,特研南麦88和荣春南麦1号的6A,南麦302、特研南麦88和荣春南麦1号的3B和1D以及特研南麦88的2D,这些染色体的主要区段可能不是来自攀早抗,而是另一个亲本。这一结果为进一步对这些材料进行分子标记分析提供了细胞学基础。
3.2 FISH核型可以反映品种间染色体的不同重组情况
本研究所用材料中,南麦302、特研麦南88和荣春南麦1号都来自同一组合16-1/攀早抗。但这3个品种的2A、6A、7A和2D染色体结构不同。南麦302的2A和6A、南麦302和荣春南麦1号的2D染色体与攀早抗的相应染色体结构相同,对于这3对染色体的重组情况不好推测。而这3个品种中的7A染色体的结构变异却比较复杂。根据Oligo-275.1和 Oligo-275.2的信号模式,可以作如下推测:亲本16-1的7A染色体两个臂的亚端部都带有 Oligo-275.1和 Oligo-275.2的信号,南麦302中的7A可能在两亲本的短臂发生了重组,特研麦南88的7A可能在短臂和长臂都发生了重组,荣春南麦1号的7A也在短臂和长臂发生了重组,但重组位点与特研麦南88的7A不同。
3.3 FISH技术在研究小麦品种(系)染色体结构演变中的作用
有不少研究表明,以串联重复序列为探针的FISH技术可以反映小麦品种(系)之间的染色体结构变异[14-15,18-20]。Jiang等[14]利用Oligo-pSc119.2-1、Oligo-pTa535-1和(AAG)6三种寡核苷酸探针对85个小麦品种(系)的染色体进行了分析,结果表明,每一个小麦品种(系)都有自己独特的FISH核型。Huang等[15]对2000多份来自中国的小麦材料进行了基于寡核苷酸探针的高分辨率的FISH分析,结果发现这些小麦材料的染色体存在易位、近着丝粒倒位和缺失等结构变异,根据FISH核型,这些小麦材料可以分为4大类。本研究结果也表明,基于寡核苷酸探针的ND-FISH技术可以用来推测小麦品种系谱,反映染色体之间的交换重组关系。因此,可以建立小麦染色体FISH核型,从染色体结构水平来反映小麦品种(系)间的遗传多样性,从而推断小麦品种(系)的系谱及演变过程。FISH技术能够真实地反映染色体的结构变异,可以为分子标记技术提供更直观的参考。此外,基于寡核苷酸探针的ND-FISH技术具有方便、快捷和经济的优点[11],可以用来规模化分析小麦亲本及其衍生后代。随着适宜于ND-FISH分析的新型寡核苷酸探针的不断开发,该技术在小麦品种(系)染色体结构演变研究中的作用将更加突出。
综上所述,利用ND-FISH技术可以检测小麦染色体的结构差异,反映染色体的重组情况,进行小麦品种系谱跟踪,明确小麦染色体组成及遗传背景,为进一步的分子标记分析提供可靠的参考。
