小麦GRAS基因家族的全基因组鉴定与分析
2019-06-14李亚飞阳文龙顾晶晶张爱民詹克慧
李亚飞,阳文龙,顾晶晶,张爱民,,詹克慧
(1.河南农业大学农学院/河南粮食作物协同创新中心, 河南郑州 450002;2.中国科学院遗传与发育生物学研究所/植物细胞与染色体工程国家重点实验室, 北京 100101)
GRAS基因是一类转录因子基因,参与调控植物的生长发育[1],由GAI(gibberellic acid insensitive)、RGA(repressor of GAI-3 mutant)和SCR(scarecrow) 3个家族成员命名。早期认为GRAS基因家族是植物特有的转录因子基因家族,然而,近期一项研究表明该基因家族最早存在于细菌基因组中,并提出GRAS基因家族归属于Rossmann折叠甲基转移酶超家族[2]。典型的GRAS蛋白至少含350个氨基酸残基[1],GRAS基因编码的蛋白通常是由可变的N末端序列和高度保守的C末端组成,其C端包含LRⅠ(leucine-rich region Ⅰ)、VHⅡD、LRⅡ(leucine-rich region Ⅱ)、PFYRE和SAW共5个高度保守的结构域[3]。VHⅡD结构域是GRAS蛋白的核心结构域,存在于所有家族成员中,其中只有组氨酸和天冬氨酸是绝对保守的。目前,对该基因家族成员的鉴定工作相继在拟南芥、水稻、杨树和葡萄等几个物种完成,在早期的拟南芥GRAS基因家族系统发育分析中,将其分为8个亚家族,分别命名为DELLA、LS、SCR、SHR、PAT1、HAM、SCL9(LISCL)和SYN4/7[4]。随后,GRAS基因家族被分为了10个亚家族,即:DELLA、AtLAS(LS)、AtSCR、AtSHR、AtPAT1、HAM、LISCL、AtSCL3、SCL4/7和DLT[5],但是,在这些系统发育分析中,来自水稻的GRAS蛋白Os4和Os19未被分配到亚家族中去。近期,利用拟南芥、水稻、杨树、桃树、葡萄和番茄等物种的GRAS蛋白,通过序列分析构建拟南芥、水稻和杨树的系统发育进化树,将GRAS基因家族分为13个亚家族,分别为AtSHR、AtPAT1、AtSCR、AtSCL4/7、 AtLAS、Os19、HAM、Os4、Pt20、DLT、AtSCl3、DELLA和LISCL,其中,Os4、Os19和Pt20是新鉴定的亚家族,Pt20是杨树特有的亚家族[1]。由于GRAS基因亚家族的多样性,导致该基因家族功能的多样性。研究表明,在植物的生长发育中,GRAS基因在植物激素、光、生物和非生物胁迫等多种生长调节和环境信号中发挥重要作用。例如,DELLA亚家族的AtGAI参与拟南芥中赤霉素的信号转导[6]。AtLAS控制拟南芥叶腋分生组织的形成[7],番茄的Ls[8]和水稻的OsMOC1[9]也是AtLAS亚家族的一员,具有相同的功能。OsMOC1作为水稻分蘖的关键控制因子,在揭示水稻高产的分子机理上具有重要意义。SCL13参与光敏色素B(phyB)的信号转导,在脱黄化过程中使胚轴拉长[10]。AtHAM1、AtHAM2和AtHAM3参与拟南芥顶端分生组织和叶腋分生组织的发育[11-13]。AtPAT1亚家族中的AtPAT1、AtSCL5、AtSCL21基因作为拟南芥光敏色素A信号转导途径的正向调控因子[14-15]。GRAS基因还参与植物对多种非生物胁迫的响应,研究表明胡杨AtSCL4/7亚家族的PeSCL7过表达增强了转基因拟南芥的抗旱和耐盐性[16];AtSCL14和TGA转录因子相互作用可促进拟南芥逆境诱导基因的表达,增强其抗逆能力[17]。
总之,目前已经对许多植物中的GRAS基因家族进行了鉴定及系统分析,如拟南芥中鉴定出34个GRAS基因[1],水稻60个[1]、杨树106个[1]、番茄53个[18]。小麦作为世界上重要的粮食作物,对其生长发育起调控作用的GRAS基因是至关重要的。但由于小麦是异源六倍体,基因组巨大且复杂,至今尚未见其GRAS基因家族的研究报道。因此,本研究利用小麦基因组信息通过生物信息学方法对小麦GRAS基因家族的组成在全基因组水平进行鉴定,并进一步对基因结构、染色体分布、启动子顺式作用元件、表达谱进行分析,以期为后续小麦GRAS基因的功能研究提供参考。
1 材料与方法
1.1 小麦GRAS基因的全基因组鉴定
参考邢光伟等对小麦LBD基因的全基因组鉴定方法[19],从Ensembl Plants 数据库(http://plants.ensembl.org/index.html)下载拟南芥、水稻和小麦蛋白序列数据库并提取已报道的拟南芥34个GRAS蛋白序列和水稻的60个GRAS蛋白序列。通过BLAST构建小麦蛋白序列本地数据库,将拟南芥和水稻的GRAS蛋白序列作为query序列进行BLASTP比对(E-value<1E-5),获得这些序列的同源序列并删除其中的重复序列。同时,在Pfam数据库(http://pfam.xfam.org/)下载GRAS保守结构域(PF03514)作为模板序列,利用HMM3.0比对得到含有保守结构域的蛋白序列。将上述两种比对方法获得的候选蛋白序列合并,去除重复序列。进一步利用Pfam序列搜索(http://pfam.xfam.org/)、SMART序列分析(http://smart.embl-heidelberg.de/)和HMMER序列分析(https://www.ebi.ac.uk/Tools/hmmer/search/hmmscan)验证候选蛋白是否含有GRAS保守结构域,删除不含或者缺失GRAS结构域的候选基因,最终获得小麦GRAS基因。
1.2 系统发育进化树的构建和基因结构分析
利用Clustal_W工具将拟南芥、水稻和小麦GRAS蛋白序列进行多重比对,将比对结果放入MEGA7.0软件,在p-distance模型、Bootstrap参数1000和成对删除选项下采用邻接法(NJ)构建系统发育进化树。从Ensembl Plants 数据库下载的小麦基因组数据用于提取小麦GRAS基因的DNA query和CDS query,利用在线软件GSDS2.0(http://gsds.cbi.pku.edu.cn/)进行基因结构分析。
1.3 小麦GRAS基因编码蛋白的理化性质分析、二级结构分析和亚细胞定位
利用Expasy提供的Protparam(https://web.expasy.org/protparam/)在线软件计算小麦GRAS蛋白的氨基酸长度、分子量和等电点。利用SOPMA在线软件(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl? page= npsa_sopma.html)预测分析α-螺旋、β-折叠和无规则卷曲。借助Cello软件[20],利用小麦GRAS基因或蛋白序列进行亚细胞定位。
1.4 小麦GRAS基因染色体定位和顺式作用元件分析
根据IWGSC(https://wheat-urgi.versailles.inra.fr/)公布的最新小麦基因组数据(CS1.0) BLASTN构建本地数据库,通过比对获得小麦GRAS基因在染色体上的物理位置。利用MG2C(http://mg2c.iask.in/mg2c_v2.0/)在线软件构建小麦GRAS基因在染色体上的物理图谱。利用Perl程序截取小麦各GRAS基因上游1.5 kb的DNA序列,并将其提交PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)和NEWPLACE在线软件(https://sogo.dna.affrc.go.jp/cgi-bin/sogo.cgi?sid=&lang=en&pj=640&action=page&page =newplace)进行启动子顺式作用元件的预测并手动整理。
1.5 串联重复和片段复制
小麦Rht-D1c(GRAS基因家族DELLA亚家族)的等位基因Rht-D1b是控制株高的关键基因,在其大于1 Mb区域发现该基因有两个拷贝且为串联重复(TSD)[21]。因此,在小麦GRAS基因大于1 Mb区域鉴定是否发生串联重复事件。利用从Ensembl Plants同源基因数据库下载的GRAS基因的共线性分析数据,鉴定小麦GRAS基因是否发生片段复制事件[22]。发生串联重复和片段复制基因的非同义替换率(Ka)和同义替换率(Ks)的比值用软件KaKs_Calculator2.0[23]进行计算,预测分析基因CDS区的适应性进化。
1.6 小麦GRAS基因组织表达和逆境表达分析及部分GRAS基因的Real-time PCR验证
利用Wheat Exp数据库(https://wheat.pw.usda.gov/WheatExp/)下载小麦5个组织或器官(根、茎、叶、穗和种子)、干旱胁迫(1 h、6 h)、热胁迫(1 h、6 h)和干旱加热胁迫(1 h、6 h)的转录组数据FPKM(fragments per kilobase of transcript per million mapped reads)值,作为表达分析数据。最后利用Morpheus(https://software.broadinstitute.org/morpheus/)在线软件构建基因表达热图。采用RNeasy Plant Mini Kit(QIANGEN,德国)提取小麦开花后15天的根、茎、叶、穗子和种子的RNA,使用FastQuant RT Kit(With g DNase)(QIANGEN,德国)进行反转录cDNA第一条链合成,用于Real-time PCR 分析。Real-time PCR仪器型号为Roche LightCycler 480(Roche,瑞士),使用试剂为Light Cycler 480 CYBR Green Mix(Roche,瑞士),以Ta4045为内参基因。PCR程序设置为:95 ℃ 5 min; 95 ℃ 10 s, 60 ℃ 10 s,72 ℃ 20 s,共45个循环;95 ℃ 10 s,65 ℃ 1 min,40 ℃ 10 s。每个样品重复3次。
2 结果与分析
2.1 小麦GRAS基因全基因组鉴定结果
根据拟南芥34个GRAS基因和水稻60个GRAS基因的蛋白序列,利用BLASTP和HMM比对搜索,在Ensembl Plants最新小麦基因组数据库中鉴定出169个GRAS候选基因。进一步利用Pfam、SMART、HMMER结构域搜索检验GRAS候选基因是否含有完整结构域,发现其中的16个GRAS基因有不完整的结构域。例如在小麦GRAS候选基因(TRIAE_CS42_3B_TGACv1_223416_AA0781900)结构域片段仅含有67个氨基酸,候选基因(TRIAE_CS42_5BL_TGACv1_408118_AA1361700) 结构域只含有319个氨基酸,而其HMM模型显示完整GRAS结构域有374个氨基酸组成,认为这些GRAS结构域严重缺失的基因不属于GRAS基因[24],那些结构域不完整的候选基因可能是假基因。最后,在小麦基因组中鉴定出153个GRAS基因,并根据基因在染色体上的位置命名。
2.2 系统发育进化树的构建和基因结构分析
为了构建小麦GRAS基因家族的进化树,分别利用拟南芥、水稻和小麦34、60、153个GRAS基因蛋白序列进行多序列比对,结果发现部分GRAS基因蛋白序列导致系统发育进化分析的可靠性降低,因此我们删除了导致可靠性降低的GRAS基因,最终利用拟南芥(33个)、水稻(50个)和小麦(134个)GRAS基因进行了系统发育进化分析。结果(图1)发现,根据进化树拓扑结构可将GRAS蛋白分为12个亚家族,这些亚家族的命名参照前人的研究结果[25-26]进行。小麦GRAS基因的12个亚家族分别为:AtSHR、AtPAT1、AtSCR、AtSCL4/7、AtLAS、Os19、HAM、Os4、DLT、AtSCL13、DELLA和LISCL。在12个亚家族中均包含拟南芥、水稻和小麦的GRAS基因,暗示GRAS基因的分化发生在单子叶和双子叶分化之前。同时在进化树拓扑结构中发现,小麦GRAS基因在亚家族中分布不均匀,亚家族LISCL中包含43个小麦GRAS基因,而亚家族DLT、Os19和AtSCL4/7中分别含有1、3、3个小麦GRAS基因。为了研究小麦GRAS基因结构的多样性和系统发生情况,仅对来自小麦的134个GRAS基因的蛋白序列构建系统发育进化树,结果也分为12个亚家族(图2)。在系统发育树末端节点鉴定出45个同源基因对或簇,例如Ta2BSGRAS37的同源基因Ta2DSGRAS45,Ta4ALGRAS77的同源基因Ta4BSGRAS93和Ta4DS111。这些同源基因在小麦GRAS基因中占到了75.8%。随后,通过比较小麦GRAS基因结构,发现高达74.5%(114/153)的小麦GRAS基因没有内含子。此外,观察发现,同一亚家族的小麦GRAS基因表现出相似的基因结构。但也有例外,如Ta3ALGRAS51和Ta3BLGRAS55、Ta4ALGRAS72和Ta4BSGRAS94、Ta4DLGRAS98和Ta5ALGRAS116等具有不同的基因结构,这可能是由于基因在进化过程中内含子的丢失或增加造成的。
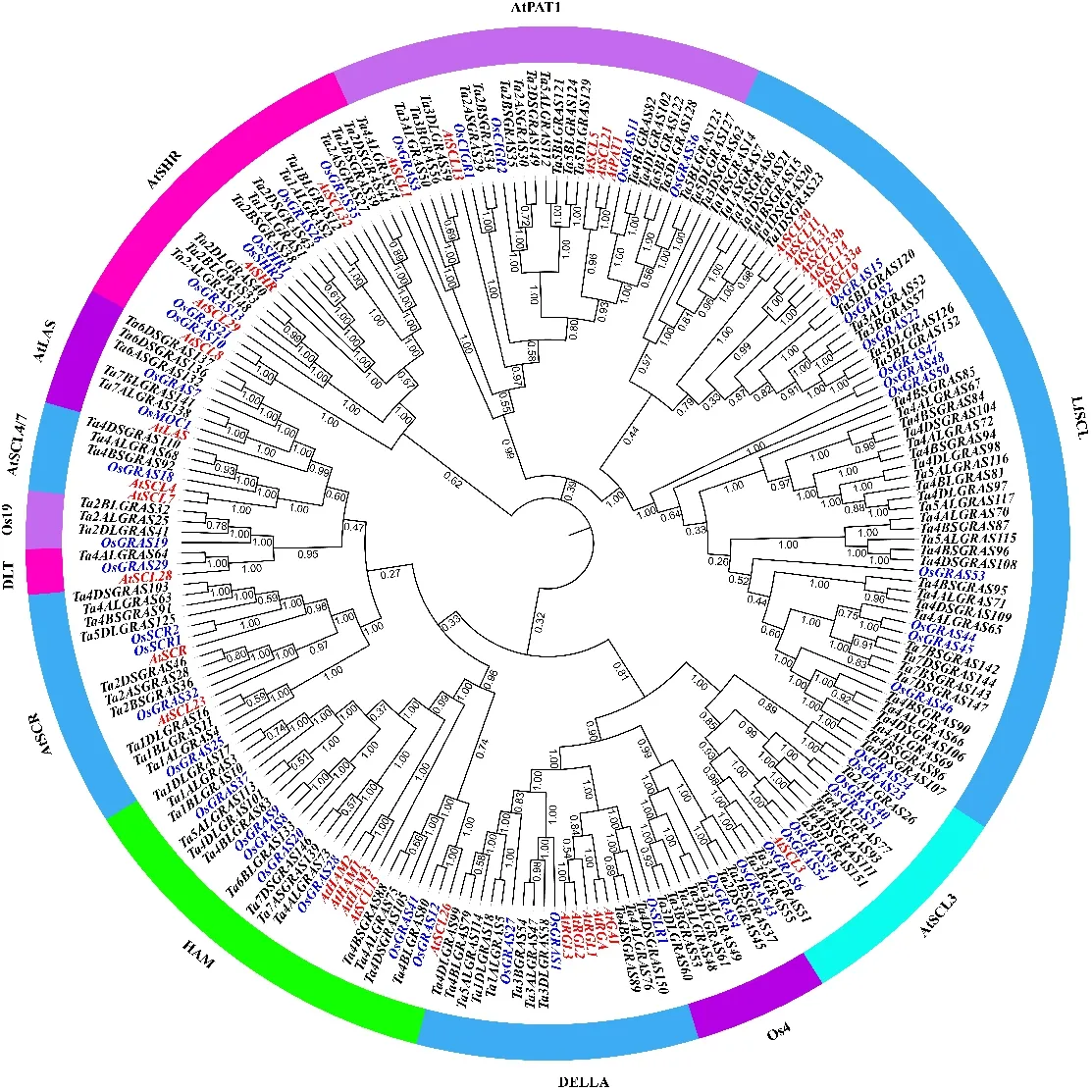
小麦、拟南芥和水稻GRAS基因分别用黑色、红色和蓝色字体表示。
GRAS genes from wheat,Arabidopsisand rice are represented in black, red and blue fonts, respectively.
图1 小麦、拟南芥和水稻GRAS基因家族的系统进化树
Fig.1 Phylogenetic tree of GRAS genes in wheat,Arabidopsisand rice

图2 小麦GRAS基因家族的系统进化和基因结构
2.3 小麦GRAS基因编码蛋白的理化性质和二级结构分析及亚细胞定位
蛋白序列一级结构理化性质分析显示,153个GRAS基因编码区长度为1 083~5 130 bp,编码含有361~1 710个氨基酸残基的蛋白质,分子量为39.9~192.9 kDa,等电点范围为 4.73~ 9.33。不同亚家族间氨基酸数目和理化性质存在一定的差异,如AtPAT1亚家族平均氨基酸残基数目最多(803个),AtSCL3亚家族的氨基酸残基数目最少(442个)。除了少数几个GARS基因氨基酸序列理论等电点在碱性范围内,其余都在酸性范围内,说明GRAS基因蛋白质分子富含酸性氨基酸。SOPMA预测蛋白质二级结构显示,小麦153个GRAS基因的氨基酸序列均含有α-螺旋、随机卷曲、扩展链和β-转角,各GRAS基因编码蛋白质二级结构中α-螺旋含量为31.1%~ 55.5%,随机卷曲含量为27.4%~57.4%,扩展链含量为7.1%~20.7%,β-转角含量为2.8%~ 9.9%。同时,亚细胞定位显示67个小麦GRAS基因定位在细胞核中,33个定位在细胞质中,26个定位在叶绿体中,14个定位在线粒体中,12个定位在质膜中,1个定位在过氧化物酶体中。
2.4 小麦GRAS基因染色体定位和顺式作用元件分析
根据定位结果(图3),在小麦的A、B、D三个同源染色体组中,均含有51个GRAS基因,暗示小麦GRAS基因在小麦两次自然杂交加倍过程中,同源染色体的保留和丢失在亚基因组间没有明显偏好性。GRAS基因在小麦A、B、D三个同源染色体组上分布是均匀的,而在不同染色体上分布不均匀,且与染色体长度无关。GRAS基因在1A、1B和1D,2A、2B和2D,3A、3B和3D,4A、4B和4D,5A、5B和5D,6A、6B和6D,7A、7B和7D的数目分别为9、6和8,8、9和8,6、5和5,16、18和16,7、8和7,2、2和3,3、3和4。其中,4A、4B和4D染色体上分布的GRAS基因最多(50个),在6A、6B和6D染色体上分布的GRAS基因最少(7个)。
利用NEWPLACE在线软件分析小麦GRAS基因启动子序列的顺式作用元件,结果共鉴定到了56种267个顺式作用元件,除了含有启动子基础作用元件CAAT-Box、GATA-Box、TATA-Box等之外,还含有多种与激素应答、组织特异性表达、胁迫诱导等相关的顺式作用元件。其中,叶肉特异表达相关元件(CACTFTPPCA1)和细胞分裂素响应元件(ARR1AT)在所有的小麦GRAS基因的启动子区域都检测到,平均每个GRAS基因分别含有17.2和9.4个。小麦151个GRAS基因含有光照响应元件GATA-Box,暗示小麦GRAS基因表达可能受光调节。
2.5 串联重复和片段复制分析
分析发现小麦中含有13个串联重复,例如Ta1ASGRAS7、Ta1ASGRAS8和Ta1ASGRAS9等基因簇存在串联重复事件(图3红色字体为串联重复基因)。每个串联重复包含2~8个GRAS基因。其中4B染色体上有最大的串联重复,包含8个GRAS基因。同时,在4D染色体上包含2个串联重复。总之,小麦中有45个GRAS基因为串联重复,表明它们的起源涉及串联重复事件。通过分析Ensembl Plants中小麦GRAS基因的同源基因,发现138个基因具有同源基因,表明GRAS基因起源于片段复制。利用KaKs_Calculator2.0 软件,将发生串联重复和片段复制的GRAS基因的CDS区进行比对、YN法计算分析。结果发现,除了Ta6DSGRAS136和Ta6DSGRAS137串联重复基因簇的Ka和Ks均为0之外,其余的12个串联重复GRAS基因簇和所有发生片段复制的同源基因簇的Ka均小于Ks。其中12个串联重复的平均Ka/Ks为0.188~ 0.535,片段复制的平均Ka/Ks为0.037~0.523。根据串联重复和片段复制的GRAS基因在密码子水平上的Ka/Ks值,可以推测,除Ta6DSGRAS136和Ta6DSGRAS137串联重复基因簇外,其余的GRAS基因受负选择作用。
2.6 小麦GRAS基因组织表达和逆境表达分析及部分GRAS基因的Real-time PCR验证
利用Wheat Exp数据库中小麦GRAS基因在种子、叶、根、穗和茎的RNA-seq数据进行分析,发现小麦GRAS基因在不同的组织或器官的表达模式有明显的差异(图4a)。其中17个GRAS基因在种子中具有较高的表达量,41个GRAS基因在叶中高表达,53个GRAS基因在根中高表达,29个GRAS基因在穗子中高表达,21个GRAS基因在茎中表达量高。这些结果表明,在叶和根中高表达的GRAS基因多于穗和茎中表达的GRAS基因。进一步分析发现,Ta2DSGRAS45和Ta2DSGRAS149在5个器官或组织中都是高表达;部分GRAS基因在某一组织或器官中明显高表达,在其他4个器官或组织中低表达或无表达,例如Ta2ALGRAS25、Ta2ALGRAS26、Ta2BLGRAS32等在根中高表达,而在其他4个组织或器官中低表达,表明这些GRAS基因可能在小麦根部的形态建成中发挥重要作用。

图3 小麦GRAS基因在染色体上的定位(红色字体代表串联基因簇)

a:热图表示小麦GRAS基因在不同组织中的表达;b:热图表示小麦GRAS基因在干旱及热胁迫下的表达。D1和D6:干旱胁迫处理1 h和6 h;H1和H6:热胁迫处理1 h和6 h;DH1和DH6:干旱加热胁迫处理1 h和6 h。
a:Heatmap showing expression of wheat GRAS genes in different organs or tissues; b:Heatmap showing expression of wheat GRAS genes under drought and heat stress conditions. D1 and D6:Treatment for 1 h and 6 h under drought stress conditions; H1 and H6:Treatment for 1 h and 6 h under heat stress conditions; DH1 and DH6:Treatment for 1 h and 6 h under drought and heat stress conditions.
图4 小麦GRAS基因在不同组织、干旱及热胁迫下的表达
Fig.4 Expression profiles of wheat GRAS genes in different organs or tissues,
and under drought and heat stress conditions

图5 根和种子发育相关的GRAS基因在不同组织或器官的相对表达量
同时,用RNA-seq数据分析小麦GRAS基因在热和干旱胁迫下的表达情况。结果显示(图4b),和对照相比,Ta3DSGRAS62、Ta1BSGRAS13、Ta1BSGRAS14、Ta1BSGRAS15、Ta5BLGRAS119、Ta5BLGRAS120和Ta5DLGR AS126在干旱处理1 h和6 h表达量没有发生明显变化,处于较低水平,当热处理1 h和6 h时表达量有所提高,当干旱加热处理6 h时,表现出明显的上调表达。Ta4BSGRAS78、Ta6ASGRAS132、Ta6BSGRAS134、Ta6DSGRAS136、Ta6DSGRAS137、Ta7ASGRAS140在干旱6 h时表达量上调,表明它们在干旱胁迫响应过程中可能发挥重要作用。此外,Ta4ALGRAS77、Ta4BLGRAS80、Ta2DSGRAS45、Ta2DSGRAS149、Ta4DLGRAS100与对照相比,干旱和热胁迫处理前后表达量均未发生明显变化,表明这些基因的表达可能不受干旱和热胁迫的影响。
同时,用Real-time PCR技术对与根和种子发育相关的6个GRAS基因进行了定量表达分析实验。结果(图5)表明,Ta1ALGRAS2、Ta1BLGRAS12、Ta1DLGRAS19、Ta2BLGRAS32和Ta5BLGRAS152在根中的表达量均高于茎、叶、穗和种子中的表达量,其结果和Wheat Exp数据库中相应小麦GRAS基因在种子、叶、根、穗和茎中的表达一致。Ta4ALGRAS77在种子中的表达量高于根、茎、叶和穗中的表达量,其结果也验证了该基因在Wheat Exp数据库中的表达情况。
3 讨 论
本研究首次对小麦的GRAS基因家族进行了综合分析,根据基因组注释,在小麦中发现了153个GRAS基因,多于拟南芥(34个)和水稻(60个)GRAS基因家族成员的数目。这主要可能是因为小麦是异源六倍体,在A、B、D染色体组通常存在同源基因,同时,小麦GRAS基因家族发生了扩张,最终导致小麦GRAS基因家族比拟南芥和水稻庞大。串联重复和片段复制是植物基因家族扩张的主要方式[22],这些基因通过串联重复和片段复制保留在植物基因组中,在对环境刺激的适应性反应中起重要作用[27-28]。早期主要是在拟南芥和水稻中对GRAS基因家族的扩张方式进行了分析[25],本研究结果也发现串联重复和片段复制是小麦GRAS基因家族主要的扩张方式。
无内含子或单个外显子基因是原核生物基因的典型特征,然而它们在真核生物基因组中也占很大的比例,例如无内含子基因在拟南芥、水稻和扁豆基因中分别占21.7%、19.9%和18.9%[29]。真核生物基因组中的无内含子基因可能来自古老原核生物基因水平转移或已存在无内含子基因的复制[30]。植物GRAS基因家族中的无内含子成员可能起源于原核生物,然后在植物中广泛复制。基因启动子区域的顺式作用元件通过响应不同外界环境信号来调节基因转录过程,进而影响植物的生长发育[31]。本研究发现,小麦GRAS基因启动子区域有56种267个顺式作用元件,主要包括叶肉特异表达相关元件CACTFTPPCA1、光响应元件GATABOX和干旱胁迫响应元件MYB2CONSENSUSAT等,表明小麦GRAS基因在调节小麦生长发育、参与光及逆境胁迫等环境调节中发挥重要作用。
Peng等[32]对拟南芥和水稻等的研究表明,DELLA亚家族的AtGAI基因参与拟南芥中赤霉素信号转导。本研究中的DELLA亚家族基因Ta4DSGRAS150和Ta4BSGRAS89在小麦的茎中高表达,而叶、穗、根和种子中表达量相对较低,暗示这两个基因在小麦茎的伸长中发挥关键作用。Ls是AtLAS亚家族的成员,在番茄的腋生分生组织中发挥关键作用[8]。本研究中,Ta6ASGRAS132、Ta6DSGRAS136、Ta6DSGRAS137、Ta7ALGRAS138和Ta7BLGRAS141属于AtLAS亚家族成员,它们在根中的表达量远远高于在其他组织或器官中的表达量,暗示这些基因在小麦分蘖上具有重要作用,但是具体的功能还需进一步的实验验证。
