基于GC-MS研究不同产地三色藜麦种子的代谢物差异
2019-06-11时羽杰李兴龙唐媛董雨邬晓勇
时羽杰 李兴龙 唐媛 董雨 邬晓勇


摘要:为了研究不同生境对藜麦发育的影响,以不同产地的藜麦种子为研究对象,筛选显著差异代谢物质和相关代谢通路,利用GC-MS平台进行非靶向代谢组学分析,分析了青海都兰县和四川盐源县两地产出的黑藜、红藜、白藜种子的代谢轮廓,将原始数据经XCMS online平台预处理后进行后续多元统计分析。结果表明,青海都兰县和四川盐源县两地的黑、红、白藜麦种子的代谢特征均不相同,其中红藜麦种子与白藜麦种子差异较大,而黑藜麦种子差异最小;红藜存在显著差异,共有特征峰570个,上调物少于下调物;黑藜差异最小,上、下调趋势与红藜一致;白藜差异较大,上、下调趋势与红藜和黑藜相反,共有特征峰486个,上调物质多于下调物质;不同生境对藜麦的影响较大,黑藜和红藜更适宜在青海都兰县种植,而白藜更适宜在四川盐源县种植。研究结果可以指导青海都兰县和四川盐源县两地藜麦的栽培育种,也为深入研究藜麦的抗逆性机理提供了理论依据。
关键词:作物栽培学;藜麦;GC-MS;XCMS;代谢差异;抗逆性
中图分类号:TS210.1文献标志码:A
Abstract:In order to study the effects of different habitats on the development of quinoa, taking quinoa seeds from different habitats as research objects, significant differences in metabolites and related metabolic pathways are screened. The metabolic profiles of the seeds of black quinoa, red quinoa and white quinoa from Dulan County, Qinghai Province and Yanyuan County, Sichuan Province are analyzed through non-targeted metabonomics analysis with GC-MS platform. The original data are pretreated by XCMS online platform for subsequent multivariate statistical analysis. The results show that the metabolic characteristics of black, red and white seeds in Dulan county and Yanyuan County are different. Among them, the difference between red and white seeds from the two counties is larger, while the difference of the black quinoa seeds is the smallest. There are 570 characteristic peaks in red seeds which have significant differences, and the up-regulated substances are less than the down-regulated. The difference between black quinoa from two habitats is the smallest, and the up- and down-regulated trends are like that of red quinoa. There is secondly significant difference between the white seeds from the two habitats, and the up- and down-regulated trends are the opposite of that of the red and black quinoas, and 486 characteristic peaks are detected, with more up-regulated substances than down-regulated. Different habitats have great impact on quinoa. Black and red quinoas are more suitable to be planted in Dulan County, Qinghai Province, while white quinoa is more suitable to be planted in Yanyuan County, Sichuan Province. The conclusions could guide the cultivation and breeding of quinoa in Dulan county and Yanyuan County, and provide theoretical basis for further study of the stress resistance mechanism of quinoa.
Keywords:crop cultivation;quinoa;GC-MS;XCMS;metabolic differences;stress resistance
代謝组学(metabonomics/metabolomics)是继基因组学、蛋白组学之后又一门新兴的学科,是系统生物学的重要组成部分,旨在研究生物体或组织,甚至单个细胞的全部小分子代谢物(MV<1 000)成分及其动态变化[1],根据分析目的的不同又可以分为靶向代谢组学和非靶向代谢组学。靶向代谢组学主要针对几种目标化合物或某条通路上涉及的部分代谢物,利用标准品,构建特异性强、灵敏度高、重复性好的检测方法,对目标物质进行分析。非靶向代谢轮廓分析方法面向所有能检测到的化合物,可以较全面地反映整体代谢状态的变化,并通过多元统计分析筛选差异代谢物,对差异代谢物进行通路分析,揭示其变化的生理机制,在转基因植物非预期效应研究[2]、非生物与生物胁迫研究中显示出了极大的潜力。
藜麦,古代印加人称之为“粮食之母”[3]。藜麦含有蛋白质、矿物质、氨基酸、纤维素、维生素等营养成分,含量明显高于大多数常见谷物[4]。联合国粮农组织于2013年正式将藜麦推荐为最适宜人类的“全营养食品”[5]。藜麦还含有丰富的多酚类、黄酮类、皂苷等活性成分[6],具有耐寒、耐旱、耐瘠薄、耐盐碱等生理特点。藜麦种子主要有白、黑、红、黄等几种颜色系,营养品质差异较大,藜麦中含有生物碱、多酚等物质。黑藜中总多酚的含量最高,明显高于红藜和白藜,而红藜和白藜中总多酚含量差异不大[7]。赵亚东[8]研究发现青海的不同色系藜麦中,红藜含皂苷最多,抗氧化性极强,白藜抗氧化性最弱。这些研究均表明,藜麦的生境对籽粒发育影响显著。鉴于目前国内从代谢组学方面对不同产地藜麦的研究甚少,本研究主要以非靶向代谢组学研究青海都兰县和四川盐源县两地的黑、白、红藜麦,以獲取较为全面的代谢物质信息,全面、准确地分析其差异物质和代谢通路,分析3种藜麦种子代谢物在不同生境下的异同,为揭示藜麦在不同栽培地区造成的差异等后续研究提供理论依据,指导藜麦的耕作与栽培。
1主要材料与仪器
1.1材料
分别选2018年采收自青海省海西蒙古族藏族自治州都兰县(平均海拔3 000 m)和四川省凉山州的盐源县(平均海拔2 000 m)的黑、红、白色藜麦种子,经发芽试验筛选,取发芽率和发芽势相同的一批进行实验,每一个样品均重复6次。文中所提到的青海都兰县黑藜、红藜、白藜均以A,C,E表示,四川盐源县黑藜、红藜、白藜均以B,D,F表示。
1.2试剂
核糖醇,甲氧氨基盐酸盐,N-甲基-N-三甲基硅基三氟乙酰胺(MSTFA),购自Sigma Aldrich公司;吡啶,甲醇,氯仿,三甲基氯硅烷均为色谱纯,成都科隆化学品有限公司提供。
1.3仪器
Agilent GC-MS 7890B 5977气质联用仪,产地美国;DB-5MS毛细管气相色谱柱(30 m×0.25 mm,0.25 μm),购自安捷伦公司(美国);THZ-82水浴恒温振荡器,常州荣华仪器制造有限公司提供;通风橱、H1850R台式高速冷冻离心机,产地湖南;ZLS-2真空离心浓缩仪,湖南赫西仪器有限公司提供。
2实验方法
2.1样品制备
分别取来自青海都兰县和四川盐源县两地的黑、红、白藜麦种子,参考LISEC等[9]的研究方法对植物样本进行提取,条件稍作优化。对样品进行6次生物学重复,并做质控样本QC,QC样本是将所有提取物的均等混合样随机插入检测的样本序列中,主要用于实验方法和仪器稳定的校正。将所有样品置于EP管,于真空离心浓缩仪中进行挥干备用。
2.2样品衍生化
在上述干燥后的样品管中加入60 μL甲氧氨基盐酸盐(20 mg/mL吡啶溶液),于37 ℃振荡反应2 h。反应完毕后再加入100 μL的N-甲基-N-三甲基硅基三氟乙酰胺和1%(体积分数,下同)三甲基氯硅烷(MSTFA+1%TMCS),于37 ℃振荡反应30 min。将衍生化完成后的样品用0.22 μm滤膜过滤[10]后转移至适合GC-MS分析的内衬管中。
2.3GC-MS分析条件
2.3.1色谱条件
采用DB-5MS(30 m×0.25 mm,0.25 μm)毛细管气相色谱柱;升温程序:起始温度保持在80 ℃,2 min后以10 ℃/min升至190 ℃,再以3 ℃/min升至220 ℃,最后以20 ℃/min升至280 ℃,保持10 min;载气:氦气(体积分数为99.999%);恒流,流速为1 mL/min;进样模式:不分流进样,进样量为1 μL。
2.3.2质谱条件
电子碰撞内能:70 eV;离子源温度:220 ℃;传输线温度:270 ℃;注射剂温度:280 ℃;扫描速度:每20 s光谱全波段扫描法捕获;溶剂延迟:420 s;质量扫描范围m/z:70~500。
2.4数据处理
将GC-MS原始数据[11]上传至XCMS online程序,进行行峰识别(peaks identification)、峰过滤(peaks filtration)、峰对齐(peaks alignment)等[12],同时设置相应参数[13],最后得到包括质核比(m/z)和保留时间(rt)及峰面积等信息的数据矩阵。将数据导出至Excel进行后续整理,用SIMCA-P(v14.0) 软件进行主成分分析(PCA)和正交-偏最小二乘判别分析(OPLS-DA),结合XCMS分析筛选差异代谢物,进行后续的差异代谢物统计分析。
3结果与分析
3.1主成分分析
原始数据经XCMS进行峰识别、过滤、对齐等处理后,结果见图1。将处理后的数据整理后导入SIMCA-P软件中进行主成分分析(PCA),用UV处理数据进行拟合,拟合后方程的R2X=0.855>0.5,说明拟合性好,且Q2=0.648,与R2X相差较小,说明拟合方程的稳定性好。图2为得到的PCA得分,由图2可知,QC的重复样本间较为聚集,说明实验方法和检测仪器较为稳定,准确性较好。此外,6组样品中除了B6,D3和E3外的所有点基本都处在95%的置信区间内,且每一组内的重复样品较为聚集,而各组间的样品又相对较为分散,组与组之间有明显的界限,说明样品组内的重复性好,数据较为准确;而样品组间的明显界限说明,各组间存在着明显的差异。还可以看出,在A组、C组、E组间,A组与C组、E组在第一主成分上被明显分开,其中A组被分在了第一主成分的左边,而C组和E组被分在了第一主成分的右边,说明A组与C组、E组间差异较大,C组和E组间差异较小。B组、D组、F组间也存在着一样的差异趋势,说明同一产地的不同色系藜麦间也存在着明显差异。用DModX和置信区间进行初步筛选,剔除B6,D3,E2这3个异常点[14],以保证后续数据分析的准确性。
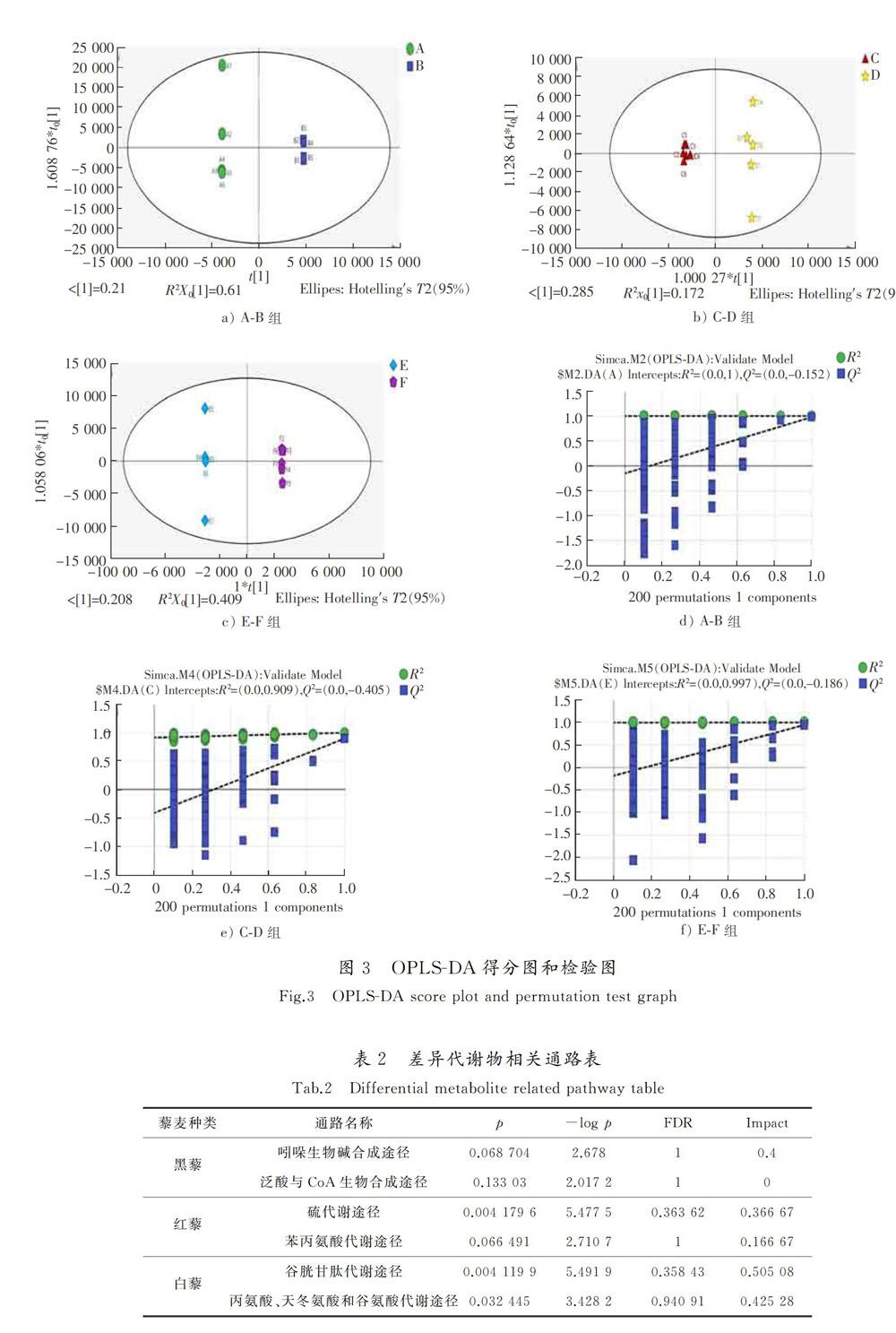
PCA只能反映样品的整体趋势,不利于发现组间差异和差异化合物,所以需要通过正交-偏最小二乘法判别(OPLS-DA)来更为准确地分析数据,寻找组间的差异[15]。首先根据种子颜色进行分组:A-B组、C-D组和E-F组,用Par算法进行数据转换,用OPLS-DA方程进行拟合,得到拟合方程的模型参数见表1。其中所有的R2X,R2Y和Q2都大于0.5且各参数间差异较小,说明拟合方程较为可靠。由方程拟合得到OPLS-DA得分图,见图3中的a),b)和c)。由图3可知,A-B组、C-D组和E-F组的分离程度较大,说明各组间有明显差异,且组内点相对较为聚集,表明样品间重复性好且较为稳定。再经过OPLS-DA置换检验来判断方程是否过拟合,得到检验图(见图3中的d),e)和f))。图3中所有的Q2点从左到右均低于最右边的原始Q2点,且Q2<0,R2和Q2的回歸线与横坐标交叉或者小于0,说明评估模型可靠有效[16]。
3.3筛选显著性差异代谢物
采用XCMS的Pairwise中的对比分析,上传A-B组、C-D组和E-F组数据进行分组分析处理,得到Metabolomic Cloud Plot图,设置参数p-value<0.01,fold change>1.5,进行显著性差异代谢物的筛选,得到代谢物的云图,见图4。图中M线上部为上调,M线下部为下调,横坐标为保留时间,纵坐标是m/z。图4显示,A-B组有140个差异代谢物,上调的有90个,下调的有50个;C-D组有570个差异代谢物,上调的有209个,下调的有361个;E-F组有486个代谢物,上调的有379个,下调的有107个。其中A-B组中上调物质占总物质的64.28%,下调物质占35.72%;E-F组中上调物质占总物质的77.98%,下调物质占总物质的22.02%,两组趋势和比例相差不大。C-D组中,上调物质占总物质的36.67%,下调物质占总物质的6333%,与A-B组和E-F组完全相反。其中C-D组间差异物质最多,差异最大,这一结果也与胡一晨等[7]的报道一致。
3.4差异代谢物通路分析
将上述筛选鉴定的差异代谢物上传至Metaboanalyst,利用MetPA数据库对其进行超几何检验和拓扑结构分析,根据代谢通路的浓缩,识别出可能的受生物扰动的代谢通路[17]。对差异代谢物和通路进行相关性分析,得到多条相关通路。根据-log p值和Impact值综合分析,每一组筛选出最为显著的2条代谢通路,如表2所示。
由表2可知,黑藜中主要是吲哚生物碱合成途径,合成的生物碱可以降血糖和血脂,有效治疗糖尿病,与房娜[18]的研究结果一致;泛酸与CoA生物合成途径与植物的碳水化合物、脂肪酸、蛋白质和能量代谢密切相关。红藜中主要是硫代谢途径,它与植物抗逆性密切相关,其途径中SO2-4经过植物一系列还原与同化反应后进入有机骨架,生成半胱氨酸(Cys),植物再以Cys为前体,合成众多具有重要生物学功能的代谢产物,直接关系到植物耐逆境胁迫和农作物的产量与品质,苯丙氨酸代谢途径与植物的防御系统密切相关。白藜中主要是谷胱甘肽代谢途径,它与植物直接和间接的抗逆功能活动密切相关[19];丙氨酸、天冬氨酸和谷氨酸代谢途径与糖代谢密切相关。
4讨论与结论
青海省海西蒙古族藏族自治州都兰县是典型的高原大陆性气候,日照时间长,太阳辐射强,昼夜温差大,常年干旱、多风、少雨,年平均气温4 ℃左右;而四川省凉山州盐源县的日温差较大,年均气温12.1 ℃,最高温度30.7 ℃,最低温度-11.3 ℃;虽然两地都较适宜藜麦的种植,但不同藜麦品种间的适应性不同。
本研究对青海都兰县和四川盐源县两地的黑、红、白藜麦种子做了代谢组学分析,发现两地种子之间都存在着明显差异,其中红藜麦种子与白藜麦种子差异较大,而黑藜麦种子差异最小。差异物质主要是脂肪酸(棕榈酸、肉桂酸、油酸)、糖类(乳糖、葡萄糖、脱氧糖苷)和氨基酸(脯氨酸、丝氨酸、组氨酸)等。通过比较发现,四川盐源县与青海都兰县的黑藜相比,差异代谢物缬氨酸表达下调,而缬氨酸是合成α-酮异戊酸的前体,α-酮异戊酸在酶作用下生成酮泛解酸,再与β-Ala合成泛酸。缬氨酸的下调导致泛酸合成受阻,使得植物体的碳水化合物、脂肪酸、蛋白质和能量代谢受阻。四川盐源县和青海都兰县的红藜相比,差异代谢物O-Acetylserine表现为显著下调,而这与半胱氨酸(Cys)密切相关。O-Acetylserine是Cys合成的前体,Cys合成受阻会导致硫代谢受阻。这些结果均表明,青海都兰县的黑藜与红藜均比四川盐源县有较强的耐受性,均能通过调节体内能量代谢通路的变化更好地去适应青海相对低温的生境。而四川盐源县和青海都兰县的白藜相比,差异代谢物谷胱甘肽(还原型)表达上调。因为谷胱甘肽有很强的抗氧化性,其上调增加直接影响到植物的抗逆性,使得四川盐源县的白藜能更快地通过代谢通路的变化去消除体内过氧化物以及自由基等,使藜麦进行正常的生长,因此四川盐源县的白藜比青海都兰县的白藜有更好的适应性,而这一研究结果也与青海都兰县白藜麦栽培的结果一致——温度的变化会导致白藜生长缓慢[21]。
本实验通过代谢组学分析四川盐源县与青海都兰县的黑、红、白藜,发现两地的同种藜麦间代谢物质有很大差异。在此基础上通过分析差异代谢物的代谢通路,发现青海都兰县的黑藜和红藜比四川盐源县具有更好的抗逆性,而青海都兰县的白藜较四川盐源县的白藜抗逆性弱。抗氧化性与抗逆性密切相关,这与赵亚东等[22]的研究结果一致。所以红藜和黑藜更适宜在青海都兰县种植,而白藜更适宜在四川盐源县种植。研究结果可为后续在代谢途径层面上深入研究藜麦的抗逆性机理提供理论依据。
本研究所选取的样本种类过少,在对不同生境下藜麦种子进行分析时只进行了代谢层面的分析,缺少对通路中关键蛋白质和关键酶的分析,对整个机理的解释存在着一定局限性。后续将结合多组学技术包括基因组学、转录组学和蛋白质组学等,对不同生境下黑、红、白藜麦的差异进行更加全面深入的分析,以期选育出优质、高产、抗逆性强的藜麦新品种,为藜麦的栽培种植提供理论依据。
参考文献/References:
[1]FIEHN O. Metabolomics the Link Between Genotypes and Phenotypes[M]. Berlin:Springer Netherlands, 2002:155-171.
[2]DUBOUZET J G, ISHIHARA A, MATSUDA F, et al. Integrated metabolomic and transcriptomic analyses of high-tryptophan rice expressing a mutant anthranilate synthase alpha subunit[J]. Journal of Experimental Botany, 2007, 58(12):3309-3321.
[3]FERREIRA D S, PALLONE J A L, POPPI R J. Direct analysis of the main chemical constituents in Chenopodium quinoa, grain using Fourier transform near-infrared spectroscopy[J]. Food Control, 2015, 48(48):91-95.
[4]VEGAGLVEZ A, MIRANDA M, VERGARA J, et al. Nutrition facts and functional potential of quinoa (Chenopodium quinoa Willd.), an ancient Andean grain: A review[J]. Journal of the Science of Food & Agriculture, 2010, 90(15):2541-2547.
[5]JAMES L E A. Chapter 1 quinoa (Chenopodium quinoa, Willd.):Composition, chemistry, nutritional, and functional properties[J]. Advances in Food & Nutrition Research, 2009, 58(9):1-10.
[6]肖正春, 张广伦. 藜麦及其资源开发利用[J]. 中国野生植物资源, 2014, 33(2):62-66.XIAO Zhengchun, ZHANG Guanglun. Development and utilization of Chenopodium quinoa Willd.[J].Chinese Wild Plant Resources, 2014, 33(2):62-66.
[7]胡一晨,赵钢,秦培友,等. 藜麦活性成分研究进展[J].作物学报,2018,44(11):1579-1591.
HU Yichen, ZHAO Gang, QIN Peiyou, et al. Research progress on bioactive components of quinoa (Chenopodium quinoa Willd.)[J].Acta Agronomica Sinica,2018,44(11):1579-1591.
[8]趙亚东. 青海藜麦资源营养品质评价及功能成分与抗氧化活性研究[D].西宁:青海大学,2018.
ZHAO Yadong. Nutritional Quality Evaluation and Study of the Functional Compositions and Antioxidant Activities in Quinoa from Qinghai[D]. Xining:Qinghai University, 2018.
[9]LISEC J, SCHAUER N, KOPKA J, et al. Gas chromatography mass spectrometry-based metabolite profiling in plants[J]. Nature Protocols, 2006, 1(1):387-396.
[10]哈婧, 王娇娇,赵帆帆,等. 石韦配方颗粒HPLC-DAD特征图谱研究[J]. 河北科技大学学报, 2017, 38(1):39-45.
HA Jing,WANG Jiaojiao,ZHAO Fanfan,et al.Study of HPLC-DAD characteristic chromatogram of Pyrrosia leaf formula granules[J]. Journal of Hebei University of Science and Technology, 2017, 38(1):39-45.
[11]COLIN A S, ELIZABETH J W, GRACE O, et al. XCMS: Processing mass spectrometry data for metabolite profiling using nonlinear peak alignment, matching, and identification[J]. Analytical Chemistry, 2006, 78(3):779-787.
[12]FORSBERG E M, TAO H, RINEHART D, et al. Data processing, multiomic pathway mapping, and metabolite activity analysis using XCMS online[J]. Nature Protocols, 2018, 13(4):633-651.
[13]POGACIC T, MAILLARD M B, LECLERC A, et al. A methodological approach to screen diverse cheese-related bacteria for their ability to produce aroma compounds[J]. Food Microbiology, 2015, 46:145-153.
[14]阿基业. 代谢组学数据处理方法——主成分分析[J]. 中国临床药理学与治疗学, 2010, 15(5):481-489.A Jiye. Analysis of metabolomic data:Principal component analysis[J]. Chinese Journal of Clinical Pharmacology and Therapeutics, 2010, 15(5):481-489.
[15]JOHAN A W, EWOUD J J, HUUB C J H, et al. Multivariate paired data analysis: Multilevel PLSDA versus OPLSDA[J]. Metabolomics, 2010, 6(1):119-128.
[16]BOULESTEIX A L, STRIMMER K. Partial least squares: A versatile tool for the analysis of high-dimensional genomic data[J]. Briefings in Bioinformatics, 2007, 8(1):32-37.
[17]OGATA H, GOTO S, SATO K, et al. KEGG: Kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Research, 2000, 27(1):29-34.
[18]房娜. 觀察藜麦饮食对2型糖尿病患者餐后血糖影响[J]. 中国药物与临床, 2017, 17(8):1180-1182.
[19]张洁琳, 付畅. 半胱氨酸蛋白酶及半胱氨酸蛋白酶抑制剂对逆境胁迫的应答[J]. 分子植物育种, 2018, 16(13):4453-4459.ZHANG Jielin, FU Chang. Response of cysteine protease and cysteine protease inhibitor to adverse stress[J].Molecular Plant Breeding, 2018, 16(13):4453-4459.
[20]孙国祥,张旭,张成国,等.谷胱甘肽在动植物领域的应用研究进展[J].安徽农业科学,2018,46(8):42-45.SUN Guoxiang, ZHANG Xu, ZHANG Chengguo,et al. Application of glutathione in animal and plant fields[J]. Journal of Anhui Agricultural Sciences, 2018,46(8): 42-45.
[21]刘洋, 闫殿海, 张玉清, 等. 青海藜麦的发展现状与发展中存在的问题[J]. 青海农林科技, 2015(4):31-34.LIU Yang, YAN Dianhai, ZHANG Yuqing, et al. The current situation and problems in development of Chenopodium quinoa in Qinghai[J].Science and Technology of Qinghai Agriculture and Forestry, 2015(4):31-34.
[22]赵亚东, 党斌, 杨希娟, 等. 青海藜麦皂苷超声提取工艺及抗氧化活性[J]. 食品工业科技, 2017,38(19):45-52.
