EGFR酪氨酸激酶区生物信息学分析
2019-06-11王贞超孟娇
王贞超 孟娇
表皮生长因子受体(Epidermal Growth Receptor, EGFR)是一种重要的跨膜受体。本文拟采用生物信息学分析方法,对其胞内区的酪氨酸激酶氨基酸序列性质进行分析,为探究EGFR与肿瘤之间的关系提供参考。
EGFR是原癌基因HER-1的表达产物,其基因位于第7号染色体上,全长200 kb,由28个外显子组成,具有酪氨酸激酶(Tyrosine Kinase, TK)活性。EGFR分为3个区域:胞内区、跨膜区和胞外区,其中胞内区包括近膜区、TK区和C末端。EGFR-TK对肿瘤细胞的形成与生长起着重要的作用,酪氨酸残基的磷酸化激活各种信号分子和信号通路。本研究将对EGFR-TK区展开研究,利用生物信息学方法研究其理化性质,为探究EGFR基因及其编码蛋白与肿瘤之间的关系提供理论基础。
一、方法
在NCBI数据库下载EGFR-TK区序列信息;采用Gendoc软件进行多重序列比对分析;采用Prot Param tool在线分析软件,探究氨基酸的理化性质;应用WoLF PSORT预测亚细胞定位;应用ProtScale分析氨基酸序列亲疏水性;采用SignalP4.1在线服务器分析EGFR的信号肽;采用TMHMM Server和SMART5.0分析软件,对EGFR跨膜结构进行分析。
二、结果与结论
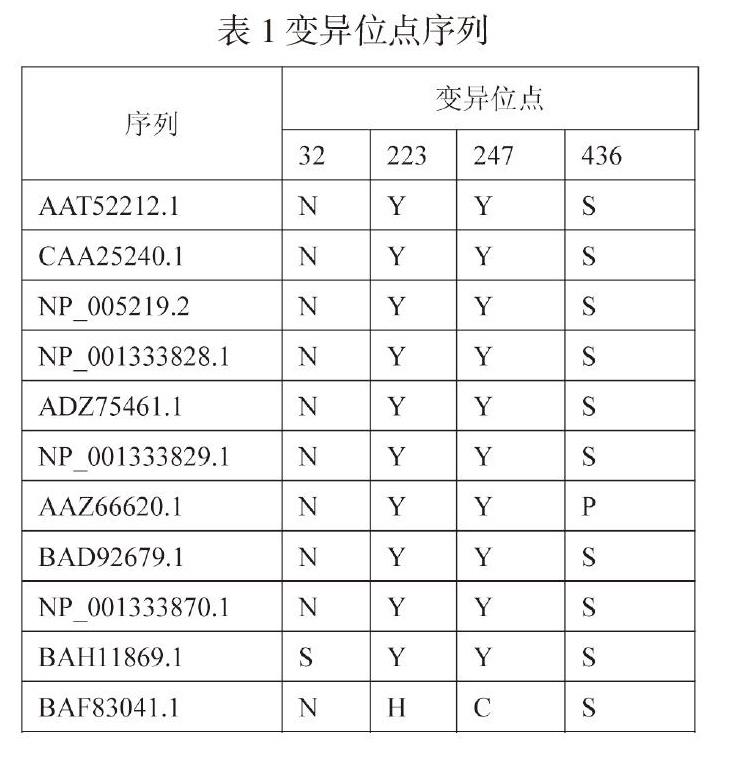
(一)多重序列比对分析
经过分析發现这些序列的保守性较强,BAH11869.1在32 aa处N突变成S,突变率8.3%;BAF83041.1有2处发生了突变,分别是223 aa处Y突变成H,247 aa处Y突变C,突变率都为8.3%;AAZ66620.1在436 aa处S突变为P,突变率8.3%。
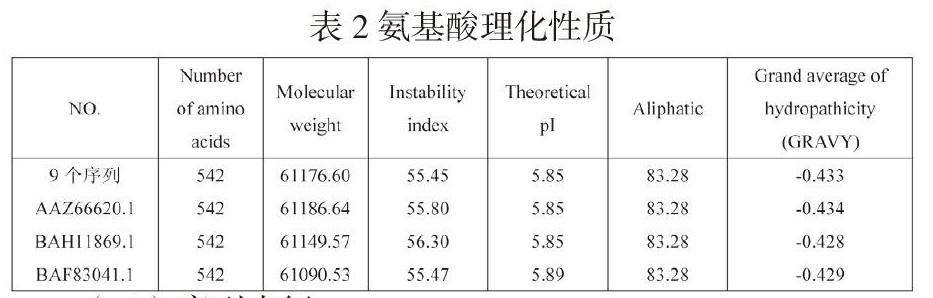
(二)理化性质分析
经过分析发现,12个氨基酸序列理化性质相似。分子量为61.09 kDa~61.17 kDa,理论等电点为5.85~5.89。脂肪系数为83.28,总平均亲水性为-0.434~-0.428。不稳定指数为55.45~56.30。说明这些氨基酸是不稳定弱疏水氨基酸。9个序列和AAZ66620.1亚细胞定位推测其位于cyto_nucl,BAH11869.1、BAF83041.1推测其位于nucl。
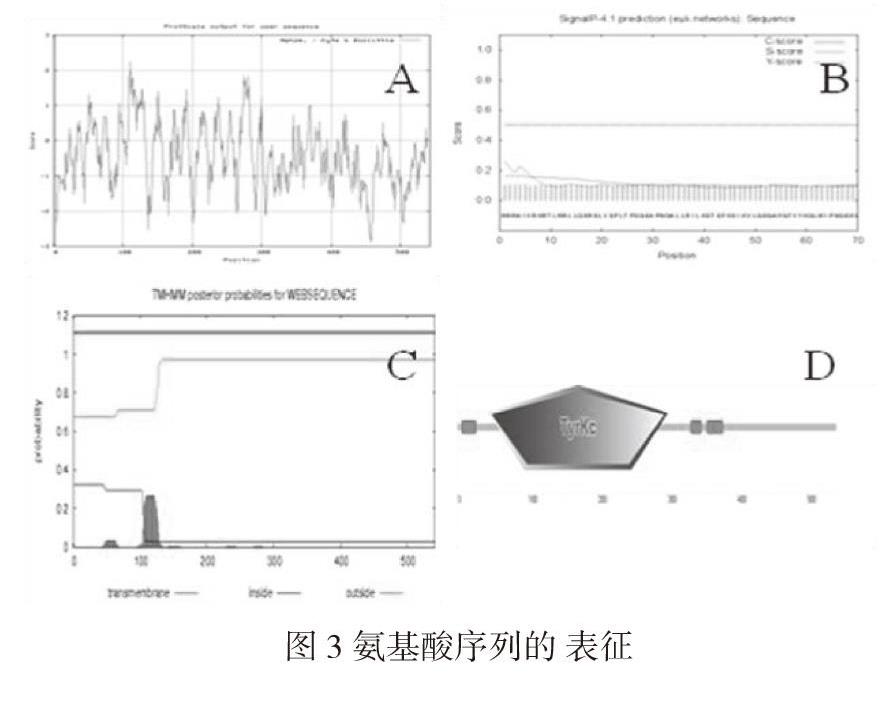
(三)序列表征
经过分析发现,12个氨基酸序列具有相同的性质:ProtScale程序分析蛋白有一个较强的亲水区域,位于445~455 aa,同时有一个较强的疏水区域,位于105~110 aa(图3-A);SignalP 4.1研究发现蛋白无信号肽(图3-B);TMHMM Server对其跨膜结构进行分析发现,有跨膜结构域,分别位于50~70 aa和110~140 aa (图3-C);SMART 5.0对其结构域分析表明,其有三个低度复杂区,在7~24 aa,334~347 aa,357~378 aa处,酪氨酸激酶活性区域在44~300 aa。(图3-D)。
3-A亲水性和疏水性;3-B信号肽;3-C跨膜结构;3-D结构域
三、展望
EGFR在肿瘤细胞中过表达导致肿瘤产生,成为国内外研究焦点。本文利用生物信息学方法对EGFR-TK区的性质进行分析,从而为选择特定部位作为靶点,研发更安全、更高效的EGFR-TKI提供新的研究思路。(作者单位:贵州大学 药学院)
