生物等效性检验在SAS及stata中的实现及对比
2019-05-24四川大学华西公共卫生学院四川大学华西第四医院610041郑倩雯李亚文李宓儿朱彩蓉
四川大学华西公共卫生学院四川大学华西第四医院(610041) 张 露 郑倩雯 李亚文 李宓儿 王 菊 朱彩蓉
【提 要】 目的 探索在SAS和stata中如何实现生物等效性检验,并且比较两个软件实现方法的异同点和结果的一致性。方法 利用现有加拿大指南中的2×2交叉设计生物等效性试验的数据集,结合生物等效性检验的原理,给出SAS实现的宏程序以及stata程序,并且比较二者结果之间的异同。结果 通过SAS宏语言能够实现生物等效性检验结果的快速输出。由于stata中“pkequiv”默认的计算原理在最后需要取反对数的过程与SAS有所不同,导致最后结果出现差异,通过添加对最终置信区间取反对数的命令后与SAS结果一致。结论 本研究可作为利用常用软件进行生物等效性检验时参考,同时提示应注意根据自身数据集分布正确使用“pkequiv”命令。
2016年以来,中国高度重视仿制药和原研药的一致性评价工作[1],出台了很多仿制药与原研药一致性评价的统计指导原则类文件。然而生物等效性评价作为仿制药与原研药一致性评价中不可缺少的一环,目前尚未出台如何利用软件实现生物等效性检验的具体操作指南。在目前能够实现生物等效性检验的众多软件中,国外更推荐在SAS中进行生物等效性检验[2]。相对于SAS程序而言,stata在进行生物等效性检验时,有专门的命令“pkequiv”,仅需要一个命令即可进行检验,十分简便。故本文将对比stata和SAS进行生物等效性检验的异同,为相关工作者利用常用软件进行生物等效性检验提供参考。
生物等效性检验原理
生物等效性试验最常用也是各国指导性文件中最推荐的试验设计是2×2交叉设计[3],下面将简单介绍在该试验设计下的生物等效性检验的原理。
生物等效性检验最常用的方法之一是置信区间法[4]。需要进行生物等效性检验的药代动力学参数包括峰浓度(cmax)、血药浓度0到t时间下面积(area under the curve from zero to t time point,AUCT)及血药浓度0到无穷大时间下面积(area under the curve from zero to infinity,AUCI),这三个参数通常情况下呈正偏态分布,在经过对数转换后呈正态分布。故首先需要对这三个参数进行对数转换。进行对数转换后的参数通过方差分解,获得残差均方MSresidual。
利用上述的残差均方计算估计的标准误,并计算试验制剂与参比制剂差值的90%置信区间[5]。
(1)
(2)
根据残差均方MSresidual计算个体内变异性[6]。
(3)
对置信区间取反对数后,获得试验制剂与参比制剂几何均值比的90%置信区间。在个体内变异性小于30%的情况下,如果这三个参数几何均值比的90%置信区间在(80%,125%)范围内,则说明具有生物等效性[7]。
软件实现
现以加拿大生物等效性试验指南中的原始数据为例[6],来展示生物等效应检验在SAS与stata两个软件中的实现。
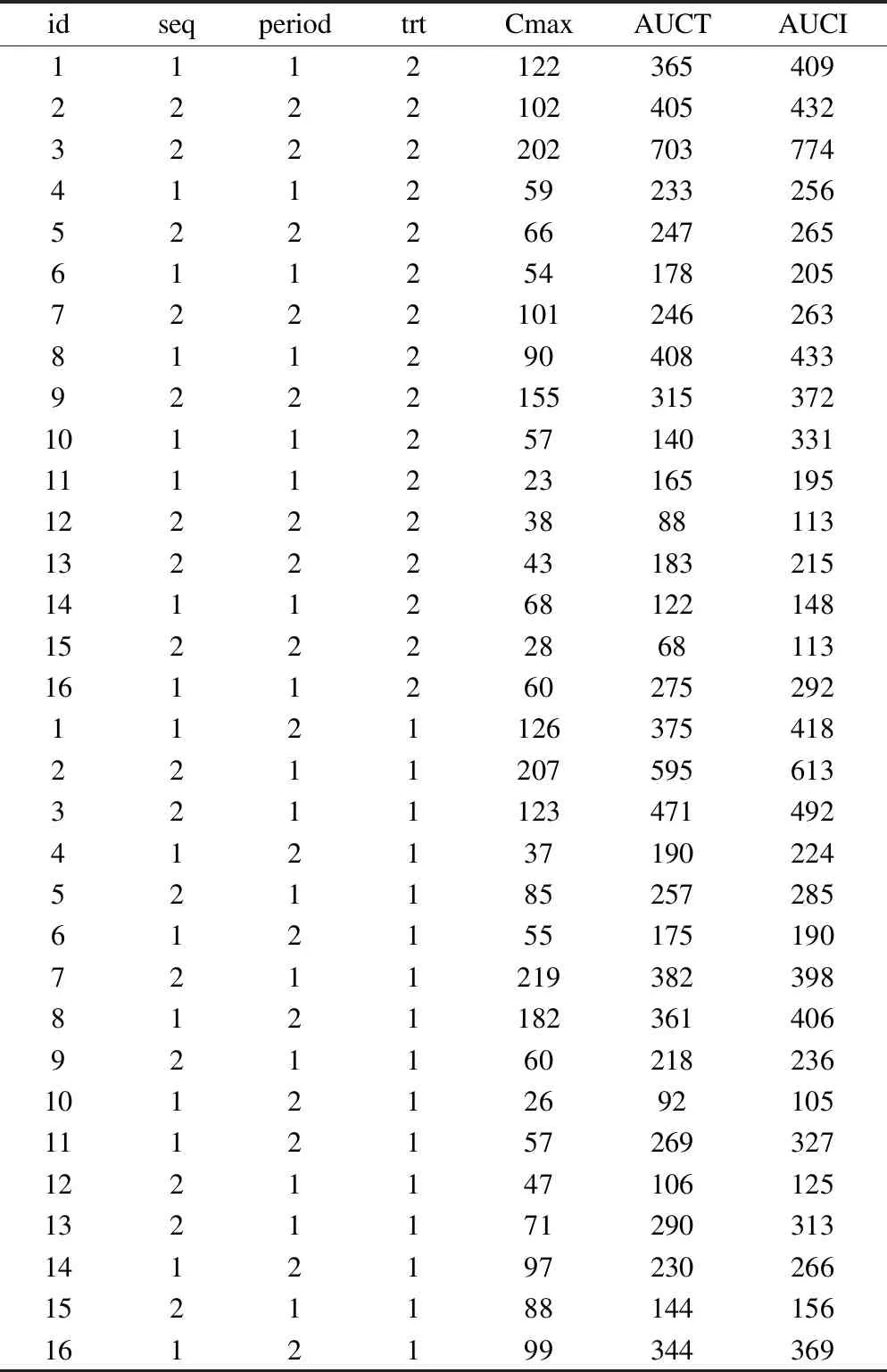
表1 原始数据格式
seq:给药顺序,1代表TR,2代表RT;period:1代表第一阶段,2代表第二阶段;trt:药物,1表示R,2表示T。
1.SAS程序 由于在进行生物等效性检验时,三个药代动力学参数Cmax、AUCT及AUCI在SAS中计算原理完全一致,故我们利用SAS宏和DO循环输出其生物等效性结果。其中产生残差的关键步骤是“proc mixed”过程。以下为SAS程序:
%macro be(data=);
/*分别建立三个参数的数据集并对参数进行对数转换*/
data a1;set&data(keep=id trt seq period cmax rename=(cmax=var));logvar=log(var);run;
data a2;set&data(keep=id trt seq period auct rename=(auct=var));logvar=log(var);run;
data a3;set&data(keep=id trt seq period auci rename=(auci=var));logvar=log(var);run;
/*对数据进行混合线性模型分析,获得方差分析差值估计、残差*/
%do i = 1 %to 3;
ods output estimates=b&i;odsoutput covparms=c&i;
proc mixed data=a&i;
class trt period seq id;
model logvar=trt period seq/cl alpha=0.1;
random id(seq);
estimate ′T-R′ trt-1 1/cl alpha=0.1;
run;
/*对试验制剂与参比制剂差值及其90%置信区间取反对数并调整几何均值比和置信区间的格式*/
data b&I;setb&I;
pe=exp(estimate)*100;lowerCI=exp(lower)*100;upperCI=exp(upper)*100;
CI=cat(′(′,strip(put(lowerCI,8.2)),′%′,′,′,strip(put(upperCI,8.2)),′%′,′)′);
GMR=cat(strip(put(pe,8.2)),′%′);
keep CI GMR;run;
/*计算个体内变异*/
data c&i;set c&i(firstobs=2);
_cv=100*sqrt(exp(estimate)-1);
cv=cat(strip(put(_cv,8.2)),′%′);
keep cv;run;
/*对其中一个参数的各个生物等效性检验结果进行横向合并*/
data d&i;merge b&i c&i;run;
%end;
/*将三个药代动力学参数的结果进行纵向合并,增加新变量z指示参数名称*/
data d;setd1 d2 d3;length z $20.;
if _n_=1 then z="Cmax";if _n_=2 then z="AUCT";if _n_=3 then z="AUCI";
run;
%mend;
/*调用宏*/
%be(data=be);
2.stata程序 在stata中,进行生物等效性检验有“pkcross”和“pkequiv” 两个关键命令,这两个命令包含的算法是在stata中预先设定好的。其中“pkcross”将产生方差分析表,获得残差均方,后者用来计算个体变异。而“pkequiv”过程则计算比值的90%置信区间。
generate outcome1=log(cmax)
pkcross outcome1,id(id)period(period)sequence(seq)treatment(trt)carryover(none)
pkequiv outcome1 trt period seq id
generate outcome2=log(auct)
pkcross outcome2,id(id)period(period)sequence(seq)treatment(trt)carryover(none)
pkequiv outcome2 trt period seq id
generate outcome3=log(auci)
pkcross outcome3,id(id)period(period)sequence(seq)treatment(trt)carryover(none)
pkequiv outcome3 trt period seq id
结果与对比
SAS生物等效性检验与stata的结果对比见表2。

表2 SAS与stata的生物等效性检验结果
根据结果对比,stata给出了试验制剂和参比制剂之间的比值及其置信区间,与SAS给出的几何均值比和置信区间之间差异巨大,但是其个体内变异却保持一致。
表3仅以Cmax为例,对比SAS、stata以及指南中给出的结果,SAS的结果与指南基本一致,出现细微差别的原因是指南在进行差值90%置信区间计算时采用的是保留四位小数的估计值。stata的结果中差值及其90%置信区间与SAS结果保持一致,而其比值及其90%置信区间差别明显。

表3 SAS与stata对Cmax进行生物等效性的检验结果对比
讨论
通过stata的帮助文献,“pkequiv”作为一个高度合成的命令,其算法在软件中是默认的,stata在方差分析、计算差值及其90%置信区间时,其原理与SAS一致,但是SAS在计算几何均值比及其90%置信区间时是将差值的结果取反对数回去后获得,而在stata中并没有计算其几何均值比,而是通过如下的方法计算试验制剂和参比制剂之间的比值:
(4)
(5)
(6)

return list
di exp(r(uci))
di exp(r(lci))
通过上述的讨论,我们可以看出,默认的stata中的生物等效性检验过程实际上是适用于原始药代动力学参数呈正态分布的数据。尽管stata的原理与SAS在最后一步略有不同而导致结果不一致,但是通过命令的补充,同样可对药代动力学参数经对数转化后呈正态分布的数据进行生物等效性检验,且命令更简单。
在本研究中,SAS利用宏可以多次调用处理同类数据,所有操作都不需要人为的复制粘贴数据;SAS在进行临床试验的统计分析时更具有权威性。stata命令相对来说更简洁,但stata的内置“pkequiv”很容易导致初次使用者由于不了解使用条件而产生误用。因此本研究更推荐SAS作为生物等效性检验的软件工具。
