基于改进的L-BFGS稀疏降噪自编码网络的MRI脑图像识别方法
2019-05-14王新颖王婉秋
王新颖,王婉秋,王 慧
基于改进的L-BFGS稀疏降噪自编码网络的MRI脑图像识别方法
王新颖,王婉秋,王 慧
(长春工业大学计算机科学与工程学院,吉林 长春 130012)
随着人类科技的飞速发展以及医学影像设备的不断更新,医学影像技术在脑部病变的辅助诊断中起到了越来越重要的作用,为此,提出一种基于改进的L-BFGS稀疏降噪自编码网络模型(ILSDAE),并将其应用于MRI脑图像的阿尔茨海默病的识别与脑部疾病的辅助诊断。实验数据源取自ADNI数据集,经过校正、配准、分割、平滑等操作,获得脑部灰质图像,随后将改进的无监督贪婪预训练方法和L-BFGS算法相结合,对深度自编码网络进行训练并通过Softmax回归训练学习特征,从而实现对病症患者脑部图像的识别。ILSDAE网络模型具有很好的鲁棒性,与堆栈式自编码和自学习方法相比,实验结果证明了所提方法的有效性。
阿尔茨海默氏症;L-BFGS;稀疏降噪自编码;MRI脑图像
1 研究背景
老年痴呆症又名阿尔茨海默病(Alzheimer’s disease,AD),是老年人中最为常见的脑神经性疾病,并且成为了美国第六大死亡原因[1]。据统计,2010年年龄大于60岁的老人当中平均有4.7%的人被诊断为具有AD[2]。这种原发性退行性脑部病变疾病起病隐匿且不可逆,由发病到死亡的平均病程大约需要8~10年。医学界认为脑神经细胞的损伤和死亡具有不可逆性,很难找到有效治愈该病的方法,也是多数研究人员对AD的早期诊断和预测关注的原因。
AD由于灰质部分的萎缩加快了神经系统的机能衰退,是一个神经退化的不可逆过程,目前对该疾病的早期诊断主要包括心理学量表测试[3]、生物标志物检测[4]、神经影像学检测和基因检测[5]等。其中核磁共振成像(magnetic resonance imaging,MRI)是较为广泛的神经影像诊断方法,其能够以多方位、多序列的方式对脑组织进行成像,分辨率高,能清楚地区分灰质和白质,并且对人体没有放射性危害,在所有的影像学检测中,具有明显优势。
随着人们对该疾病的不断认识,使得近年来基于磁共振(magnetic resonance,MR)成像的AD辅助诊断算法受到了广大研究者的关注。陈斯鹏等[6]提出了一种基于MR图像纹理特征的AD的分类模型,从MR图像中提取脑部海马区域纹理特征参数,进而建立AD的早期分类预测模型。KAMATHE和JOSHI[7]提出了一种基于独立成分分析(independent component analysis,ICA)的脑脊液MR图像分类方法。ALTAF等[8]提出了一种AD检测与分类算法,该方法使用一种基于视觉词的方法提高灰度共生矩阵(gray-level co-occurrence matrix,GLCM)、尺度不变特征变换(scale invariant feature transformation,SIFT)、局部二值模式和梯度直方图等纹理特征的有效性,并将图像数据与提供的临床数据相结合从而辅助对疾病的诊断。
本文针对ADNI (Alzheimer’s disease neuroimaging initiative database)脑图像数据,提出了一种基于改进的LBFGS (limited-memory Broyden fletcher Goldfarb Shanno,L-BFGS)稀疏降噪自编码网络模型(improved L-BFGS sparse denoising autoencoder, ILSDAE),并将其应用于MRI脑图像的AD识别。通过对MRI脑图像灰质特征的提取及深层次的训练,论证了本文的方法具有很好的鲁棒性,提高了AD早期诊断分类的准确率。
2 相关介绍
2.1 自编码
自编码神经网络属于无监督学习算法,主要用于数据降维或特征抽取,其使用反向传播算法,最小化重构误差项,自编码神经网络代价函数

其中,为样本的训练集;()=s(W+)为编码函数;()=s(ʹ+)为解码函数,s和s为激活函数[9]。
2.2 稀疏降噪自编码
稀疏降噪自编码神经网络是在自编码神经网络的基础上,给待训练的数据加入噪声,让自动编码器去学习如何去除噪声以获得没被污染的数据,这样做就使得编码器的泛化能力增强且能够获得对输入数据更加鲁棒性的表达。再基于稀疏性的思想,对抽象出的特征稀疏表示,抑制了大部分的输出神经元,使用较少的激活单元表示特征,有效的用于对数据的分类[10]。
稀疏降噪自编码神经网络(sparse denoising auto-encoder,SDAE)的总体代价函数为

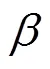
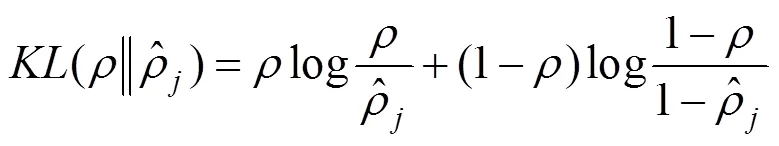
3 图像预处理
3.1 样本数据
实验采用标准数据集ADNI[11]提供的脑图像数据。ADNI数据库于2003年启动,由美国加利福尼亚大学放射学和生物医学成像中心创建,主要目标是为了更早的识别AD患病的高危人群并尽快的找到治疗该病的有效措施。有关ADNI研究和患者纳入/排除标准的详细信息,请参阅ADNI通用程序手册[11]。本研究使用的数据下载于2018年2月至2018年4月。
本文选取55名AD患者和63名正常对照组(NL)的MRI脑图像数据进行实验。表1列出了本文选择的测试数据的基本资料信息。

表1 被试者资料信息(均值±标准差)
本次实验使用的MR图像是由Wyman提出的标准化数据集中的“ADNI1 standardized 1.5T:screening 1.5T”,实验中所用图像为T1加权图像,所有数据都经过一系列预处理,包括Gradwarp (由于梯度非线性导致的图像几何失真的系统特定校正),B1非均匀性强度校正,N3 (减少由于波或介电效应引起的强度不均匀性)和尺度缩放。关于图像的协议以及这部分预处理流程的详细介绍可以参看ADNI网站(http://adni.loni.usc.edu/data-samples/ mri/)。使用Signa1.5T MRI系统进行三维快速梯度回波翻转恢复(FSPGRIR)脉冲序列(T1W1)进行扫描,行结构像横段位扫描,扫描范围从颅顶至枕骨大孔,翻转角FA=8,射频重复时间TR=1 000 ms,TI=3 000 ms,回波时间TE=3.6 ms,层厚1.2 mm,无间隔连续扫描,矩阵=192×192,层数160。
3.2 MRI图像及预处理
本文采用SPM12[12]与MRIcron[13]共同处理MR图像数据。首先将从ADNI标准数据库中获得的数据用SPM12进行预处理,其步骤如下:
步骤1.时间层校正(Slice Timing)。其用来校正1个扫描帧中层与层之间采集时间的差异;Number of Slices:输入每祯图像的层数(如本文使用的图像Dimensions:192×192×160, 160即为层数);TR:为2 s;TA:每祯图像获取第一层开始到获取最后一层图像的时间间隔,公式为TR-TR/nslice,本实验中为”2-2/160”;Slice order:输入[1:2:159, 2:2:160](图像是隔层扫描所得到的),采用的是交叉扫描模式(Interleaved),在交叉EPI扫描中,先采集所有奇数图层,再采集所有偶数图层;Reference Slice:输入”159”,选择参考层,其他图层校正时以此为对照,通常选择nslice/2,这样能使数据的时间校正最小化。
步骤2. 头动校正(Realignment)。其主要为了处理被试在做检测时的一些微小的晃动,尽管是微小的晃动和一些细小的情况,也会对实验造成影响。实验中使用特定的一些算法将所有的MR图像数据均与选中的第一个的MR图像对齐。
步骤3. 配准(Coregister)。其功能采用刚体变换以匹配不同模态扫描所得的图像。
Reference Image:选择新生成的mean*.nii文件;Source Image:基于Reference Image进行处理的图像;Other images:执行与Source Image相同处理的图像。
步骤4. 分割(Segment)。其功能用于分辨解剖图像中的组织类型,可分割为5部分:灰质、白质、脑脊液、头骨和软组织。
图1是正常对照组022_S_0014分割后所对应的5部分。
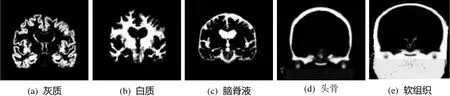
(a) 灰质(b) 白质(c) 脑脊液(d) 头骨(e) 软组织
步骤5. 标准化(Normalise)。将不同容积和形状被试者的大脑放置在标准空间中,并使用共同的坐标系描述特定位置。空间标准化通过生成”deformation field” (变形项)来实现。变形项是在3D空间中对每个位置的位移程度进行定量描述的图像。
步骤6.平滑(Smooth)。其功能用于对图像进行模糊处理来减少噪声和失真。以对不同受试者的解剖/功能图像的轻微残余差异进行校正。MR图像由于像素点数较大的原因经过平滑处理后能够得到较多的特征数据。
图2是经过处理后,灰质正常图像与异常图像对比。
导出本文用SPM12预处理过后的MR脑图像,再用MRIcron对图像进行切片处理,从而获得更大的输入数据集和更多的特征信息。为了更准确的对图像进行识别,切片时尽量保留特征较为明显的部分。图3是AD病人和正常对照组的切片后的图像示例。
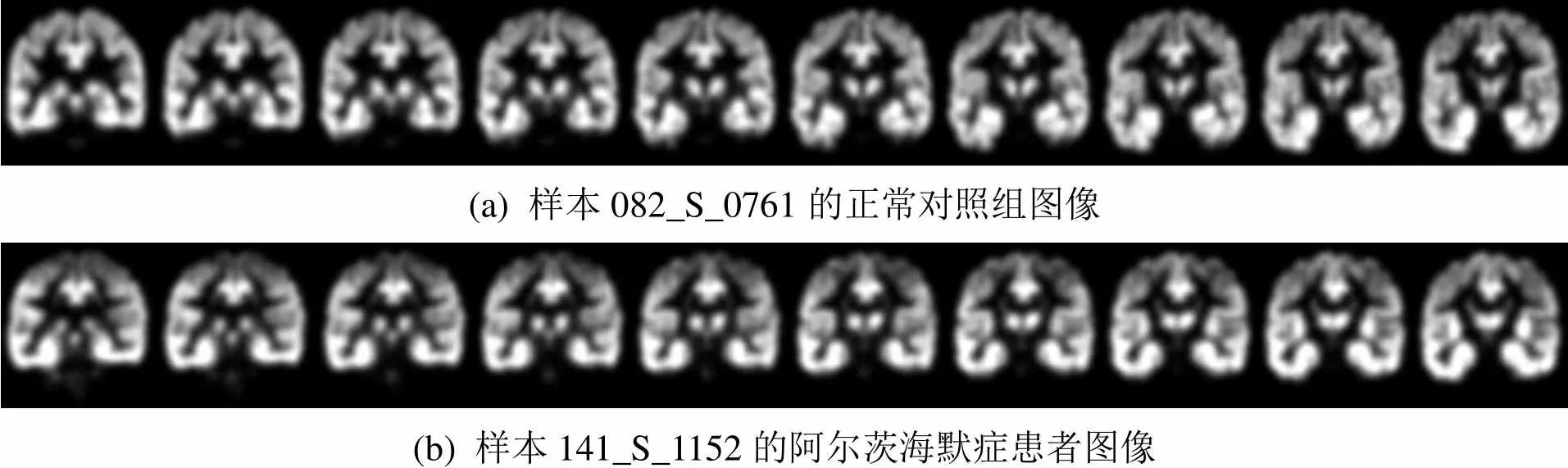
(a) 样本082_S_0761的正常对照组图像 (b) 样本141_S_1152的阿尔茨海默症患者图像
4 改进的L-BFGS稀疏降噪自编码方法
本文提出的ILSDAE方法包含2部分:稀疏降噪自编码器和softmax分类器。为了防止数据量少引起处理时的过度拟合问题,本文采用改进的L-BFGS算法计算J和隐含层权重,即

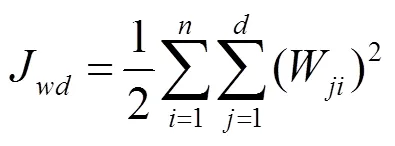
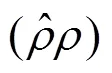
将参数更新为
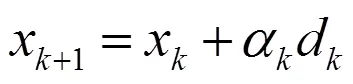
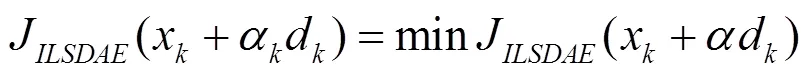
4.1 算法描述
步骤1. 选择脑图像的初始节点0,误差>0,设置最近几次的迭代次数(实验取6)。
步骤2.计算
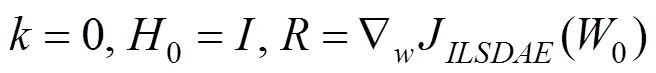
步骤4.此次迭代的可行性方向为
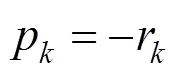
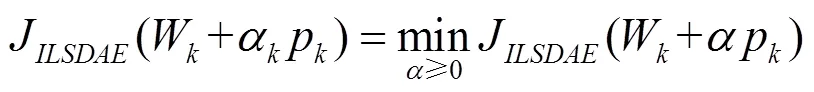
步骤6.对权重进行更新
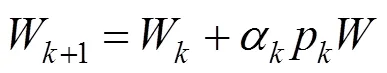
步骤7.若>,保存近几次的次的矢量值,删除(s-,y-)。
步骤8.计算
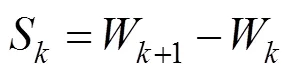
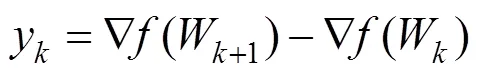
步骤9.递归得到
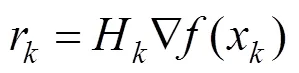
若=+1,执行步骤3。
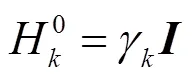
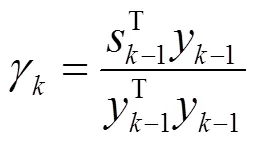
为减小代价函数J的值,不断重复以上算法的迭代步骤,训练神经网络模型。模型中参数的更新可以作为softmax分类器的输入,最终达到分类的目的。
4.2 网络结构
模型的网络结构如图4所示,根据MRI脑图像的灰质特征,首先获取网络结构中的隐藏层参数1,同时得到隐藏层的特征表示。并将作为softmax分类器的输入,然后利用有污染的或插入噪声的原始信息来获得softmax分类器的参数0。将权值参数0和1作为网络模型的初始化参数,通过网络模型,最终得到整个模型的代价函数J微调过程。采用受限的拟牛顿法神经网络调整函数J,输出层和隐含层权重参数。
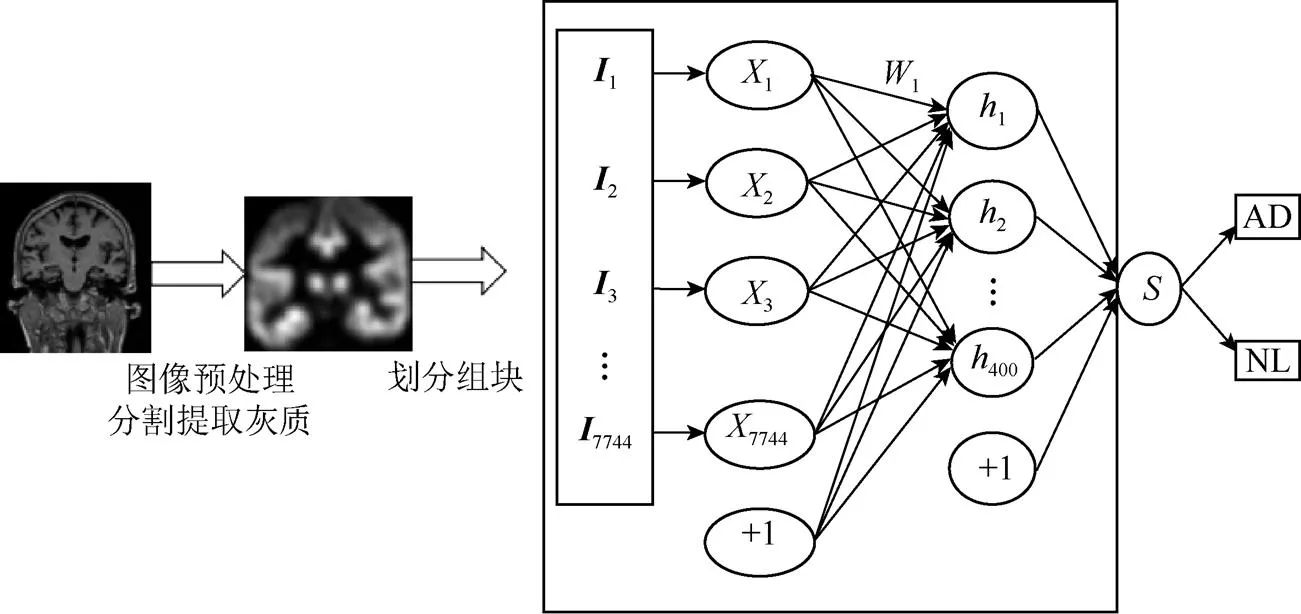
图4 基于ILSDAE的网络结构
5 实验与结果
5.1 脑图像特征可视化表示
实验中选取了118个ADNI脑图像的数据,其数据分为2组:AD患者组和正常对照组。将实验数据分为测试集(59人)和训练集(59人)。对于ADNI数据,先经过SPM预处理,获取到经过平滑后的特征明显的灰质图像,再将得到的MR灰质图像导入MRIcron软件中进行切片处理可获得大量输入数据,得到了灰质图像为88×88像素图片,可视化特征如图5所示。
图5 特征的可视化表示
5.2 识别性能比较
将实验数据分别放入堆栈式自编码神经网络(stacked autoencoder,SAE)[14]、自学习神经网络(self-taught learning,SL)[15]和本文提出的ILSDAE方法中进行训练,最后得出分类结果。表2列出了几种方法的识别效果,其中每类实验6次,取平均值。

表2 被图像识别效果比较(%)
由表2可见,本文方法优于传统的SAE和SL。
下面对于多种灰质图像特征提取方法,与本文所提直接使用数据源进行实验。
图6显示了当前灰质脑图像,经过基本像素点组成的特征,与LBP特征[16],HOG[17]特征分别经过SAE、 SL和ILSDAE对数据的分类识别准确率比较的结果。由图6可见,本文模型在LBP特征及HOG特征下也优于其他模型。
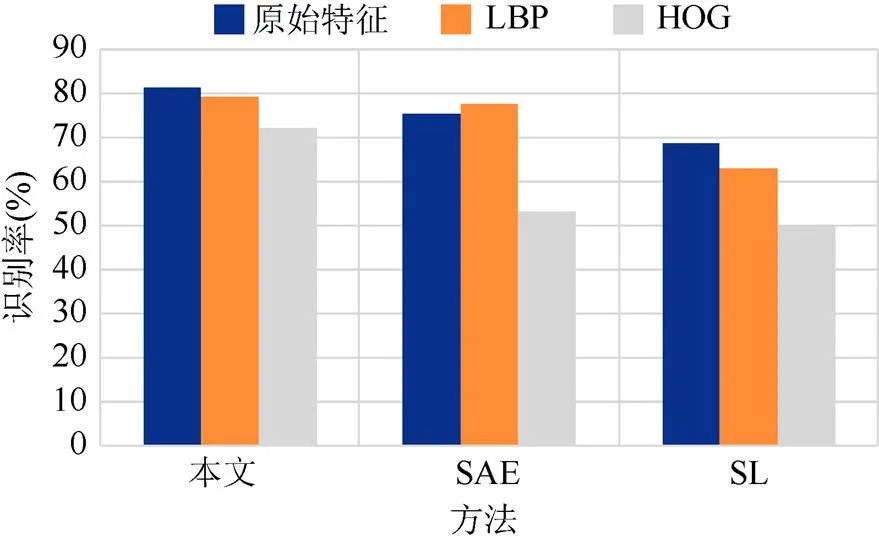
图6 不同图像特征提取方法识别率比较
6 结束语
本文提出了一种基于改进的ILSDAE的稀疏降噪自编码方法,构造了一个深度网络模型。首先,利用无监督贪婪预训练方法和ILBFGS方法对神经网络进行训练;然后,使用Softmax训练所学习的有关脑图像灰质的特征;最后,将构建好的分类器应用于脑图像的识别。实验结果表明,与SAE和SL相比,该方法具有更好的识别性能。如何缩短特征学习的时间,调整算法的相关参数,实现并行处理是本文的下一步工作。
[1] PRINCE M, WIMO A, GUERCHET M, et al. World Alzheimer Report 2015: The global impact of dementia [R]. London: ABI, 2015: 6-9.
[2] ASSOCIATION A. 2013 Alzheimer’s disease facts and figures [J]. Alzheimers and Dementia. 2013, 9(2): 208-245.
[3] PETRELLA J R, COLEMAN R E, DORAISWAMY P M. Neuroimaging and early diagnosis of Alzheimer disease: A look to the future. [J]. Radiology, 2003, 226(2): 315-336.
[4] FJELL A M, WALHOVD K B, FENNEMANOT ESTINE C, et al. CSF biomarkers in prediction of cerebral and clinical change in mild cognitive impairment and Alzheimer’s disease [J]. Journal of Neuroscience, 2010, 30(6): 2088-2101.
[5] BUIZZA L, CENINI G, LANNI C, et al. conformational altered p53 as an early marker of oxidative stress in Alzheimer’s disease [EB/OL].[2018-10-30].http://med. wanfangdata.com.cn/Paper/Detail/PeriodicalPaper_PM22242180.
[6] 陈斯鹏, 高妮, 田思佳, 等. 基于MR图像纹理特征的阿尔茨海默病分类模型[J]. 北京生物医学工程, 2017, 36(2): 134-138.
[7] KAMATHE R S, JOSHI K R. A novel method based on independent component analysis for brain MR image tissue classification into CSF, WM and GM for atrophy detection in Alzheimer’s disease [J]. Biomedical Signal Processing and Control, 2018, 40: 41-48.
[8] ALTAF T, ANWAR S M, GUL N, et al. Multi-class Alzheimer’s disease classification using image and clinical features [J]. Biomedical Signal Processing and Control, 2018, 43: 64-74.
[9] LIOU C Y, CHENG W C, LIOU J W, et al. Autoencoder for words [J]. Neurocomputing, 2014, 139: 84-96.
[10] NG A. CS294A lecture notes sparse autoencoder. stanford university, 1-19 [EB/OL]. [2018-06-15]. https://doi.org/10.1371/journal.pone.0006098.
[11] JR C R J, BERNSTEIN M A, FOX N C, et al. The Alzheimer’s disease neuroimaging initiative (ADNI): MRI methods [J]. Alzheimers and Dementia, 2010, 27(4): 685-691.
[12] ASHBURNER J. SPM12 Manual. [EB/OL]. [2018-06-15]. https://www.fil.ion.ucl.ac.uk/spm/doc/manual.pdf.
[13] RORDEN C. MRIcron [EB/OL]. [2018-06-15]. https://www.nitrc.org/projects/mricron.
[14] SHIN H C, ORTON M R, COLLINS D J, et al. Stacked auto-encoders for unsupervised feature learning and multiple organ detection in a pilot study using 4D patient data [J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2013, 35(8): 1930-1943.
[15] RAINA R, BATTLE A, LEE H, et al. Self-taught learning: Transfer learning from unlabeled data [C]// Proceedings of the 24th International Conference on Machine Learning. New York: ACM Press, 2007: 759-766.
[16] AHONEN T, HADID A, PIETIKÄINEN M. Face description with local binary patterns: application to face recognition [J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2006, 28(12): 2037-2041.
[17] NAVNEET D, TRIGGS B. Histograms of oriented gradients for human detection [C]//IEEE Computer Society Conference on Computer Vision and Pattern Recognition. New York: IEEE Press, 2005: 886-893.
MRI Brain Image Recognition Method Based on Improved L-BFGS Sparse Denoising Autoencoder
WANG Xin-ying, WANG Wan-qiu, WANG Hui
(College of Computer Science and Engineering, Changchun University of Technology, Changchun Jilin 130012,China)
With the rapid development of human science and technology and the continuous updating of medical imaging equipment, medical imaging technology plays an increasingly important role in the auxiliary diagnosis of brain lesions. An improved L-BFGS (limited memory Broyden-Fletcher- Goldfarb-Shanno) sparse denoising self-coding network model is proposed and applied to the recognition of Alzheimer’s disease (AD) in MRI brain images. The experimental data source is taken from the ADNI data set, and the original data is corrected, registered, segmented, smoothed, etc. to obtain gray matter images of the brain. Then, the improved unsupervised greedy pre-training method and L-BFGS algorithm are combined to train the deep self-coding network as well as the learning features through Softmax regression training, so as to realize the brain mapping of patients with symptoms image recognition. ILSDAE network model is of good robustness, and compared with the stack self-coding and self-learning methods, the experimental results show the effectiveness of the proposed method.
Alzheimer’s disease (AD); L-BFGS; sparse denoising autoencoder; MRI brain image
TP 391
10.11996/JG.j.2095-302X.2019020261
A
2095-302X(2019)02-0261-06
2018-09-03;
2018-10-30
国家自然科学基金项目(61303132);吉林省教育厅“十三五”科学技术项目(JJKH20170574KJ)
王新颖(1979-),女,吉林长春人,副教授,博士,硕士生导师。主要研究方向为图形图像处理、数据挖掘等。 E-mail:wang_xinying1979@163.com
