基于高通量测序的江西特色发酵豆豉中微生物群落多样性及其特征分析
2019-03-28,,,,
, ,, ,
(1.江西中医药大学基础医学院,江西南昌 330004; 2.厦门医学院,福建厦门 361023; 3.南昌大学中德联合研究院,江西南昌 330047)
豆豉是一类利用微生物发酵黄豆或黑豆等原料而成的传统调味副食品。豆豉历史悠久,可追溯至两千余年前的先秦时代,且分布广泛,至今依然在我国江西[1]、云南[2]、湖南[3]、湖北[4]等地区及日本[5]、韩国[6]、印度[7]、泰国[8]、马来西亚[9]等国家被人们制作食用。其中,江西豆豉精选优质黑豆,经洗豆、泡豆、沥干蒸制、冷却接种、制曲及二次发酵而成,以江西南昌稻香园豆豉为典型代表,具有“颗粒完整、乌黑油亮、豉肉酥松、豉香浓郁”等特点,历来是我国江南百姓青睐的地方风味特产,也是药食两用淡豆豉的典型代表。然而,由于气候、水土环境、风味偏好等因素不同,各地的豆豉微生物种类组成差异明显,极具地方特色。按照制曲及发酵过程及终成品的微生物区分,豆豉大致分为细菌、曲霉、毛霉、根霉和脉孢菌型等[10]。
传统发酵类食品的微生物群落组成之前通常采用富集分离培养鉴定的方式分析,由于技术本身的局限,即使选择更多的培养基和分离条件,也仅能分离出少量优势菌群,不能反映其微生物群落的确切组成[2]。随着低成本高通量测序服务的飞速发展,其在传统发酵类食品如豆豉中也得以越来越多地应用。第二代测序技术采用稳定的可逆终止法边合成边测序反应,即生成新DNA互补链时,加入的dNTP通过酶促级联反应催化底物激发出荧光,或直接加入被荧光标记的dNTP或半简并引物,在合成或连接生成互补链时释放出荧光信号;捕获的光信号并转化为一个测序峰值,获得互补链序列信息[11]。不仅确保了测序的高精确性和高顺序性,而且排除了由重复序列和同聚物导致的测序错误。如通过高通量测序,李晓然等[2]研究了云南地区豆豉中细菌群落多样性,石聪等[3]对比湖南浏阳豆豉在不同发酵阶段的微生物多样性,董蕴等[4]对湖北当阳细菌型豆豉细菌类群进行了评价。然而,作为经典的江西特产中华豆豉,仅有陈廷涛、汪孟娟、杨林等[12-15]近年来结合传统活菌计数、PCR-DGGE和高通量测序法对江西豆豉制曲和发酵阶段的菌群动态变化进行了一定的监测,而基于高通量测序的成品微生物群落多样性分析却少见报道。
本研究分别对细菌16S rDNA V4区和真菌ITS1区进行PCR扩增及MiSeq高通量测序,进而分析江西特色发酵豆豉中微生物群落多样性,从而更准确、全面地掌握其菌群特征,以了解和提高产品质量和食用安全性,也为后续豆豉生产的优化改进提供理论指导。
1 材料与方法
1.1 材料与仪器
豆豉 南昌稻香园调味食品有限公司,于实验室-80 ℃超低温冰箱保存备用;基因组DNA提取试剂盒 天根生化科技(北京)有限公司;Phusion ® High-Fidelity PCR Master Mix with GC Buffer NEB(北京)有限公司;高效高保真酶 NEB(北京)有限公司;Next® UltraTMDNA Library Prep Kit for Illumina建库试剂盒 NEB(北京)有限公司;GeneJET 胶回收试剂盒 赛默飞世尔科技(北京)有限公司。
5427 R高速冷冻离心机、Mastercycler X50 PCR扩增仪 艾本德中国有限公司;Miseq高通量测序仪 Illumina公司。
1.2 实验方法
1.2.1 gDNA提取 采用CTAB法[2]提取豆豉样品中所有微生物的gDNA,经1%琼脂糖凝胶电泳(电压:100 V时间:40 min)选择无明显降解、无杂质,质量合格,无菌水稀释浓度至1 ng/μL,备用。
1.2.2 PCR扩增 以1.2.1得到的gDNA为模板,选择Phusion® High-Fidelity PCR Master Mix with GC Buffer及高效高保真酶,采用带Barcode的特异引物,进行PCR扩增。30 μL PCR体系:2× Phusion® High-Fidelity PCR Master Mix 15 μL,2 μmol/L Primer 3 μL,1 ng/μL gDNA 10 μL,ddH2O 2 μL。PCR条件:98 ℃ 1 min;98 ℃ 10 s,50 ℃ 30 s,72 ℃ 60 s,30个循环;72 ℃ 5 min。引物及序列信息:16S rDNA(515F:5′-GTGCCAGCMGCCGCGGTAA-3′;806R:5′-GGACTACNNGGGTATCTAAT-3′),ITS1(ITS5-1737F:5′-GGAAGTAAAAGTCGTAACAAGG-3′;ITS2-2043R:5′-GCTGCGTTCTTCATCGATGC-3′)。
1.2.3 扩增产物的混样及纯化 得到的PCR扩增产物经2%琼脂糖凝胶电泳检测后,根据电泳结果将PCR产物调节成等浓度后进行混匀,再用GeneJET胶回收试剂盒纯化回收[16]。
1.2.4 高通量测序及α-多样性分析 由北京诺禾致源科技股份有限公司测序及物种注释、丰富度、复杂度等数据分析。采用Next® UltraTMDNA Library Prep Kit for Illumina建库,经定量和检测合格后MiSeq测序。截去Barcode和引物扩增序列,FLASH拼接,过滤、去嵌合体序列,得到有效Tags。Uparse聚类OTUs,细菌采用Mothur方法与Silva数据库(阈值设定0.8~1),真菌采用Qiime Blast方法与Unite_INSDC数据库进行物种注释(阈值设定0.6~1),统计各分类水平组成。基于物种注释结果进行物种丰富度聚类及α-多样性分析。以稀释曲线、chao1指数和Shannon指数评估α-多样性。
1.3 数据处理
物种注释、丰富度等数据分析结果均采用GraphPad Prism 5软件统计及绘图。
2 结果与分析
2.1 测序数据统计
经鉴定,gDNA无明显降解、无杂质,确认PCR产物目的条带大小正确,样品合格后进行后续建库测序分析,如图1所示。豆豉样品中细菌及真菌测序数据统计结果如表1所示。原始上机数据经拼接、过滤后,分别得到细菌31563条及真菌46834条有效序列;以97%的一致性经Uparse软件将有效序列聚类分析,分别得到细菌OTUs为114个,真菌OTUs为8个。

表1 豆豉样品中细菌及真菌测序数据统计Table 1 Bacterial and fungal sequencing data statistics in the Douchi
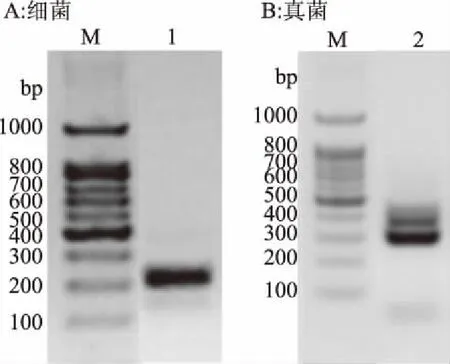
图1 豆豉样品中细菌及真菌PCR结果Fig.1 PCR result of bacteria and fungi in the Douchi注:M为100 bp ladder,上样量为2 μL;1为细菌PCR产物, 2为真菌PCR产物,上样量均为3 μL。
2.2 物种注释
豆豉样品中细菌物种注释结果如图2所示。可知豆豉样品中细菌的组成较丰富,以占比58%的Cyanobacteria Chloroplast Unclassified(蓝细菌门未分类属)和34%的Firmicutes Bacilli(厚壁菌门芽孢杆菌纲)为主。厚壁菌门芽孢杆菌纲中,94%为Bacillales(芽孢杆菌目)和约6%的Lactobacillales(乳杆菌目)。芽孢杆菌目中以Staphylococcus(葡萄球菌属)绝对优势(99%),约1%为Bacillus(芽胞杆菌属),以及极少量的Paenibacillus(类芽孢杆菌属)。尤其值得关注的是乳杆菌目,分布较为复杂,主要为Lactobacillaceae(乳杆菌科),含51%的Lactobacillus(乳杆菌属)和5%的Pediococcus(片球菌属);其次为Streptococcaceae(链球菌科),含12%的Streptococcus(链球菌属)和0.5%的Lactococcus(乳球菌属);另有12%的Enterococcus(肠球菌属);以及Leuconostocaceae(明串珠菌科),含7%的Leuconostoc(明串珠菌属)和5%的Weissella(魏斯氏菌属);0.2%的Aerococcus(气球菌属)。
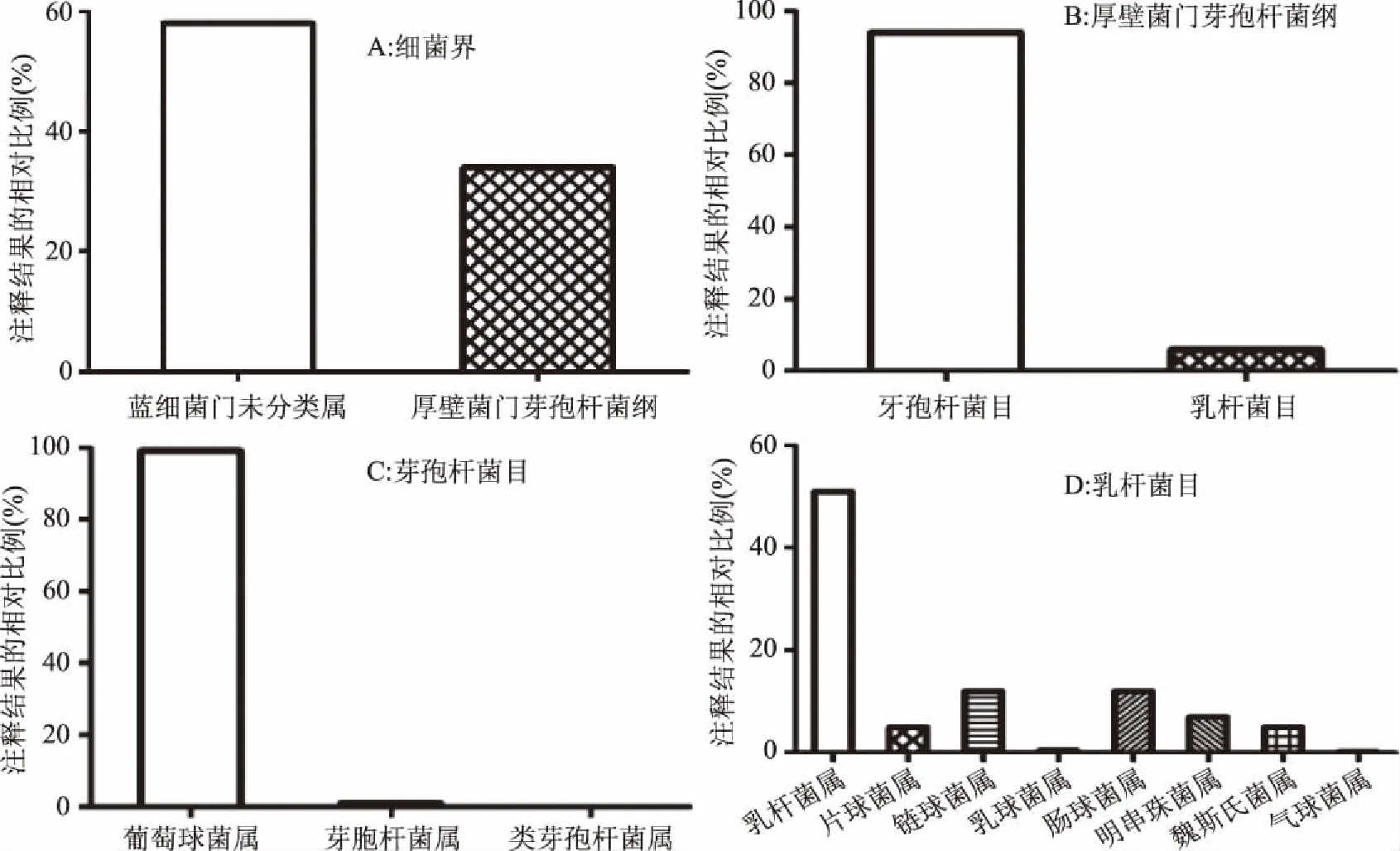
图2 豆豉样品中细菌的物种注释结果Fig.2 Species annotation result of bacteria in the Douchi
豆豉样品中真菌物种注释结果如图3所示。可知豆豉样品中真菌的组成极为单一,几乎全为ZygomycotaLichtheimia(接合菌门横梗霉属)。
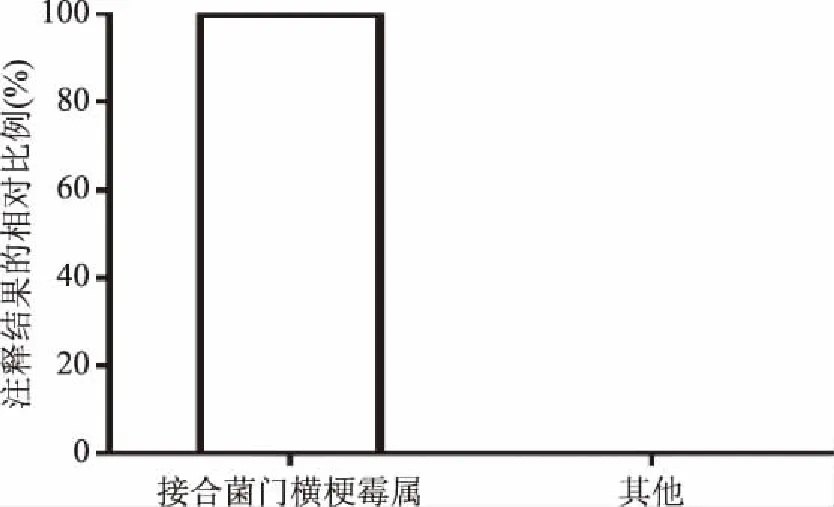
图3 豆豉样品中真菌的物种注释结果Fig.3 Species annotation result of fungi in the Douchi
2.3 物种丰富度聚类
根据物种注释结果,统计豆豉样品的细菌、真菌在门分类水平上物种相对丰度分布,如图4所示。可知占比最高为Cyanobacteria(蓝细菌门),达58%;其次Firmicutes(厚壁菌门),为34%;分列三、四位的Actinobacteria(放线菌门)和Proteobacteria(变形菌门),各约占4%;其余6门均低于总丰度的0.1%。而豆豉样品的真菌组成中,Zygomycota(接合菌门)占据绝对优势,比例高达99.94%,而Ascomycota(子囊菌门)仅为0.06%。

图4 豆豉样品在细菌、真菌门水平上的物种相对丰度分布Fig.4 Bacterial and fungal relative abundance on the phylum level in the Douchi
2.4 样品复杂度(α-多样性)分析
豆豉样品内细菌、真菌α-多样性如图5、图6和表2所示。根据稀释曲线、Chao1曲线、Shannon曲线结果可知,随着测序量增加细菌和真菌均趋向平坦,说明此次实验测序量合理,可涵盖豆豉样品中物种总数,反映样品内细菌、真菌等微生物信息全貌。而且,由表2可知,豆豉样品中细菌的物种总数(102)远大于真菌的物种总数(8)。细菌的Shannon指数(2.106)大于真菌的Shannon指数(0.774),说明豆豉样品中细菌的群落多样性较真菌更高。
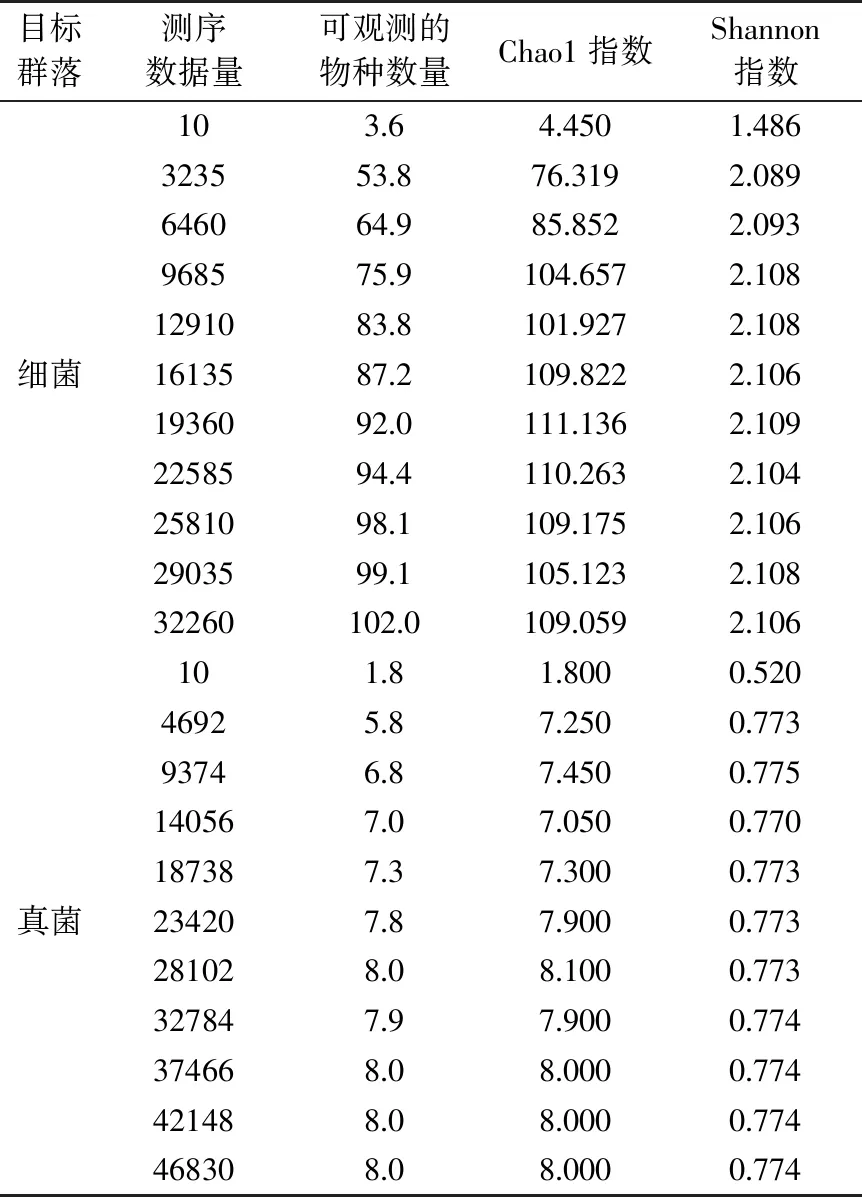
表2 豆豉样品中细菌及真菌α-多样性指标统计Table 2 Alpha diversity statistics of bacteria and fungi in the Douchi
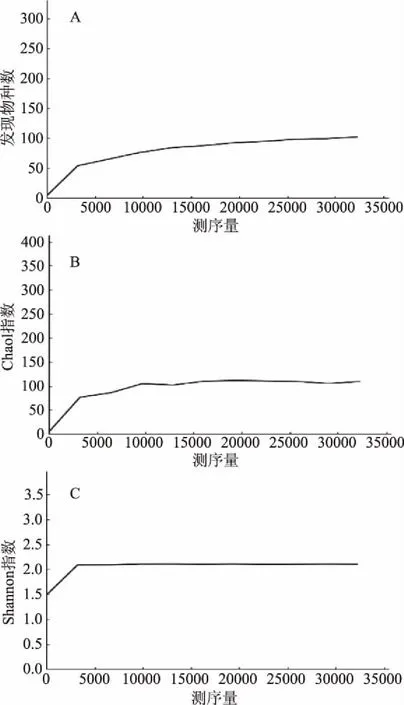
图5 豆豉样品中细菌α-多样性分析Fig.5 Alpha diversity analysis of bacteria in the Douchi注:A:稀释曲线;B:Chaol指数;C:Shannon指数;图6同。
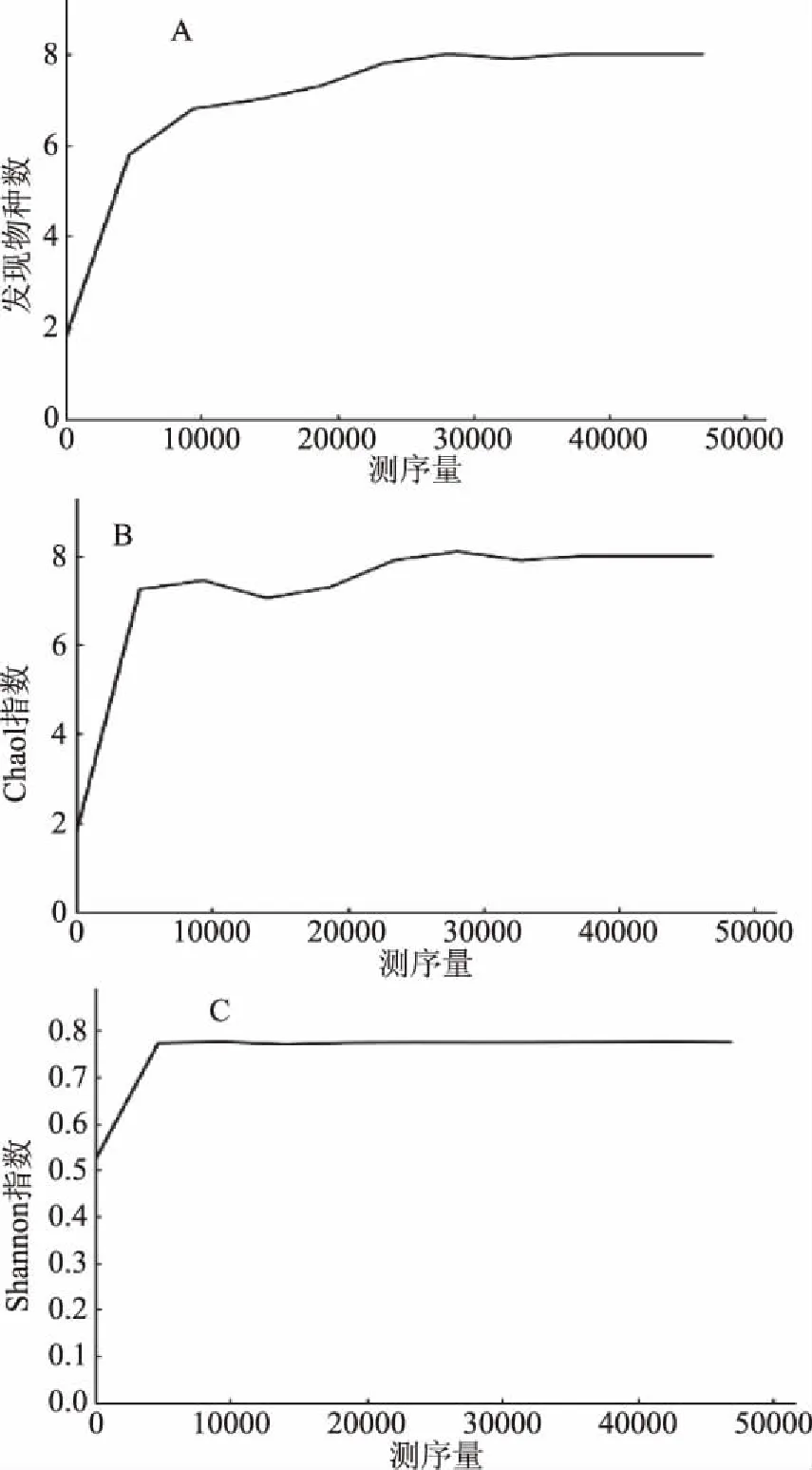
图6 豆豉样品中真菌α-多样性分析Fig.6 Alpha diversity analysis of fungi in the Douchi
3 讨论
传统发酵食品以其独特的风味、丰富的营养及优越的益生功能等特点被人们广泛喜爱,在很多地区已演化成宝贵的代表性文化遗产,如绍兴黄酒、镇江香醋、桂林腐乳、江西豆豉、郫县豆瓣酱、日本清酒、韩国泡菜、意大利乳酪等。基于传统培养和分子微生态学方法,结合基因组学技术,愈加证明其地域、风味特色与所处环境的特征微生物的参与密切相关[17-18]。其中豆豉作为一种我国特色的药食两用发酵豆制品,拥有复杂的微生物多样性及潜在的优良发酵益生菌株资源,近年来也愈加为科学工作者所关注[19-22]。由于多采用传统工艺开放式生产,各地区的环境气候、原料工艺、风味偏好等差异明显,豆豉制曲、发酵过程中及成品间的微生物种类组成差异极大,从而造就了其极具地方特色的特点。前期已有不少科研工作者采用高通量测序技术对云南[2,23-25]、湖南[3,26]、湖北[4]、甘肃[22]等国内各地区豆豉的微生物多样性进行了分析。然而,作为药食两用的地方风味特产江西豆豉,其微生物群落多样性及特征却较少被关注。少数报道也仅采用了分析能力较局限的传统活菌计数和PCR-DGGE法[12-14]。在本研究中,利用MiSeq高通量测序对江西豆豉成品中细菌、真菌均进行了表征,并与国内其他典型地区进行比较。结果表明,江西豆豉成品中细菌组成较为丰富,主要以蓝细菌门未分类属、葡萄球菌属、乳杆菌属为主,含少量的肠球菌属、链球菌属、明串珠菌属、片球菌属、魏斯氏菌属,以及极少量的乳球菌属、气球菌属;而真菌组成较为单一,几乎全为接合菌门横梗霉属;相比传统活菌计数和PCR-DGGE结果显然分析更为全面准确,与制曲发酵阶段的菌群组成也有较大差异[1,12-15]。与其他地区豆豉样品特征差异明显,如云南[24,25]、甘肃[22]样品中假丝酵母属均为优势菌群,而江西未检出;湖南浏阳[3]样品中虽以横梗霉属为主,但真菌组成更为复杂,同时存在丝衣霉菌属、嗜热子囊菌属、毛孢子菌属、毕赤酵母属、德巴利酵母属等[4]。而细菌类别中基本都存在葡萄球菌属、乳杆菌属、肠球菌属、明串珠菌属、魏斯氏菌属;但比例区别较大,如云南景洪[24]样品以明串珠菌属为主,云南普洱[25]样品以嗜盐四联球菌、湖北当阳[4]样品以芽孢杆菌属为绝对优势菌群。
4 结论
江西豆豉成品中细菌组成较为丰富,以蓝细菌门未分类属、葡萄球菌属、乳杆菌属为主;真菌组成较为单一,大都为接合菌门横梗霉属。因此,本研究结果进一步揭示了环境对豆豉中微生物群落多样性的影响,同时证实了江西豆豉具有丰富的微生物群落多样性及特色的菌群组成结构,这为后续豆豉工业化生产的优化改进提供理论指导,以进一步提高产品质量和食用安全性;同时也为深入挖掘具有我国传统地方特色的发酵菌种和基因资源提供基础数据和导向。
