应用剩余杂合体衍生群体定位水稻粒重粒形QTL
2019-03-22朱安东孙志超朱玉君张荟牛小军樊叶杨张振华庄杰云
朱安东 孙志超 朱玉君 张荟 牛小军 樊叶杨 张振华 庄杰云, *
应用剩余杂合体衍生群体定位水稻粒重粒形QTL
朱安东1孙志超1朱玉君1张荟2, 3牛小军1樊叶杨1张振华1庄杰云1, *
(1中国水稻研究所 水稻生物学国家重点实验室/ 国家水稻改良中心, 杭州 310006;2福建农林大学 作物科学学院, 福州 350002;3福建农业科学院 水稻研究所, 福州 350018;*通讯联系人, E-mail: zhuangjieyun@caas.cn)
【目的】粒重粒形是影响水稻产量和品质的重要因素,由大量数量性状座位(QTL)控制,其作用变异极大,但以往研究主要着眼于效应大的QTL。本研究在剔除主效QTL影响的基础上,开展微效粒重粒形QTL分析。【方法】在前期研究基础上,从原群体挑选出1个剩余杂合体单株,构建了在主效QTL区间纯合、在其余区域中13个区间分离的群体,种植于浙江杭州和海南陵水,测定千粒重、粒长和粒宽。【结果】采用Windows QTL Cartographer 2.5,检测到22个QTL,分布于10条染色体的12个区间,其中,10个区间在两地均呈显著作用,2个区间仅在杭州试验中呈显著作用。进一步从该群体筛选出1个只在其中4个QTL区间杂合的单株,自交构建分离群体,验证了这4个区间对粒重粒形的效应。【结论】排除主效QTL有利于提高微效粒重粒形QTL的检测功效;虽然微效QTL可能易受环境和遗传背景影响,但仍可具有稳定表现。这些结果为进一步开展粒重粒形QTL的精细定位、克隆和分子标记辅助选择奠定了基础。
数量性状座位; 千粒重; 粒形; 剩余杂合体; 水稻
水稻(L.)是人类最重要的粮食作物之一,水稻单产的提高对粮食安全发挥了至关重要的作用。随着生活水平的提高,人们对稻米品质的要求越来越高,优质和高产均至关重要。千粒重是水稻产量的重要构成因素,在正常生长状态下,主要决定于粒长、粒宽和粒厚;粒形是评价稻米外观品质的重要指标之一,同时还影响稻米的加工、蒸煮和食味品质[1],该指标一般由粒长和长宽比确定。揭示水稻千粒重、粒长和粒宽的遗传控制机理,具有重要的生物学意义和育种应用价值。
水稻千粒重属于多基因控制的数量性状,其数量性状座位(QTL)研究一直得到广泛重视,定位的QTL分布于水稻所有12条染色体,并已有16个获得克隆[2-6]。这些研究大大丰富了人们对水稻千粒重分子调控机制的了解,并为水稻高产优质育种提供了宝贵的基因资源。然而,这些克隆研究均着眼于效应较大的QTL,加性效应最弱的也达到了0.8 g[7]。有研究表明,微效QTL在水稻重要农艺性状调控中扮演着重要角色[8];就千粒重、粒长和粒宽而言,仅应用珍汕97/密阳46这1个籼籼交组合,就在第1染色体长臂分解出6个微效QTL[9]。可以推测,水稻基因组中存在大量影响千粒重的微效QTL,它们对水稻产量潜力的贡献和对品质性状的影响不容忽视,因此,微效QTL的发掘、验证和克隆,对于全面解析千粒重调控网络、丰富可供育种利用的优异基因资源,具有重要意义。
微效QTL研究一直都面临挑战,其中一个重要原因是易受遗传背景影响。在主效和微效位点同时分离的初级定位群体中,效应较小的QTL往往因主效基因的掩盖而难以得到有效检测[10-12],排除主效QTL干扰是解决该问题的一条有效途径。在排除前期检测到的6个水稻种子活力QTL影响的基础上,Ye等[13]利用剩余杂合体衍生的分离群体,检测到3个控制种子活力的微效QTL;Xu等[14]利用主效基因纯合的次级群体,定位到6个控制稻米蒸煮与食味品质的微效QTL。在前期研究中,我们应用由籼稻材料特青和IRBB品系配组构建的重组自交系群体(简称TI群体),检测到13个控制粒形的QTL,分布于9条染色体的11个区间;其中,第3染色体区间的和第5染色体区间的呈主效作用,分别解释了56.71%的粒长变异和59.51%的粒宽变异,而其它QTL的贡献率均不足5%[15]。在本研究中,我们从特青/IRBB52剩余杂合体衍生的材料中,挑选出1个在和区间纯合、在其他部分染色体区域分离的群体,检测控制千粒重、粒长和粒宽的QTL,进而发展1个遗传背景更为一致的分离群体,验证了其中4个QTL区间的作用。
1 材料与方法
1.1 水稻材料
本研究应用了2个水稻分离群体。第1个群体衍生于籼稻组合特青/IRBB52的1个F7剩余杂合体,称为Ti52-2;该群体已应用于检测微效抽穗期QTL[16],含251个株系,存在13个分离区间,分布于除第2和第7染色体外的所有水稻染色体,分离区间共覆盖约84.0 Mb (图1)。在前期应用TI群体检测到的11个粒长和粒宽QTL区间中[15],Ti52-2群体的纯合区域覆盖2个主效QTL区间和6个效应较小的区间,分离区域仅覆盖3个QTL(、和)。第2个群体衍生于Ti52-2的1个单株,该单株仅在4个区间杂合,自交构建了1个新的S1:2群体,含179个家系,称为ZC8。
1.2 田间试验与表型鉴定
Ti52-2群体的S1:2和S1:3家系分别种植于浙江杭州(2016年5−9月)和海南陵水(2016年12月−2017年4月),ZC8群体的S1:2家系种植于杭州(2017年5−9月)。试验采用完全随机区组设计,2个重复,每重复每株系种1行12个单株,株行距16.7 cm × 26.7 cm,正常田间管理。成熟后每行混收中间10株,晒干。遵循Zhang等[17]的方法进行饱满籽粒的挑选,采用SA-G型种子数粒仪(万深)测定千粒重、粒长和粒宽,以2个重复的平均值为基础进行数据分析。
1.3 标记检测
Ti52-2群体的标记数据已具备,连锁图谱已构建[16]。对于ZC8群体,从每个S1单株取幼叶约2 cm,采用微量法[18]提取DNA。共采用9个多态性标记,分别为位于第6染色体的RM20731、第8染色体的RM22755和RM210、第10染色体的RM6100、RM1108和RM7300以及第12染色体的RM3246、和RM511。SSR标记信息源自Gramene数据库 (http://www.gramene.org/)。PCR扩增遵循Chen等[19]方法,扩增产物应用6%非变性聚丙烯酰胺凝胶分离,银染检测。
1.4 数据分析
分别计算Ti52-2群体杭州试验和陵水试验、ZC8群体杭州试验的基本统计数据,包括平均值、标准差、变异系数、变异范围、峰度和偏度。
对于Ti52-2群体,连锁图谱前期已构建[16];对于ZC8群体,利用Mapmaker/Exp 3.0软件[20]进行连锁分析,采用Kosambi函数将标记间的重组率转换成遗传图距(cM)。所有群体的QTL检测均运用Windows QTL Cartographer 2.5[21],采用复合区间作图法(composite interval mapping, CIM),经置换检验(permutation test)1000次计算,取全基因组< 0.05水平的LOD值为阈值。QTL命名遵循文献[22]制定的规则。
2 结果与分析
2.1 表型变异
Ti52-2和ZC8群体的千粒重、粒长和粒宽表现如表1所示。在各个试验中,3个性状均呈连续分布,偏度和峰度亦均较小,呈数量性状的遗传特点。
ZC8群体衍生于Ti52-2群体的1个剩余杂合体单株,群体同质性前者比后者高。2个群体都在杭州试验点进行性状鉴定,并在性状变异上呈现出ZC8低于Ti52-2的特点:千粒重、粒长和粒宽的标准差前者分别为0.27、0.053和0.016,后者分别为0.42、0.094和0.028;变异系数前者分别为0.012、0.007和0.006,后者分别为0.019、0.012和0.011。该结果与ZC8群体遗传背景同质性高于Ti52-2群体的特点是一致的。
2.2 在Ti52-2群体中检测到的千粒重、粒形QTL
采用Windows QTL Cartographer 2.5分别对Ti52-2群体杭州和陵水两地的千粒重、粒长和粒宽数据进行分析,共检测到22个QTL,它们位于10条水稻染色体的12个区间(表2和图1)。
在12个区间中,有10个在两地均检测到QTL,其中4个区间检测到的QTL在两地间完全一致。在第4染色体RM16252-RM335区间,检测到和,增效等位基因均来自特青,对千粒重和粒宽变异的贡献率在杭州分别为6.66%和11.50%,在陵水分别为7.14%和9.02%;在第8染色体RM22755−RM210区间,检测到和,增效等位基因均来自IRBB52,对千粒重和粒长变异的贡献率在杭州分别为3.55%和4.09%,在陵水分别为6.29%和5.70%;在第1染色体RM12210区间检测到,增效等位基因来自IRBB52,在杭州和陵水对粒长变异的贡献率分别为4.51%和4.76%;在第12染色体RM3246−区间检测到,增效等位基因来自特青,在杭州和陵水对粒长变异的贡献率分别为2.31%和11.67%。
在两地均检测到QTL的其余6个区间中,有4个在两地均对粒长具显著作用,它们分别位于第3、5、6和10染色体上的RM14302−RM14383、RM18927−RM3321、RM469−RM587和RM6704−RM6100区间,增效等位基因均来自IRBB52,对粒长变异的贡献率为3.52%~40.16%。此外,RM14302−RM14383区间还在陵水检测到,增效等位基因来自特青,对粒宽变异的贡献率为6.93%;RM18927−RM3321和RM6704−RM6100区间还在杭州分别检测到和,增效等位基因均来自IRBB52,对千粒重变异的贡献率分别为27.44%和6.44%;RM469−RM587区间还在杭州和陵水分别检测到和,增效等位基因分别来自特青和IRBB52,对粒宽和千粒重变异的贡献率分别为14.78%和3.98%。
在两地均检测到QTL的其余2个区间为位于第6染色体的RM20731区间和第11染色体的RM1233−RM5926区间,它们在两地均对粒宽呈显著作用,增效等位基因均来自特青,对粒宽变异的贡献率为4.79%~10.09%。这2个区间还分别在杭州和陵水对千粒重呈显著作用,增效等位基因均来自特青,对千粒重变异的贡献率分别为4.62%和6.13%。
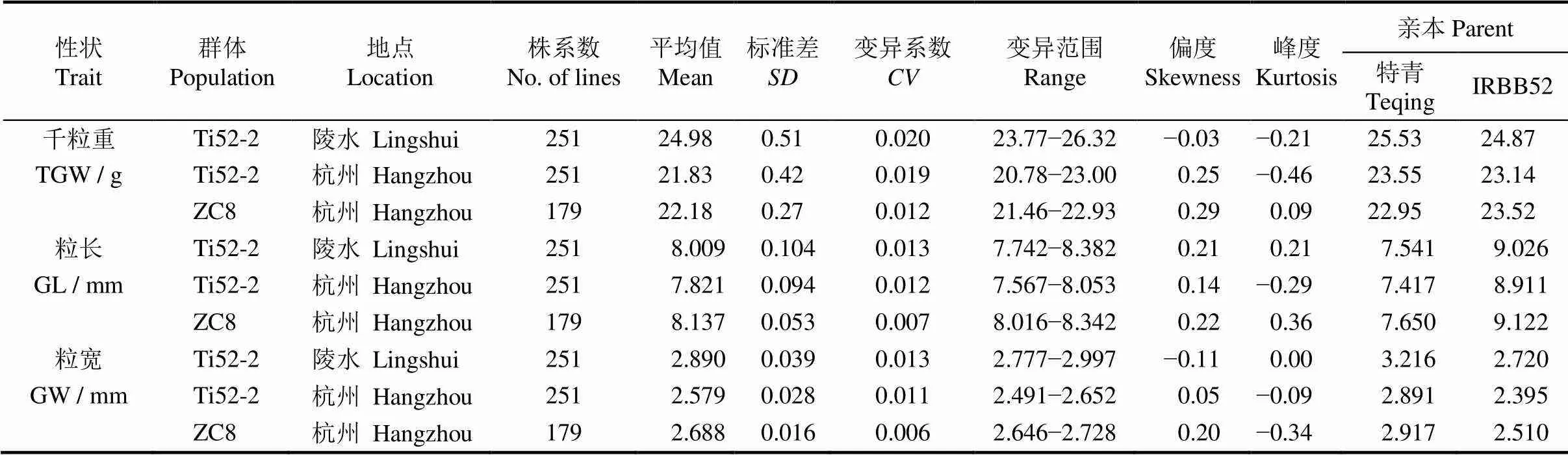
表1 Ti52-2群体的千粒重、粒长和粒宽表现
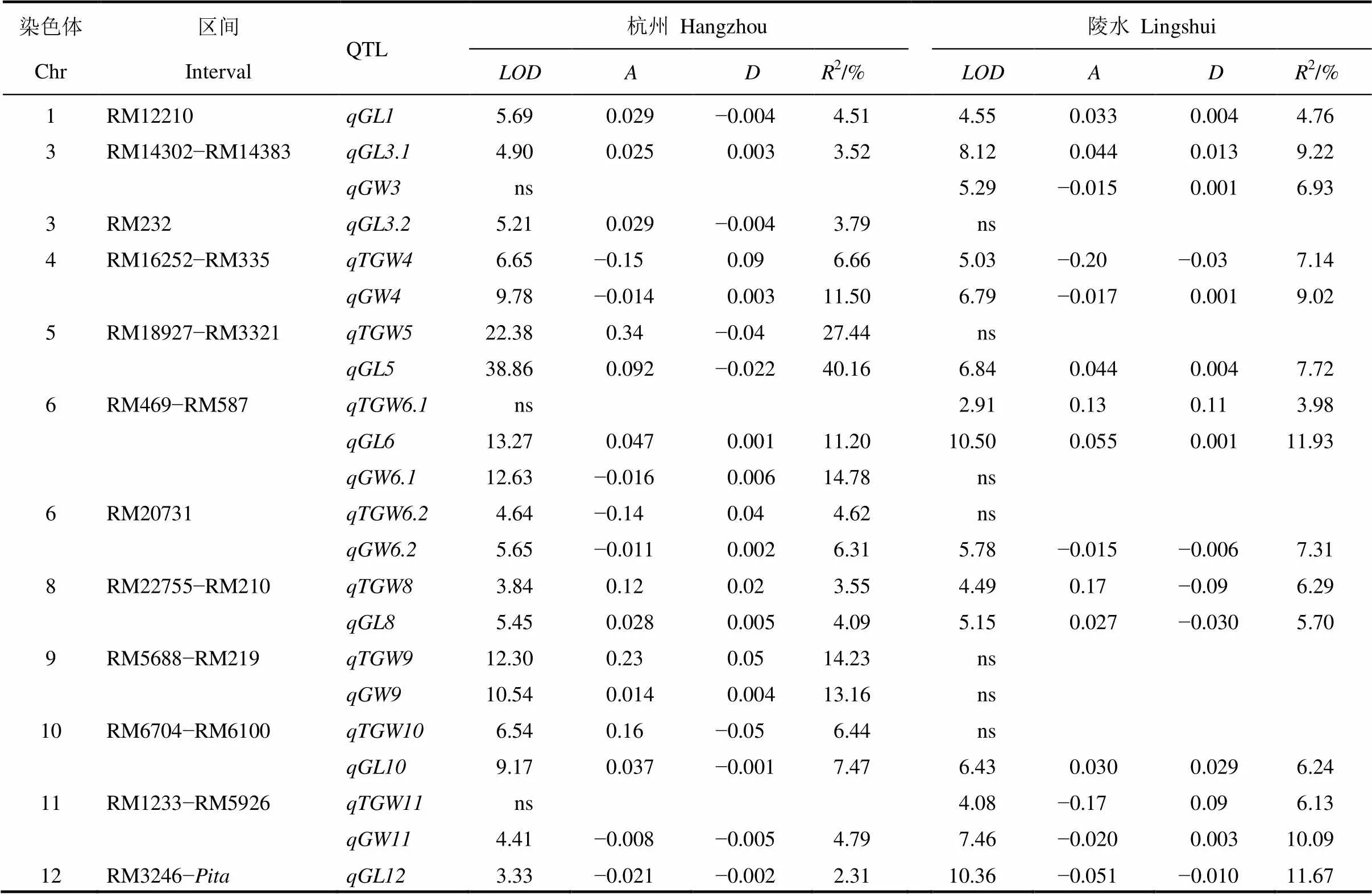
表2 在Ti52-2群体检测到的千粒重、粒长和粒宽QTL
−加性效应,指一个IRBB52等位基因取代特青等位基因所产生的遗传效应;−显性效应;2−贡献率,指相应QTL所解释的群体表型方差的比例;ns−不显著。表3中的缩写与此表相同。
, Additive effect measured as the genetic effect when the Teqing allele is replaced with the IRBB52 allele;, Dominance effect;2, Proportion of phenotypic variation explained by the QTL effect. ns, Not significant. Abbreviations are the same as in Table 3.
其余2个QTL区间为位于第3染色体的RM232区间和第9染色体的RM5688−RM219区间,均仅在杭州呈显著作用,增效等位基因均来自IRBB52,其中,对粒长变异的贡献率为3.79%。和对千粒重和粒宽变异的贡献率分别为14.23%和13.16%。
2.3 对4个QTL区间的验证
从Ti52-2群体挑选出1个单株,该单株除4个区间外均呈纯合状态,杂合区间为分别位于第6、8、10和12染色体的RM20731、RM22755−RM210、RM6100−RM7300和RM3246−RM511区间。将由该单株衍生的S1:2家系种植于杭州,进行千粒重、粒长和粒宽的QTL分析,共检测到8个QTL(表3)。与应用Ti52-2群体在这些区间检测到的QTL相比,性状和等位方向高度一致,对表型变异的贡献率普遍提高。
在Ti52-2群体中,位于第6染色体RM20731区间的和在杭州均被检测到,增效等位基因均来自特青;在ZC8群体中,这2个QTL亦被检测到,增效等位基因仍来自特青,对千粒重和粒宽变异的贡献率从Ti52-2群体的4.62%和6.31%分别提高到5.08%和8.03%。两群体均未在RM20731区间检测到对粒长的显著作用。
在Ti52-2群体中,位于第8染色体RM22755− RM210区间的和在杭州均被检测到,增效等位基因均来自IRBB52。在ZC8群体中,这2个QTL亦被检测到,增效等位基因来自IRBB52,对千粒重和粒长变异的贡献率分别从Ti52-2群体的3.55%和4.09%提高到16.32%和11.92%。两群体均未在该区间检测到对粒宽的显著作用。
在Ti52-2群体中,位于第10染色体RM6704− RM6100区间的和在杭州均被检测到,增效等位基因均来自IRBB52,其中,的显著性(LOD值)和贡献率均高于。在ZC8群体中,仅被检测到,增效等位基因仍来自IRBB52,对粒长变异的贡献率从Ti52-2群体的7.47%提高到9.27%。两群体均未在RM6704− RM6100区间检测到对粒宽的显著作用。

图1 在Ti52-2群体检测到的千粒重、粒长和粒宽QTL在染色体上的位置
Fig. 1.Chromosomal regions of QTL for the 1000-grain weight (TGW), grain length (GL) and grain width (GW) detected in the Ti52-2 population.
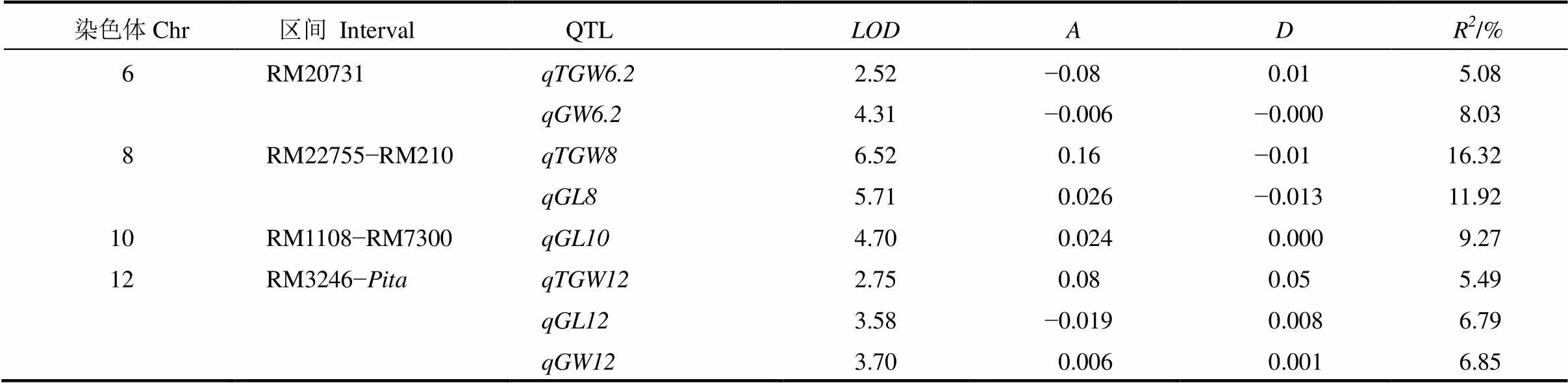
表3 在ZC8群体检测到的千粒重、粒长和粒宽QTL
在Ti52-2群体中,位于第12染色体RM3246−区间的在杭州被检测到,增效等位基因来自特青。在ZC8群体中,亦被检测到,增效等位基因仍来自特青,对粒长变异的贡献率从Ti52-2群体的2.31%提高到6.79%;同时,还检测到和,增效等位基因均来自IRBB52,对千粒重和粒宽变异的贡献率分别为5.49%和6.85%。
3 讨论
千粒重是水稻重要的农艺性状,与稻米产量和品质密切相关。本研究根据前期粒长、粒宽QTL初定位结果[15],从原群体后代株系中筛选材料构建群体,开展千粒重、粒长和粒宽QTL分析。首先,利用剔除了主效QTL变异的Ti52-2群体,在12个区间检测到22个QTL;然后,进一步构建了1个仅在4个QTL区间分离的ZC8群体,验证了这4个区间对千粒重、粒长和粒宽作用。
本研究所用的Ti52-2群体来源于特青/IRBB品系重组自交系群体(简称TI群体)的后代单株。在TI群体检测到的13个QTL[15],仅有3个处于Ti52-2的分离区域中,包括、和。和在Ti52-2群体中检测到,加性效应方向保持不变,来自特青的等位基因分别降低粒长和增加粒宽;未在Ti52-2群体检测到,但该QTL在TI群体定位于RM3773-RM3123区间,而Ti52-2群体的分离区间覆盖RM3773,未覆盖RM3123,两个群体检测结果的差异,是源于QTL与环境或遗传背景的相互作用,还是因为本身不位于Ti52-2群体的分离区域中,尚待进一步研究。除和所处区间外,在Ti52-2群体中还检测到10个QTL区间;挑选Ti52-2群体的1个单株构建了仅在其中4个区间分离的ZC8群体,这些区间对粒重、粒形的作用都得到良好验证,且对性状表型变异的贡献率有较大程度提高。这些表明,排除主效QTL分离、提高遗传背景纯合度,都有利于提高微效QTL的检测功效。
本研究检测到的12个QTL区间中,除第4染色体的RM14302−RM14383区间外,前人均有粒重粒形QTL报道。在第1染色体上,RM12210区间的粒长QTL与Huang等[23]所定位的粒长QTL具有类似的染色体位置;在第3染色体上,检测到粒长和粒宽QTL的RM14302−RM14383区间包含已克隆的[3],而RM232附近的与邢永忠等[24]定位的具有类似位置;在第5染色体上,RM18927−RM3321区间的粒长和千粒重QTL,与Li等[25]和姜恭好等[26]定位的千粒重QTL、与王军等[27]定位的粒长QTL具有类似位置;在第6染色体上,RM469−RM587区间检测到控制千粒重、粒长和粒宽的QTL,在多个研究中有报道[28-31],而RM20731附近的千粒重和粒宽QTL与Gao等[32]定位的和王军等[27]定位的具有类似位置;在第8染色体上,检测到千粒重和粒长QTL的RM22755−RM210区间包含Kang等[33]精细定位的;在第9染色体上,RM5688−RM219区间的千粒重和粒宽QTL与Tian等[34]定位的和张亚东等[35]定位的具有类似位置;在第10染色体上,RM6704−RM6100区间的千粒重和粒长QTL与前人多个报道类似[23, 36-38];在第11染色体上,检测到千粒重和粒宽QTL的RM1233−RM5926区间包含Oh等[39]精细定位的;在第12染色体上,RM3246−区间的粒长QTL与周梦玉等[40]定位的具有类似位置。对这些QTL区间的进一步鉴定,将有助于完善我们对水稻千粒重和粒形遗传基础的认识。
4 结论
在固定主效千粒重QTL基因型的基础上,应用1个涵盖13个分离区间的群体,两地鉴定千粒重、粒长和粒宽,检测到12个呈显著作用的区间,其中,10个在两地均呈显著作用,2个仅在杭州呈显著作用。与前期重组自交系群体定位结果相比,10个区间为新检测到,表明剔除主效QTL的影响有利于提高微效千粒重QTL的检测功效。继续构建仅在其中4个QTL区间分离的群体,经QTL分析,这些区间对粒重粒形性状的作用均得到良好验证,表明微效千粒重QTL的作用亦可高度稳定,这些结果为开展微效千粒重QTL的精细定位、克隆和分子标记辅助育种奠定了基础。
[1] 李一博, 赵雷. 水稻品质性状的遗传改良及其关键科学问题. 生命科学, 2016, 28(10): 1168-1179.
Li Y B, Zhao L. Genetic improvement and key scientific questions of grain quality traits in rice.2016, 28(10): 1168-1179. (in Chinese with English abstract)
[2] Li N, Xu R, Li Y. Control of grain size in rice., 2018, 31(3): 237-251.
[3] Yu J, Xiong H, Zhu X, Zhang H, Li H, Miao J, Wang W, Tang Z, Zhang Z, Yao G, Zhang Q, Pan Y, Wang X, Rashid M A R, Li J, Gao Y, Li Z, Yang W, Fu X, Li Z.contributing to rice grain length and yield was mined by Ho-LAMap., 2017, 15(1): 28.
[4] Hu Z, Lu S J, Wang M J, He H, Sun L, Wang H, Liu X H, Jiang L, Sun J L, Xin X, Kong W, Chu C, Xue H W, Yang J, Luo X, Liu J X. A novel QTLencodes the GSK3/SHAGGY-like kinase OsGSK5/OsSK41 that interacts with OsARF4 to negatively regulate grain size and weight in rice., 2018, 11(5): 736-749.
[5] Yu J, Miao J, Zhang Z, Xiong H, Zhu X, Sun X, Pan Y, Liang Y, Zhang Q, Rashid M A R, Li J, Zhang H, Li Z. Alternative splicing ofcontrols grain length and yield inrice., 2018, 16(9): 1667-1678.
[6] Zhao D S, Li Q F, Zhang C Q, Zhang C, Yang Q Q, Pan L X, Ren X Y, Lu J, Gu M H & Liu Q Q.acts as a transcriptional activator to regulate rice grain shape and appearance quality., 2018, 9(1): 1240.
[7] Li Y, Fan C, Xing Y, Jiang Y, Lou L, Sun L, Shao D, Xu C, Li X, Xiao J, He Y, Zhang Q. Natural variation inplays an important role in regulating grain size and yield in rice., 2011, 43: 1266-1269.
[8] Noriko K, Masayuki K, Kei K, Takuya K, Tsutomu N, Yuji H, Itsuro T, Takashi S, Kiyoaki K. Identification of quantitative trait loci for rice grain quality and yield-related traits in two closely relatedL. subsp.cultivars grown near the northernmost limit for rice paddy cultivation., 2017, 67: 191-206.
[9] Dong Q, Zhang Z H, Wang L L, Zhu Y J, Fan Y Y, Mou T M, Ma L Y, Zhuang J Y. Dissection and fine-mapping of two QTL for grain size linked in a 460-kb region on chromosome 1 of rice., 2018, 11: 44.
[10] Yamamoto T, Yonemaru J, Yano M. Towards the understanding of complex traits in rice: Substantially or superficially?, 2009, 16(3): 141-154.
[11] Takai T, Ikka T, Kondo K, Nonoue Y, Ono N, Arai-Sanoh Y, Yoshinaga S, Nakano H, Yano M, Kondo M, Yamamoto T. Genetic mechanisms underlying yield potential in the rice high-yielding cultivar Takanari, based on reciprocal chromosome segment substitution lines., 2014, 14(1): 295.
[12] Nagata K, Ando T, Nonoue Y, Mizubayashi T, Kitazawa N, Shomura A, Matsubara K, Ono N, Mizobuchi R, Shabaya T, Ogisotanaka E, Hori K, Yano M, Fukuoka S. Advanced backcross QTL analysis reveals complicated genetic control of rice grain shape in a×cross., 2015, 65(4): 308-318.
[13] Ye H, Foley M E, Gu X Y. New seed dormancy loci detected from weedy rice-derived advanced populations with major QTL alleles removed from the background., 2010, 179(6): 612-619.
[14] Xu F F, Sun C X, Huang Y, Chen Y L, Tong C, Bao J S. QTL mapping for rice grain quality: A strategy to detect more QTLs within sub-populations., 2015, 35(4): 105.
[15] Wang Z, Chen J Y, Zhu Y J, Fan Y Y, Zhuang J Y. Validation of, a quantitative trait locus for grain size on the long arm of chromosome 10 in rice (L.)., 2017, 16(1): 16-26.
[16] Sun Z C, Zhu Y J, Chen J Y, Zhang H, Zhang Z H, Niu X J, Fan Y Y, Zhuang J Y. Minor-effect QTL for heading date detected in crosses between indica rice cultivar Teqing and near isogenic lines of IR24., 2018, 6(3): 291-298.
[17] Zhang H W, Fan Y Y, Zhu Y J, Chen J Y, Yu S B, Zhuang J Y. Dissection of theregion into two tightly-linked minor QTLs having stable effects for grain weight in rice., 2016, 17(1): 98.
[18] Zheng K L, Huang N, Bennett J, Khush G S. PCR-based marker-assisted selection in rice breeding//IRRI Discussion Paper Series No.12. Manila, Los Banos, Philippines: International Rice Research Institute, 1995.
[19] Chen X, Temnykh S, Xu Y, Cho Y G, McCouch S R. Development of a microsatellite framework map providing genome-wide coverage in rice (L.)., 1997, 95(4): 553-567.
[20] Lander E S, Green P, Abrahamson J, Barlow A, Daly M J, Lincoln S E, Newberg L A. MAPMAKER: An interactive computer package for constructing primary genetic linkage maps of experimental and natural populations., 1987, 1(2): 174-181.
[21] Wang S, Basten C J, Zeng Z B. Windows QTL Cartographer 2.5. Raleigh, NC, USA: Department of Statistics, North Carolina State University, 2012.
[22] McCouch S R, CGSNL. Gene nomenclature system for rice., 2008, 1(1): 72-84.
[23] Huang N, Parco A, Mew T, Magpantay G, McCouch S, Guiderdoni E, Xu J, Subudhi P, Angeles E R, Khush G S. RFLP mapping of isozymes, RAPD and QTLs for grain shape, brown planthopper resistance in a double haploid rice population., 1997, 3(2): 105-113.
[24] 邢永忠, 谈移芳, 徐才国, 华金平, 孙新立. 利用水稻重组自交系群体定位谷粒外观性状的数量性状基因. 植物学报, 2001, 43(8): 840-845.
Xing Y Z, Tan Y F, Xu C G, Hua J P, Sun X L. Mapping quantitative trait loci for grain appearance traits of rice using a recombinant inbred line population., 2001, 43(8): 840-845. (in Chinese with English abstract)
[25] Li J X, Yu S B, Xu C G, Tan Y F, Gao Y J, Li X H, Zhang Q. Analyzing quantitative trait loci for yield using a vegetatively replicated F2population from a cross between the parents of an elite rice hybrid., 2000, 101: 248-254.
[26] 姜恭好, 徐才国, 李香花, 何予卿. 利用双单倍体群体剖析水稻产量及其相关性状的遗传基础. 遗传学报, 2004, 31(1): 63-72.
Jiang G H, Xu C G, Li X H, He Y Q. Characterization of the genetic basis for yield and its component traits of rice revealed by doubled haploid population., 2004, 31(1): 63-72. (in Chinese with English abstract)
[27] 王军, 朱金燕, 周勇, 杨杰, 范方军, 李文奇, 梁国华, 仲维功. 基于染色体单片段代换系的水稻粒形QTL定位. 作物学报, 2013, 39(4): 617-625.
Wang J, Zhu J Y, Zhou Y, Yang J, Fan F J, Li W Q, Liang G H, Zhong W G. Mapping of QTLs for grain shape using chromosome single segment substitution lines in rice (L.)., 2013, 39(4): 617-625. (in Chinese with English abstract)
[28] 林荔辉, 吴为人. 水稻粒型和粒重的QTL定位分析. 分子植物育种, 2003, 1(3): 337-342.
Lin L H, Wu W R. Mapping of QTLs underlying grain shape and grain weight in rice., 2003, 1(3): 337-342. (in Chinese with English abstract)
[29] Liang Y S, Zhan X D, Gao Z Q, Lin Z C, Yang Z L, Zhang Y X, Shen X H, Cao L Y, Cheng S H. Mapping of QTLs associated with important agronomic traits using three populations derived from a super hybrid rice Xieyou9308., 2012, 184(1): 1-13.
[30] Marathi B, Guleria S, Mohapatra T, Parsad R, Mariappan N, Kurungara V K, Atwal S S, Prabhu K V, Singh N K, Singh A K. QTL analysis of novel genomic regions associated with yield and yield related traits in new plant type based recombinant inbred lines of rice (L.)., 2012, 12: 137.
[31] Gao F Y, Zeng L H, Qiu L, Lu X J, Ren J S, Wu X T, Su X W, Gao Y M, Ren G J. QTL mapping of grain appearance quality traits and grain weight using a recombinant inbred population in rice (L.)., 2016, 15(8): 1693-1702.
[32] Gao Y, Zhu J, Song Y, He C, Shi C, Xing Y. Analysis of digenic epistatic effects andinteraction effects QTL controlling grain weight in rice., 2004, 5(4): 371-377.
[33] Kang Y J, Shim K C, Lee H S, Jeon Y A, Kim S H, Kang J W, Yun Y T, Park I K, Ahn S N. Fine mapping and candidate gene analysis of the quantitative trait locusassociated with grain length in rice., 2018, 40(4): 389-397.
[34] Tian F, Li D J, Fu Q, Zhu Z F, Fu Y C, Wang X K, Sun C Q. Construction of introgression lines carrying wild rice (Griff.) segments in cultivated rice (L.) background and characterization of introgressed segments associated with yield-related traits., 2006, 112(3): 570-580.
[35] 张亚东, 张颖慧, 董少玲, 陈涛, 赵庆勇, 朱镇, 周丽慧, 姚姝, 赵凌, 于新, 王才林. 特大粒水稻材料粒型性状QTL检测. 中国水稻科学, 2013, 27(2): 122-128.
Zhang Y D, Zhang H Y, Dong S L, Chen T, Zhao Q Y, Zhu Z, Zhou L H, Yao S, Zhao L, Yu X, Wang C L. Identification of QTL for rice grain traits based on extra-large grain material., 2013, 27(2): 122-128. (in Chinese with English abstract)
[36] Hittalmani S, Shashidhar H E, Bagali P G, Huang N, Sidhu J S, Singh V P, Khush G S. Molecular mapping of quantitative trait loci for plant growth, yield and yield related traits across three diverse locations in a doubled haploid rice population., 2002, 125(2): 207-214.
[37] Li S, Cui G, Guan C, Wang J, Lian G. QTL detection for rice grain shape using chromosome single segment substitution lines., 2011, 18(4): 273-278.
[38] 杨占烈, 戴高兴, 翟荣荣, 林泽川, 王会民, 曹立勇, 程式华. 多环境条件下超级杂交稻协优9308重组自交系群体粒形性状QTL定位. 中国水稻科学, 2013, 27(5): 482-490.
Yang Z L, Dai G X, Zhai R R, Lin Z C, Wang H M, Cao L Y, Cheng S H. QTL analysis of rice grain shape traits by using recombinant inbred lines from super hybrid rice Xieyou 9308 in multi-environments., 2013, 27(5): 482-490. (in Chinese with English abstract)
[39] Oh J M, Balkunde S, Yang P, Yoon D B, Ahn S N. Fine mapping of grain weight QTL,using near isogenic lines from a cross betweenand., 2011, 33(3): 259-265.
[40] 周梦玉, 宋昕蔚, 徐静, 付雪, 李婷, 朱雨晨, 肖幸运, 毛一剑, 曾大力, 胡江, 朱丽, 任德勇, 高振宇, 郭龙彪, 钱前, 吴明国, 林建荣, 张光恒. 籼稻C84和粳稻春江16B重组自交系遗传图谱构建及籽粒性状QTL定位与验证. 中国水稻科学, 2018, 32(3): 207-218.
Zhou M Y, Song X W, Xu J, Fu X, Li T, Zhu Y C, Xiao X Y, Mao Y J, Zeng D L, Hu J, Zhu L, Ren D Y, Gao Z Y, Guo L B, Qian Q, Wu M G, Lin J R, Zhang G H. Construction of genetic map and mapping and verification of grain traits QTLs using recombinant inbred lines derived from a cross betweenC84 andCJ16B., 2018, 32(3): 207-218. (in Chinese with English abstract)
Identification of QTL for Grain Weight and Grain Shape Using Populations Derived from Residual Heterozygous Lines ofRice
ZHU Andong1, SUN Zhichao1, ZHU Yujun1, ZHANG Hui2, 3, NIU Xiaojun1, FAN Yeyang1, ZHANG Zhenhua1, ZHUANG Jieyun1, *
(State Key Laboratory of Rice Biology / Chinese National Center for Rice Improvement,;College of Crop Science,;Rice Research Institute, Fujian Academy of Agricultural Science,,;*,:)
【Objective】Grain weight and grain shape are important factors influencing grain yield and quality in rice. There are quantitative traits controlled by a large number of quantitative trait loci (QTL) that vary greatly in effect, but not much attention has been paid to minor QTL. This study was conducted to detect minor QTL for grain weight and shape in the absence of major-QTL segregation. 【Method】Following results of a previous QTL mapping study, a residual heterozygous plant was identified from the original population. The new population derived was homozygous in regions where major QTL for grain weight and shape were detected in the previous study, but segregated in 13 segments of other genomic regions. The population was planted in Hangzhou, Zhejiang and Lingshui, Hainan. Thousand-grain weight, grain length and grain width were tested and used for QTL analysis using Windows QTL Cartographer 2.5. 【Result】A total of 22 QTL were detected, distributing on 12 intervals of 10 chromosomes. Ten of them were significant in both locations, and the other two were detected in Hangzhou only. One plant that was only heterozygous in four of the 12 QTL regions was identified, from which one new population was constructed and tested. The QTL effects of the four regions were well validated. 【Conclusion】Our results indicate that the power of detecting minor QTL could be efficiently increased by eliminating the influence of major-QTL segregation. It is also suggested that minor QTL could have a stable effect though it is believed that they are generally subjected to large influence of the genetic background and environmental conditions. Our results also lay a foundation for fine mapping, cloning and marker-assisted selection of minor QTL for grain weight and grain shape in rice.
quantitative trait locus; 1000-grain weight; grain shape; residual heterozygous line; rice
10.16819/j.1001-7216.2019.8120
Q343.1+5; S511.032
A
1001-7216(2019)02-0144-08
2018-11-06;
2018-12-14。
国家自然科学基金资助项目(31521064); 中央级公益性科研院所所基本业务费专项(2017RG001-2)。
