DNA甲基化抑制剂5-氮脱氧胞苷对水稻基因组甲基化及幼苗生长发育的影响
2019-03-22邓卉鄂志国牛百晓王磊陈忱
邓卉 鄂志国 牛百晓 王磊 陈忱, *
DNA甲基化抑制剂5-氮脱氧胞苷对水稻基因组甲基化及幼苗生长发育的影响
邓卉1鄂志国2牛百晓1王磊2陈忱1, *
(1扬州大学 江苏省作物遗传生理重点实验室/植物功能基因组学教育部重点实验室/江苏省作物基因组学与分子育种重点实验室/江苏省粮食作物现代产业技术协同创新中心, 江苏 扬州 225009;2中国水稻研究所 水稻生物学重点实验室, 杭州 311401;*通讯联系人, E-mail: chenchen@yzu.edu.cn)
【目的】DNA甲基化是高等植物中普遍存在的一种表观修饰形式,其在调节基因表达、维持基因组稳定以及调控植物生长发育等方面发挥重要作用。本研究拟对基因组甲基化如何影响水稻发育进行解析。【方法】利用DNA甲基化抑制剂5-氮脱氧胞苷(AZA, 5-Aza-2′-deoxycytidine)处理水稻幼苗,研究DNA甲基化抑制剂对水稻幼苗生长发育及相关基因表达的影响。【结果】AZA处理后水稻基因组甲基化水平下降、植株发育迟缓,但种子萌发并不受AZA处理的影响;DNA和组蛋白表观修饰相关基因的表达受到AZA处理抑制。此外,防卫反应和光合通路相关基因表达也受到AZA处理的影响,暗示DNA甲基化在这些基因的表达调控中可能发挥作用。【结论】这些结果表明AZA是一种有效的DNA甲基化抑制剂,AZA处理可以破坏水稻基因组甲基化水平的正常状态,从而影响水稻的正常发育。
DNA甲基化;幼苗生长;基因表达;水稻
表观遗传是指在核苷酸序列不发生改变的情况下,基因表达和功能发生的可遗传的变化[1]。表观遗传调控对于植物生长发育过程起着重要作用[2],如组蛋白甲基化修饰的位点及其修饰丰度均能参与基因的表达调控[3];植物DNA甲基化也参与植物基因表达调控,调节植物的生长发育[4]。
DNA甲基化在高等生物中普遍存在。与动物不同,高等植物基因组DNA甲基化除了发生在对称序列CG区,也会发生在非对称位点CHG和CHH(H=A、C或T)序列中[5]。植物DNA甲基化的发生和维持主要依赖3种在结构和功能上不同的甲基转移酶,即甲基转移酶1(MET1)、结构域重排甲基转移酶(DRM)和染色质甲基化酶(CMT)[6]。正常的DNA甲基化状态对于植物生长发育是必需的。DNA甲基化可以参与植物的逆境响应。植物通过调节相关基因的DNA甲基化状态或甲基化程度影响基因表达,以适应或减轻生物胁迫或非生物胁迫对植物造成的伤害[7-11]。此外,甲基化修饰或甲基化水平异常,会直接影响植株的正常生长发育[12-13]。DNA甲基化抑制剂是一类能使基因组甲基化水平降低、抑制DNA甲基化发生的化学物质。目前在植物生理研究上常用的DNA甲基化酶抑制剂包括5-氮胞苷(5-azacytidine)和5-氮脱氧胞苷(5-Aza-2′-deoxycytidine, AZA),其本质是一种胞嘧啶的类似物,它们能够与DNA甲基化转移酶共价结合,降低DNA甲基化转移酶的催化活性,从而降低DNA甲基化水平[14]。与此同时,另外一种胞苷类似物折布拉林(zebularine)也被广泛应用于植物的DNA甲基化研究中。对拟南芥等植物的研究表明,这些DNA甲基化抑制剂能够影响植物生长发育进程以及植株表型[15-18]。但是,对于DNA甲基化抑制剂处理影响生长发育的机制仍有待深入研究。本研究尝试利用DNA甲基化抑制剂AZA处理水稻幼苗,以探索DNA甲基化调控水稻生长发育过程的可能机理。
1 材料与方法
1.1 材料培养与处理
取粳稻品种Kitaake干燥、饱满的当季籽粒去壳,先用70%的酒精消毒1 min,10%次氯酸钠溶液浸泡60 min,再用灭菌蒸馏水清洗3次,每次1 min,最后分别播种于常规1/2 MS培养基(对照,CK)和AZA浓度为40 mg/L的1/2 MS培养基,放置于25℃恒温培养箱中生长。
1.2 方法
1.2.1 叶绿素含量测定
采用热乙醇浸提叶绿素的方法。对照苗和AZA处理苗生长7 d后,分别称取100 mg水稻根部以上部位,剪成长度约为20 mm的条状,放入盛有7 mL 95%乙醇、避光的试管中,加塞,置于60℃的温箱中保温浸提1 h,直至组织变白后取出,将浸提液浸入另一试管,再用2 mL 95%乙醇冲洗叶片3次,将冲洗液一并倒入试管,稀释3倍后,分别测定663 nm、645 nm和652 nm处吸光值,按Arnon公式计算叶绿素含量[19]。
1.2.2 叶片半薄树脂切片
采集AZA处理和对照生长7 d的幼苗叶片固定于2.5%戊二醛溶液,浸泡2 d;用0.1 mol/L磷酸缓冲液(pH 7.2)清洗3次,20 min/次;接着用不同浓度酒精(25%-50%-70%-90%-100%-100%)逐级脱水,20 min/次;再用不同浓度伦敦白胶(25%-50%-75%- 100%-100%)渗透,12 h/次。以上步骤均在4℃下进行。包埋样品,60℃下聚合2 d,样品切2 μm厚度半薄切片后用1%甲苯胺蓝染色后进行显微观察。
1.2.3 叶片表皮细胞显微观察
取长度2 cm左右叶片材料,放入1%纤维素酶R10溶液中,50℃下消化8 h后用1%的甲苯胺蓝溶液染色,再用刀片慢慢刮取叶片表皮,封片、压片后镜检。
1.2.4 RNA抽提和反转录
采集AZA处理和对照生长7 d的幼苗各3个生物学重复,液氮速冻后利用OMEGA植物RNA抽提试剂盒抽提RNA,再使用TaKaRa的反转录试剂盒对RNA进行反转录。
1.2.5 荧光定量PCR
利用Bio-RAD公司的SYBR Green荧光定量试剂盒对基因表达情况进行分析,20 μL反应体系中加入2 μL cDNA模板,以作为内参基因,利用ΔΔC法对基因表达水平进行比较(表1)。
1.2.6 DNA提取与重亚硫酸盐测序
分别采集AZA处理和对照生长7 d的幼苗样本,利用全式金DNA快速抽提试剂盒抽提DNA,抽提的DNA用ZYMO甲基化试剂盒进行亚硫酸氢盐处理。所有步骤均按照试剂盒推荐流程进行。根据CpG岛预测设计引物(表1)扩增目的片段,目的片段连进T载体,挑选至少10个单克隆测序后进行甲基化分析。
1.2.7 叶绿体透射电镜观察
取边长为1 cm的正方形叶片固定于2.5% GA溶液中,用注射器抽气去除叶片表面空气后4℃下过夜浸泡。用0.1 mol/L磷酸缓冲液(pH 7.2)清洗样品3次,20 min/次,4℃下进行。再用1%锇酸溶液固定20 min。用不同浓度丙酮(30%-50%-70%-80%- 90%-100%)逐级脱水,20 min/次。再用不同浓度树脂进行渗透包埋后切片观察。

表1 本研究所用PCR引物
2 结果与分析
2.1 5-氮脱氧胞苷(AZA)对水稻幼苗基因组甲基化水平的影响
为了研究甲基化抑制剂AZA对水稻幼苗基因组甲基化水平的影响,利用CG甲基化敏感的限制性内切酶Ⅰ对AZA处理后的幼苗基因组DNA进行酶切。Ⅰ能酶切未甲基化的限制性位点,而不能对甲基化的性位点进行酶切。与对照相比,在不同酶切反应条件下,AZA处理使植株基因组DNA对Ⅰ更敏感,其酶切条带大小整体小于未用AZA处理的幼苗(图1-A)。这一结果表明,AZA处理使基因组的CG形式甲基化水平下降。是水稻内源转座子,通常条件下处于高度甲基化状态以抑制其活性[20]。利用重甲硫酸盐转化的方法,对用AZA处理和未用AZA处理的水稻幼苗体内chr7(AP005292, nt 186 486−190 599, in GenBank)甲基化程度也进行了分析。发现在AZA处理的幼苗中chr7的CG、CHG和CHH甲基化均有不同程度下降,其中CG甲基化和CHG甲基化水平降低尤为显著(图1-B~C)。这些表明AZA处理能够有效降低水稻基因组的甲基化水平。
同时,为了研究AZA处理导致的基因组低甲基化状态对DNA甲基化酶相关基因表达的影响,我们利用荧光定量PCR对DNA甲基化酶、、、和的表达情况进行了分析。结果表明,除了的表达被显著上调外,其他4个基因的表达均明显受到AZA的抑制(图2),暗示AZA处理除了直接影响抑制基因组甲基化外,还能影响基因组甲基化相关酶编码基因的表达活性。
2.2 AZA处理对水稻幼苗生长发育的影响
在1/2 MS培养基中加入40 mg/L AZA处理水稻种子,种子的萌发与对照相比并无显著影响,其萌发速率和萌发率均与对照无明显差异(图3-A)。在AZA处理2 d后,幼苗生长状况与对照相比无明显差异,但随后幼苗的生长受到显著抑制。在AZA处理7 d后,水稻幼苗出现矮化表型(图3-B)。同时,AZA处理的植株叶片明显小于对照,叶色黄化,叶片相对卷曲,不能完全展开(图3-C)。

A−基因组DNA甲基化;B−转座子Tos17chr7的甲基化水平;C−转座子Tos17chr7的甲基化模式。
Fig. 1. Effects of AZA treatment on genomic methylation.

图中所显示数据为平均值±标准差(n=3)。
Fig. 2. Relative expression level of DNA methylation related genes in AZA-untreated and -treated rice seedlings.
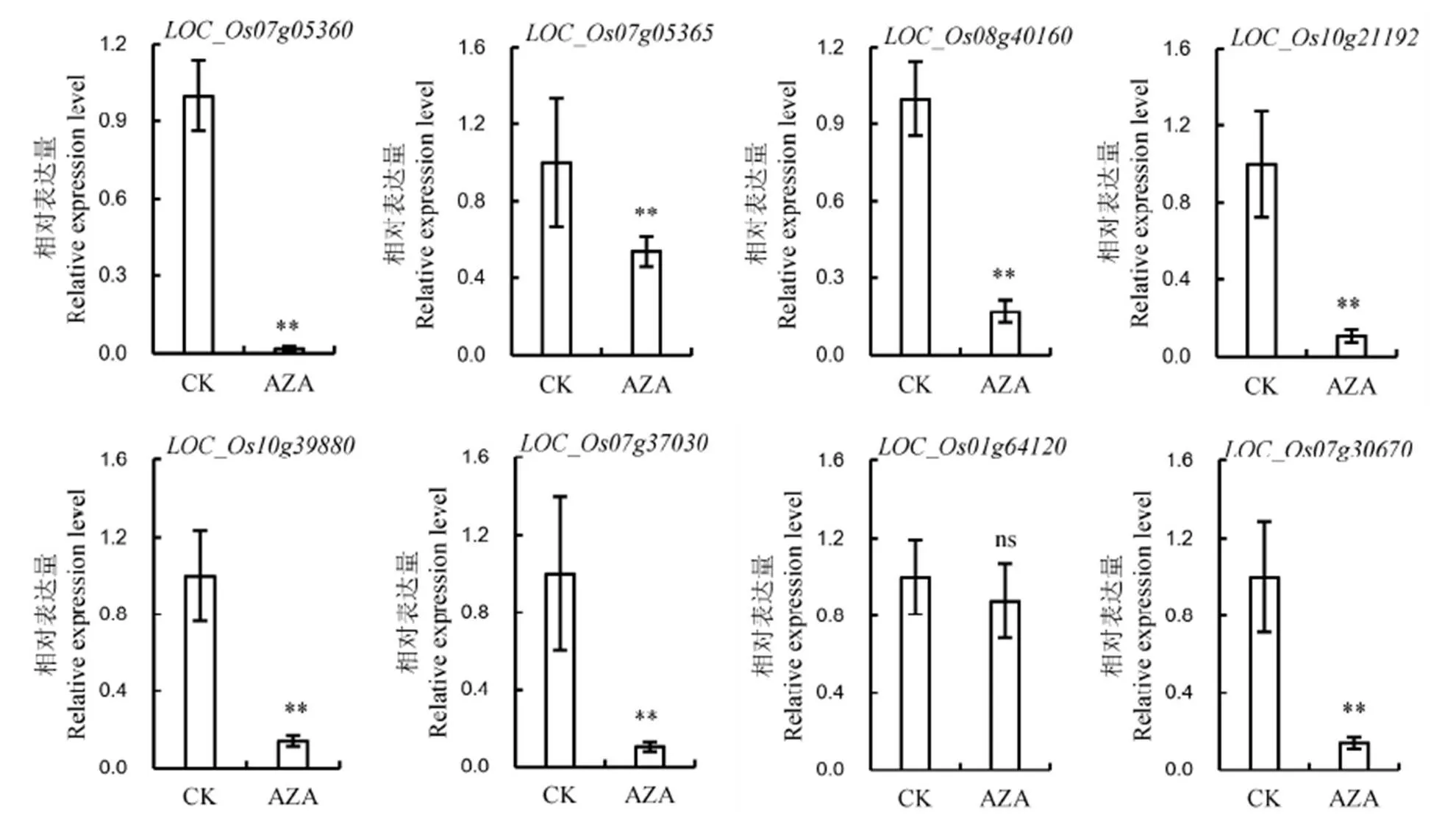
进一步对水稻幼苗叶片中叶绿素的含量进行测定,发现AZA处理后植株的叶绿素a、叶绿素b和总叶绿素含量均呈显著下降趋势(图3-D),表明AZA能够抑制水稻叶片中叶绿素的积累。随后我们通过透射电镜,比较了AZA处理和未用AZA处理植株叶片中叶绿体的超微结构。结果显示,AZA处理并未显著改变叶片中叶绿体的形态和结构,叶绿体的基粒和垛叠均无明显差异(图3-E)。同时,利用定量PCR分析,我们对参与光合反应相关基因表达量进行了分析。选取了8个在光合作用中参与了光合电子传递的基因包括光合反应中心蛋白编码基因,光合系统Ⅰ复合体蛋白编码基因,光合系统Ⅱ复合体蛋白编码基因以及细胞色素b6/f复合体蛋白编码基因。在AZA处理后,这些基因中除了没有变化外,其他基因的表达均受到显著抑制(图4)。

A−萌发率动态曲线,图中所显示数据为平均值±标准差(n=50);B−生长2 d后及7 d后植株表型;C−叶片表型;D−叶绿素含量测定,图中所显示数据为平均值±标准差(n=3),**表示差异达0.01显著水平;E−叶绿体结构观察,标尺为0.5 μm。
Fig. 3. Phenotypic analysis of the AZA-treated rice seedlings.
为了进一步探讨AZA抑制水稻幼苗发育的细胞学基础,我们对AZA处理和未处理的幼苗叶片进行了显微观察。半薄树脂切片结果表明AZA处理后植株叶片维管束数目减少4个左右(图5-A~B)。与对照相比,运动细胞结构异常,运动细胞的过度弯曲导致AZA处理幼苗叶片卷曲(图5-C~D)。通过对叶表皮细胞大小的量化分析,我们发现AZA处理后叶片表皮细胞的长度和宽度均显著降低(图5-F)。另外我们发现,AZA处理后,叶片上的硅细胞数目明显将少(图5-E)。因而,我们对硅转运蛋白基因的表达也进行了分析。结果显示3个重要的水稻硅转运蛋白基因、和的表达均受到AZA的显著抑制(图5-G),这与叶片中硅细胞数目减少的表型一致。
图中所显示数据为平均值±标准差(n=3)。**表示差异达0.01显著水平, ns表示无差异。
Fig. 4. Relative expression level of photosynthetic genes in AZA-untreated and AZA-treated plants.
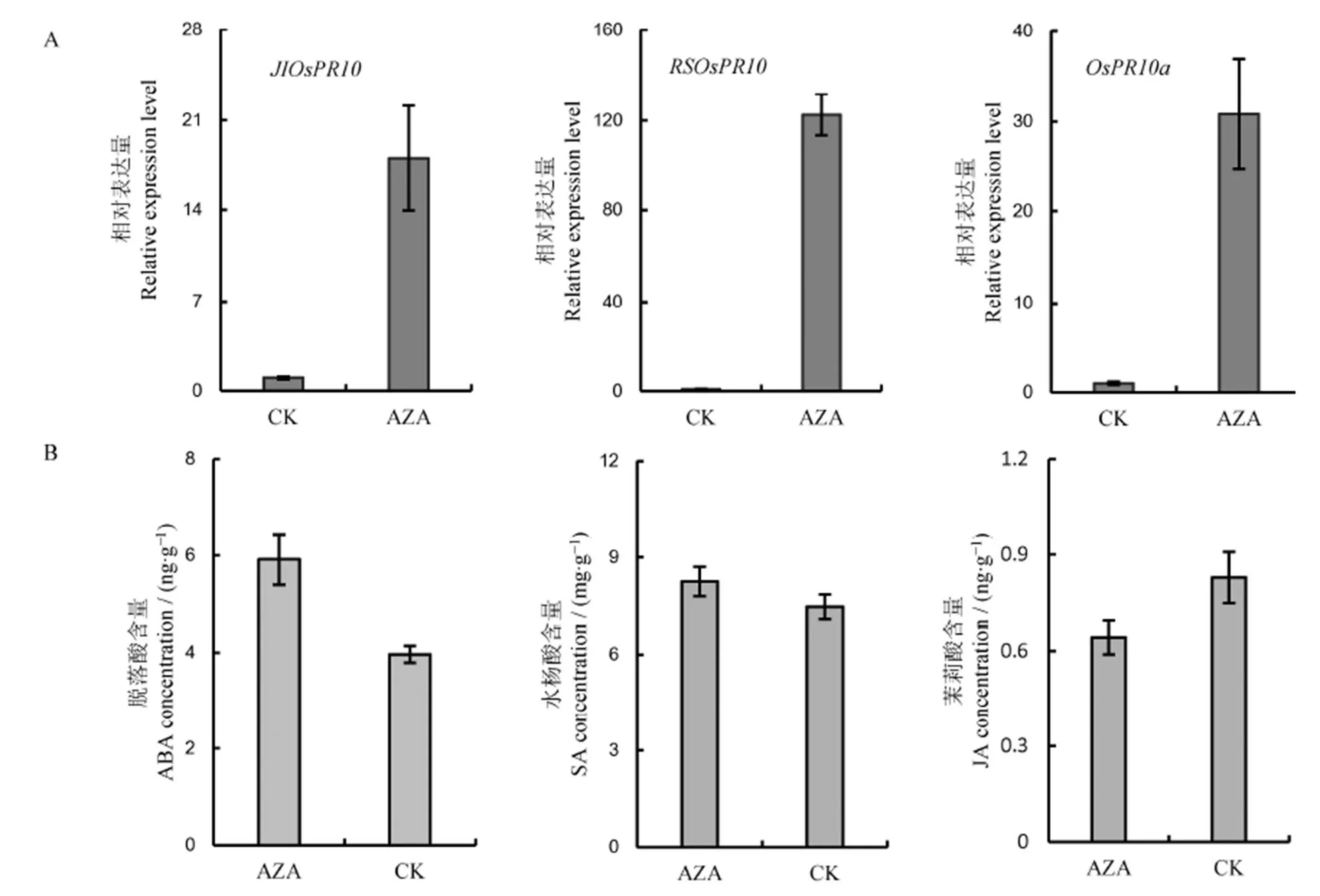
2.3 AZA对于病程相关基因表达以及内源激素含量的影响
近年来有研究显示DNA甲基化参与逆境胁迫下的基因表达调控[21-22],如病原菌侵袭时,植物病程相关基因表现出低甲基化,从而激活基因的表达,有助于激活植物对病原菌的防卫反应[23]。AZA处理能够降低基因组的甲基化水平,因此我们对AZA处理的幼苗中病程相关基因的表达也进行了分析。定量PCR结果显示,随机筛选的3个病程相关基因、和的表达在AZA处理苗中极显著激活,表达量增加了20~120倍(图6-A)。脱落酸(abscisic acid, ABA)、水杨酸(salicylic acid, SA)和茉莉酸(jasmonic acid, JA)是参与调控植物生物胁迫和非生物胁迫的主要激素,因而对ABA、SA和JA含量进行测定。结果表明在AZA处理后,ABA和SA的积累显著增加,但JA的含量却有所下降(图6-B)。这暗示AZA处理后植株呈现的低甲基化状态,除了导致病程相关基因的高表达,也导致内源激素的含量变化。
3 讨论
本研究以粳稻品种Kitaake为研究对象,对甲基化抑制剂AZA处理后植株的DNA甲基化状态、表型以及差异表达基因进行了详细的分析。结果发现AZA处理后,植株的CG形式甲基化水平(图1-A)和DNA甲基化酶相关基因表达降低(图2),基因组的CG形式甲基化水平下降,植株幼苗矮化,叶片较小且叶绿素含量的显著下降(图3-B~D)。这与之前发现的DNA甲基化水平异常会影响植株形态特征的结论一致。当植物中维持CG甲基化的DNA甲基化转移酶MET突变后,水稻种子严重萎缩,幼苗发育迟缓,甚至死亡[24];拟南芥植株呈现矮小的表型等[25]。这些结果表明甲基化抑制剂AZA处理可以降低水稻体内的DNA甲基化水平,进而影响植株的正常生长和发育。根据前人报道,突变后没有可见表型,而突变后纯合致死,因此可以推测起着更重要的作用[24]。已知突变水稻植株的DNA甲基化水平降低[24],这和AZA处理后的DNA低甲基化水平结果一致。我们发现大多数表观修饰相关的基因在AZA处理的幼苗中表达量降低,但是的表达却被显著激活。有意思的是,在突变植株中,的表达量也是上升的[24],我们推测当受到抑制后,能通过激活其表达,部分补偿突变带来的不利效应。由于和表现出不同的AZA响应形式,我们推测这两个基因的表观调控可能存在差异,AZA处理的植株的低甲基化状态能够抑制的表达,但是却能激活的表达。其背后的具体作用机理需要进一步深入研究。

A−AZA未处理植株叶片半薄切片,*表示维管束,标尺为200 μm;B−AZA处理植株叶片半薄切片,*表示维管束,标尺为200 μm;C−AZA未处理植株叶片放大;D−AZA处理植株叶片放大;E−叶片表皮细胞观察,标尺为25 μm;F−叶片表皮细胞长度和宽度统计,图中所显示数据为平均值±标准差(n=100),0.01 Fig. 5. Leaf phenotype and relative expression level of silicon transporter. A−病程相关基因JIOsPR10、RSOsPR10和OsPR10a的相对表达量, 图中所显示数据为平均值±标准差(n=3);B−AZA处理植株和未处理植株的脱落酸、水杨酸及茉莉酸含量, 图中所显示数据为平均值±标准差(n=3)。 Fig. 6. Relative expression level of pathogenesis-related genes and contents of resistance-related hormones. 前人研究发现,DNA甲基化作用可以通过调节杂交种中某些基因的表达去实现杂种优势。这一结论在水稻、高粱等杂交种中得以证实[26-27]。水稻杂交种转录组分析发现差异表达基因显著富集在光合作用生物学路径上[28],同时玉米杂交种、小麦杂交种都呈现较高的光系统活化能力及电子传递效率[27]。表明在杂交种中DNA甲基化通过调节基因表达可以影响光合系统通路。在我们的研究中,也发现AZA处理植株的光合系统通路相关基因表达被显著抑制了(图4),即基因组DNA甲基化水平降低会抑制光合基因的表达,光合反应过程对于DNA甲基化水平敏感。 叶片表皮细胞观察发现AZA处理之后,叶片上的“乳突”结构数目明显减少(图5-E),“乳突”是水稻叶片硅质化过程中形成的一种硅质细胞。同时,3个重要的水稻硅转运蛋白基因,和的表达均受到AZA的显著抑制(图5-G),这暗示DNA甲基化抑制剂降低了硅转运蛋白的活性,使植物对硅的吸收效率降低,影响了硅在水稻叶片上的积累和分布。 已有研究表明DNA甲基化与植物的抗性相关。在AZA处理水稻幼苗中,病程相关基因、的表达均被显著激活了(图6-A)。拟南芥中病程相关基因的表达也会被基因组低甲基化水平所诱导表达[29],同时DNA的低甲基化能够激活植物防御反应,拟南芥突变体对病原菌DC3000表现出比野生型更强的抗性[30]。水稻基因组DNA甲基化水平下降后,对于病原菌的抗性增强[31],即植物在应对胁迫时,会引发了大量的甲基化修饰改变去控制基因的表达,从而帮助植物抵御病原菌入侵[32]。在植物抗病反应中,内源信号分子如ABA、SA和JA等负责抗病信号的传递。我们发现在AZA处理后,水稻幼苗中这些内源激素水平也受到影响,ABA和SA的积累显著增加,JA的含量下降(图6-B),暗示DNA甲基化不仅能调控抗性相关基因表达,也能调控内源分子的抗病信号传递,从多个方面参与植物的抗病防御。 DNA甲基化涉及植物生长过程的各个方面,我们的研究表明DNA甲基化抑制剂处理使植物基因组甲基化水平下降后,植物的光合作用、抗逆性及硅吸收等方面都会受到影响。这些发现为今后研究DNA甲基化调控水稻生长发育提供了有用信息。 [1] Springer N M, Schmitz R J. Exploiting induced and natural epigenetic variation for crop improvement., 2017, 18(9): 563. [2] Shi J, Dong A, Shen W H. Epigenetic regulation of rice flowering and reproduction., 2015, 5: 803. [3] 洪苓苓, 马旭东. 组蛋白甲基化修饰的研究进展. 临床血液学杂志, 2010, 23(1): 54-56. Hong L L, Ma X D. Advances in histone methylation modification., 2010, 23(1): 54-56. (in Chinese) [4] 关录飞, 吴笑女, 徐启江. DNA甲基化及其对植物发育的调控. 生物技术通讯, 2008, 19(4): 632-634. Guan L F, Wu X N, Xu Q J. DNA methylation and its regulation effect during plant development., 2008, 19(4): 632-634. (in Chinese with English abstract) [5] 郭广平, 袁金玲, 吴晓丽, 顾小平. DNA甲基化在植物研究中的应用现状与前景. 植物遗传资源学报, 2011, 12(3): 425-430. Guo G P, Yuan J L, Wu X L, Gu X P. DNA methylation and its application in plant research., 2011, 12(3): 425-430. (in Chinese with English abstract) [6] Finnegan E J, Kovac K A. Plant DNA methyltransferases., 2000, 43(2/3): 189-201. [7] Dowen R H, Pelizzola M, Schmitz R J, Lister R, Dowen J M, Nery J R, Dixon J E, Ecker J R. Widespread dynamic DNA methylation in response to biotic stress., 2012, 109(32): E2183-91. [8] Wang W S, Zhao X Q, Pan Y J, Zhu L H, Fu B Y, Li Z K. DNA methylation changes detected by methylation- sensitive amplified polymorphism in two contrasting rice genotypes under salt stress., 2011, 38(9): 419-424. [9] 赵云雷, 叶武威, 王俊娟, 樊保香, 宋丽艳. DNA甲基化与植物抗逆性研究进展. 西北植物学报, 2009, 29(7): 1479-1489. Zhao Y L, Ye W W, Wang J J, Fan B X, Song L Y. Review of DNA methylation and plant stress tolerance., 2009, 29(7): 1479-1489. (in Chinese with English abstract) [10] Ou X F, Zhang Y H, Xu C M, Lin X Y, Zang Q, Zhuang T T, Jiang L L, Wettstein D V, Liu B. Transgenerational inheritance of modified DNA methylation patterns and enhanced tolerance induced by heavy metal stress in rice (L.)., 2012, 7(9): e41143. [11] Goll M G, Bestor T H. Eukaryotic cytosine methyltransferases., 2005, 74: 481-514. [12] Kakutani T, Jeddeloh J A, Flowers S K, Munakata K, Richards E J. Developmental abnormalities and epimutations associated with DNA hypomethylation mutations., 1996, 93(22): 12406-12411. [13] Asao H, Eun S H, Okano Y, Moritoh S, One A, Okano Y, Yamaguchi K, Shimatani Z, Koizumi A, Terada R. Targeted disruption of an orthologue of DOMAINS REARRANGED METHYLASE 2, OsDRM2, impairs the growth of rice plants by abnormal DNA methylation., 2012, 71(1): 85. [14] Baubec T, Pecinka A, Rozhon W, Scheid O M. Effective, homogeneous and transient interference with cytosine methylation in plant genomic DNA by zebularine., 2010, 57(3): 542-554. [15] 袁建民, 木万福, 麻继仙, 杨龙, 李易蓉, 但忠, 苏银玲. DNA甲基化抑制剂5-azaC对花椰菜生长发育的影响. 江西农业学报, 2018, 30(2): 42-45. Yuan J M, Mu W F, Ma J X, Yang L, Li Y R, Dan Z, Su Y L. The effects of DNA methylation inhibitor 5-azaC on growth and development of cauliflower., 2018, 30(2): 42-45. (in Chinese with English abstract) [16] 汪媛媛. 两种DNA甲基化抑制剂对菊花表型和DNA甲基化的影响. 开封: 河南大学, 2012. Wang Y Y. The effect of two different DNA methylation inhibitors on curcumin phenotype and DNA methylation. Kaifeng: Henan University, 2012. (in Chinese with English abstract) [17] 刘秋香. DNA甲基化抑制剂5-Aza-2′-deoxycytidine处理后水稻的DNA甲基化研究. 杭州: 浙江大学, 2014. Liu Q X. The research on DNA methylation of rice treated with DNA methylation inhibitor 5-Aza-2′- deoxycytidine. Hangzhou: Zhejiang University, 2014. (in Chinese with English abstract) [18] Sano H, Kamada I, Youssefian S, Katsumi M, Wabiko H. A single treatment of rice seedlings with 5-azacytidine induces heritable dwarfism and undermethylation of genomic DNA., 1990, 220(3): 441-447. [19] 段光明. 叶绿素含量测定中Arnon公式的推导. 植物生理学报, 1992(3): 221. Duan G M. Derivation of Arnon formula in determination of chlorophyll content., 1992(3): 221. (in Chinese) [20] Cheng C, Daigen M, Hirochika H. Epigenetic regulation of the rice retrotransposon Tos17., 2006, 276(4): 378. [21] 黄菲, 李雪梅, 王文生, 傅彬英. DNA甲基化在植物抗逆反应中的研究进展及其育种应用. 中国农业科技导报, 2013, 15(6): 83-91. Huang F, Li X M, Wang W S, Fu B Y. Research progress of DNA methylation in stress response and breeding in plant., 2013, 15(6): 83-91. (in Chinese with English abstract) [22] Boyko A, Kovalchuk I. Epigenetic control of plant stress response., 2008, 49(1): 61-72. [23] 彭海, 席婷, 张静, 吴先军.胁迫条件下植物DNA甲基化的稳定性. 中国农业科学, 2011, 44(12): 2431-2438. Peng H, Xi T, Zhang J, Wu X J. Stability of stress-induced DNA methylation in plant., 2011, 44(12): 2431-2438. (in Chinese with English abstract) [24] Hu L J, Li N, Xu C M, Zhong S L, Lin X Y, Yang J J, Zhou T Q, Yuliang A, Wu Y, Chen Y R, Cao X F, Zemach A, Rustgi S, Wettstein D V, Liu B. Mutation of a major CG methylase in rice causes genome-wide hypomethylation, dysregulated genome expression, and seedling lethality., 2014, 111(29): 10642-10647. [25] Kankel M W, Ramsey D E, Stokes T L, Flowers S K, Haag J R, Jeddeloh J A, Riddle N C, Verbsky M L, Rechards E J.MET1 cytosine methyltransferase mutants., 2003, 163(3): 1109. [26] Wang Z, Gerstein M, Snyder M. RNA-Seq: A revolutionary tool for transcriptomics., 2010, 10(1): 57-63. [27] 仪治本, 孙毅, 牛天堂, 梁小红, 刘龙龙, 赵威军, 李炳林. 高粱基因组DNA胞嘧啶甲基化在杂交种和亲本间差异研究. 作物学报, 2005, 31(9): 1138-1143. Yi Z B, Sun Y, Niu T T, Liang X H, Liu L L, Zhao W J, Li B L. Patterns of DNA cytosine methylation between hybrids and their parents in sorghum genome., 2005, 31(9): 1138-1143. (in Chinese with English abstract) [28] Song G S, Zhai H L, Peng Y G, Zhang L, Wei G, Chen X Y, Xiao Y G, Wang L L, Chen Y J, Wu B, Chen B, Zhang Y, Chen H, Feng X J, Gong W K, Liu Y, Yin Z J, Wang F, Liu G Z, Xu H L, Wei X L, Zhao X L, Ouwerkerk P, Hankemeier T, Reijmers T, Heijden R, Lu C M, Wang M, Greef J, Zhu Z. Comparative transcriptional profiling and preliminary study on heterosis mechanism of super- hybrid rice., 2010, 3(6): 1012-1025. [29] Dowen R H, Pelizzola M, Schmitz R J, Lister R, Dowen J M, Nery J R, Dixon J E, Ecker J R. Widespread dynamic DNA methylation in response to biotic stress., 2012, 109(32): E2183. [30] Yu A, Lepere G, Jay F, Wang J Y, Bapaume L, Wang Y, Abraham A L, Penterman J, Fischer R L, Voinnet O, Navarro L. Dynamics and biological relevance of DNA demethylation inantibacterial defense., 2013, 110(6): 2389-2394. [31] Akimoto K, Katakami H, Kim H J, Ogawa E, Sano C M, Wada Y, Sano H. Epigenetic inheritance in rice plants., 2007, 100(2): 205-217. [32] Deleris A, Halter T, Navarro L. DNA methylation and demethylation in plant immunity., 2016, 54(1): 579-603. Influence of DNA Methylation Inhibitor 5-Aza-2′-deoxycytidine on DNA Methylation and Seedling Development of Rice DENG Hui1, E Zhiguo2, NIU Baixiao1, WANG Lei2, CHEN Chen1,* (Jiangsu Key Laboratory of Crop Genetics and Physiology / Key Laboratory of Plant Functional Genomics of the Ministry of Education / Jiangsu Key Laboratory of Crop Genomics and Molecular Breeding / Jiangsu Co-Innovation Center for Modern Production Technology of Grain Crops,,;State Key Laboratory of Rice Biology, China National Rice Research Institute, Hangzhou 311401,;Corresponding author, E-mail: chenchen@yzu.edu.cn) 【Objective】DNA methylation is an epigenetic modification broadly existed in higher plants. DNA methylation plays an important role in regulating gene expression, maintaining the genomic stability and modulating plant development. Here, we used a DNA methylation inhibitor, 5-Aza-2′-deoxycytidine (AZA), to study how the DNA methylation status influences rice development. 【Method】We applied AZA to the rice seedlings to explore its influence on seedling development. 【Results】AZA-treatment resulted in overall hypomethylation of the rice genome. AZA-treated plants were strikingly shorter than the control, indicating AZA treatment disturbed normal development of the plants. However, the seed germination was not significantly affected. Expression of several key genes involved in DNA methylation could be repressed by AZA treatment. In addition, some genes involving defense and photosynthesis pathways were also down-regulated. 【Conclusion】Our findings confirm that AZA is a potent DNA methylation inhibitor and suggest that to maintain proper DNA methylation status is important for rice development. DNA methylation; seedling growth; gene expression; rice 10.16819/j.1001-7216.2019.9001 Q755; S511.01 A 1001-7216(2019)02-0108-10 2019-01-01; 2019-01-15。 国家自然科学基金资助项目(31571623, 31771744);植物功能基因组学教育部重点实验室开放课题资助项目(ML201805)。