酱油酿造过程中微生物多样性分析方法研究进展
2019-03-18张书泰周斌童星周其洋
张书泰,周斌,童星,周其洋*
(1.佛山市海天(高明)调味食品有限公司,广东 佛山 528500;2.广东海天创新技术有限公司,广东 佛山 528500)
酱油起源于中国,至今已有2500年的历史,在中国、日本、韩国及东南亚国家作为一种重要的食品调味品[1,2]。酱油酿造最重要的两个工艺环节是制曲和酱醪发酵[3,4]。其中,制曲阶段主要是以曲霉(如米曲霉、酱油曲霉)代谢活动为中心进行固态发酵,生成的物质是构成酱油风味的源泉[5,6];酱醪发酵主要是以耐盐性乳酸菌和耐盐性酵母菌为主进行液体发酵,在这个阶段生成大量的乙醇、甘油和醇类物质以及芳香族化合物,赋予酱油特殊的风味[7-9]。在酱油发酵过程中,微生物对酱油色、香、味和体态的形成和品质起到了极其重要的作用[10,11]。
自20世纪60年代开始,研究者开始对酱油发酵过程中的微生物多样性进行研究,研究方法主要是传统的平板分离培养技术[12]。后续随着生物化学和分子生物学技术的发展应用,使微生物多样性的研究方法不再局限于传统的分离培养技术,运用非培养的生理生化方法和分子生物技术实现了对环境中89%~99%不可培养或未培养微生物的检测,对微生物进行更全面而深入的了解[13]。本文介绍了研究传统发酵食品微生物多样性的多种方法,简要地综述了各技术的原理、优缺点及应用概况,以期为研究酱油酿造过程中微生物群落结构组成、演替和功能代谢及其对酱油风味的形成机理和对酱油品质的影响开拓新思路。
1 传统微生物培养法
传统微生物培养法根据目标微生物对不同碳源、氮源等营养物质的需求,在培养基中加入相应比例的营养成分,进行定时定温培养,然后通过形态观察,并结合生理生化特征等进行鉴定,分析不同可培养微生物的种类和数量。Tanaka等、Wei等[14]和Yan等[15]利用传统微生物培养法对中国传统酱油和日本酱油发酵过程中微生物变化分析得出,酱油制曲阶段中乳酸菌为优势菌群,其次为酵母菌和霉菌;酱醪发酵中虽然乳酸菌仍为优势菌群,但数量与制曲阶段相比降低了3~5倍。Cui等[16]研究发现酱油发酵整个阶段中,乳酸菌、酵母菌和霉菌总微生物数量在春季最高,夏季最低;其中,乳酸菌和酵母菌数量变化与总微生物变化趋势相一致;而霉菌在冬季数量最多,夏季数量变化最大。Tanasupawat等[17]利用微生物培养方法对泰国酱油酱醪中的乳酸菌数量和种类进行了分析,共分离纯化得到44株乳酸菌,其中以Lactobacillusacidipisis和Tetragenococcushalophilus为主,其次为Lactobacilluspentosus,Lactobacillusplantarum和Lactobacillusfarciminis;同时,结合生理生化反应发现Lactobacilluspentosus可利用D-glucose产生DL-lactic,Lactobacillusplantarum细胞内含2,6-二氨基庚二酸,Lactobacillusfarciminis为异型发酵乳酸菌。这些研究揭示了酱油酱醪发酵中乳酸菌群落的结构和功能,为酱油发酵中乳酸菌的应用提供了参考依据。传统微生物培养法有利于了解分离纯化得到的微生物的形态特征和生理特性,对有价值的微生物进行保藏、应用等,但该方法在实际应用中工作量大、流程繁琐,且人工模拟的环境与酱油发酵过程中微生物的生长环境存在一定偏差,不能准确反映微生物群落组成、分布及变化特征[18-20]。
2 磷脂脂肪酸(PLFA)分析法
不同的微生物细胞膜中含有不同种类和数量的磷脂脂肪酸,通过提取微生物细胞内的磷脂脂肪酸,利用气相、气质联用等色谱技术测定其种类和含量并进行分类鉴定。目前,该方法多用于土壤微生物的研究,在酱油发酵过程中微生物多样性的研究中运用较少,在白酒大曲、酸菜中有较多报道[21-24]。赵金松等采用PLFA技术分析了清香型大曲、浓香型大曲和酱香型大曲的微生物群落结构及其多样性,结果表明不同类型的大曲微生物都以真菌(18:2ω6,9、18:1ω9)为优势菌群,占脂肪酸总量的90%以上;细菌以G+(a14:0、i14:0、i15:0、a16:0、i16:0)为主;不同工艺大曲微生物群落结构存在明显差异,随制曲温度升高,大曲微生物多样性降低。Wu等利用PLFA分析了Lactobacillusplantarum和Zygosaccharomycesrouxii对中国酸菜发酵过程中微生物群落结构多样性及酸菜风味形成的影响,检测到酸菜发酵过程中微生物以G+(a14:0、i16:0)和真菌(18:2ω6,9、18:1ω9)为优势菌群。Zhang等[25]运用PLFA方法对中国传统发酵酱油不同发酵模式下酱醪发酵过程中微生物群落结构多样性进行分析,包括高盐稀态发酵(HSDS)和低盐液态发酵(LSSS),实验结果表明酱醪发酵中以G+((a14:0、i15:0、a15:0、a16:0、i16:0))和真菌(18:2ω9,11、18:1ω9、18:3ω9,12,15)为优势菌群;发酵期间,HSDS酱醪真菌生物量高于LSSS酱醪,且在发酵中期(60天)生物量达到最大,之后降低。
由于某些磷脂脂肪酸在不同微生物类群中都有存在,造成不同微生物类群的磷脂脂肪酸出现重叠;且磷脂脂肪酸含量也会受到不同因素的影响而发生改变,比如温度、pH值等;同时,微生物细胞内的磷脂脂肪酸也会随着生长期变化而发生变化[26,27]。因此,磷脂脂肪酸分析法适用于对微生物生物量和群落结构的动态分析,但不适用于微生物群落种群分析。
3 分子生物学方法
随着DNA提取技术、聚合酶链式反应(Polymerase Chain Reaction,PCR)技术的不断发展,分子生物学技术越来越广泛地应用于微生态学的研究,为发酵食品微生物群落多样性的研究带来了良好的契机[28,29]。
分子生物学以核糖体基因序列(rRNA)为研究对象,16S rRNA基因具有高度的保守性和高变异性,保守性能够反映生物物种的亲缘关系,高变异性则能反映生物物种特征核酸序列的差异性,以及拥有丰富的数据库等优点,广泛应用于原核微生物多样性研究[30-32]。真核微生物多样性研究主要基于18S rRNA基因、26S rRNA基因或5.8S rDNA间隔区的分析(ITS间隔区)的扩增和分析[33-36]。
3.1 基因指纹图谱分析法
基因指纹图谱技术是基于核酸分子成分、大小和构型上的差异,利用电泳方法将不同的核酸分子分离,形成不同分子量的基因条带图谱,根据图谱上条带信息进行基因分析的技术。基因条带图谱中,每个条带代表不同的操作分类单位(Operational Taxonomy Unit,OTU),条带数量反映微生物种群的多样性和相对丰度。目前,基因指纹图谱技术在发酵食品中应用较广泛,有以下几种类型。
3.1.1 DNA长度多态性图谱分析
DNA长度多态性图谱分析法是基于DNA长度多态性的研究方法,包括限制性片段长度多态性(Restriction Fragment Length Polymorphism,RFLP)分析法、扩增核糖体DNA限制性分析法(Amplified Ribosoma DNA Restriction Analysis,ARDRA)、末端限制片段多态性(Terminal Restriction Fragment Length Polymorphism,T-RFLP)技术、随机扩增多态性DNA(Randomly Amplified Polymorphic DNA,RAPD)技术、扩增片段长度多态性(Amplified Fragment Length Polymorphism,AFLP)技术。
限制性片段长度多态性分析法(RFLP)是根据不同微生物种群基因限制性片段长度的差异,先将基因组DNA进行PCR扩增,利用限制性内切酶进行消化,经电泳分离形成长度多态图谱,再根据图谱条带分子量大小和数量分析微生物群落结构和组成[37]。末端限制片段多态性(T-RFLP)技术和扩增核糖体DNA限制性分析法(ARDRA)是在RFLP基础上发展而来的。末端限制片段多态性(T-RFLP)技术是在其中一个PCR扩增引物5'末端上加上荧光标记,将基因组DNA进行PCR扩增后,经限制性内切酶消化后进行电泳分离,分析其带荧光标记的末端限制性片段多样性。扩增核糖体DNA限制性分析法(ARDRA)是对扩增后的16S rDNA进行限制性酶消化,再根据电泳分离得到的多态图谱分析微生物群落结构、组成。王彩虹[38]采用RFLP技术建立了汾酒清香型大曲、泸州老窖浓香型大曲和郎酒酱香型大曲细菌和真菌群落结构,比较了不同类型大曲细菌和真菌多样性;研究发现随着制曲品温的升高,耐温性芽孢杆菌、高温放线菌、嗜热或耐热霉菌比例也增加,同时也检测到了传统培养方法未检测到的微生物,如枝芽孢杆菌(Virgibacillus)、糖多孢菌(Saccharopolyspora)、扫帚状曲霉(Aspergilluspenicillioides)等。李艳等[39]从羊羔美酒大曲中分离出14种不同形态类型的酵母共474株,采用ITS-RFLP分析酵母菌多样性,经基因序列发现14株不同形态的酵母分属于6个属的6种酵母菌,其中以酿酒酵母为优势菌群。但是,RFLP、T-RFLP和ARDRA技术都存在一定的缺陷:操作繁琐,对操作人员技术和仪器检测精度要求高,以及图谱中的每个条带不一定对应一个菌种,影响微生物群落多样性的评估[40]。
随机扩增多态性DNA(RAPD)技术是以非限制性的随机核苷酸为引物,对基因组DNA进行多态扩增,扩增产物经电泳分离,根据图谱分析微生物基因组多态性。该技术的优点是对整个未知的基因组进行多态性分析,在物种分类和鉴定上应用较广泛[41,42]。但是,由于引物筛选过于繁琐,且随机引物扩增的条带过于复杂,不易分析。
3.1.2 DNA成分多态性分析
DNA成分多态性分析是利用双链DNA在变性剂梯度或者温度梯度下,部分解链形成单链DNA在聚丙烯酰胺变性凝胶上的迁移速率随解链程度的增大而减小,形成不同序列的DNA图谱条带。DNA成分多态性图谱分析技术包括时间温度梯度凝胶电泳(Temporal Temperature Gradient Gel Electrophoresis,TTGE)、温度梯度凝胶电泳(Temperature Gradient Gel Electrophoresis,TGGE)和变性梯度凝胶电泳(Denaturing Gradient Gel Electrophoresis,DGGE)。DGGE已成为研究发酵食品微生物多样性最常用的手段之一,也是酱油酿造过程中微生物多样性的研究中应用最广泛的技术,表1展示了部分近些年DGGE技术在酱油酿造过程中微生物多样性研究的概况。利用DGGE对酱油酿造过程中微生物多样性的研究结果表明,酱油制曲过程中微生物多样性程度较高,细菌主要以Weissellasp.,Staphylococcussp.,Bacillussp.,Lactobacillussp.为优势菌群,真菌以Aspergillus,Candida,Trichosporonasahii,Pichia等为优势菌群[43]。酱油发酵过程中,主要优势微生物由制曲期间不耐盐性和不耐酸性微生物演替成盐水落黄发酵之后的耐盐性和耐酸性微生物。其中细菌主要有:Weissellasp.,Staphylococcussp.,Lactobacillusfermentum,L.plantarum,Tetragenococcushalophilus[44]。真菌主要有:Zygosaccharomycesrouxii,Candidaetchellsii,Candidaversatilis,Rhodotorulasp.等。另外,还检测到部分不可培养的微生物,如uncultured yeast。DGGE和TGGE检测的准确性和灵敏性都很高,能够分离出1 bp碱基差异的DNA片段。但是,对操作人员技术要求高,由于部分微生物存在异源双链现象和对相似度近的微生物检测性差异等因素,无法对微生物进行定量分析。

表1 部分DGGE技术在酱油酿造过程中微生物多样性研究概况Table 1 Research status of microbial diversity in soy sauce brewing process by DGGE technology

续 表
3.2 宏基因组学
宏基因组是1998年Handelsman首次提出,将宏基因组定义为环境中全部微小生物遗传物质的总和[45,46]。随着高通量测序技术的不断发展,宏基因组学被广泛应用于自然环境微生物群落结构(如土壤、海洋等)、人体和动物肠道微生物群落结构、食品微生物群落结构的研究[47-52]。表2列举了近几年部分与食品微生物相关的宏基因组学研究,主要应用在奶制品、泡菜、普洱茶等发酵食品研究中。分析方法包括利用细菌和古细菌16S rDNA、真菌18S rDNA、28S rDNA和ITS分析食品微生物多样性及对食品微生物宏基因进行测序,分析其群落功能和代谢途径[53,54]。目前,宏基因组学在酱油酿造微生物的研究中应用较少,本文简要介了宏基因组学在其他发酵食品中的应用进展,以供利用宏基因组学分析酱油酿造微生物研究做参考依据。

表2 部分与食品微生物相关的宏基因组学研究Table 2 Some metagenomic researches related to food microorganisms

续 表
注:项目信息由MG-RAST(http://metagenomics.anl.gov/)搜索获得。
3.2.1 宏基因组学技术基本方法
3.2.1.1 提取总基因组DNA
DNA的提取方法可分为两种,即原位裂解法和间接提取法(细胞分离提取法,见图1)。原位裂解法主要是通过酶解法、变性剂等方法直接从样品中提取微生物总DNA,该方法操作简便,提取率高,但是提取的DNA片段小(1~50 kb),适用于构建小片段文库的DNA提取。间接提取法是先用物理方法从样品中分离微生物,然后采用温和的方法提取微生物总DNA,该方法的操作较繁琐,在分离微生物过程中可能造成部分微生物丢失,但是获得的DNA片段大(20~500 kb),且纯度高,适用于构建大片段文库的DNA提取。
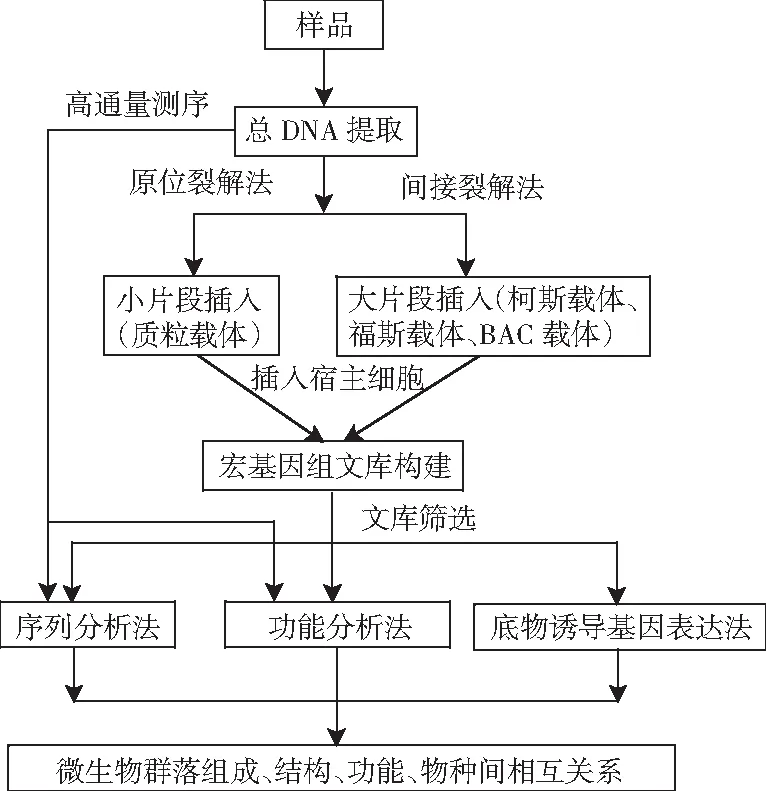
图1 环境微生物宏基因组学研究方法Fig.1 Research methods for environmental microbial metagenomics
3.2.1.2 宏基因组文库的构建
根据插入片段大小,将总基因组DNA克隆到质粒载体、柯斯载体、福斯载体或BAC载体中。再将克隆后的载体转化到宿主细菌,构建基因组文库,见图1。
3.2.1.3 宏基因组文库的筛选
由图1可知,宏基因组文库的筛选方法可分为:基于基因序列特异性差异的序列筛选法;基于基因活性的功能筛选法;基于底物诱导基因表达的筛选法[55]。
3.2.1.4 宏基因组学的分析
宏基因组学的分析方法主要是基于高通量测序技术和数据处理分析平台对扩增子测序和全基因组测序结果进行分析。其中,以对16S rDNA扩增进行测序分析的方法最常用[56]。表3列举了宏基因组学研究中部分常用的生物信息学平台。通过数据处理分析平台可以对序列进行前处理和分析,包括序列去噪、质量控制、序列拼接、分类学分析和序列功能预测等,进而分析微生物群落组成、群落功能、遗传多样性、微生物代谢的相互作用及其与环境因素的相互关联等[57-59]。

表3 常用的生物信息学平台及其主要工具Table 3 Commonly used bioinformatics platforms and their main tools

续 表
3.2.2 宏基因组学在发酵食品微生物研究中的应用
Almeida等[60]采用鸟枪法宏基因组学研究传统发酵奶酪微生物群落功能基因库,实验分离出142株细菌。基于宏基因组序列分析,这些细菌分属于67个属、137个种,并构建117个基因组草图,使得奶酪中细菌基因组增加2倍,为乳制品微生物群落研究提供了更多的基因数据源。Chen等[61]利用宏基因组学分析中国甘肃浆水菜发酵过程中真菌群落结构组成,研究发现浆水菜中真菌分属4个菌门、10个种。其中Candidaxylopsoci为优势菌群(占23.96%),其次为Sporopachydermiasp.,Cystofilobasidiaceae等,以及大量的未鉴定的真菌序列(占44.44%)。这些研究结果揭示了浆水菜中微生物群落结构组成,而大量的未鉴定菌群也暗示着浆水菜发酵过程中微生物多样性丰富、代谢途径复杂,有待进一步研究。Lyu等[62]运用宏基因组学分析普洱茶在渥堆发酵过程中微生物群落组成和群落功能,研究发现普洱茶在渥堆发酵中以细菌为优势菌群,细菌以变形菌门(Proteobacteria)、放线菌门(Actinobacteria)和厚壁菌门(Firmicutes)为主要菌群,真菌以子囊菌门(Ascomycota)为优势菌群。经KEGG代谢途径分析,与萜类、酮类及其他次生代谢产物相关的69种酶基因分布于16种代谢途径中,包括氨基酸代谢途径、碳水化合物代谢途径、核酸代谢途径等。
传统发酵酱油酿造过程微生物多样性研究中,Wang等[63]利用宏基因组学分析中国传统发酵酱油制曲到酱醪发酵过程细菌多样性的变化,研究发现制曲阶段细菌存在29个不同种属,酱醪发酵阶段有34个不同种属。由制曲转化至酱醪发酵有7个不同种属的细菌消失,12个不同新种属出现。Sulaiman等[64]运用鸟枪法宏基因组学对中国传统发酵酱油为期6个月的酱醪发酵过程中微生物群落结构多样性及微生物功能进行分析,实验发现Weissella在发酵初期为优势菌群,随后Candida为优势菌群,整个发酵过程中酵母数量增加,真菌丰度降低;而细菌丰度在发酵过程中先降低,到第6个月则增加。通过宏基因组的代谢重建,揭示了酱油发酵过程中蛋白质和碳水化合物异氧发酵的特征,与检测到的发酵过程中乙醇含量和pH值下降情况相符合。其中,对编码碳水化合物代谢途径的基因进行分析,包括三羧酸循环(TCA循环)和磷酸戊糖途径,发现从酱醪发酵第1个月开始代谢途径由同型发酵转为异型发酵,且与异型发酵乳酸菌相关性高。对编码支链氨基酸基因进行分析,包括异亮氨基酸、亮氨酸和缬氨酸,发现Candida可能通过Ehrlich途径代谢支链氨基酸,在发酵过程中由支链氨基酸转氨酶、脱羧酶和乙醇脱氢酶作用产生芳香化合物。这些研究结果有助于我们更好地了解中国传统发酵酱油发酵过程中微生物群落结构、功能演替和潜在的代谢能力。
另外,发酵食品安全也是发酵食品工业中的一个重要研究方向,宏基因组学技术在分析发酵食品安全问题中也发挥着重要作用。Park等[65]运用鸟枪法宏基因组学技术分析自然发酵的虾、韩国泡菜和德国泡菜中的病毒种类,发现37%~50%的新序列,以双螺旋DNA病毒Caudovirales为主,包括Myoviridae,Podoviridae和Siphoviridae。Jung等[66]运用宏转录组学技术研究朝鲜泡菜发酵过程中乳酸菌基因表达时也发现了大量噬菌体DNA序列。这些研究表明发酵食品中微生物种类和功能复杂,为发酵食品安全研究提供了更丰富的基因数据库。但是,宏基因组学的研究及数据的分析很大程度依赖于现有微生物物种数据库的丰富度,且生物信息分析对数据处理系统要求高,不同的算法对研究结果会产生很大的影响,这限制了对微生物新品种和新功能基因的研究和开发利用。
4 展望
随着分子生物学技术的发展,我们对酱油酿造过程中微生物群落结构的研究不再受传统微生物纯培养技术的限制。近几年,基于细菌16S rDNA和真菌18S rDNA(或25S rDNA~28S rDNA)或rDNA间隔区的分析,不仅克服了纯培养的限制,丰富了酱油酿造过程中微生物多样性,也提高了分析效率。特别是高通量测序和宏基因组学技术的不断革新,以及生物信息学工具和大数据处理平台的完善,不仅更真实地反映了酱油发酵过程中微生物组成、分布及演替,也让我们进一步了解了酱油酿造过程中微生物代谢和功能演替。但是,每种微生物多样性研究方法都有其优缺点,在研究工作中我们应当根据研究目的,选择合适的一种或结合几种研究方法进行研究,才能更有效地挖掘和利用微生物多样性信息。
在未来的工作中,我们应该考虑酱油工业生产中,酱油发酵过程的主要优势微生物和酱油生产工艺及生产环境之间的相互关系;了解酱油发酵过程中主要优势微生物间的相互作用,探索如何通过微生物数据预测酱油发酵过程中存在的工艺问题,为酱油生产工艺优化和酱油质量控制提供参考依据。
