转基因棉花连续种植对土壤AM真菌群落结构的影响
2019-03-08刘瑞华陈静怡王丽丽刘惠芬杨殿林赵建宁
刘瑞华,陈静怡,王丽丽,李 静,刘惠芬,杨殿林,赵建宁*
(1.天津农学院农学与资源环境学院,天津300384;2.农业农村部环境保护科研监测所,天津300191;3.东北农业大学资源与环境学院,哈尔滨150030;4.农业部产地环境污染防控重点实验室/天津市农业环境与农产品安全重点实验室,天津300191)
转基因作物在21 年的全球商业化进程中,种植面积从1996 年的170 万hm2上升至2016 年的1.85 亿hm2。棉花作为我国种植面积最大的转基因作物,2016 年种植面积约为280 万hm2,占棉花总播种面积的96%以上[1]。转基因作物的大规模商业化种植在给人们带来收益的同时,其对土壤微生物的影响及对土壤生态系统带来的风险越来越引发人们的关注[2]。
土壤是植物与其生长环境之间物质循环和能量转化的重要场所。丛枝菌根真菌是其菌丝与植物根系形成的共生体[3],也是土壤生态系统中一种同时具有植物根系和微生物特性的互惠共生体[4]。其容易受到土壤环境变化的影响,是一种较好的指示微生物,因此近年来吸引了越来越多的研究者对其进行研究和探讨。卢鑫萍等[5]研究发现,土壤盐分组成类型的不同会对AM 真菌孢子的密度和侵染率有影响;Ndoye 等[6]研究发现,土壤pH 值及各种无机速效养分含量均对AM 真菌的种属分布有显著影响;张海波等[7]研究发现,AM 真菌物种丰富度、Shannon 多样性指数和侵染率受土壤类型与植物种类交互作用的显著影响。
转基因作物的残体及根系分泌物皆有可能对土壤中的AM 真菌群落产生影响。王凤玲等[8]研究表明转Bt 棉叶片腐熟物抑制了AM 真菌在植物根部的定植,降低了AM 真菌的共生效应;Turrini 等[9-10]发现,转Bt 基因的玉米会影响共生体菌丝生长和侵染,并危害到其附着胞的发育,最终导致转Bt 基因品种的侵染率远低于其非转基因亲本;但是梁晋刚等[11]对转基因高蛋氨酸大豆的研究并未发现AM 真菌群落结构与非转基因大豆间存在显著性差异;Pasonen 等[12]研究发现转基因植物对AM 真菌的定植可能会有一定的影响。目前关于转基因作物种植对AM 真菌的影响还没有定论[13],关于长期种植转基因作物是否会对AM 真菌产生影响的研究还鲜见报道。因此,本研究利用PCR-DGGE 技术,以转基因棉花013011(抗旱)、SGK321(抗虫)和非转基因棉花TH2(抗旱受体)、石远321(抗虫受体)为研究材料,探究它们长期种植后对土壤AM 真菌群落多样性的影响,旨在为转基因棉花种植的土壤环境安全评价提供理论基础。
1 材料与方法
1.1 研究区概况
试验地位于天津市武清区梅厂镇周庄村(39°36′71.21″N,117°22′69.65″E),海拔6.3 m。地处华北平原东北部,地势平缓。年平均气温为11.6 ℃,年平均降水量为606 mm,无霜期212 d。供试土壤为潮土,2016 年棉花播种前试验地基本理化性质:全磷含量0.77±0.05 g·kg-1,全氮含量0.65±0.04 g·kg-1,有机质含量18±1.02 g·kg-1,pH值8.26±0.06。
1.2 供试材料
供试棉花品种为4 个品种,分为2 组对照试验。第1 组:TH2(抗旱受体)和013011(抗旱);第2 组:石远321(抗虫受体)和SGK321(抗虫)。供试的转基因材料013011 抗旱棉尚属于研究阶段,因此其所转基因不便公布;SGK321为我国自主研发的双价抗虫棉,是将双价抗虫基因(Bt+CpTI)导入石远321 后育成的新品种。供试品种均由中国农业科学研究院棉花研究所提供。
1.3 实验设计
本试验为大田试验,每个小区100 m2(20 m×5 m),每种棉花种植3 个小区即3 次重复,宽窄行种植,行距分别为90 cm和40 cm。试验地自2011年开始种植供试品种至今,每个小区固定种植同一棉花品种,一直采用覆膜种植的方式,每个小区间种植宽度为5 m 的玉米保护行。施肥量为:氮肥200 kg·hm-2,钾肥100 kg·hm-2,磷肥60 kg·hm-2。其中氮肥基施60%,追肥40%。磷钾肥全部作基肥施用,棉花其他田间管理按照常规管理,不施农药。
1.4 土壤样品采集
本试验分别在棉花的花铃期(2016 年8 月16 日)和吐絮期(2016 年9 月27 日)采集土样。采用随机取样法每个小区选取3 个点,每个点选5 株相距最近的棉花,去除表面杂草和枯枝落叶,每株距离主根2 cm位置处用直径3.5 cm 土钻取20 cm 深土样,并将3 个采样点的样品等比例混合,作为一个重复放入灭菌塑封袋带回实验室放入低温冰箱(-20 ℃)保存。
1.5 土壤总DNA提出
称取0.35 g土壤样品后采用BBI公司(Canada)的EZ-10 Spin Soil DNA Extraction kit 按操作说明提取总DNA,获得的总DNA 使用Biophotometer(Eppendorf,Germany)进行定量分析,并在1.0%琼脂糖凝胶中进行电泳,检测总DNA质量。
1.6 PCR扩增
采用巢式PCR(Nested PCR)方法针对AM 真菌的ITS片段进行扩增,引物序列及反应程序见表1。
第一轮PCR 反应体系:1 μL 每种引物,25 μL Premix Ex Taq(Loading dye mix),2 μL 的模板DNA,终体积为50 μL;第二轮PCR 反应体系:1 μL 每种引物,25 μL Premix Ex Taq(Loading dye mix),以2 μL 第一轮PCR 产物为模板,补充无核酸酶水(Nuclease-Free water,Promega)至50 μL。
1.7 变性梯度凝胶电泳(DGGE)检测
采用DcodeTM通用突变检测系统(Bio-Rad,USA)按照操作说明进行DGGE分析。丙烯酰胺凝胶(37.5∶1)浓度为8%,变性剂梯度为25%~50%[100%变性剂含有7 mol·L-1尿素和40%(V/V)去离子甲酰胺],电泳缓冲液为1×TAE。将25 μL PCR 产物和5 μL 6×loading buffer 混合后用微量进样器加入胶孔中,100 V、60 ℃条件下电泳16 h。电泳结束后,小心取出凝胶,放在SYBRTMGreen I(1∶10 000)(Invitrogen,USA)染液中染色30 min,然后用Gel Dox XR 凝胶成像系统(Bio-Rad,USA)观察与拍照。
1.8 条带回收及测序对比
选取主要条带割胶回收,用不带GC 夹子的引物进行扩增,PCR产物经过电泳分析确定为单一条带后送出进行克隆测序。测序结果在NCBI 上经过Blast对比分析。将其中同源性最高的序列确定为参照菌株。相似性≥97%的序列则视为同一序列型。
1.9 数据分析
采用Excel 2007 和SPSS 17.0(Duncan′s test)对试验数据进行分析,采用Quantity One 4.6.2 软件进行数字化处理并进行聚类分析。各样品用香农-维纳指数(Shannon-Wiener index,H)、均匀度(Evenness index,EH)和丰富度(Richness,S)评价AM 真菌多样性的变化,其计算公式如下:
H=-ΣPilnPi
EH=H/lnS
式中:H 代表香农-威纳指数;Pi代表第i 条带占总强度的比值;EH代表均匀度;S代表条带数量或丰富度。
测序结果采用Chromas 2.0 软件进行序列分析,登陆NCBI(National Center for Biotechnology Information)网站下载最相似的菌株序列作为系统发育树的参考序列。然后采用MEGA 6.0 软件,Neighbor-Joining法构建系统发育树,自展数(Bootstrap)为1000。
2 结果与分析
2.1 土壤总DNA的提取及PCR扩增
各土壤样品的总DNA 经过1.0%的琼脂糖凝胶电泳(图1),所有土壤样品总DNA 均在9416~23 130 bp,表示所获得的土壤DNA 质量很好。将获得的土壤DNA 直接用于PCR 扩增(图2)。扩增后所获得的片段集中在230 bp左右且条带清晰片段大小均一,可用于后续DGGE分析。
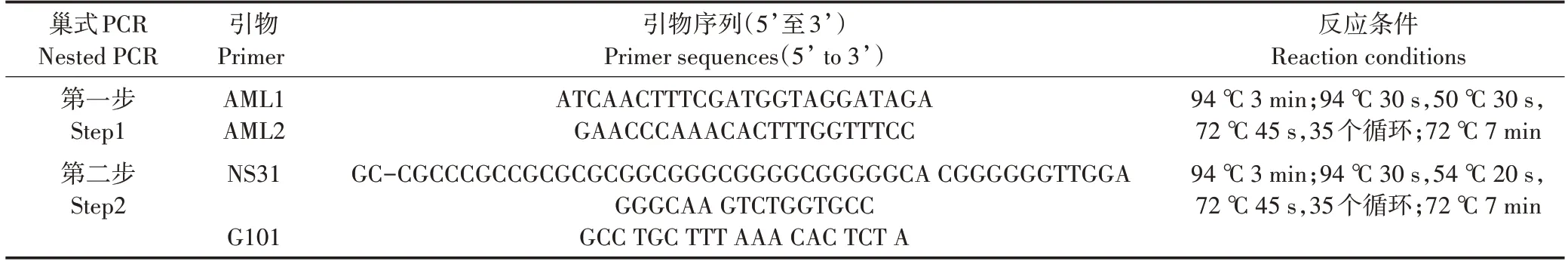
表1 PCR反应的引物及反应条件Table 1 Primers and conditions used for PCR
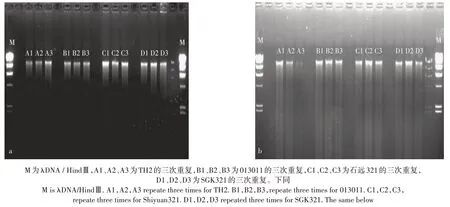
图1 棉花土壤样品花铃期(a)和吐絮期(b)DNA提取Figure 1 DNA samples extracted from the cotton soil at flowering and boll-setting stage(a)and boll opening stage(b)
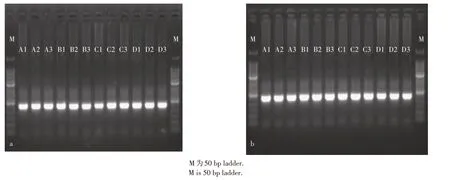
图2 棉花土壤样品花铃期(a)和吐絮期(b)AM真菌的ITS片段PCR结果Figure 2 AM fungi ITS fragment PCR results of the cotton samples at flowering and boll-setting stage(a)and boll opening stage(b)
2.2 DGGE指纹图谱和AM真菌基因测序结果分析
DGGE 图谱能够直观地反映出不同棉花品种土壤中AM 真菌群落结构的多样性。在DGGE 图谱中,不同位置的条带代表不同的AM 真菌类群,不同泳道同一横向位置的不同条带一般被认为是同一AM 真菌类群。花铃期共有24条不同条带,显示出AM真菌的种类,其中非转基因棉TH2 有18 个条带,转基因棉013011有20个;非转基因棉石远321有21个,转基因棉SGK321 有21 个。吐絮期共有27 条不同条带,显示出AM真菌的种类,其中非转基因棉TH2有26个条带,转基因棉013011 有25 个;非转基因棉石远321 有27个,转基因棉SGK321有26个。
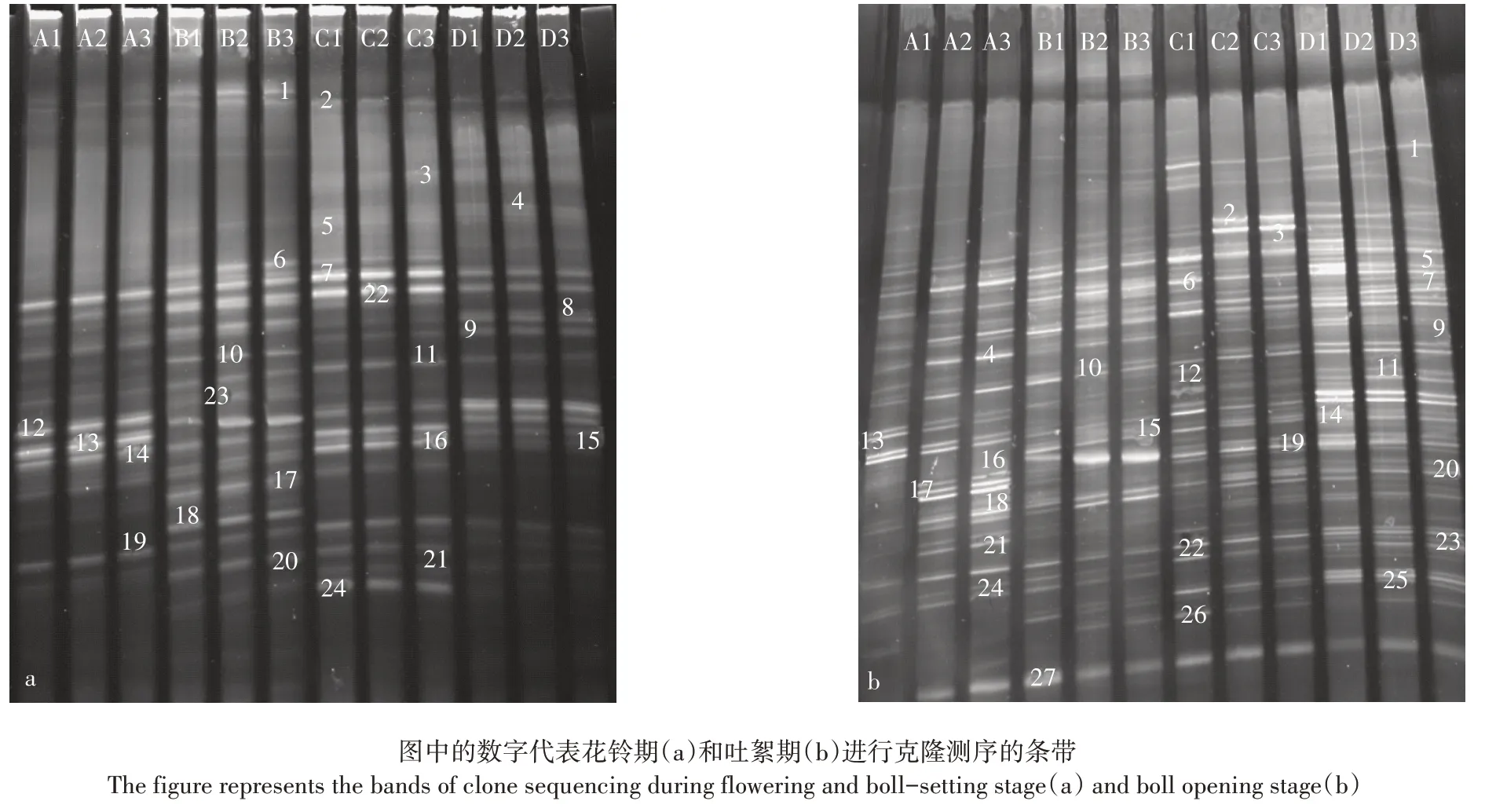
图3 转基因棉和非转基因棉在花铃期(a)和吐絮期(b)土壤中AM真菌群落结构DGGE图谱分析Figure 3 DGGE fingerprint analyses of AM fungi communities planted with GM verus Non-Gm cotton at flowering and boll-setting stage(a)and boll opening stage(b)
根据AM 真菌DGGE 指纹图谱(图3)选择DGGE胶上易于区分的条带进行克隆测序。在花铃期样品中选取24 条条带,在吐絮期样品中选取27 条条带,分别进行测序。测序结果登陆美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI),得到条带所代表的AM 真菌类型。分析所得序列的归属(表2、表3)以及样品的指纹图谱(图3)发现,花铃期测序的样品条带中两组对照TH2 和013011(抗旱)、石远321 和SGK321(抗虫)多为共有条带,均包含有Uncultured Glomus(球囊霉属)、Uncultured Glomeraceae(球囊霉科)、Uncultured Glomeromycota(球囊菌门)、Uncultured Rhizophagus(根孢囊霉属)和Glomus sp.,且亮度较高的条带大多为Uncultured Glomus(球囊霉属),因此Uncultured Glomus(球囊霉属)为共同优势属。对比TH2 和013011(抗旱)样品指纹图谱(图3)发现条带18 和条带24 为转基因棉013011(抗旱)特有条带,分别属于Uncultured Glo⁃mus(球囊霉属)和Uncultured Glomeromycota clone(球囊霉科);条带13 为非转基因棉TH2 特有条带,属于Uncultured Rhizophagus(根孢囊霉属)。对比石远321和SGK321(抗虫)样品指纹图谱(图3)发现条带13为转基因棉SGK321 特有条带,属于Uncultured Rhizoph⁃agus(根孢囊霉属);条带24为非转基因棉石远321特有条带,属于Uncultured Glomeromycota(球囊菌门)。未能在GenBank 收录相似的AM 真菌分类中找到与条带17 相似的分类。吐絮期测序的样品条带中两组对照TH2 和013011(抗旱)、石远321 和SGK321(抗虫)多为共有条带,均包含有Uncultured Rhizophagus(根孢囊霉属)、Uncultured Glomeraceae(球囊霉科)、Uncultured Glomus(球囊霉属)、Rhizophagus sp.、Uncultured Diversispora(多孢囊霉属)、Uncultured Glomeromycotina 和Uncultured Claroideoglomus(幼 套近明囊属),且亮度较高的条带大多为Uncultured Clo⁃mus(球囊霉属),因此Uncultured Clomus(球囊霉属)为共同优势属。对比TH2 和013011(抗旱)样品指纹图谱(图3)发现条带18为非转基因棉TH2特有条带,属于Uncultured Glomus(球囊霉属),条带15为转基因棉013011 特有条带,属于Rhizophagus sp.(根孢囊霉属)。对比石远321 和SGK321(抗虫)样品指纹图谱(图3)发现全为共有条带。未能在GenBank收录相似的AM 真菌分类中找到与条带16、20、21、23、24 相似的分类。

表2 花铃期序列鉴定和同源性分析结果Table 2 Sequence identification and homology analysis of flowering and boll-setting period
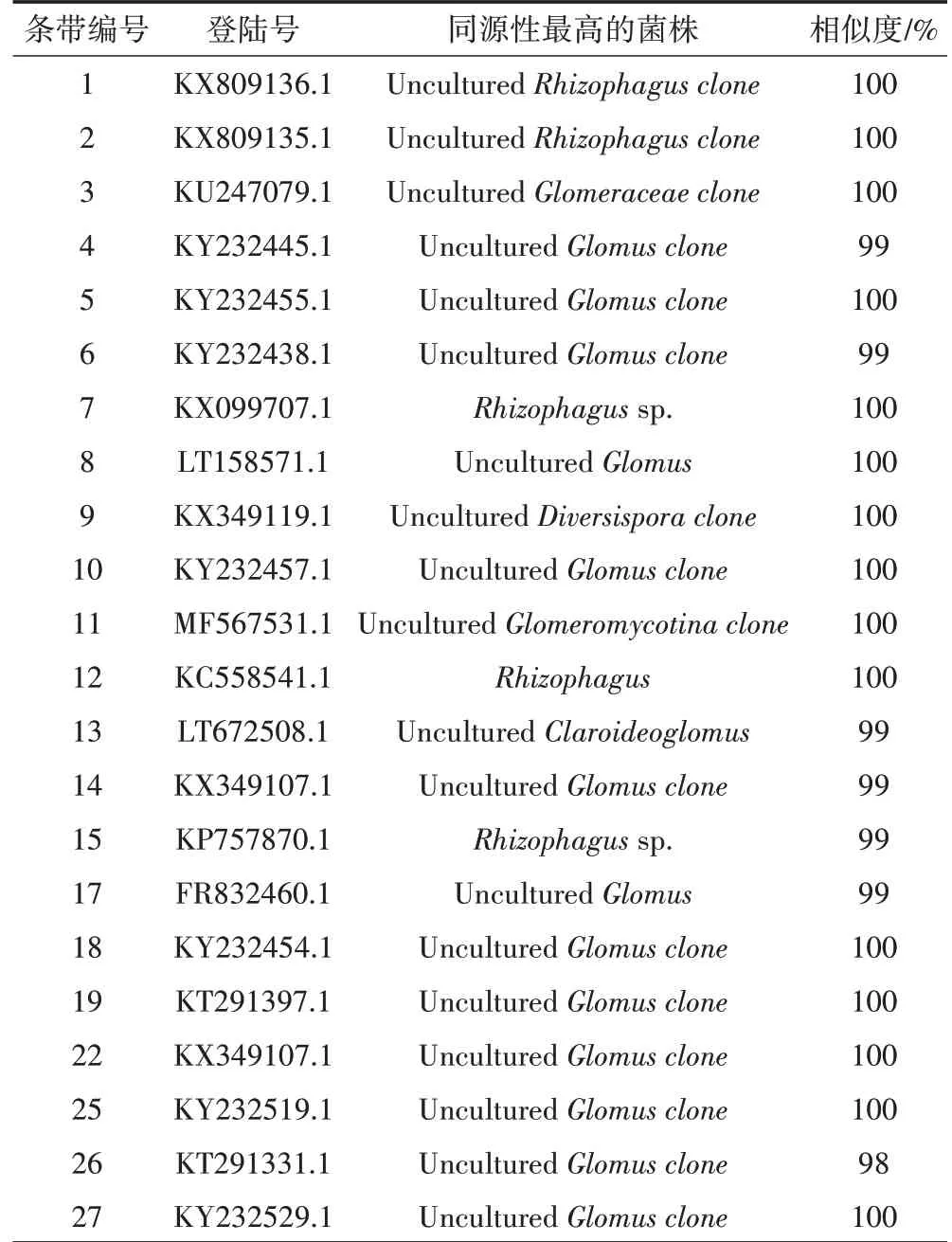
表3 吐絮期序列鉴定和同源性分析结果Table 3 Sequence identification and homology analysis of boll opening stage
2.3 土壤AM真菌多样性分析
AM 真菌的多样性指数是研究其群落物种数、个体数以及均匀度的综合指标。一般可用香农-威纳指数(H),均匀度(EH)和丰富度(S)表现。根据DGGE指纹图谱中每条条带的灰度比率对香农-威纳指数(H)、均匀度(EH)和丰富度(S)进行分析。
结果表明土壤中AM 真菌的香农-威纳指数仅在吐絮期石远321 显著低于SGK321,其余均未发现显著性差异;TH2、013011 和石远321、SGK321 的土壤AM 真菌丰富度在各生长期内均未发现显著性差异;土壤中AM 真菌的均匀度仅在吐絮期TH2 显著高于013011,其余均未发现显著性差异(表4)。
2.4 条带相似性分析及系统发育树分析
利用相似性矩阵数据,通过未加权算术平均对群法(The unweighted pair-group method with arithmetic averages,UPGMA)进行聚类分析。一般认为相似度大于0.60 就说明两个群体具有较好的相似性。本研究中花铃期4 种不同土壤样品的最小相似度为0.65(图4),在吐絮期4 种不同土壤样品的最小相似度为0.61(图4)。说明转基因棉与常规棉土壤中AM 真菌群落结构在花铃期和吐絮期差异不显著。
结果表明,花铃期测序序列与数据库中的已知序列相似度在99%~100%;吐絮期测序序列与数据库中的已知序列相似度在98%~100%。将测序获得的基因序列与相似序列对比,采用MEGA6 软件Neighbor-Joining 法构建系统发育树并进行系统发育分析(图5和图6)。花铃期样品根据进化上亲缘关系的相似度,条带1、3、4、6、7、9、10、11、12、14、21、22 形成了第1 类群;条带2、5、15 形成了第2 类群;条带8、16、18、19、20、23、24 形成了第3 类群;条带13 为第4 类群。由图6 可知,吐絮期样品根据进化上亲缘关系的相似度,条带4、5、6、7、9、10、18、19 形成第1 类群;条带1、2、3、12、15、17 形成第2 类群;条带8、11、13、14、22、25、26、27形成了第3类群。
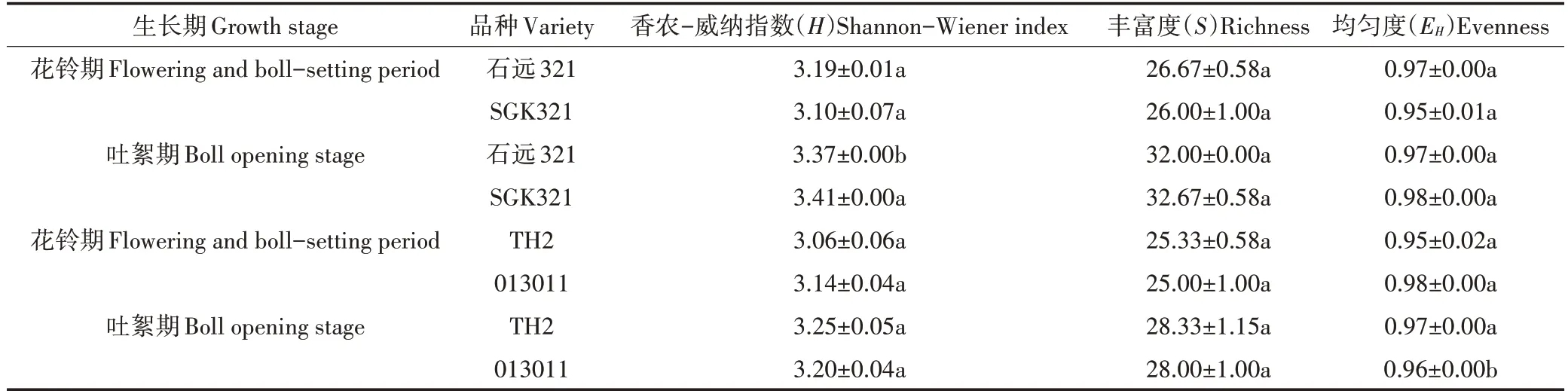
表4 DGGE图谱多样性指数分析Table 4 DGGE profiles diversity index analysis
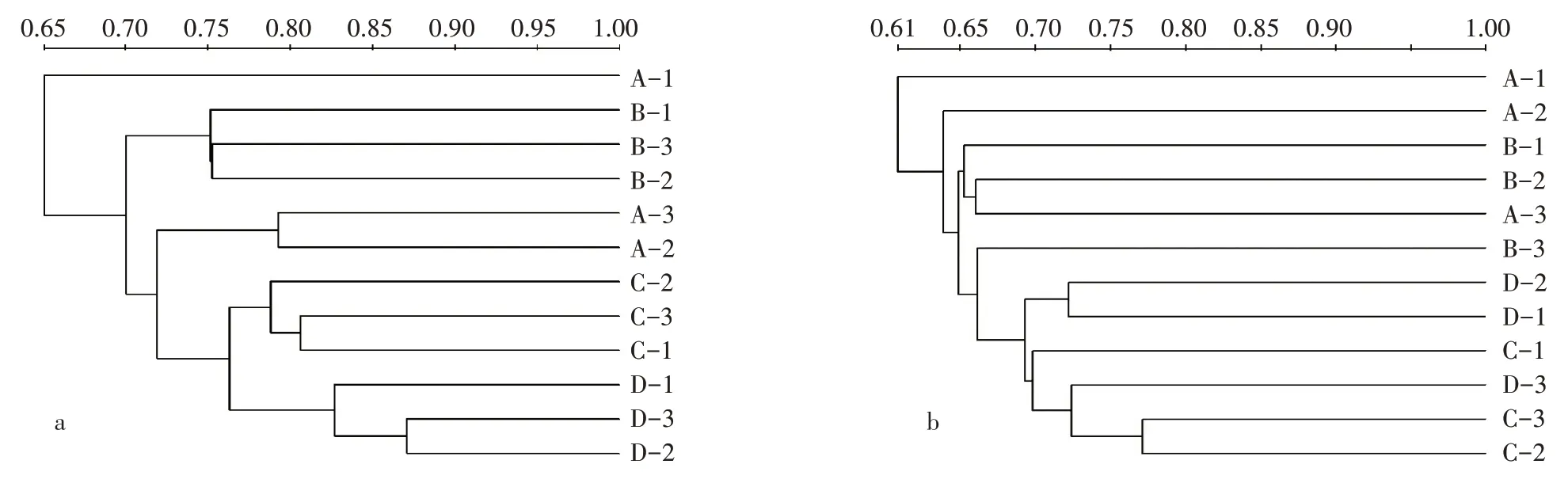
图4 转基因棉与非转基因棉在花铃期(a)和吐絮期(b)土壤样品AM真菌群落结构聚类分析Figure 4 Dendrogram of AM fungi communities at flowering and boll-setting(a)and boll opening stage(b)based on DGGE fingerprint
3 讨论
本研究采用了PCR-DGGE 技术分析转基因棉与非转基因棉土壤中AM 真菌群落结构,直观地反映出花铃期和吐絮期两组对照样品中AM 真菌群落结构的动态变化。结果发现同一时期转基因样品与非转基因样品之间DGGE 指纹图谱有很强的相似性,且各条带的亮度差异较小。一般认为条带的多少代表群落的多样性,条带的亮度代表微生物的量[14]。这说明同一生育时期AM 真菌的群落多样性和群落构成比较稳定,没有因为转基因棉花的种植而发生变化。而Castaldini 等[15]和Turrini 等[10]的研究表明转基因作物的种植会对AM 真菌造成负面的影响,Blackwood等[16]采用Biolog 方法发现转Bt 玉米种植的土壤中真菌的含量要高于常规玉米,这些结果与本研究结果存在一定的差异,可能是由于所选生境及植物的种类不同而造成的。本研究中,花铃期和吐絮期土壤样品中AM 真菌群落结构的最小相似度均大于0.6,优势属均为Glomus(球囊霉属),进一步证明了转基因棉的种植未对AM 真菌群落产生显著影响,这一结果与Knox等[17]的研究结果相似,其结果也表明了转基因植物的种植对AM 真菌无影响或影响甚微,Daniell 等[18]的研究结果表明Glomus(球囊霉属)是根际丛枝菌根真菌群落的重要组分,这也与本研究结果相一致。同时还可以发现两组棉花对照土壤样品并不是以转基因与非转基因分布在聚类图的上下两侧,而是根据品种的不同分在了聚类图的上下两侧,这说明品种间的差异要大于转基因与非转基因之间的差异。另外同一组对照之间转基因棉与非转基因棉聚类结果大多没有完全分开,可能是由于转基因的导入使棉花根系分泌物发生了一定变化,从而导致土壤中AM 真菌的生长发育过程受到了一定的影响[29]。
对土壤样品AM 真菌的香农-威纳指数、丰富度、均匀度研究发现吐絮期的数值均要高于花铃期,这种微生物数量与植株生长发育呈正相关的现象在其他文献中也有相似报道[20-21]。香农-威纳指数仅在吐絮期石远321 显著低于SGK321,均匀度仅在吐絮期TH2显著高于013011,这种显著性差异从整体结果来看相差甚微,且其余结果均未发现显著性差异。由于在自然环境中影响微生物群落多样性的因素很多,作物的生长期的不同、作物和土壤类型的差异、土壤养分因子、作物根系分泌物和农业管理等都会影响微生物的群落结构[22]。因此对于这种短暂的、不持续的差异性,本研究认为是由于生育期的不同和种植地点的差异引起的。这也与Meyer等[23]发现影响真菌群落结构的主要因素是作物生育期、种植地点及季节的观点相符合。本研究还发现,同一生育期同一特有条带在两组对照实验中出现在一组的转基因棉中同时也出现在另一组非转基因棉中,因此认为这种特异性并不是由于转基因棉的种植而引起的,可能是由于品种的不同而造成,Bouffaud 等[24]对玉米土壤微生物的研究也表明不同玉米品种的种植会对其根际微生物群落组成产生一定的影响。在不同的生育期两组对照实验的特异性条带差异较大。因此可以认为生育期的不同对土壤AM 真菌的群落结构有一定的影响[25],类似地,Liu 等[26]和Guadarrama 等[27]的研究也认为季节变化以及生育期的不同对AM 真菌群落结构有较大的影响。从系统发育树结果分析可知吐絮期AM 真菌的类群与花铃期相比发生了变化,这与本研究认为AM 真菌的类群差异是由于生育期的不同而造成的相一致。
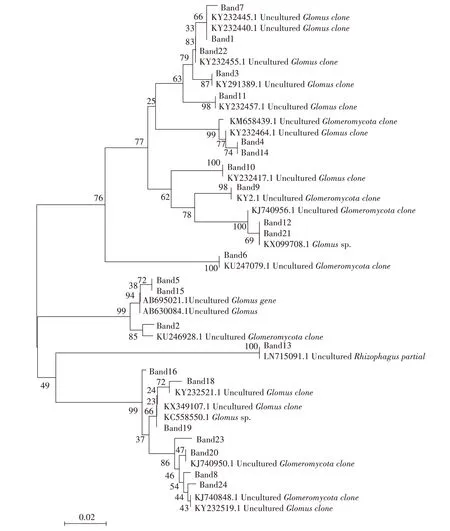
图5 花铃期系统发育树Figure 5 Phylogenetic tree of flowering and boll-setting period
任馨[28]对转Bt 基因水稻种植的土壤研究发现,转Bt 基因水稻的种植没有对土壤中的AM 真菌造成影响,刘华等[29]对多年种植转基因棉的研究也未发现转基因的种植对土壤微生物群落结构造成影响。本实验室其他研究者用不同方法对与本研究相同试验地种植的转基因棉与非转基因棉土壤中酶活性、养分含量、微生物群落、细菌群落以及古菌群落结构进行研究,其研究结果均未发现转基因棉的种植会对以上指标造成显著性的影响[30-34]。这也与本研究结果认为转基因棉的种植并未对土壤AM 真菌群落结构造成显著性影响相一致。另外,本研究检测出的大多数AM 真菌属于不可培养微生物,且大部分分类地位仍未知,要确定其具体种属和功能特性还需要进一步研究。
4 结论
(1)转基因棉与非转基因棉土壤AM 真菌DGGE指纹图谱在同一生长时期多为共有条带,同一生长时期土壤AM 真菌群落结构相似性较高,花铃期和吐絮期优势属均为Glomus(球囊霉属)。
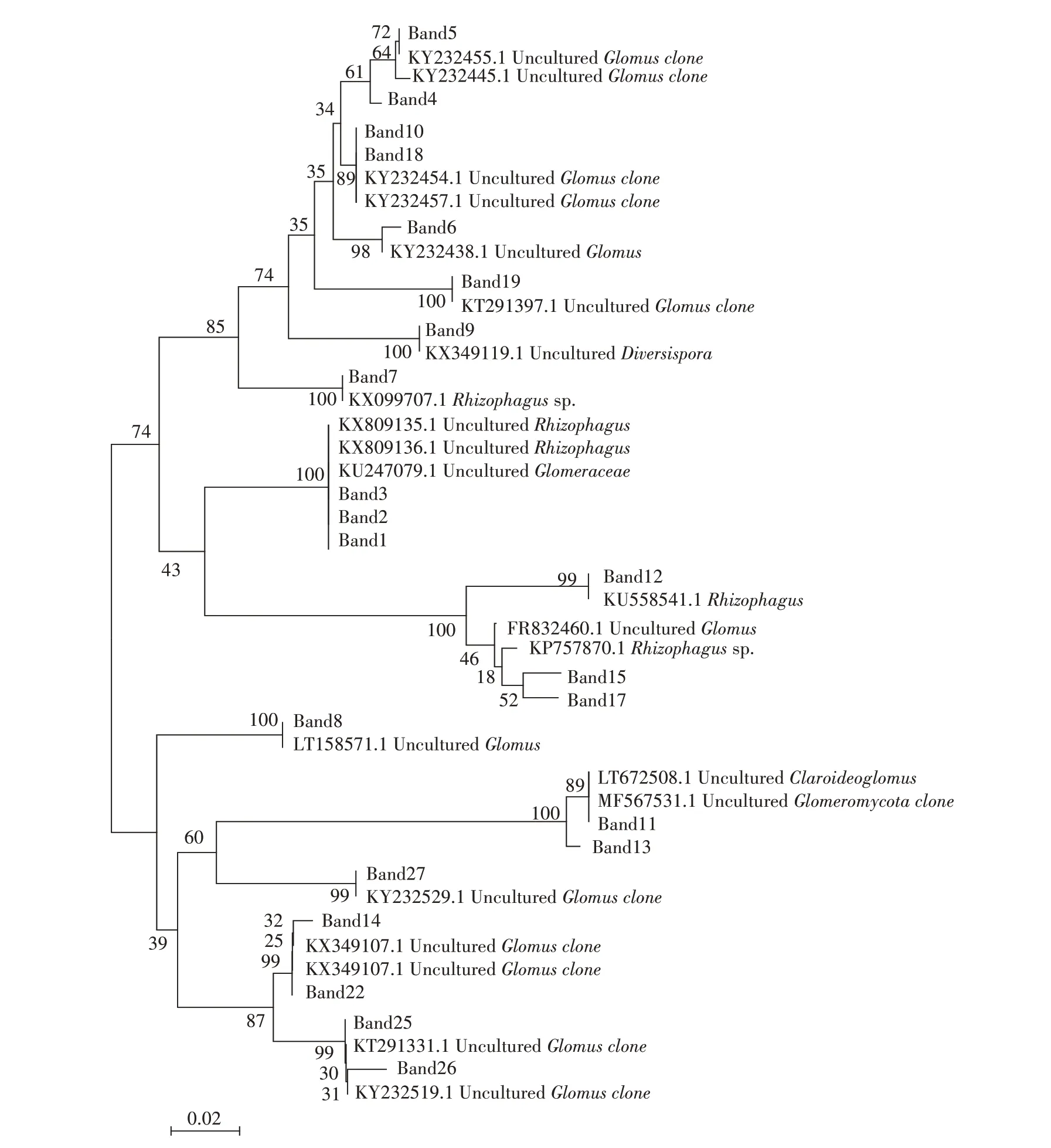
图6 吐絮期系统发育树Figure 6 Phylogenetic tree of boll opening stage
(2)转基因棉与非转基因棉的种植对土壤AM 真菌香农-威纳指数(H)、均匀度(EH)和丰富度(S)几乎没有产生显著性影响。
因此,本研究认为转基因棉的种植对土壤AM 真菌群落多样性没有产生显著影响。
