一种基于极大特征点的三维椎骨分割方法
2019-03-02檀结庆
王 浩,邵 堃,霍 星,杨 鹏,檀结庆
一种基于极大特征点的三维椎骨分割方法
王 浩1,邵 堃1,霍 星2,杨 鹏1,檀结庆2
(1. 合肥工业大学计算机与信息学院,安徽 合肥 230000;2.合肥工业大学数学学院,安徽 合肥 230000)
椎骨的精确分割对于椎骨形态学研究和脊柱疾病的诊断和治疗有重要意义。通过对正常人脊柱CT图序列的变化规律进行研究,提出了一种基于CT图像序列并利用椎骨面积变化规律进行分割的椎骨分割算法。该方法通过对预先处理后的CT图像序列进行椎骨区域面积统计,找出用于分割的显著极大特征点,并利用连续图像相似性筛选出椎骨实际分割点,最后从序列中提取图像并进行三维重建。实验表明,该算法对正常人体腰椎和胸椎下部的椎骨CT图像序列有良好地分割效果,自动化程度较高。对脊柱形态学研究和矫正手术模拟有重要意义。
CT图像序列;椎骨分割;面积变化规律;三维重建
随着计算机图像技术的不断发展,当今社会数字图像处理技术在各个领域都有重要地应用,展现在医学领域的应用也越来越广泛。医学影像设备的不断升级,三维造型技术的不断进步,数字诊疗技术的不断提升,都离不开医学图像处理技术的发展。如今,医学图像处理已成为计算机科学和生物医学工程领域研究的热点。
椎骨分割一直是医学图像处理领域中研究的重点和热点。一个良好的椎骨分割结果对人们研究椎骨形态、计算机辅助诊断及治疗等都具有重大的意义[1]。但是,由于脊柱空间结构较为复杂、各椎骨相互勾连,存在较大的局部噪声[2],使得椎骨分割成为一项极具挑战性的任务,同时也吸引了大批研究者从事相关问题的研究。
近年来,随着三维手术导航技术的开发与应用,如何从整体三维脊柱模型中快速分割出完整的单块脊椎骨并生成三维模型也成为研究热点之一。
当今医学图像分割的方法主要有:基于图像、纹理和边缘3种[3]。目前椎骨分割方法大致有:基于阈值和区域增长的分割方法[4];基于主动轮廓模型检测椎骨区域的分割方法[5];基于图割与均值漂移算法,利用椎骨中心线进行分割的方法[6];基于深度学习的椎骨定位与分割方法[7];基于统计模型和刚性配准的分割方法[8];基于标准脊柱边缘轮廓检测框架的分割方法[9]等。可将上述分割方法归为2类:①从CT或MR三维图像的矢状面入手,主要集中于椎体的定位、识别与分割;②从主要轴心面入手,在二维图像上将椎骨区域分割出来。其中,第一类方法得到的分割结果往往不够精确,椎骨模型的完整性较差;第二类方法虽然在二维图像上有较好的区域分割结果,但往往没有将椎骨进行分离。
由此可见,目前椎骨分割方法,仍存在以下不足:①所用的算法和数据过于复杂,导致时间复杂度很高,效率较低;②实现分割的限制条件较多,例如只能分割腰椎区域的椎骨;③自动化程度较低,需要的人工干预较多。因此需要对分割方法进一步进行优化[3]。
通过研究人体脊柱CT图序列,发现在正常脊柱图像序列中,椎骨区域面积的大小呈现周期性变化的规律。基于此本文提出了一种从正常人体脊柱CT图序列中快速分割完整椎骨图像的方法,利用正常脊柱的CT断层扫描图,经过预处理去除大部分干扰因素后得到二值图像;然后对预处理后图像序列进行面积统计和数据处理,并根据面积变化曲线寻找其波谷特征点,利用前后相邻图片的面积相似度检测及筛选这些特征点;最后得到椎骨分割点并利用分割点提取图像数据进行三维重建,得到单块脊椎骨模型。
1 分割算法
1.1 流程、算法概述
本文的分割方法分为预处理、椎骨区域面积检测、波谷特征点提取与筛选和单块椎骨图像提取。4个步骤为:①将CT图像转换成bmp图像并进行基础处理,凸显椎骨部分的图像特征;②计算图像序列中每张图像的椎骨区域的面积,生成面积变化曲线,提取曲线上的波谷特征点并进行筛选,确定最终的椎骨分界点;③利用分界点在图像序列中进行图像提取,得到单块椎骨的图像序列;④进行三维建模,得到单块椎骨模型。
1.2 图像预处理
由于CT图像不易直接进行处理,本文对DICOM医学图像的格式和操作进行了调研与分析[10-13]并确定了处理方法。首先将CT图转换为容易处理的bmp图像,同时将一些参数以文本的形式保存下来。
然后对bmp灰度图像序列进行二值化,以增强骨组织区域特征,为后续分割提供基础,并对图像进行中值滤波,其选取方法为
其中,为以当前像素为中心的滑动窗口;和均为中的相对像素坐标;和为图像中实际的像素坐标;为映射到滑动窗口的像素值;为图像的实际像素值。滤波原理是先将滑动窗口覆盖的图像中的像素按照大小进行排序并得到中值,然后让窗口中心位置的像素等于计算出的中值。中值滤波对椒盐噪声的抑制效果好,在抑制随机噪声的同时能有效保护边缘,减少模糊。中值滤波窗口大小应为奇数,本文选用9×9的窗口。图1为图像进行过预处理的结果。
图1 经过中值滤波后得到的图像
为了去除大部分由肋骨带来的干扰,中值滤波后,使用矩形框将椎骨所在区域框选出来,完成预处理操作。
1.3 极大特征点提取
经过预处理可以得到一系列包含椎骨主体的二值图像,并对其进行观察,研究发现正常的人体脊柱表现的变化规律为:图像序列中的椎骨骨质区域的面积呈现波浪形变化,并且当图像处于两块椎骨过渡的位置时,其中骨质区域的面积在相邻几张图片中是最小的。利用此规律,可以在面积变化曲线上寻找波谷点作为极大特征点,其包含了所有的椎骨分割点;接下来运用相邻图像间的相似性,从特征点中筛选出2块椎骨的分割点;最后,以该分割点为界,从整体图像序列中提取出单块椎骨的图像,并重建成三维模型。
1.3.1 骨质区域面积计算
在含有椎骨的二值图像中,白色区域即为骨质区域,但由于骨质区域的不规则形状,无法直接计算其面积,但可以通过统计像素灰度值为255的像素点个数作为骨质区域的面积值。本文以此计算图像中骨质区域面积的大小。
通过遍历序列中的每一幅图像,计算图像中白色像素点的个数,最终得到所有图像的骨质区域的面积值。图2是正常脊柱CT图序列得到的脊柱面积变化曲线。
1.3.2 计算曲线波谷特征点
经过对椎骨面积变化曲线的观察和验证可知:所有的椎骨分割点都处在椎骨面积变化曲线的波谷点上。曲线上的波谷特征点可表示为
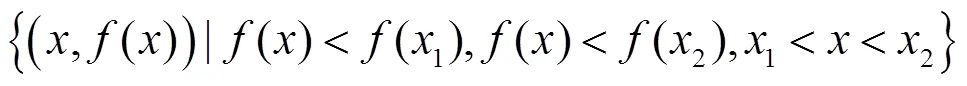
其中,为曲线上的横轴坐标;()为面积值,波谷特征点即面积值小于其前后面积值的那些点。
由于存在噪声,定位不准,对寻找波谷特征点有一定干扰,为此设置了一个步长间隔,代表2个波谷点之间最小的距离。初步过滤掉一些干扰点,加快计算的速度。图3是识别波谷点的结果。
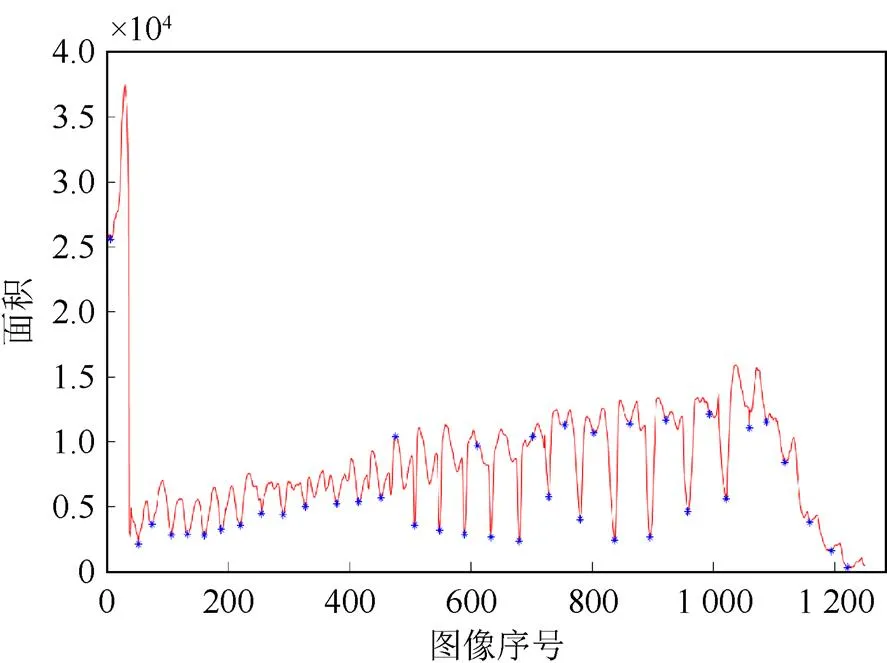
图3 椎骨面积变化曲线中寻找波谷点
1.3.3 筛选波谷特征点
由于并不是所有波谷点都是椎骨分割点,所以需要对波谷特征点进行筛选,为此采用了一种基于面积相似度的方法[14]。该方法通过比较每个波谷点代表的图像b和其前后相邻图像a和c中的椎骨面积,来检测波谷特征点是否为最终的椎骨分割点。步骤如下:
(1) 根据图像序列的间隔距离大小,确定需要比较的3张图像之间的图像间隔数量。若图像间隔距离大于1,则取波谷点前后相邻的3张图像进行比较;若间隔小于1,则取波谷点前后相隔一幅图像的3张图像进行比较,其原因是若图像之间的间距太小,则前后相邻图像之间的相似度很高,不利于筛选。然后,分别计算图像a和c中椎骨区域area_a与area_c和图像b的椎骨区域area_b的重合区域的大小,得到重合区域的大小分别占图像a和c的椎骨区域的比例,即
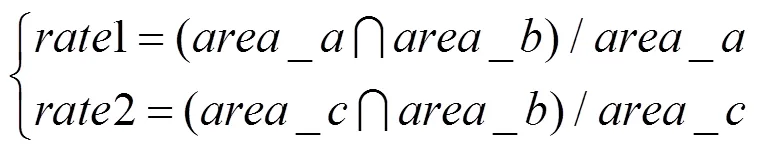
(2) 得到2个比例后,利用阈值来判断1和2是否符合条件,低于说明此处图片相似度低,处于2块椎骨的过渡位置,则需要保留此点;反之说明相似度高,还在一块椎骨内部,需去除此点。这里阈值是根据图像之间的间距来决定的,经测试发现,2幅图像间隔越小,值越大才能得到良好的分割效果,根据多组图像的分割测试结果,拟合了取值的公式,其中,为序列中图像之间的间隔距离,即
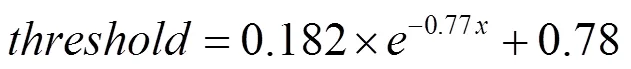
图4是最终得到的分割点的示意图。

图4 波谷点筛选后的结果
1.4 图像提取和三维模型重建
得到图像序列中椎骨之间交界处的分割点后,利用分割点抽取分割点之间的椎骨图像区域,最后使用Marching-Cubes算法,对分割后提取出的图像序列进行三维重建,得到分割后的三维椎骨模型。
2 实验结果
2.1 正常椎骨分割情况
向医院申请获取了几组DICOM切片数据,并选取了相对正常脊柱的一组数据,利用本文方法进行分割操作,图5是分割所得的结果。对分割后的图像和原始图像分别进行三维重建,并将单块椎骨模型重新组合成完整脊柱,得到了分割结果与实际结果的对比。
图5(a)中每种颜色为一个分割结果,每2条直线之间是一块椎骨。从建模的结果来看,发现在胸椎上部T6之前,分割的效果不是很理想,分割点将一块椎骨分成了上下两部分或者未找到正确分割点导致两块椎骨被看成一块。但在胸椎下部和腰椎区域,分割的效果明显更好,能够正确找到分割点并将椎骨完整分割出来,其原因如下:由于在胸椎上部区域,各椎骨之间间距很小,且有肋骨干扰,所以仅靠面积相似度难以准确判断波谷点是否为椎骨分割点,而胸椎下部和腰椎区域,各椎骨之间的间距变大,且肋骨干扰逐渐减小,所以面积相似度可以作为判断是否为分割点的依据。
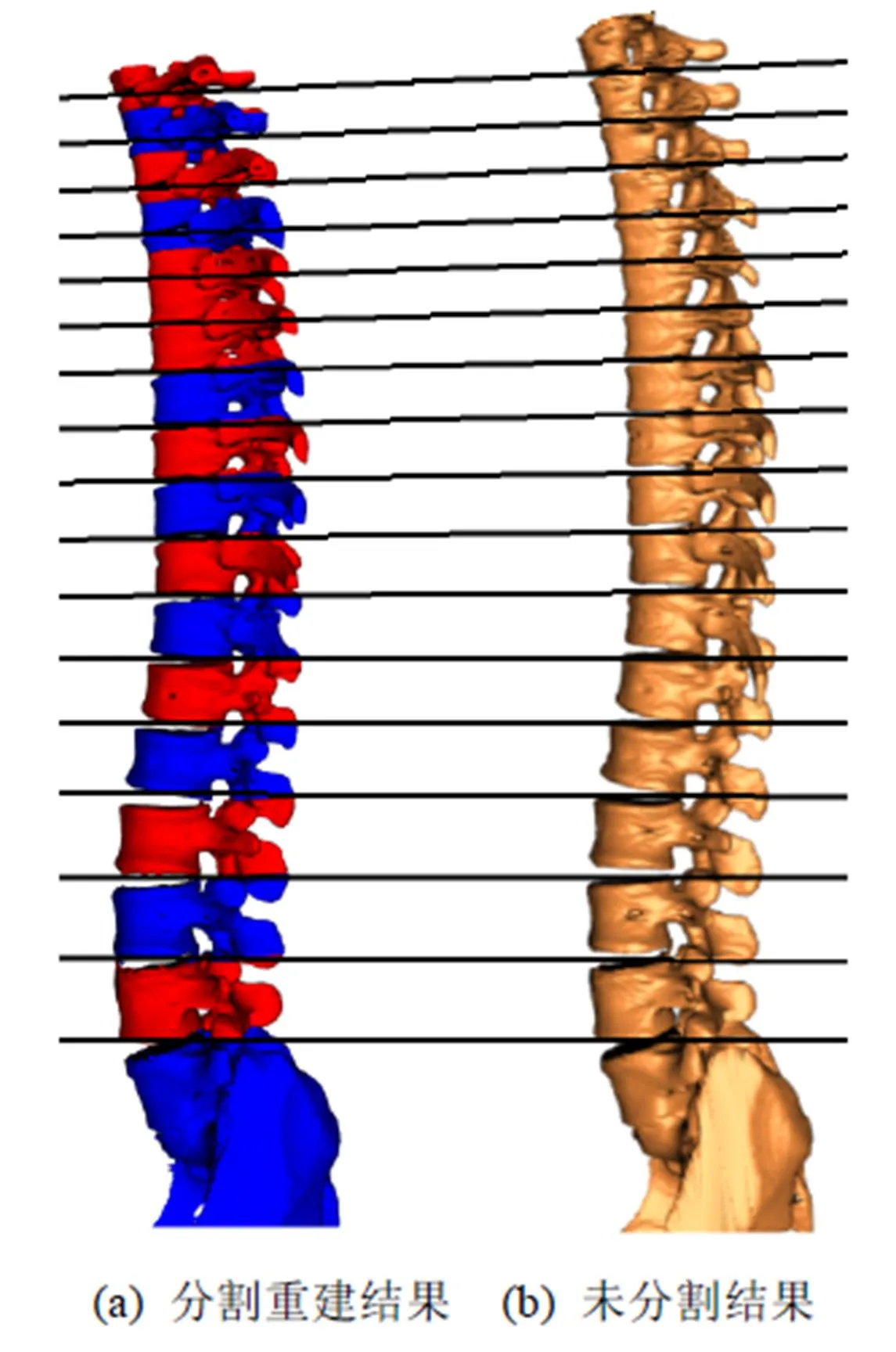
图5 正常脊柱的分割建模效果
2.2 在椎骨数据集上的分割情况
B. Glocker等人为本研究提供了一些开放的脊柱CT图像数据集,其图像的清晰程度较从医院获取的图像相比稍有逊色,但不影响使用此方法进行分割。图像效果对比如图6所示。

图6 2种数据图像质量情况对比
开放数据集中包含138位病人的部分脊柱序列图,从中选取了45组相对正常的脊柱序列图并利用本文方法对其进行了分割,并按照数据中有无腰椎数据将45组数据分为2组,一组含有腰椎,另一组不含腰椎。并按照每组数据中实际含有椎骨数量和实验分割出的椎骨数量对实验后的数据进行统计。图7和图8是2组数据的统计直方图。
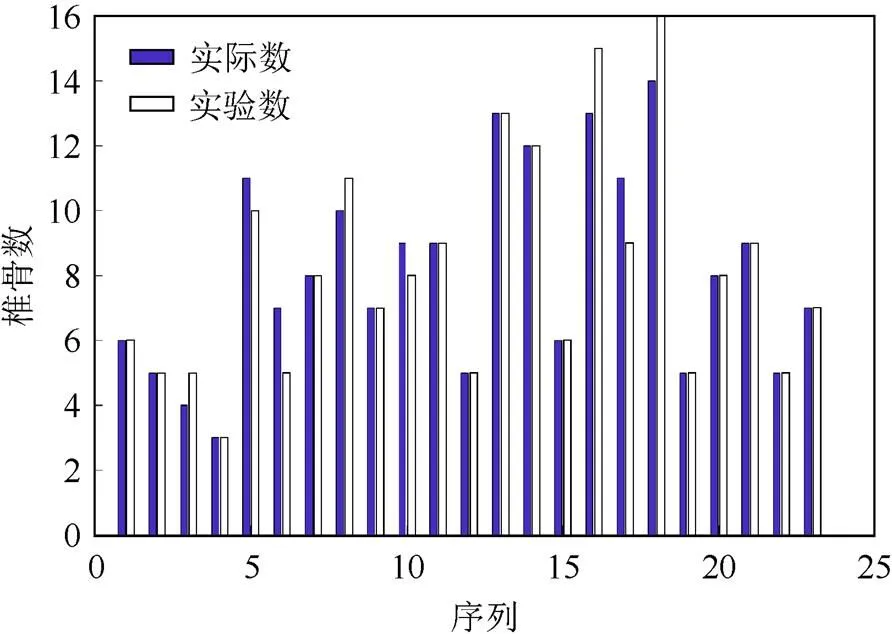
图7 含有腰椎的实验组分割情况
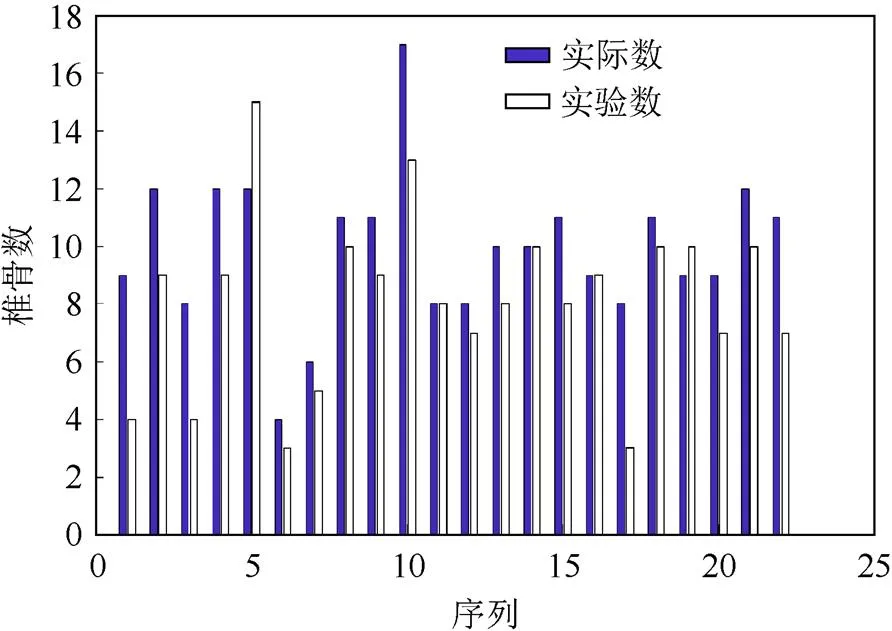
图8 不含腰椎的实验组分割情况
图7和图8中蓝色代表实际的椎骨数量,白色代表实验分割出的椎骨数量。从图中可以看出,含有腰椎的实验组分割椎骨数量与实际椎骨数量差异不大,而不含腰椎的实验组差异较大。本文用皮尔逊相关系数量化了2组实验组中实际含有的椎骨数量和分割出的椎骨数量之间的相关性,含有腰椎及不含腰椎的实验组相关系数分别为0.961 9,0.759 0。由此可见,含有腰椎的比不含腰椎的实验组的分割情况要好得多。
2.3 在侧弯脊柱上的分割结果
为了验证本方法的适用范围,本文挑选了脊柱侧弯患者的CT图序列,此患者在T4-L2部位的脊柱侧弯比较严重。利用本文方法对其脊柱CT序列进行了分割,并将分割结果与未分割的相应部分脊柱进行了对比,结果如图9所示。

图9 2种数据图像质量情况对比
图9(a)是分割结果,每种颜色代表一个分割结果,图9(b)是未分割的整体脊柱。由图可知,本文的方法在弯曲严重的脊柱区域分割效果不理想,没有分割出一块完整的椎骨,只有在相对位置正常的L2-L4腰椎部分,才分割出了相对完整的椎骨。其原因是:弯曲脊柱表现在二维CT图上的形状复杂多变,且面积变化规律不明显,因此只有在位置相对正常的椎骨处才能得到良好的分割效果。
2.4 与同类分割方法的比较
经文献查阅,发现文献[3]与本文方法和目的均较相似。文献[3]第一步首先对包含椎骨的CT图像进行聚类并进行分割,然后对聚类后的区域进行遍历,遍历时分为图像内遍历和间遍历,图像内遍历得到单张图像中的椎骨区域,利用图像间遍历来确定椎骨的边界;第二步将标记后的椎骨区域重建为3D粗模型,然后利用3D插值,将粗模型细化得到最终的分割效果。
与本方法相比,文献[3]在第一步对图像进行处理时,聚类并进行3D图像椎骨分割的时间复杂度较高,随后进行3D插值时,算法时间复杂度也较高;而本方法仅对图像进行了基础的二值化以及滤波操作,然后利用像素统计的方法进行单块椎骨图像抽取,并且重建后的三维数据不需要进一步操作,所以本方法在时间效率上有一定优势。
因文献[3]将图像进行了聚类,并以聚类区域为操作单位,而本文方法以整张图像作为操作单位,因此在椎骨分割的精度、完整性以及方法适用性上,文献[3]的方法较本文方法更好。
3 结束语
本文提出的是一种基于波谷特征点的椎骨自动分割方法,利用椎骨区域在CT图像序列中的变化规律,用检测面积变化曲线中波谷特征点的方式对椎骨分割点进行定位。经测试发现,该方法能够有效地应用于相对正常的脊柱CT切片图序列并对其中的椎骨进行提取,对于正常脊柱的胸椎下部以及腰椎部分有较好地分割效果,能够分割出完整的单块脊椎骨图像并生成三维模型。相比于传统的手工分割方法,本文方法大大提高了分割的效率且避免了繁琐的劳动,能够良好地分离正常脊柱中的椎骨。分割出的图像经三维重建后的模型表面光滑且数据量少,在脊柱分析、手术模拟、有限元分析等方面有着良好地应用价值。
但是本文方法在正常脊柱的胸椎上部以及侧弯脊柱中的弯曲部分上分割效果较差,原因是这些区域中椎骨区域形状复杂,导致前后椎骨面积变化不够明显,仅从面积上难以确定椎骨间的分割点。并且本文方法只能在竖直方向上分离椎骨图像,对于倾斜状态的椎骨不能完整地提取。
针对本文方法存在的不足,在后续的研究中,将拟利用空间邻域算法,以得到的椎骨分割点为基础,对3D图像进行处理,以提高椎骨分割的完整性以及对弯曲脊柱的适用性。
(感谢:B. Glocker等人为本研究提供了一些开放的脊柱CT图像数据集!)
[1] BAZILA, MIR A H. Segmentation of lumbar intervertebral discs from spine MR images [C]//2014 Innovative Applications ofComputational Intelligence on Power, Energy and Controls with Their Impact on Humanity (CIPECH). New York: IEEE Press, 2014: 85-91.
[2] ZHAO L K, ZHENG S Y, WEI H T. Adaptive active contour model driven by global and local intensity fitting energy for image segmentation [J]. Optik - International Journal for Light and Electron Optics, 2017, 140: 908-920.
[3] COURBOT J B, RUST E, Monfrini E. Vertebra segmentation based on two-step refinement [J]. Journal of Computational Surgery, 2016, 4(1): 1.
[4] KIM Y, KIM D. A fully automatic vertebra segmentation method using 3D deformable fences [J]. Computerized Medical Imaging and Graphics: the Official Journal of the Computerized Medical Imaging Society, 2009, 33(5): 343-352.
[5] ATHERTYA J S, SARAVANA K G. Automatic segmentation of vertebral contours from CT images using fuzzy corners [J]. Computers in Biology and Medicine, 2016, 72: 75-89.
[6] CHENG E K, LIU Y X, WIBOWO H, et al. Learning-based spine vertebra localization and segmentation in 3D CT image [C]//IEEE 13th International Symposium onBiomedical Imaging (ISBI 2016). New York: IEEE Press, 2016: 160-163.
[7] SUZANI A, RASOULIAN A, SEITEL A. Deep learning for automatic localization, identification, and segmentation of vertebral bodies in volumetric MR images [EB/OL]. (2015-03-18)[2018-06-29]. https://doi.org/10.1117/12.2081542.
[8] RASOULIAN A, ROHLING R, ABOLMAESUMI P. Lumbar spine segmentation using a statistical multi-vertebrae anatomical Shape+Pose model [J]. IEEE Transactions on Medical Imaging, 2013, 32(10): 1890-1900.
[9] KOREZ R, IBRAGIMOV B, LIKAR B. A Framework for automated spine and vertebrae interpolation-based detection and model-based segmentation [J]. IEEE Transactions on Medical Imaging, 2015, 34(8): 1649-1662.
[10] LI C, MANTELL S, POLLA D. Design and simulation of an implantable medical drug delivery system using microelectro-mechanical systems technology [J]. Sensors and Actuators A Physical, 2001, 94(1): 117-125.
[11] ESCOTT E J, RUBINSTEIN D. Free DICOM image viewing and processing software for your desktop computer: What’s available and what it can do for you [J]. Radiographics, 2003, 23(5): 1341-1357.
[12] ROSSET A, SPADOLA L, RATIB O. OsiriX: An open-source software for navigating in multidimensional DICOM images [J]. Journal of Digital Imaging, 2004, 17(3): 205-216.
[13] CHUNG H COBXAS D, BIRDSELL L, et al. Automated segmentation of muscle and adipose tissue on CT images for human body composition analysis [C]// Medical Imaging 2009: Visualization, Image-Guided Procedures, and Modeling.Bellingham: International Society for Optics and Photonics, 2009: 72610K-72610K-8.
[14] 姚沛延, 赵宏安, 周明全, 等. 基于CT图像的脊椎骨组织分割[J]. 计算机工程与应用, 2009, 45(27): 237-239.
A Method of 3D Vertebra Segmentation Based on Maximum Feature
WANG Hao1, SHAO Kun1, HUO Xing2, YANG Peng1, TAN Jie-qing2
(1. School of Computer Science and Engineering, Hefei University of Technology, Hefei Anhui 230000, China; 2. School of Mathematics, Hefei University of Technology, Hefei Anhui 230000, China)
Accurate segmentation of vertebrae has become more and more important for the diagnosis and treatment of spinal diseases. After studying the change rule of some spinal CT image sequences of normal people, we proposed a new vertebral segmentation algorithm based on vertebral area change regulation. This method first calculates the area of vertebral region in the image after pre-processing, then finds out the significant features for segmentation, uses image similarity of continuous images to select the actual segmentation points, and finally extracts vertebral images from image sequence for 3D reconstruction. The innovation of the method is that a new segmentation algorithm based on the maximum feature and correlation of CT image sequence is proposed. The experimental results show that the algorithm has a good segmentation effect on the CT image sequence of lumbar vertebrae and the lower thoracic vertebrae of normal human bodies and it has a high degree of automation. This study is of great significance to the morphological study of the spine and the simulation of the scoliosis operation.
computed tomography image sequence; vertebra segmentation; area change regulation; three-dimensional reconstruction
TP 391
10.11996/JG.j.2095-302X.2019010040
A
2095-302X(2019)01-0040-06
2018-06-19;
2018-06-27
国家自然科学基金面上项目(61472466,61572167,61271123);国家自然科学基金青年项目(61502136)
王 浩(1994-),男,山东日照人,硕士研究生。主要研究方向为图形图像处理、软件工程。E-mail:2013214020@mail.hfut.edu.cn
霍 星(1979-),女,安徽合肥人,副教授,博士,硕士生导师。主要研究方向为图形图像处理等。E-mail:huoxing@hfut.edu.cn
