嗜碱盐单胞菌X3中异化型硝酸盐还原酶编码基因簇的功能验证及蛋白结构预测❋
2019-02-21李秋芬
王 越, 李秋芬, 张 艳
(1.上海海洋大学水产与生命学院, 上海 201306;2.农业部海洋渔业可持续发展重点实验室中国水产科学研究院黄海水产研究所,山东 青岛266071)
细菌的氮代谢是海洋生态系统中氮循环的主要驱动者之一[1],也是建立生物脱氮技术的主要依据[2],其中硝酸盐还原是氮代谢中最重要的步骤之一[3]。细菌的硝酸盐还原分为异化型和同化型,大多数脱氮细菌同时具有同化硝酸盐还原途径和异化硝酸盐还原途径[4],如海杆菌(Marinobactersp. )NY-4[5]。异化型的硝酸盐还原又包括硝酸盐异化还原成铵和微生物在厌氧条件下将硝酸盐还原为N2、N2O或NO的反硝化作用,这2种途径的第一步都是在异化型硝酸盐还原酶(Dissimilatory nitrate reductase,Nar)的作用下将硝酸盐还原为亚硝酸盐,进而进行后续步骤,可见异化型硝酸盐还原酶是生物脱氮过程中的关键酶[6-7]。据报道大肠杆菌(Escherichiacoli)和鸡肠血清沙门氏杆菌(Salmonellaentericaserovar gallinarum)中将硝酸盐转化成亚硝酸盐的酶是由基因簇narGHJI编码的[8-10],它们分别编码组成膜内Nar的α、β、γ、和δ四个亚基,其中narG编码膜内Nar的最大结构基因,通常有3 600 bp[11-12],其活性位点位于向着细胞质一面的细胞膜表面[13]。Pérezrodríguez 等[14]使用narG基因作为硝酸盐还原菌的诊断标记,来研究深海热液区的微生物群落。Oikawa等[15]通过构建narG的缺失变体确定深海脱氮菌假单胞菌(Pseudomonassp.) MT-1中该基因编码异化硝酸盐还原酶,但不同的菌中,构成该基因簇的基因也不尽相同,如:有的菌中narH代替了narY、narI代替了narV[16]。基因的不同也可能会影响该菌的异化硝酸盐还原酶的结构和功能。
本实验室筛选获得一株海水细菌X3,具有脱氮功能[17],后鉴定为嗜碱盐单胞菌(Halomonasalkaliphila),研究其脱氮特性和环境适应性,发现菌株X3可同时去除氨氮、亚硝酸氮和硝酸氮[18],但尚未对该菌株氮代谢路径的相关功能基因做进一步研究,也未见有针对Halomonasalkaliphila功能基因的相关报道。本文在前期对该菌的全基因组测序(GenBank登录号: cp024811)预测到其氮代谢通路中含有narGYJV基因簇的基础上,报告了对该菌株氮代谢途径中nar基因簇功能的验证及编码的Nar蛋白结构预测的结果,以期为下一步研究该菌的氮代谢机理和调控奠定基础。
1 材料和方法
1.1 实验材料和仪器
1.1.1 菌株与培养基 实验所用菌株为由本实验室从海水中分离纯化所得的嗜碱盐单胞菌X3,采用甘油冷冻法保存于-80℃冰箱[19]。
培养基:硝酸盐还原培养基,参照李洪鹏等的配方[17],并有所改进。具体如下:NH4Cl 0.5g,MgSO40.1g,NaH2PO40.2g,K2HPO40.5g,CaCO31g,FeSO40.1g,KNO31g,Fe3PO40.1g,葡萄糖1g,pH=7.8,灭菌海水1 000 mL。
1.1.2 主要药品与试剂 主要化学试剂均购于国药集团化学试剂有限公司,均为分析纯;所采用的试剂盒包括:GENMED Bradford 蛋白质浓度定量试剂盒、GENMED 细菌异化型硝酸盐还原酶活性比色法定量检测试剂盒、TIANamp Bacteria DNA Kit 细菌基因组DNA提取试剂盒、天根RNAprep Pure培养细胞/细菌总RNA提取试剂盒、荧光定量的试剂盒Takara One Step PrimeScript RT-PCR Kit(Perfect Real Time)等。
1.2 试验方法
1.2.1 酶活测定 首先在硝酸盐还原培养基中,菌株X3扩大培养至OD600(Optical density在600 nm处的吸光值)为0.4~0.8 (即1×107~2×107细胞/mL),然后用Bradford 蛋白质浓度定量试剂盒测定样品溶液中的蛋白质浓度,测定方法参照试剂盒的说明书,用96孔板微量测定。再运用细菌异化型硝酸盐还原酶定量检测试剂盒测定Nar的酶活,测定方法按照说明书中的酶标仪测定法,先按照操作步骤,利用试剂盒中的标准样品,建立纵坐标为吸光值OD540、横坐标为标准亚硝酸盐浓度的标准曲线,对样品进行测定后,根据标准曲线获得样品对应的亚硝酸盐浓度,进行下列公式的计算获得酶活。其中异化型硝酸盐还原酶的活性单位定义为37 ℃、pH=7.5 条件下,每分钟内能够还原1nmol硝酸盐所需要的酶量为一个活性单位,计算公式如下:
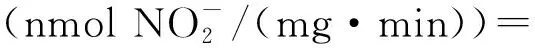
1.2.2 反转录荧光定量PCR(qRT-PCR) 以硝酸盐还原培养基培养菌株X3,运用RNA提取试剂盒提取该菌株的总RNA,用0.7%琼脂糖凝胶电泳和超微量核酸蛋白分析仪检测RNA的质量。引物选用NarG1F、NarG1R (见表1)。实时荧光定量PCR的反应体系采用25 μL:2×One Step RT-PCR Buffer III 12.5 μL,TaKaRa Ex TaqHS 0.4 μL,PrimeScript RT Enzyme Mix II 0.4 μL,引物(10 μmol/L) 各0.5 μL,Eva green 0.8 μL,模板1 μL(其中阳性对照为菌株X3的基因组 DNA,阴性对照为无菌水,实验组为总RNA),RNase Free dH2O 8.9 μL。反应时间为42 ℃,10 min;95 ℃,1 min;95 ℃,10 s;55 ℃,30 s。

表1 实验所用引物Table 1 Primers used in this study
1.2.3 生物信息学分析 运用DNAMAN翻译得到Nar四亚基的各氨基酸序列,通过ORF Finder (https://www.ncbi.nlm.nih.gov/ Structure/cdd/wrpsb.cgi) 在线预测得到Nar开放阅读框,然后运用在线软件SignalP 3.0 Server (http://www.cbs.dtu.dk/ services/ SignalP-3.0/) 对信号肽和分泌蛋白的有无进行预测;并运用TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM/) 预测氨基酸序列有无跨膜结构,用在线软件ProtParam tool (http://web.expasy.org/protparam/) 分析氨基酸序列组成以及等电点和分子量。运用在线软件NPS Network ProteinSequenceAnalysis (https://npsa-prabi.ibcp.fr/cgibin/npsaautomat.pl?page=/NPSA/ npsa_sopma.html) 进行蛋白质二级结构的分析[20]。最后运用Phyre2对四个基因编码蛋白的三维结构进行预测,并在蛋白质库PDB中检索同源蛋白[21];通过BLAST比对同源氨基酸序列,运用MAGE 5.0构建基于narG和narY编码蛋白的系统发育树。
2 结果
2.1 酶活验证结果
通过酶活性测试验证了narGYJV基因簇存在的可能性及其功能。培养菌株X3至OD600=0.7时,测得蛋白质浓度为0.013 μg/μL,且在OD540处的吸光度为0.082,根据图1标准曲线计算得样品中亚硝酸盐浓度为5.618 μmol/L,将数值代入1.2.1的酶活力公式进行计算,并根据对酶活单位的定义,得出菌株X3的异化硝酸盐还原酶为每克蛋白21.415 U。

图1 异化型硝酸盐还原酶酶活标准曲线Fig.1 Standard curve of the enzyme activity of the dissimilatory nitrate reductase
2.2 转录水平上的验证结果
对菌株X3的qRT-PCR验证结果 (见图2)显示菌株X3的RNA可以扩增得到目的片段,且CT值约为27,证明了narGYJV基因簇在X3中存在并具有表达活性。图3中的熔解曲线具有单一峰,证明所用引物具有特异性。

(a.阳性对照;b.样品;c.阴性对照。a.Positive control; b. Sample; c.Negative control.)
图2 qRT-PCR结果
Fig.2 qRT-PCR results
2.3 生物信息学分析
据全基因组测序获得的菌株X3的异化型硝酸盐还原酶基因簇中4个基因narG、narY、narJ和narV的序列(GenBank登录号:MG776025~MG776028),全序列翻译得到2 280个氨基酸,在该氨基酸序列中检测到4个蛋白功能域,1~1 246氨基酸属于NarG超家族,编码Nar的α亚基;1 261~1 752氨基酸属于DMSOR-beta-like 超家族,编码Nar的β亚基;2 061~2 279氨基酸 编码γ亚基,1 802~2 018氨基酸编码δ亚基(见图4)。经软件TMHMM Server和ProtParam tool分析,此氨基酸序列没有信号肽但有跨膜结构,等电点为5.86,分子量256 453.72 Da,带负电荷的氨基酸(Asp + Glu) 289个,占总蛋白的12.7%,带正电荷的氨基酸(Arg + Lys) 242个,占总蛋白的10.6%,蛋白序列中有36个半胱氨酸,不稳定系数是40.20,是一种不稳定蛋白,脂溶性为79.77,总平均亲水性为-0.341。NPS Network Protein Sequence Analysis预测的二级结构显示无规卷曲占37.59%,α螺旋占34.43%,延伸链占18.33% (见图5)。
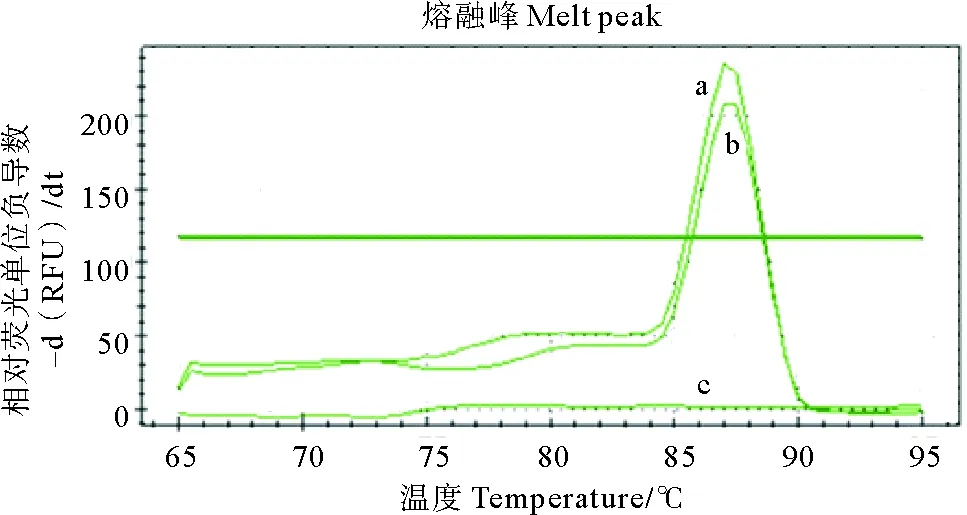
(a.阳性对照;b.样品;c.阴性对照。 a.Positive control; b. Sample; c.Negative control.)
图3 qRT-PCR熔解曲线
Fig.3 Melting curve of qRT-PCR
结合图6和表2分析NarGYJV的蛋白3D结构预测,Phyre2预测的NarG、NarY和NarV蛋白3D结构(见图6(a)、(b)、(c)),在PDB中同源关系最近的是1Q16,来自大肠杆菌三亚基与1Q16之间均具有6%~100%的覆盖率,54%~74%的一致性,各对应区域如图6(d)所示;图6(e)是NarJ的蛋白3D结构预测图,在PDB中预测的唯一同源性3D结构来自家牛(BosTaurus)的蛋白细胞色素B5(Cytochrome B5)(见图6(f)),但其覆盖率仅13%,相似性仅44%(见表2),可能因为PDB库中相应蛋白质数据不全导致比对结果不理想。

(查询序列 Query seq.;特定匹配 Specific hits;超家族 Superfamilies)
(蓝色:α螺旋;红色:延伸链;绿色:β折叠。Blue: α helix; Red: Extended strand; Green: β turn.)
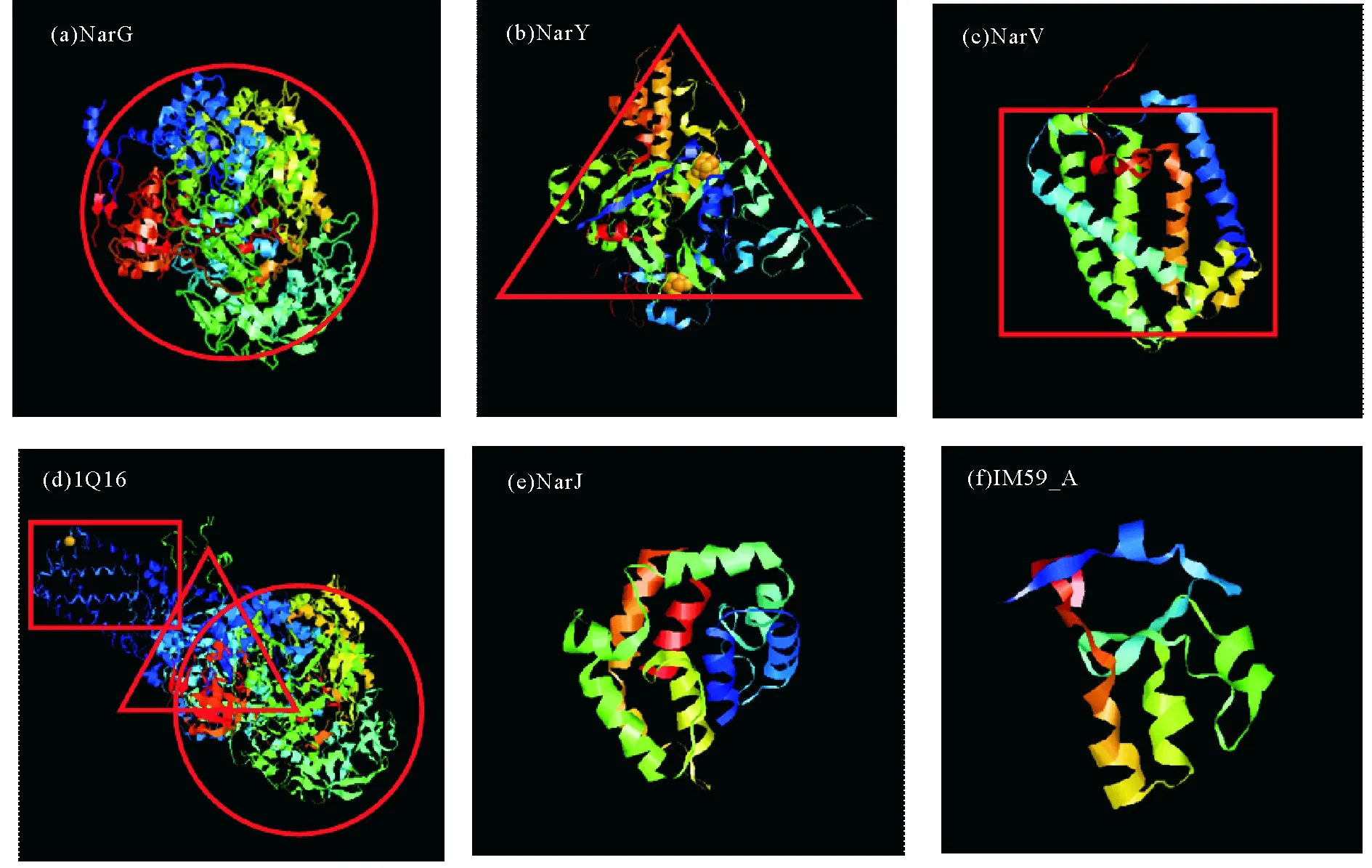
((a)运用Phyre2 预测的NarG的3D模型 3D model of NarG protein predicted using Phyre2;(b)运用Phyre2 预测的NarY的3D模型 3D model of NarY protein predicted using Phyre2; (c)运用Phyre2 预测的NarV的3D模型 3D model of NarV protein predicted using Phyre2; (d)从PDB中检索的与NarG、NarY和NarV同源性较高的3D模型 3D model with high homology to NarG、NarY andNarV retrieved from PDB; (e)运用Phyre2 预测的NarJ的3D模型 3D model of NarJ protein predicted using Phyre2; (f)从PDB中检索的与NarJ同源性较高的3D模型 3D model with high homology to NarJ retrieved from PDB.)

图6 预测的蛋白三级结构Fig.6 Predicted protein tertiary structure
2.4 系统进化树
基于NCBI网站的BLAST比对结果,根据narG和narY编码的氨基酸序列同源性分别构建了系统发育树,可见菌株X3的narG编码氨基酸与坎帕尼亚盐单胞菌(Halomonascampaniensis)等的遗传距离最近,同源性最高,其次是同属的其他菌,但与其他属的菌之间同源性较低(见图7)。由此可见,不同属的细菌中NarG具有相同的功能,但氨基酸序列差异性较大,同源性差;菌株X3的narY编码氨基酸与大肠杆菌中的硝酸盐还原酶的同源性较高(见图8)。综上可知,菌株X3的异化型硝酸盐还原酶Nar的四个亚基在系统发育树中的进化程度不同。

图7 基于NarG同源性的系统发育树Fig.7 Phylogenetic tree based on the homology of the NarG
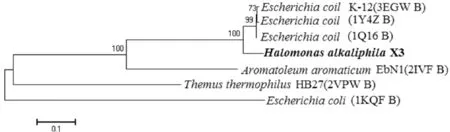
图8 基于NarY同源性的系统发育树Fig. 8 Phylogenetic tree based on the homology of the NarY
3 讨论
现发表的细菌硝酸盐还原系统包括膜结合呼吸性硝酸盐还原酶(Nar)[22],周质异化硝酸盐还原酶(Nap)[23]和细胞质同化硝酸盐还原酶(Nas)[24]三种系统。Nar锚定于细胞质膜上,主要参与细菌在厌氧条件下的硝酸盐呼吸通路,主要在厌氧条件下高水平表达;Nap位于细胞周质中,不仅具有硝酸盐的清除功能,还参与细菌厌氧呼吸下氨化作用和反硝化作用等;Nas位于细胞质中,生理结构比较单一,只参与硝酸盐的同化还原作用[25]。杨航等[26]发现全食副球菌(Paracoccuspantotrophus) ATCC 35512中同时存在Nar和Nap两种硝酸盐还原酶,在厌氧条件下Nar占主要地位,而好氧条件下Nap是主要存在形式。有的菌同时含有三种酶系统,只是在不同条件下,不同的系统发挥作用。硝酸盐还原过程因细菌不同而异,如大肠杆菌等仅使硝酸盐还原为亚硝酸盐;假单胞菌等能使硝酸盐或亚硝酸盐还原成氮(反硝化作用),而有的细菌则可以使其还原为亚硝酸盐和离子态铵[25]。菌株X3的 KEGG氮代谢途径显示其同时具有同化型和异化型硝酸盐还原途径,异化型反应的第一步依靠于Nar的催化,因此研究Nar的编码基因及其蛋白结构是下一步研究其酶的性质和氮代谢路径选择机制的基础。
本文通过测定异化型硝酸盐还原酶的酶活和qRT-PCR检测到目的片段的产生,验证了菌株X3中含有异化型还原酶Nar,但不同的细菌编码Nar的基因簇中所包含的基因也有所不同,全基因组测序预测菌株X3中nar基因簇包括narG、narY、narJ、narV四个基因,这与大肠杆菌和SalmonellaentericaserovarGallinarum的narGHJI不同[9-10]。对菌株X3narGYJV基因簇进行生物信息学分析,预测到其编码的四个蛋白结构域,其中1~1 246氨基酸属于NarG超家族,编码Nar的α亚基;1 261~1 752氨基酸属于DMSOR-beta-like 超家族,编码Nar的β亚基;2 061~2 279氨基酸编码γ亚基,1 802~2 018氨基酸编码δ亚基。检测到该氨基酸无信号肽,这与Nar是附着于胞质侧膜上的三亚基复合酶[27]描述相符。在蛋白质3D结构预测中,NarG、NarY和NarV的三级结构模型均与大肠杆菌中的硝酸盐还原酶A相似度较高,但NarJ相似度很低,需要进一步研究,这也与Blasco等主张的大肠杆菌的硝酸盐还原酶是由NarG、NarH、和NarI三亚基组成的,但第四个亚基NarJ是该酶连接钼因子的特殊结构,与编码该酶的活性部位不一致[28]。系统发育树分析可知,虽然NarG在许多菌属的细菌中存在,并执行相同的催化功能,但不同菌属间该基因的同源性不高,相同菌属间的遗传距离较近,其中与Halomonascampaniensis和盐单胞菌属(Halomonassp.) WN018的一致性均高达99%,但不同的菌属间该基因的同源性并不高;而NarY的高同源性主要表现在大肠杆菌中,可能是因为Nar中各亚基的进化程度不同,或与PDB数据库中与本菌Nar相关的基因及其蛋白相关的蛋白质结构数据不足有关,需要进一步研究,扩充PDB数据库。本文的研究结果也为进一步研究菌株X3及具有同类功能细菌的酶性质、氮代谢机理和氮代谢路径的选择机制奠定了基础。
4 结语
HalomonasalkaliphilaX3氮代谢通路中存在异化型硝酸盐还原酶Nar,它催化硝酸盐还原为亚硝酸盐。Nar由四个亚基构成,分别由基因narG、narY、narJ和narV编码。HalomonasalkaliphilaX3的异化型硝酸盐还原酶NarG的氨基酸序列在系统发育树中显示与同菌属的NarG同源关系较近,尤其与Halomonascampaniensis和Halomonassp. WN018的一致性均高达99%,且不同亚基的系统发育地位不同。研究结果为进一步深入研究嗜碱盐单胞菌的氮代谢通路选择和调控机制提供了依据。
