紫花苜蓿MsGAI的克隆、表达及遗传转化
2019-01-28张涵王学敏刘希强马琳温红雨王赞
张涵,王学敏,刘希强,马琳,温红雨,王赞
紫花苜蓿的克隆、表达及遗传转化
张涵,王学敏,刘希强,马琳,温红雨,王赞
(中国农业科学院北京畜牧兽医研究所,北京 100193)
【目的】DELLA蛋白属于GRAS家族,是赤霉素信号转导途径中重要的转录因子,负向调节GA转导途径。克隆获得紫花苜蓿,分析其基因生物信息学特征并预测蛋白结构域。明确紫花苜蓿组织表达特征及不同处理下的表达模式,构建该基因超表达载体并转入紫花苜蓿,以探究DELLA蛋白基因在紫花苜蓿赤霉素(GA)信号转导途径及胁迫条件下的作用机理。【方法】利用同源克隆的方法,从紫花苜蓿中克隆得到。利用生物信息学方法分析该基因的序列特征,使用MEGA7.0对MsGAI蛋白序列及同源序列进行多序列比对,构建同源物种间的系统发育树。利用实时荧光定量PCR检测紫花苜蓿各组织表达量以及在PEG、NaCl、GA、ABA和黑暗处理下,的表达变化。同时对转基因株系表达水平进行分析,选择表达量高、中、低株系(L5、L8、L11)分别进行PEG和NaCl处理,分析的表达变化。以pBI121为基础载体,采用双酶切-连接的方法构建植物超表达载体35S:MsGAI-gus。将重组载体转入农杆菌GV3101菌株中,以紫花苜蓿叶片为外植体,采用农杆菌介导的愈伤组织转化法转化紫花苜蓿,经PCR检测和GUS组织化学染色,得到转基因阳性苗。【结果】该基因序列包含有一个1 818 bp的开放阅读框,编码605个氨基酸。生物信息学分析结果显示,蛋白具有GRAS家族的典型结构域和保守区,其中包含N端保守结构域DELLA和TVHYNP,C端保守结构域SAW。多序列比对及系统进化树分析表明,该序列与其他物种的DELLA蛋白序列相似度均高达80%以上,将其命名为。该基因与蒺藜苜蓿亲缘关系最近,其次与鹰嘴豆、红三叶等双子叶豆科植物亲缘关系较近,与大麦等单子叶植物较远。实时荧光定量PCR分析表明,在紫花苜蓿各组织中均有表达,根中的表达量最高。经PEG、NaCl、GA以及ABA处理后,均有明显响应;黑暗处理显著抑制的表达。转基因株系经PEG、NaCl处理后,表达量均上调。对构建完善的35S:MsGAI-gus植物超表达载体进行双酶切检测,琼脂糖凝胶电泳显示,条带大小与预期一致。对转基因植株进行GUS组织染色验证,结果表明,阳性植株呈现蓝色,对照组为白色。对超表达载体携带的和序列进行PCR检测均呈阳性。【结论】紫花苜蓿DELLA蛋白基因的克隆和超表达载体构建成功,对逆境胁迫有响应。
紫花苜蓿;DELLA蛋白;赤霉素;表达分析;同源克隆;遗传转化
0 引言
【研究意义】紫花苜蓿(L.)是一种多年生优质豆科牧草,具有抗逆性强、适应性广、产量高、适口性良好、营养物质丰富等优点,享有“牧草之王”的美誉[1]。此外,紫花苜蓿根系发达,不仅具有固氮作用[2],还可以改善土壤结构、促进微生物活性和营养循环[3]。2011—2015年中国苜蓿干草年平均产量为7.1×108kg,消费量年平均1.42×109kg(数据来源:http://m.askci.com/),供需不平衡的严峻形势下,提高中国紫花苜蓿的产量已亟不可待。【前人研究进展】赤霉素(gibberllin,GAs)属于四环二萜生长因子,是种子萌发、胚轴伸长、茎的伸长、以及花的形成等植物发育过程中的基本调节剂[4]。在提高植物生物量方面发挥重要作用,DELLA蛋白是赤霉素信号转导途径中关键的负调节因子,参与GA信号传导并在其中发挥负调控的作用;赤霉素通过泛素蛋白酶降解的途径,来解除DELLA蛋白对其转导途径中的抑制作用。植物体内GA含量升高时,与赤霉素受体(Gibberllin Insensitive Dwarf1)GID结合形成一个二聚体,再与DELLA蛋白形成一个三元复合结构,从而引起DELLA构象发生变化,使SLY1元件能与GRAS域结合,被F-BOX蛋白识别,导致DELLA蛋白通过泛素-蛋白酶途径快速降解,从而使GA发挥正常效应[5]。DELLA蛋白的这种抑制作用在农业生产中起到有利作用,20世纪60—70年代育种学家选育出新的谷物品种,该品种共同特点为植株低矮且高产,比其他品种更具抗倒伏的作用,还具有耐风沙的重要特点[6-7]。这些新品种都是赤霉素合成或应答相关基因发生突变导致[8-10]。DELLA蛋白属于转录调控因子,具有保守的C端GRAS结构域,同时含有聚合Ser、Thr区域[11-13]和VHIID、SH2、SAW阻遏结构域[14]。在DELLA蛋白结构的中部,出现DELLA和TVHYNP 2个保守的酸性结构域和核定位信号NLS[15];当在转基因植物中表达时,DELLA-GFP融合蛋白定位在细胞核中[16]。拟南芥()中的5个DELLA蛋白存在功能冗余现象,其中,与对植物的营养生长和开花时间有明显影响[17];而与在种子萌发过程中发挥重要作用[18]。小麦()Rht-B1/Rht-D1和玉米()dwarf-8(d8)是拟南芥赤霉素不敏感的直系同源基因[19]。这些基因编码类似核转录因子和蛋白质,含有类似于SH2结构域,表明磷酸酪氨酸可能参与赤霉素信号传导。含有等位基因的水稻()植株对赤霉素的反应降低并且出现矮化的现象,表明突变的同源物可广泛增加作物产量[20]。突变会影响GA接收以及随后的信号转导,但不会引起GA缺乏。先前对玉米d8和小麦突变体的生理和遗传分析表明,这些突变都属于功能获得性突变[21],降低对GA的反应同时增加内源GA水平[22]。Fu等[23]和Dill等[24]指出过表达会导致植株低矮以及GA含量降低,并且这种效应的强度与转基因表达的水平相关。Foster等[25]克隆了苹果()DELLA蛋白的6个基因,将转入拟南芥,转基因植株出现开花延迟、节间变短等特征。【本研究切入点】目前,关于DELLA蛋白家族的研究多集中在作物及一些模式植物中,牧草中少有报道,且对DELLA蛋白在植物组织中表达模式和胁迫条件下的表达变化探索较少。紫花苜蓿作为牧草中的典型代表,DELLA蛋白在紫花苜蓿中的分子机理尚不明确。【拟解决的关键问题】本研究利用同源克隆的方法得到紫花苜蓿,并对该基因编码蛋白的理化性质、组织表达特异性、不同处理下的表达模式进行分析,同时构建了植物超表达载体,并获得了转基因紫花苜蓿材料,以期为解析调控紫花苜蓿生物量的机理,进而开展分子遗传改良奠定基础。
1 材料与方法
1.1 试验材料
1.1.1植物材料 试验于2016年7月至2017年12月进行,试验材料为紫花苜蓿‘中苜1号’(L.Zhongmu No.1)品种,由中国农业科学院北京畜牧兽医研究所保存。
1.1.2 试验试剂 PROMEGA总RNA提取试剂盒, 购自普洛麦格公司,TransScript Green One-Step qRT-PCR SuperMix、pEASY-Blunt Simple Cloning Kit、Trans5α Chemically Competent Cell、EasyPure Quick Gel Extraction Kit、DNA凝胶试剂盒,购自北京全式金生物技术有限公司。试验所用的内切酶、连接酶购自NEB(北京)有限公司。DNA提取试剂盒,购自天根生化科技(北京)有限公司。
1.2 紫花苜蓿MsGAI的克隆
使用Eastep Super提取紫花苜蓿总RNA;利用TransScript Green One-Step qRT-PCR反转录体系合成cDNA。
根据蒺藜苜蓿()(GenBank登录号XM_013605137.1)的cDNA序列,设计特异引物_F/R(表1),以紫花苜蓿cDNA为模板进行PCR扩增,反应体系为25 μL:cDNA 1 μL、KOD-plus 1 μL、10×Buffer for KOD 2.5 μL、25 mmol·L-1MgSO41 μL、2 mmol·L-1dNTPs 2.5 μL、引物各0.75 μL和PCR grade water 15.5 μL。反应程序为94℃ 5 min;94℃ 30 s,60℃ 30 s,72℃ 2 min,34个循环;72℃ 5 min。产物经1%琼脂糖凝胶电泳检测、纯化,并与Peasy-T1-Blunt载体连接,转化,挑取单克隆进行菌液PCR验证,阳性克隆送上海英潍捷基生物技术有限公司测序。

表1 试验中所用引物序列
黑体部分为限制性内切酶Ⅰ与HⅠ的位点
The primers blacked are restriction sitesⅠandHⅠ
1.3 MsGAI的生物信息学分析
利用SnapGene软件分析开放阅读框及其氨基酸序列;通过Protparam(http://strbio.sourceforge. net/javadocs/org/strbio/net/ProtParam.html)分析的理化性质;利用TMHMM(http://WWW.cbs.dtu.dk/ services/TMHMM)对MsGAI蛋白进行跨膜结构的预测分析;利用SOPMA(http://www.sopma.org/)在线预测该蛋白的二级结构;利用SingnalP(http://WWW. cbs.dtu.dk/services/SingnalP-4.1)分析MsGAI信号肽;利用TargetP(http://WWW.cbs.dtu.dk/services/TargetP)程序预测MsGAI蛋白的亚细胞定位。
使用ClustalX软件与其他物种进行多序列比对;利用MEGA7.0软件构建分子进化树,同时利用Neighbor-Joining(NJ)法构建系统发育树,bootstrap值设定为500,通过Bootstrap对生成的系统进化树进行校正;利用SWISS-MODEL(https://swissmodel. expasy.org/)在线预测MsGAI的三级结构;利用DNAMAN软件进行氨基酸同源序列比对;利用PlantCARE(http://www.plantcarescience.com/)预测分析启动子。
1.4 MsGAI启动子的克隆和分析
为了深入分析的表达,将克隆得到的序列进行比对(https://www.alfalfatoolbox.org/ doblast/),挑选得分最高的基因组序列,选择其起始密码子前2 000 bp左右的基因序列,设计特异性引物Promoter(表1),以‘中苜1号’DNA为模板,进行PCR扩 增。利用PlantCARE,对得到的启动子序列进行分析。
1.5 MsGAI的组织特异性表达分析
提取‘中苜1号’根、茎、叶、花瓣、花萼、雄蕊和雌蕊的总RNA,反转录成cDNA后,进行实时荧光定量PCR。根据得到的序列设计特异引物QGAI(表1),以紫花苜蓿为内参基因,利用ABI7500 Real-Time PCR system(美国ABI公司),依照TaKaRa SYBY Premix Ex Taq说明书进行PCR扩增。反应程序为95℃ 5 min;95℃ 5 s,58℃ 35 s,共40个循环。3次生物学重复,2次技术重复。采用2-∆∆CT方法[26]计算的表达量。
1.6 不同处理下MsGAI的表达分析
‘中苜1号’种子经氯气消毒,平铺在有滤纸的培养皿中,用无菌水将滤纸浸湿,在光照培养箱(16 h光照,8 h黑暗)中培养至种子露白,发芽后的种子移至MS培养基上,培养4周。分别用0.3 mol·L-1NaCl、15%的PEG、0.1x10-3mol·L-1GA3和ABA处理0、2、4、8、12和24 h;黑暗处理0、12、24、48和72 h。
1.7 MsGAI超表达载体的构建
根据的ORF序列设计带有HⅠ和Ⅰ双酶切位点的上下游引物121-(表1)。以含有的T载体质粒为模板,扩增含有酶切位点的目的片段,用Ⅰ/HⅠ双酶切目的片段和pBI121空载体,连接、转化。筛选阳性克隆送上海英潍捷基生物技术有限公司测序。超表达载体构建示意图如图1。
Less snow in winter and less rain in spring, spring comes earlier and much snow in autumn.
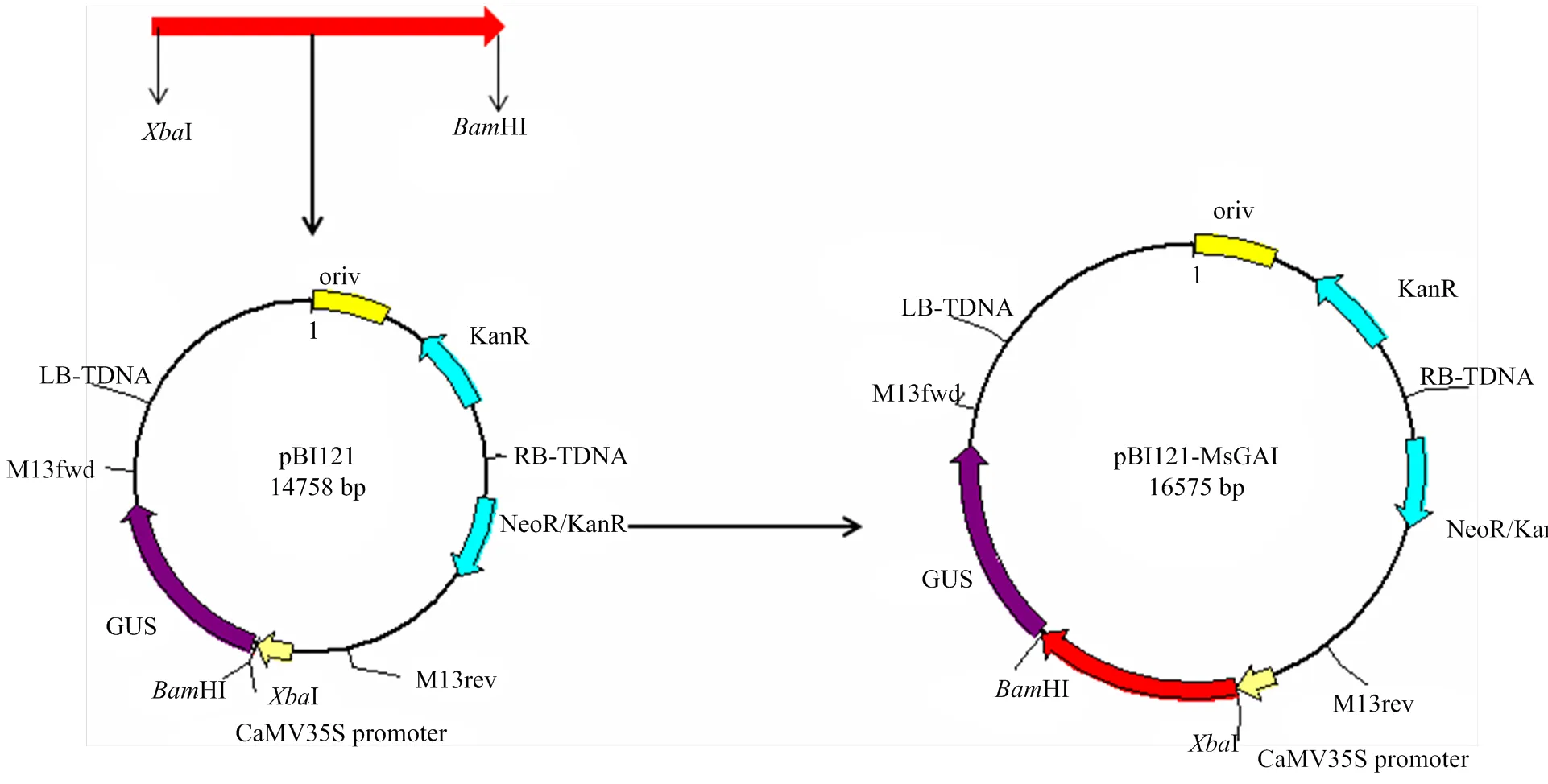
图1 35S:MsGAI-gus超表达载体的构建
1.8 紫花苜蓿的转化
以‘中苜1号’无菌苗为材料,挑选颜色为深绿色,肉眼观测无虫眼和病斑的叶片。经70%酒精消毒2 min,5%次氯酸钠消毒5 min,无菌水清洗3次后置于灭菌培养皿中。消毒后的外植体,在农杆菌溶液(OD600为0.6—0.8)重悬液中重悬15—30 min。在共培养基上培养48 h,然后转到诱导培养基,2—3周后将愈伤组织转入分化培养基,分化培养1—2个月后,愈伤组织将发育成丛生芽,经继代培养后,生成不定苗,将不定苗转至生根培养基,待不定苗生根后,移栽到营养土与蛭石比例1﹕1的花盆中,置人工气候室中培养。各类培养基的制备及具体操作流程,参照李俊等[27]方法。
1.9 转基因植株的鉴定
提取转基因植株DNA,以121-和35s/GUS为引物进行PCR扩增。将检测为阳性的转基因植株叶片浸泡在GUS染色液中,37℃过夜,次日脱色处理,观察结果。根据PCR验证及GUS染色结果,挑选长势良好的6个(L5、L7、L8、L9、L11和L12)转基因株系,利用特异引物QGAI进行qRT-PCR分析。
1.10 不同处理下转基因植株的表达分析
在选取的6个转基因株系中,分别挑选出高(L5)、中(L8)、低(L11)3个株系,移入基质为营养土的花盆中生长。剪取植株下部成熟茎,通过扦插法进行扩繁。每个株系选取6个单株分别进行NaCl和PEG处理,即用0.3 mol·L-1NaCl溶液和15%PEG均匀喷洒植物叶片,溶液分别在叶片正反面呈现挂珠状态。分别于0、2、4、8、12和24 h取样待用。
2 结果
2.1 MsGAI的获得及生物信息学分析
PCR扩增得到2 005 bp的目的片段(图2),利用SnapGene软件对序列进行分析,该基因片段包含一个1 818 bp的开放阅读框,共编码605个氨基酸。该序列编码的蛋白理论等电点为4.71,相对分子量为71.11 kD,不稳定系数为40.28,属于不稳定蛋白,该蛋白平均亲水系数为-0.43,该蛋白为亲水性蛋白。利用DNAMAN软件将MsGAI与其他物种氨基酸序列进行同源比对,发现该蛋白序列含有GRAS家族典型的DELLA和GRAS结构域(图3),包含大量与鹰嘴豆、蒺藜苜蓿、陆地棉()、小麦、玉米等物种DELLA蛋白同源的氨基酸序列。MsGAI氨基酸序列与豆科植物的相似度很高,与蒺藜苜蓿、鹰嘴豆、豌豆()的相似度分别为96%、78%和79%。证明该序列为紫花苜蓿的,故命名为。
通过对MsGAI蛋白进行分析,结果表明,该蛋白有46.06%的α-螺旋,7.11%的β转角,34.62%的无规则卷曲,12.21%延伸链(图4)。该蛋白没有跨膜结构,可能为非跨膜蛋白,存在信号肽段的可能性很小。MsGAI可能定位在叶绿体,cTP(chloroplast transit peptide)得分为0.634(表2)。
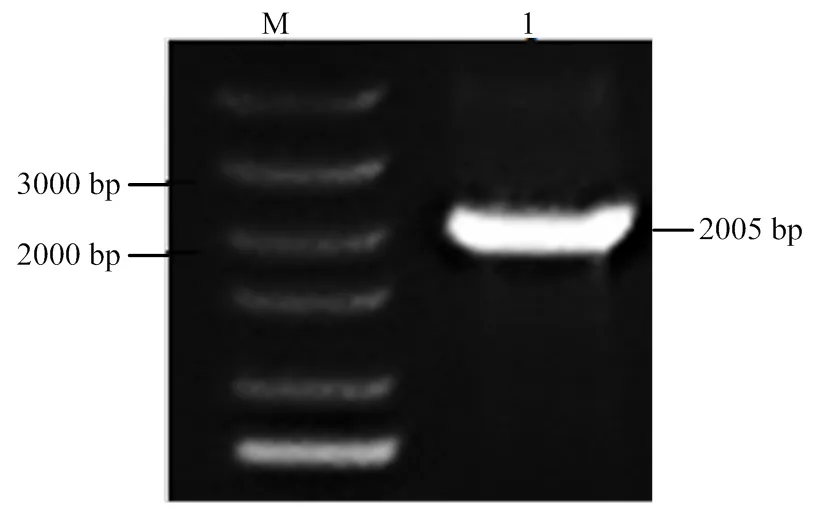
M:DL5000分子标记;1:PCR扩增产物
通过MEGA7.0软件,利用NJ法构建系统发育树,并且用Bootstrap进行校正,结果表明,与蒺藜苜蓿亲缘关系最近,其次与红三叶()等双子叶豆科植物亲缘关系较近,与单子叶植物亲缘关系较远(图5)。

表2 亚细胞定位预测结果
cTP:叶绿体转运肽;mTP:线粒体靶向肽;SP:信号肽;Loc:定位结果;RC:得分
cTP: chloroplast transit peptide; mTP: mitochondrial targeting peptide; SP: signal peptide; Loc: Subcellular localization result; RC: Score


蓝色:α-螺旋;红色:延伸链;绿色:β转角;橙色:无规则卷曲 Blue: α-helix; Red: Extended chain; Green: β-turn; Orange: Random coil
2.2 MsGAI启动子的生物信息学分析
利用PlantCARE启动子预测元件分析GAI启动子(表3)。结果显示,该基因启动子包含许多顺式作用元件,其中有光响应、昼夜节律响应元件,也存在与植物生长发育相关的元件,例如与分生组织表达相关的作用元件。同时还包含与干旱胁迫、热应激效应及低温响应相关的顺式元件。
2.3 MsGAI的组织特异性表达及不同处理条件下的表达分析
通过对不同处理条件下紫花苜蓿的表达进行分析(图6)。GA3处理后,表达量总体呈现下降的趋势,在8 h有一个小幅的上升,24 h几乎检测不到表达。ABA处理后,的表达量在4 h达到峰值,之后逐渐降低。NaCl处理下,前期表达水平变化幅度不大,但在24 h表达量显著升高。PEG模拟干旱胁迫,的表达量逐渐升高,到24 h时表达量升高到对照的30倍。黑暗处理后,表达量迅速下降,在处理2 h后表达量降低了近10倍,并一直保持较低的表达水平。
2.4 35S:MsGAI-gus超表达载体的构建及紫花苜蓿的遗传转化
利用Ⅰ和HⅠ对构建完善的pBI121植物超表达载体质粒进行双酶切检测,获得条带大小与预期一致(图7)。证明已正向插入pBI121表达载体中,超表达载体35S:MsGAI-gus构建成功。

表3 PlantCARE启动子预测结果
采用农杆菌介导的愈伤组织培养法对紫花苜蓿进行遗传转化(图8)。经过4个月的组织培养,目的片段成功转入紫花苜蓿,再生苗经GUS组织染色,呈现蓝色(图8-E),后将转基因幼苗移入人工气候室培养。
待植株生长1月后,提取转基因植株DNA,进行PCR检测(图9)。对载体中的进行PCR检测,结果显示,3对引物均扩增出大小相符的PCR片段。(图9)。
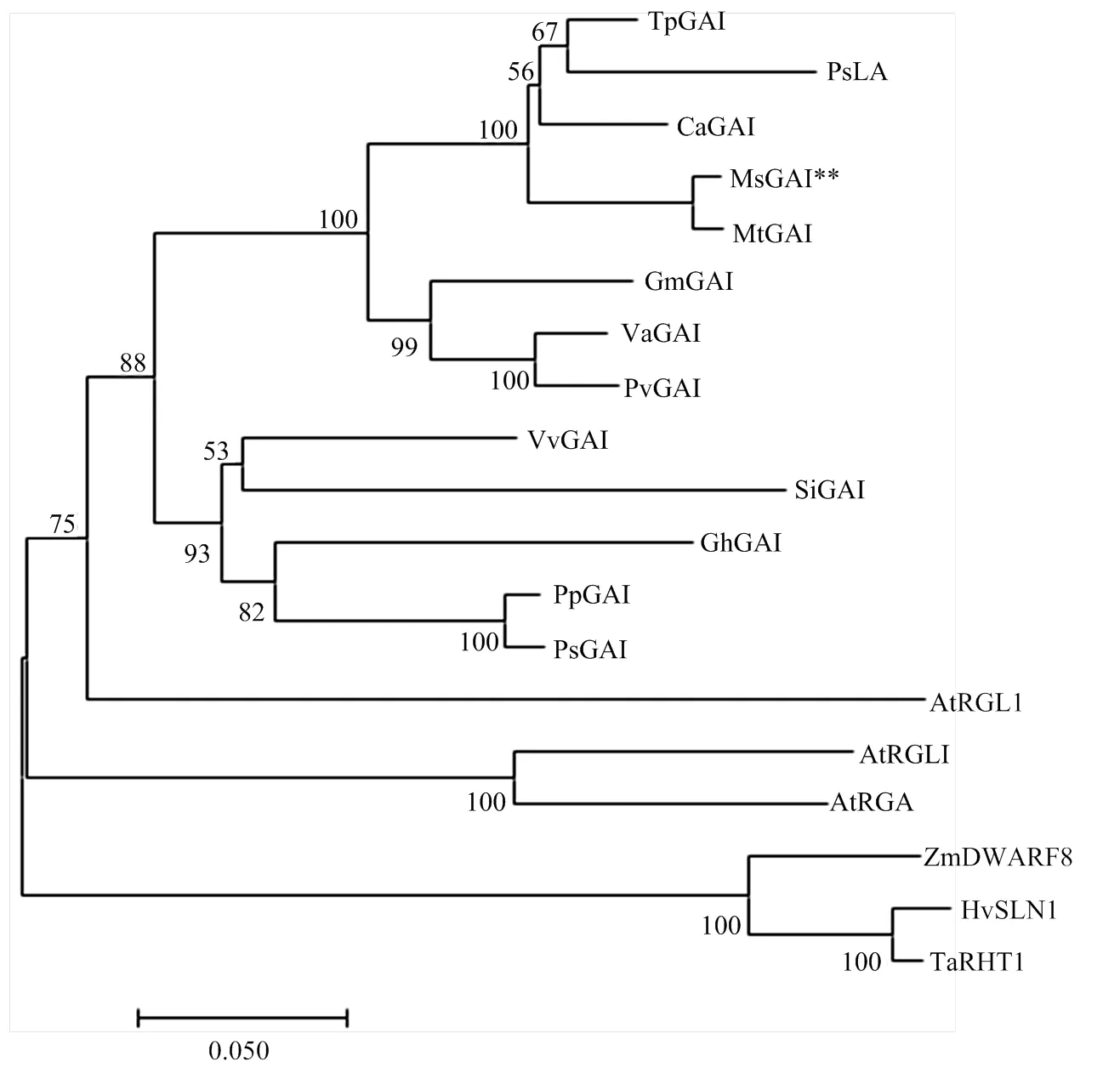
TpGAI:红三叶Trifolium pratense,PNY03470.1;PsLA:豌豆Pisum sativum,ABI30654.1;CaGAI:鹰嘴豆Cicer arietinum,XP_004503135.1;MsGAI:紫花苜蓿Medicago sativa;MtGAI:蒺藜苜蓿Medicago truntula,XP_013460591.1;GmGAI:大豆Glycine max,XP_003552980.1;VaGAI:赤豆Vigna angularis,XP_017418978.1;PvGAI:菜豆Phaseolus vulgaris,BAF62637.1;VvGAI:葡萄Vitis vinifera,ARR27421.1;SiGAI:芝麻Sesamum indicum,XP_011097451.1;GhGAI:陆地棉Gossypium hirsutum,XP_016744906.1;PpGAI:沙梨Pyrus pyrifolia,ANJ78486.1;PsGAI:李子Prunus salicina, AQQ12221.1; AtRGL1:拟南芥Arabidopsis thaliana,NC_003070.9;AtGAI:拟南芥Arabidopsis thaliana,NC_003070.9;AtRGA:拟南芥Arabidopsis thaliana,NC_003071.7;ZmDWARF8:玉米Zea mays,NC_024459.2;HvSLN1:青稞Hordeum vulgare,Q8W127.1;TaRHT1:小麦Triticum aestivum,Q9ST59.1。节点上的数值表示自举值,标尺上的数字表示遗传距离Numbers at node indicates bootstrap value derived from 1000 replicates. The scale bar indicates genetic distance
2.5 转基因植株表达分析
对转基因植株进行qRT-PCR检测,结果表明,与对照相比,转基因紫花苜蓿中的表达量较高,其中L5株系表达量最高,L11表达量相对较低(图10)。在15%PEG处理下,L5、L8、L11表达量上调,其中L5株系的表达量明显高于L11株系;与对照组相比,转基因植株各时间段表达量均提高。300 mol·L-1NaCl处理后,3个株系的表达量均出现不同程度的提高,L5最为显著。
3 讨论
3.1 MsGAI在豆科物种中高度保守
DELLA蛋白定位在细胞核中,蛋白序列结构特征分析以及蛋白核定位表明,DELLA蛋白属于一类潜在的转录因子[28]。在没有GA的情况下,DELLA蛋白阻遏植物生长发育;当DELLA蛋白上的GA信号感知域接收到信号以后,阻遏作用被抑制[29]。DELLA蛋白基因在许多植物中被成功克隆,功能研究表明,DELLA蛋白对植物种子萌发、开花时间、植株生物量方面均有重要影响[30]。
本研究利用同源克隆的方法从紫花苜蓿中成功分离基因,该基因编码的蛋白具有GRAS家族的典型结构域和保守区,其中包含DELLA蛋白N端保守结构域DELLA和TVHYNP,C端保守结构SAW。通过构建不同物种的DELLA蛋白系统发育树结果表明,紫花苜蓿基因与蒺藜苜蓿、鹰嘴豆、红三叶、大豆()、豌豆等豆科植物的相似度较高,其中与蒺藜苜蓿的相似度高达96%。说明这类基因在进化过程中高度保守,具有相似的功能。基于此理论,参考模式物种同源基因的作用机制,探索出紫花苜蓿所特有的功能,不失为一种有效的基因功能研究方法。

图6 MsGAI的组织特异性表达及不同处理下的表达模式

M:DL 5000 DNA marker;1:pBI121载体;2:35S:MsGAI-gus双酶切片段

A:外植体;B:愈伤组织;C:成熟小胚;D:阳性苗;E:GUS染色;F:成活的转基因植株

A:pBI121载体引物检测结果(1—4);B:MsGAI引物检测结果(5—8);C:上游载体引物下游基因引物检测结果(9—12)
3.2 MsGAI响应紫花苜蓿非生物胁迫
在紫花苜蓿的多个组织中均有表达,在根中的表达量最高,花中次之,茎中最低(图6)。因此推测,在紫花苜蓿各个组织的生长发育过程中均可发挥作用。本研究结果显示,GA3处理后,的表达量迅速降低,24 h后表达量降到最低,这有可能是因为外施赤霉素后,赤霉素与受体结合,然后与DELLA蛋白结合形成三元结构,最终DELLA蛋白被降解,导致编码DELLA蛋白的基因表达量下降。这一结果也与DELLA蛋白作为赤霉素信号通路 中的阻遏因子的结论一致[31]。
资料表明,ABA可通过影响细胞质子泵的生物活性来调节植物体的生长发育,植物受到外界胁迫时,ABA通过增强质子泵活性,从而刺激多种逆境蛋白的基因表达[32]。Zentella等[33]提出DELLA蛋白能够诱导E3连接酶基因XERICO表达上调来促进ABA的积累。对谷物糊粉层的研究表明,ABA通过作用于DELLA蛋白下游来抑制GA反应[34]。因此,ABA可能参与了DELLA蛋白调控GA的信号途径,DELLA蛋白与ABA在GA调控植物生长发育过程中可能存在协同作用。本研究中,ABA处理初期,基因表达量变化不显著,4 h后,表达量迅速下降。同时,将克隆得到的启动子进行序列分析,结果显示,在启动子区域含有大量与ABA抗逆相关的作用元件。模拟干旱胁迫处理后,紫花苜蓿对照组与转基因组,的表达量逐渐上升并一直处于较高水平,同时盐胁迫也诱导该基因的表达,由此推断,可能通过与ABA相互协同参与紫花苜蓿对盐及干旱的胁迫响应。
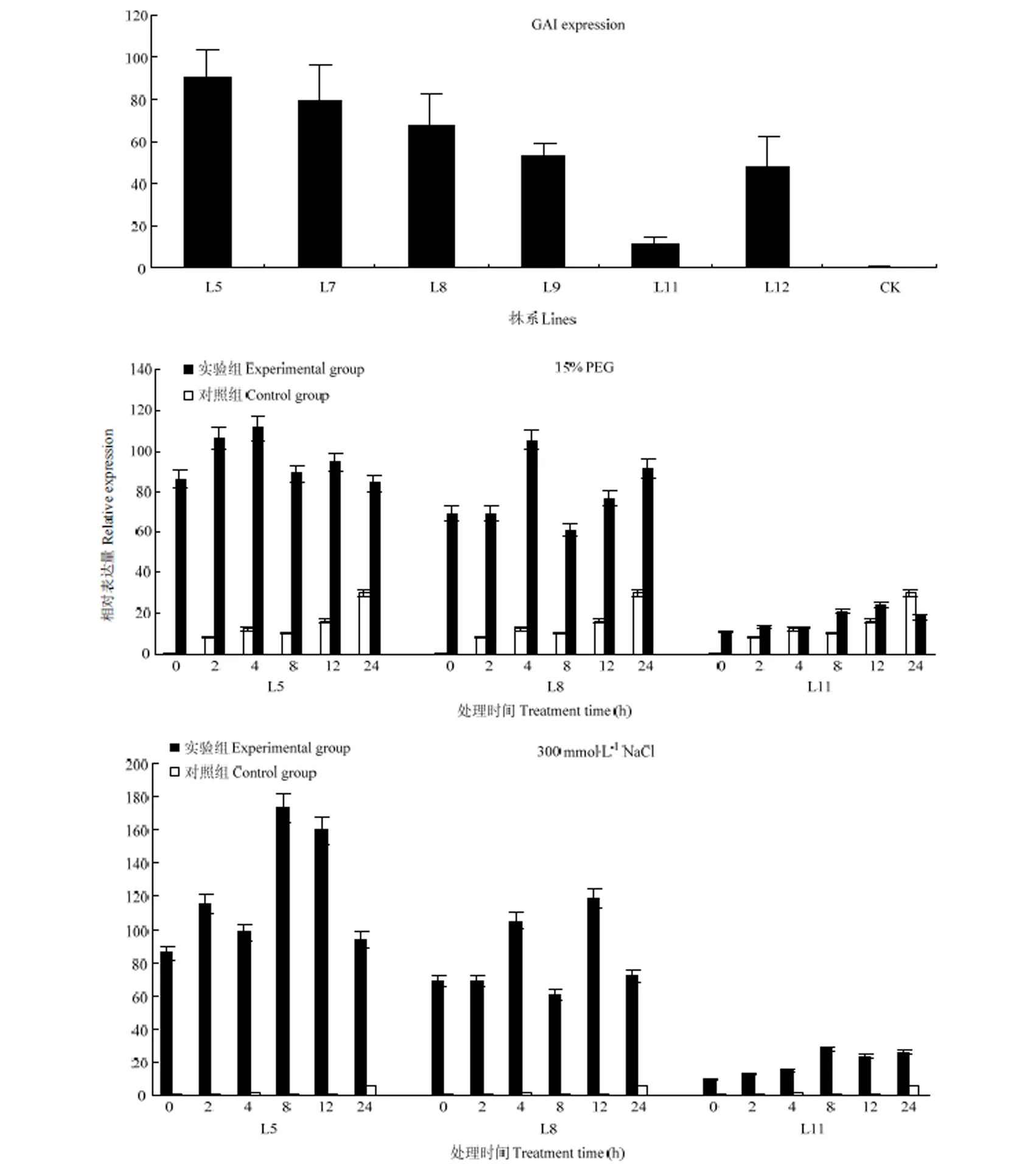
图10 转基因紫花苜蓿中MsGAI表达量及不同处理下的表达模式
4 结论
从紫花苜蓿中分离1个DELLA蛋白基因,包含1 818 bp的开放阅读框,该基因序列具有响应赤霉素的DELLA和VHYNP结构域,与蒺藜苜蓿的亲缘关系最近。在根中表达量最高。转基因紫花苜蓿对干旱、盐胁迫处理具有明显响应。
[1] 陈敏. 紫花苜蓿赤霉素受体基因的克隆及功能分析. 北京: 中国农业科学院, 2017.
CHEN M. Cloning and functional analysis of gibberellin receptor genefromL. .Beijing: Chinese Academy of Agricultural Sciences, 2017. (in Chinese)
[2] BLUMENTHAL J M, RUSSELLE M P. Subsoil nitrate uptake and symbiotic dinitrogen fixation by alfalfa., 1996, 88(6): 909-915.
[3] RUSSELLE M P, LAMB J F, MONTGOMERY B R, ELSENHEIMER D W, MILLER B S, VANCE C P. Alfalfa rapidly remediates excess inorganic nitrogen at a fertilizer spill site., 2001, 30(1): 30-36.
[4] HEDDEN P, PHILLIPS A L. Gibberellin metabolism: new insights revealed by the genes., 2000, 5(12): 523-530.
[5] SUN T. Gibberellin metabolism, perception and signaling pathways in., 2008: e0103.
[6] 董玉琛, 郑殿升. 中国小麦遗传资源. 北京: 中国农业出版社, 2000: 101-107.
DONG Y C, ZHENG D S.. Beijing: China Agricultural Press, 2000: 101-107. (in Chinese)
[7] AUSTIN R B. Yield of wheat in the United Kingdom: recent advances and prospects., 1998, 39(6): 1604-1610.
[8] MONNA L, KITAZAWA N, YOSHINO R, SUZUKI J, MASUDA H, MAEHARA Y. Rice “Green Revolution Gene” encodes a mutant enzyme involved in gibberellin synthesis., 2002, 9(1): 11-17.
[9] SASAKI A, ASHIKARI M, UEGUCHI-TANAKA M, ITOH H, NISHIMURA A, SWAPAN D. Green revolution: a mutant gibberellin- synthesis gene in rice., 2002, 416: 701-702.
[10] SPIELMEYER W, ELLIS M H, CHANDLER P M. Semidwarf (sd-1), “green revolution” rice, contains a defective gibberellin 20-oxidase gene., 2002, 99(13): 9043-9048.
[11] PYSH L D, WYSOCKA-DILLER J W, CAMILLERI C, BOUCHEZ D, BENFEY P N. The GRAS gene family in: Sequence characterization and basic expression analysis of the SCARECROW- LIKE genes., 1999, 18(1): 111-119.
[12] PENG J, RICHARDS D E, HARTLEY N M, MURPHY G P, DEVOS K M, FLINTHAM J E. 'Green revolution' genes encode mutant gibberellin response modulators., 1999, 400(6741): 256
[13] SILVERSTONE A L, JUNG H S, DILL A, KAWAIDE H, KAMIYA Y, SUN T P. Repressing a repressor: gibberellin-induced rapid reduction of the RGA protein in., 2001, 13(7): 1555-1566.
[14] GUBLER F, CHANDLER P M, WHITE R G, LLEWELLYN D J, JACOBSEN J V. Gibberellin signaling in barley aleurone cells. Control of SLN1 and GAMYB expression., 2002, 129(1): 191-200.
[15] ITOH H, UEGUCHI-TANAKA M, SATO Y, ASHIKARI M, MATSUOKA M. The gibberellin signaling pathway is regulated by the appearance and disappearance of SLENDER RICE1 in nuclei., 2002, 14(1): 57-70.
[16] PENG J, CAROL P, RICHARDS D E, KING K E, COWLING R J, MURPHY G P. Thegene defines a signaling pathway that negatively regulates gibberellin responses., 1997, 11(23): 3194-3205.
[17] TYLER L, THOMAS S G, HU J, DILL A, ALONSO J M, ECKER J R. DELLA proteins and gibberellin-regulated seed germination and floral development in., 2004, 135(2): 1008-1019.
[18] LEE S, CHENG H, KING K E, WANG W, HE Y, HUSSAIN A. Gibberellin regulatesseed germination via RGL2, a GAI/RGA-like gene whose expression is up-regulated following imbibition., 2002, 16(5): 646-658.
[19] FU X, SUDHAKAR D, PENG J, RICHARDS D E, CHRISTOU P, HARBERD N P. Expression ofin transgenic rice represses multiple gibberellin responses., 2001, 13(8): 1791-1802.
[20] TALON M, KOORNNEEF M, ZEEVAART J A D. Accumulation of C19-gibberellins in the gibberellin-insensitive dwarf mutant gai of(L.)., 1990, 182(4): 501-505.
[21] TALON M, KOORNNEEF M, ZEEVAART J A. Endogenous gibberellins inand possible steps blocked in the biosynthetic pathways of the semidwarf ga4 and ga5 mutants., 1990, 87(20): 7983-7987.
[22] WILSON R N, SOMERVILLE C R. Phenotypic suppression of the gibberellin-insensitive mutant gai of., 1995, 108(2): 495-502.
[23] FU X, HARBERD N P. Auxin promotesroot growth by modulating gibberellin response., 2003, 421(6924): 740.
[24] DILL A, SUN T. Synergistic derepression of gibberellin signaling by removing RGA andfunction in., 2001, 159(2): 777-785.
[25] FOSTER T, KIRK C, JONES W T. Characterization of the DELLA subfamily in apple (Borkh.). Tree Genetics & Genomes, 2007, 3(3): 187-197.
[26] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2− ΔΔCTmethod., 2001, 25(4): 402-408.
[27] 李俊. 东方山羊豆赤霉素受体和水通道蛋白基因功能研究. 北京: 中国农业科学院, 2014.
LI J. Construction of plant expression Vector ofgene and transformation ofL.. Beijing: Chinese Academy of Agricultural Sciences, 2014. (in Chinese)
[28] 黄先忠, 蒋才富, 廖立力. 傅向东. 赤霉素作用机理的分子基础与调控模式研究进展. 植物学通报, 2006, 23(5): 499-510.
HUANG X Z, JIANG C F, LIAO L L, Fu X D. Progress on molecular foundation of GA biosynthesis pathway and signaling., 2006, 23(5): 499-510. (in Chinese)
[29] FUKUDA A, TANAKA Y. Effects of ABA, auxin, and gibberellin on the expression of genes for vacuolar H+-inorganic pyrophosphatase, H+-ATPase subunit A, and Na+/H+antiporter in barley., 2006, 44(5/6): 351-358.
[30] ZENTELLA R, ZHANG Z L, PARK M. Global analysis of DELLA direct targets in early gibberellin signaling in., 2007, 19(10): 3037-3057.
[31] DU H, CHANG Y, HUANG F, XIONG L.modulates stomatal response and submergence tolerance involving abscisic acid and gibberellic acid signaling in rice., 2015, 57(11): 954-968.
[32] DJAKOVIC-PETROVIC T, DE WIT M, VOESENEK L. DELLA protein function in growth responses to canopy signals., 2007, 51(1): 117-126.
[33] ZENTELLA R, ZHANG Z L, PARK M, THOMAS S G, ENDO A, MURASE K. Global analysis of DELLA direct targets in early gibberellin signaling in, 2007, 19(10): 3037-3057.
[34] GÓMEZ-CADENAS A, ZENTELLA R, WALKER-SIMMONS M K, HO T H. Gibberellin/abscisic acid antagonism in barley aleurone cells: Site of action of the protein kinase PKABA1 in relation to gibberellin signaling molecules., 2001, 13: 667-679.
Cloning expression analysis and transformation ofGene fromL
ZHANG Han, WANG XueMin, LIU XiQiang, MA Lin, WEN HongYu, WANG Zan
(Institute of Animal Sciences, Chinese Academy of Agricultural Sciences, Beijing, 100193)
【Objective】DELLA, one of GRAS family proteins, is an important transcription factor, which negatively regulates the gibberellin (GA) signal pathway. In this study, theL. DELLA genewas cloned, and its secondary structure was also predicted through bioinformatics. To illustrate the molecular function ofinL. gibberellin (GA) signal transduction pathway, we clarify the spatio-temporal expression pattern ofand its response to different treatments; and the transgenic alfalfa over-expressingwere obtained by-mediated transformation to explore gene function as well.【Method】was cloned by homologous cloning and sequence characteristics ofwere analyzed by online bioinformatics tools. The phylogenetic tree ofand its homologous genes from other species was constructed by MEGA 7.0. The real-time quantitative PCR (qRT-PCR) was used to detect the spatio-temporal expression pattern ofand its response to abiotic including PEG, NaCl, GA, ABA and dark. The plant overexpression vector 35S:MsGAI-gus was transferred into Agrobacterium GV3101 strain, and transgenic alfalfa were obtained by Agrobacterium-mediated callus transformation. The transgenic positive seedlings were verified by PCR and GUS histochemical staining. Three transgenic line (L5, L8, L11) were selected to analysisexpression under PEG, and NaCl treatment.【Result】Sequence analysis showed that 605 amino acids constituteand it contains the conserved DELLA and VHYNP sequences in N-terminal regulatory region, and SAW in C-terminal. Multiple sequence alignment and phylogenetic tree analysis showed that the sequence similarity with other DELLA protein was as high as 80%, and it has the closest relationship withGAI, followed by the dicotyledonous such asand, and is far away from monocotyledon such as. Real-Time PCR analysis showed thathad the highest expression in roots and after PEG, Nacl, GA and ABA treatment, the response was obvious while dark treatment significantly inhibited the expression of. The GUS tissue staining showed that the positive plants were blue and the control group was white and the expression ofwas upregulated in positive plants under PEG and NaCl treatments.【Conclusion】The cloning and overexpression vector of DELLA protein in alfalfa was successfully constructed and thecould respond to stress treatments.
L.; DELLA protein; GA; expression analysis; homologous cloning; transformation
10.3864/j.issn.0578-1752.2019.02.002
2018-07-18;
2018-09-07
国家自然科学基金(31761143013、31272495)、国家现代农业产业技术体系(CARS34)
张涵,E-mail:793607453@qq.com。通信作者王赞,E-mail:wangzan@caas.cn
(责任编辑 李莉)
