PCR反应程序对土壤微生物PCR-DGGE分析的影响
2018-12-05武丽娟陈月星刘小凤张战凤樊永亮
武丽娟, 陈月星, 刘小凤, 宋 月, 张战凤, 樊永亮
(1.西北农林科技大学植物保护学院,陕西杨凌 712100; 2.西北农林科技大学农学院,陕西杨凌 712100)
土壤微生物是陆地生态系统中重要的组成部分,积极参与土壤养分循环、有机质分解及肥力形成等重要的生态过程[1]。它们的生存很容易受到很多环境因子的影响,如污染物、地表植被、土地利用方式、土壤类型等,因此常常被认为是土壤生态系统变化的指示者。除了对与土壤微生物密切相关的土壤酶和土壤微生物量的研究外,近年来,对土壤微生物多样性的研究密集起来,这主要得益于不依赖微生物培养的分子生物学技术[脂肪酸甲酯/磷脂脂肪酸(FAME/PLFA)、扩增的rDNA限制性分析(ARDRA)、随机扩增多态性DNA(RAPD)、末端限制性片段长度多态性(T-RFLP)、肠杆菌重复基因间一致序列PCR(ERIC-PCR)、单链构象多态性(SSCP)、16S rDNA序列分析]的发展和DNA测序技术的日益成熟。自Muyzer等将聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE)技术引入到微生物多样性分析以来[2],该技术被广泛应用于检测各种环境中微生物群落结构多样性、监测微生物群落动态变化等方面[3-6]。然而在试验操作中,该技术各个环节均需完善才能准确而科学地反映试验结果,以往研究主要集中于DNA提取[7-8]、靶序列选择[9]及变性梯度摸索等方面,而对于PCR反应程序的系统比较研究还未见报道,因此,本试验针对该技术的目的基因片段扩增(PCR)环节,以16S rDNA基因V3区片段扩增为例,比较4种不同的PCR反应程序对土壤微生物PCR-DGGE多样性分析的影响,从而评价和筛选出适合于土壤微生物多样性PCR-DGGE分析的反应程序,以期为PCR-DGGE技术在土壤微生物群落结构和种群多样性分析中的应用和完善提供理论基础。
1 材料与方法
1.1 主要试验仪器和试剂
仪器:基因扩增仪(美国伯乐公司),涡旋仪、水浴锅、高速冷冻离心机(德国艾本德生命科学公司),核酸突变检测系统(美国伯乐公司),核酸电泳仪(美国伯乐公司),凝胶成像系统(美国伯乐公司),蓝盾®621可见光凝胶透射仪。
试剂:小量土壤DNA提取试剂盒(美国Omega公司)、DNA回收纯化试剂盒(北京天根生化科技有限公司)、琼脂糖,GenFinder核酸染料(厦门百维信生物科技有限公司)、GelRed核酸染料(美国Biotium生物技术公司),rTaqDNA聚合酶和dNTPs[宝生物工程(大连)有限公司],40%丙烯酰胺溶液(丙烯酰胺 ∶甲叉丙烯酰胺=37.5 ∶1)、四甲基乙二胺(TEMED)、过硫酸铵(西陇)、PCR引物[生工生物工程(上海)股份有限公司]。
1.2 研究区概况和土壤样品的采集
本试验所用土样采集于延安市果业研究发展中心苹果试验基地,该区域位于渭北旱塬核心地段,属于温带半湿润大陆性季风气候,年均气温9.2 ℃,平均海拔1 100 m,多年平均降水量622 mm,主要集中于6—9月;地貌以塬、梁、沟相间,以塬为主;土壤为黄土母质发育而成的疏松黑垆土。
于2011年10月19日苹果成熟期,分别在苹果生育期内种植三叶草(TrifoliumrepensL.)和覆盖玉米秸秆(30 000 kg/hm2)的园区采用多点采样法采集土样(地表下5~15 cm土层),土样均匀混合后装入标记好的无菌塑封袋中带回实验室,取出石块、草根、秸秆及其他肉眼可见的杂物,过2 mm钢筛,于-80 ℃保存,7 d内完成土壤微生物总DNA的提取。
1.3 土壤微生物总DNA的提取
土壤微生物总DNA采用小量土壤DNA提取试剂盒,参照试剂盒操作说明进行提取,得到的DNA提取液经0.8%琼脂糖凝胶电泳检测。
1.4 16S rRNA基因V3区片段的扩增
本研究采用引物对27F[10]/1492R[11]和GC-338F/518R[2]分别对16S rDNA基因全长和V3区片段进行扩增。16S rDNA基因全长扩增体系和程序参照Nicomrat等的方法[12];V3区片段扩增体系如下:10×PCR缓冲液(含Mg2+)5 μL,dNTPs(各2.5 mmol/L)4 μL,正反向引物(各 5 μmol/L)4 μL,模板DNA 2 μL,TaKaRa rTaq(5 U/μL)0.5 μL,补充双蒸水至50 μL。反应程序为
A:降落式PCR。以提取的微生物总DNA为模板,进行V3区片段扩增,参考Kihara等的方法,反应条件为94 ℃预变性5 min; 94 ℃ 变性30 s,65~55 ℃(每个循环退火温度降低 0.5 ℃,直至退火温度降至55 ℃)复性30 s,72 ℃延伸1 min,20个循环;94 ℃ 变性30 s,55 ℃复性30 s,72 ℃延伸 1 min,10个循环;72 ℃延伸10 min[13]。
B:巢式-降落式PCR。以27F/1492R引物扩增产物为模板,进行V3区片段扩增,反应程序同A。
C:普通PCR。以提取的微生物总DNA为模板,进行V3区片段扩增,参考Kimura等的方法,反应条件为94 ℃预变性5 min;94 ℃变性1 min,55 ℃复性1 min,72 ℃延伸1 min,30个循环;72 ℃ 延伸8 min[14]。
D:巢式PCR。以27F/1492R引物扩增产物为模板,进行V3区片段扩增,反应程序同C。
每个土样PCR均设置3个重复,合并后采用琼脂糖凝胶DNA回收和纯化试剂盒进行回收纯化,依据试剂盒使用说明进行操作,浓缩产物保存于-20 ℃,以备DGGE分析。
1.5 DGGE分析
应用美国伯乐公司核酸突变检测系统进行DGGE分析,凝胶的变性梯度为45%~65%(100%变性剂为7 mol/L尿素和40%去离子甲酰胺的混合物),凝胶浓度为8%,在60 ℃、120 V条件下,凝胶在1×TAE缓冲液中电泳510 min。电泳完毕,将凝胶浸在含Gel Red(体积分数为1 ∶10 000)的 1×TAE 溶液中避光染色10~15 min,采用Bio-Rad凝胶成像系统进行拍照。
1.6 数据分析
应用Quantity One 4.6.2软件对DGGE图谱进行数字化分析,用以计算香农指数(H)、丰富度指数(R)、均匀度指数(E)和戴斯系数(D),并利用SPPSS 16.0软件应用单因素方差分析方法检测处理间的差异显著性。相关指数计算公式如下:
(1)
E=H/lnS;
(2)
(3)
式中:Pi为某一条带的峰强度与该泳道中所有条带峰强度和的比值;S为某一泳道的DNA条带数。Sx为泳道x中的条带数;Sy为泳道y中的条带数;Sc为泳道x和泳道y所共有的条带数。
2 结果与分析
2.1 土壤微生物总DNA提取和16S rDNA V3区扩增
2.1.1 土壤微生物总DNA提取 本试验采用土壤DNA提取试剂盒提取土壤微生物总DNA,所提取的DNA溶液D260 nm/D280 nm介于1.8~2.0之间,其D260 nm/D230 nm介于0.1~0.5之间;经8%琼脂糖凝胶电泳后得到单一、清晰的条带,大小约为23 kb,说明所提取的DNA溶液在纯度和长度上均满足后续试验的要求。
2.1.2 PCR扩增结果 应用通用引物对27F/1492R扩增16S rDNA基因全长,并经0.8%琼脂糖凝胶电泳检测。由图1可知,通过应用27F/1492R引物对的扩增,2份土样均成功扩增出16S rDNA基因的全长序列,长度约为1 500 bp。将扩增得到的3份PCR反应液进行合并,经DNA回收纯化试剂盒回收纯化后,于-20 ℃冰箱保存备用。

应用同样的反应体系,不同的反应程序扩增16S rDNA基因V3区片段,并经3%琼脂糖凝胶电泳检测。由图2-A和图2-B可知,用4种不同的PCR反应程序,2份土样的所有处理样品均扩增出正确大小的目的片段,约为220 bp;重复之间和不同反应程序之间,电泳条带的大小和强度没有明显差异,而在2份土样之间,植草覆盖果园土样(G)比秸秆覆盖果园土样(S)亮度暗。同一处理的PCR反应液合并后经DNA纯化回收试剂盒回收纯化,于-20 ℃冰箱保存,以备DGGE分析。
2.2 DGGE图谱分析
应用PCR-DGGE分析自然环境中细菌、古菌、真核生物及病毒群落的生物多样性,通过添加化学变性剂使DNA在电泳过程中发生解链变性,降低迁移率,从而使不同DNA分子得以分离。理论上,DGGE图谱上1个条带代表1个微生物优势菌群或操作分类单,条带数目的多寡反映细菌的多样性高低,对应条带的强度反映该优势菌群的相对丰度,因此可以依据图谱中的条带信息分析样品中微生物的群落结构和微生物种群多样性。
4种不同PCR反应程序下土壤微生物多样性DGGE指纹图谱见图3,通过对DGGE图谱的初步分析,可以发现,2个土样DGGE图谱中的各泳道的条带数目比较丰富、迁移率及条带亮度间存在一定差异,说明2份土样所含微生物多样性较高,且样品间微生物种类和数量差异较大;同时2个土样图谱中存在若干共同条带,说明供试土样中存在多种共有细菌;样品内不同反应程序处理间各泳道的条带数目、亮度及迁移率也存在一定差异,说明不同的PCR反应程序对DGGE多样性分析存在一定的影响,且明显可以看出,A与B、C、D差异较为明显,而B、C、D之间条带类型较为相似。

2.3 不同PCR反应程序下细菌群落结构分析
应用Quantity One 4.6.2软件对DGGE图谱进行数字化处理,并计算样品中各泳道的多样性指数、丰富度指数和均匀度指数。由表1可知,2个土样不同处理间多样性指数相似,A处理的多样性指数最低,与B处理间差异显著(P<0.05);B、C、D处理多样性指数均大于等于3.10,且C、D处理间无显著差异。丰富度指数与多样性指数比较结果相似,2个土样中A处理的丰富度最低,分别为25(G)和24(S),且与C、D处理间差异显著(P<0.05),B、C、D丰富度指数无显著性差异。结果表明,单纯应用降落式PCR在提高扩增特异性的同时降低了扩增效率,从而导致DGGE图谱条带数目较少,反映样品中微生物种群多样性较低;在巢式-降落式PCR、普通PCR和巢式PCR扩增条件下,DGGE图谱多样性较好、丰富度指数较高, 能够较好地反映土壤微生物的群落结构和种群多样性,并且表现出较好的一致性。

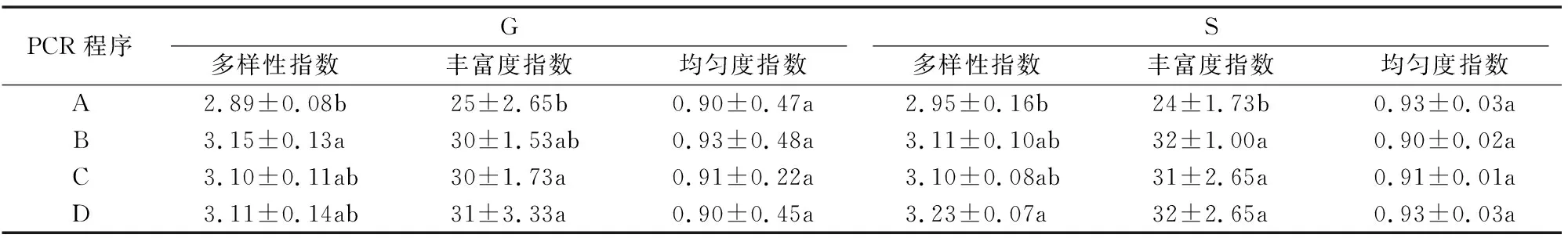
表1 不同PCR反应程序下土壤微生物多样性指数
注:同列数据后不同小写字母表示经Duncan’s检验在0.05水平上差异显著。A表示降落式PCR;B表示巢式-降落式PCR;C表示普通PCR;D表示巢式PCR。下表同。
2.4 群落相似性分析
DGGE图谱经Quantity One 4.6.2软件分析,通过戴斯系数公式计算2份土样在4个不同PCR反应程序下各泳道的戴斯系数,得到相似性矩阵(表2、表3)。2个土样中降落式PCR与其他3个处理相似度普遍较低,最低为0.78;普通PCR与其他3个PCR处理相似度比较低,主要因为普通PCR在扩增复杂模板时存在一定的非特异性扩增,表现在DGGE图谱上为出现不代表任何细菌种群的特异条带,从而夸大样品中微生物的种群多样性和丰富度;巢式-降落式PCR和巢式PCR处理间一致性较高,具有相似的微生物群落结构,尤其S的巢式-降落式PCR和巢式PCR处理反映的土壤微生物群落结构基本一致,相似度为0.97,说明巢式PCR不仅确保了扩增的特异性,且克服了降落式PCR扩增效率低的问题。

表2 不同反应程序下植草覆盖果园土壤样品微生物群落结构相似性分析

表3 不同反应程序下秸秆覆盖果园土壤样品微生物群落结构相似性分析
3 讨论
DGGE凝胶电泳技术于1979年被提出,是一种用于DNA突变检测的分子生物技术[15],该技术被Muyzer等[2]引入到微生物生态学研究领域,并证实了该技术在自然界微生物生态学研究领域的实用性和优越性。这是因为(1)DGGE是一种不依赖于纯培养的DNA片段分离技术,能够较全面反映样品中微生物的群落结构、种类和数量;(2)DGGE凝胶电泳可以同时运行多个样品,适用于研究环境样品中微生物群落结构的演替规律、种群动态变化等;(3)DGGE凝胶电泳具有重现性高、可靠性强及操作简单等优点,因此DGGE自诞生以来被广泛应用于各种环境(土壤、活性污泥、生物膜、动物肠胃以及水生生态环境)微生物多样性的检测及群落动态变化规律的研究。
然而,与其他分子生物技术一样,DGGE技术也存在一定的局限性,如只能分离500 bp以下的DNA片段,序列信息量较小;具有相同解链行为的微生物DNA序列在DGGE图谱上迁移率一致,无法准确区分;DGGE只能检测环境中优势菌群的种类和数量(1%以上),且无法提供代谢活性和基因表达水平方面的信息。除此之外,DGGE图谱分析还受到各操作环节的影响,包括总DNA的提取、PCR扩增引物选择、凝胶和变性剂梯度的设置、电泳温度和时间的确定、染色方法的选择[16-17]以及后续条带的测序步骤[18]。
本研究针对PCR扩增环节,采用目前DGGE分析中比较常用的4种扩增程序(A:降落式PCR;B:巢式-降落式PCR;C:普通PCR;D:巢式PCR)对16S rDNA基因V3区进行扩增,并进行DGGE图谱分析,结果表明,与普通PCR和降落式PCR相比,巢式PCR(巢式PCR和巢式-降落式PCR)所获得的DGGE条带较为清晰,条带数目较丰富。雷娟利等在研究不同蔬菜连作对土壤细菌DNA分子水平多态性影响中也发现,巢式PCR所获得的DGGE条带比直接PCR更清晰,可分辨的条带数量也更多[19]。同样,江芸等在研究真空包装冷却猪肉冷藏过程中菌相变化时发现,巢式PCR(巢式-降落式PCR)能够增加PCR反应的灵敏性[20]。表明在其他技术操作相同的情况下,巢式PCR(巢式PCR和巢式-降落式PCR)比其他PCR更适用于土壤及其他微生物群落结构比较复杂的样品的PCR-DGGE分析。
然而,PCR只是PCR-DGGE技术流程的一个环节,保证PCR扩增的特异性和效率是整个试验的一部分,其他环节对DGGE图谱分析同样也存在一定影响,因此,在应用PCR-DGGE技术对土壤微生物群落结构进行分析时,须摸索和完善各个环节才能得到全面和科学的结果。
4 结论
综上所述,应用PCR-DGGE分析土壤微生物群落结构和种群多样性时,不同的PCR反应程序获得的DGGE图谱分析结果存在明显差异。本试验结果表明,巢式PCR(巢式PCR和巢式-降落式PCR)反应处理下的DGGE图谱条带丰富,多样性高,能够比较全面和科学地反映样品中微生物群落结构和种群多样性。
