三个远交系豚鼠微卫星遗传结构分析
2018-12-03刘迪文谭海明陈雁虹
刘迪文,谭海明,陈雁虹,卫 振
(浙江大学实验动物中心,杭州 310058)
豚鼠由于其特有的生物学性状,成为药理学、毒理学、免疫学、眼耳科疾病研究及疫苗研发等领域常用的实验动物,目前我国用于实验的豚鼠主要是英国种花色豚鼠及哈脱莱DHP远交系白色豚鼠[1]。本课题组用上述两种豚鼠为亲本,培育出白色Zmu-1: DHP及黑色Zmu-2: DHP远交系豚鼠[2 - 3],其特征是融合了英国种豚鼠遗传杂合性和DHP豚鼠遗传稳定性的特点。由于育种过程中采取了近交及选择措施,则同一位点的等位基因进行分离,重组到不同品系中,也可能某些性状与毛色基因连锁的缘故,因此,Zmu-1: DHP、Zmu-2: DHP品系及英国种三种不同毛色的豚鼠,遗传及承载了不同的遗传特性[4-6],成为研究某些正常生物学或疾病发生机理较理想的动物模型资源。
远交系动物遗传质量常需要借助于遗传标记,通过计算群体遗传结构的数量遗传指标来评估。微卫星是一种稳定可靠的第二代DNA分子遗传标记,其序列长度的变化引起群体或个体遗传标记出现差异,表现为同一微卫星位点上,远交系动物基因呈现遗传多态性,而近交系动物则呈现一致性。优良品质的远交系动物应具备基因多态性及遗传稳定性,利用多个微卫星标记可以评价动物遗传质量,另外,筛选出与性状连锁的微卫星位点能够通过遗传定位分析,辅助定位目的性状基因。相对大小鼠,将微卫星技术用于豚鼠遗传结构分析的文章较少,目前仅见朱亮[7]、李芳芳[8]、刘迪文[9]及Burgos-Paz[10]等人报道。本文拟用45对多态性引物,分析本中心三个远交系豚鼠种群遗传结构的多态性,并从中筛选出与豚鼠特征性状连锁的微卫星位点,为评估豚鼠遗传质量,豚鼠品质资源维持及应用选择提供相关的遗传背景依据,同时为豚鼠优势性状基因定位及其机理研究奠定基础。
1 材料和方法
1.1 实验动物
英国种花色豚鼠,于70年代引自浙江农村,引进后长期封闭远交繁殖;Zmu-1: DHP白色豚鼠,2000年由英国种与HDP远交系白色豚鼠(1987年引自中检所)通过杂交-近交-远交方式育成的远交系;Zmu-2: DHP黑色豚鼠,Zmu-1: DHP豚鼠培育同时分离出的另一远交系。三个种群豚鼠年龄约为30日龄,体重约350 g,均为普通级,性别不论,使用数量分别为15只、20只及15只[SCXK (浙) 2012 - 0052]。豚鼠采用屏障设施开放饲养[SYXK (浙) 2012 - 0178],0.9 m × 0.6 m × 0.25 m塑料笼盒加铺灭菌木片及稻草作为垫料,饲喂颗粒饲料及自来水,并按实验动物使用的3R原则给予人道主义关怀。
1.2 主要试剂与仪器
DNA提取试剂盒(Takara)、PCR试剂盒(Promega)、聚丙烯酰胺(Sigma)、硝酸银(国产);冷冻离心机(Eppondorf)、Nanodrop-one(Thermo)、PCR仪(ABI)、电泳仪(Bio-Rad)、凝胶成像仪(Bio-Rad),等等。
1.3 实验方法
1.3.1 DNA模板提取
取三个种群豚鼠,每只心脏取血1 mL,肝素抗凝。用试剂盒提取豚鼠DNA,定量,ddH2O浓度调整至30 ng/μL。
1.3.2 模板PCR扩增
选取文献[11]介绍的其中45对引物,由Invitrogen公司合成。PCR配方成分:10× PCR Buffer(Mg2+)2 μL,dNTP(2.5 mmol/L)1 μL,rTaq(5 U/μL,Takara)0.2 μL,Primer F(10 mmol/L)0.5 μL,Primer R(10 mmol/L)0.5 μL,Template DNA(约50 ng)1 μL,ddH2O补足至20 μL。PCR反应条件:95℃预变性5 min;然后进入45个循环,每循环包括95℃变性30 s,退火30 s,72℃延伸30 s,各引物退火温度见文献[11];最后72℃延伸5 min,4℃维持扩增产物。
1.3.3 PCR产物电泳及条带分析
将PCR产物用12%聚丙乙酰胺凝胶电泳,电泳条件:电压120 V,电泳时间2 ~ 3 h,0.1% AgNO3溶液染色,对电泳凝胶照像。按照小鼠电泳条带标记法,标记豚鼠微卫星PCR产物电泳条带,从快到慢条带分别标记为a、b、c、d等。
1.4 统计学方法
根据每对引物对三个豚鼠种群扩增产物电泳条带的不同位置确定基因型,利用POPGENE 3.2软件统计三个群体在45个微卫星位点上的平均等位基因数(Number of alleles)、平均有效等位基因数(Number of effective alleles)、平均观测杂合度(Observed heterozygosity)、平均期望杂合度(Expected heterozygosity)、平均多态信息含量(Polymorphism information content)和平均Hardy-Weinberg平衡指数(Hardy-Weinberg Equilibrium index)。同时,采用Nei方法计算群体间的遗传距离和遗传分化指数Fst。使用MEGA 3.0软件,采取UPGMA方法根据三个群体的遗传距离进行聚类。
2 结果
2.1 微卫星位点PCR产物电泳条带
45对引物中两对引物扩增的PCR产物电泳图谱见图1。可见三个豚鼠种群的微卫星位点DNA条带清晰、分辨率较高,在种群及个体间呈现遗传多态性,说明这些位点可作为豚鼠遗传结构分析及遗传质量检测的标记。
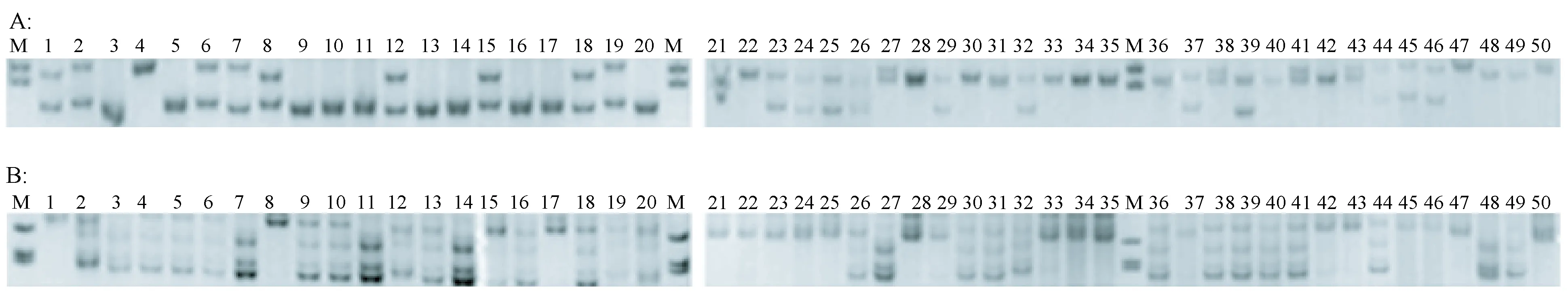
注:A:L74引物;B:D86引物。M:250 bp分子标记;泳道1 ~ 20:Zmu-1: DHP远交系;泳道21 ~ 35:英国种;36 ~ 50:Zmu-2: DHP远交系。图1 三个品系豚鼠微卫星PCR产物的电泳图谱Note. A: L74 Primer. B: D86 Primer. M: 250 bp Marker; Lane 1-20: Zmu-1: DHP outbred strain; Lane 21-35; England species; Lane 36-50: Zmu-2: DHP outbred strain.Figure 1 Electrophoretogram of PCR analysis of microsatellites in the three guinea pig groups
2.2 豚鼠微卫星位点基因型频率及数量统计
根据引物扩增产物作为电泳标记,统计豚鼠等位基因型频率、等位基因数及有效等位基因数,见表1。从表中可见,三个豚鼠种群的大部分微卫星位点呈多态性变化,所有位点电泳条带在2~7条之间,其中1个位点有7个等位基因,1个位点有4个等位基因,6个位点有3个等位基因,37个位点有2个等位基因。45个微卫星位点共有等位基因103个,平均等位基因数Na为2.29,平均有效等位基因数Ne为1.74。
2.3 三个豚鼠种群遗传多样性分析
计算得到的观测杂合度、期望杂合度及PIC见表2。从中可见,三个种群的总平均期望杂合度He为0.394,总平均观测杂合度Ho为0.212。另外,三个豚鼠种群之间的期望杂合度及观测杂合度差异无显著性(P> 0.05)。所有位点的多态信息含量PIC的可信度范围为0.0739 ~ 0.6443,平均为0.319,其中可信度> 0.5的高度多态性位点有2个,占4.44%,0.5 ~ 0.25的中度多态性位点有31个,占68.89%,< 0.25的低度多态性位点有12个,占26.67%。
2.4 豚鼠种群的哈代-温伯格遗传平衡分析、豚鼠群体间遗传分化
Hardy-Weinberg(HW)平衡卡方显著性分析、群体间遗传分化分析结果见表3。从表中可见,三个豚鼠种群总的HW平衡P值为0.1734,三个种群各自的平均P值均> 0.05,群体偏离遗传平衡程度不显著。但对每个位点的基因平衡状态进行分析,三个种群共至少有20个位点极显著偏离HW平衡,其中种群1有16个,种群2有13个,种群3有14个。平均偏离指数D为-0.407,表明群体存在HW遗传平衡偏离,同时又存在杂合子缺失现象。
三个种群的平均分化指数Fst为0.136,提示由遗传分化引起种群差异的因素是13.6%,86.4%的差异是种群内遗传不同引起,种群间属于中度分化;种群近交程度Fis为0.318,且大多数位点呈正值,说明这些种群内部的近交现象较严重;基因流Nm平均为5.835,表明育种过程中种群间存在基因交流,也说明基因交流是形成不同种群的主要原因,而遗传分化引起的因素较小。
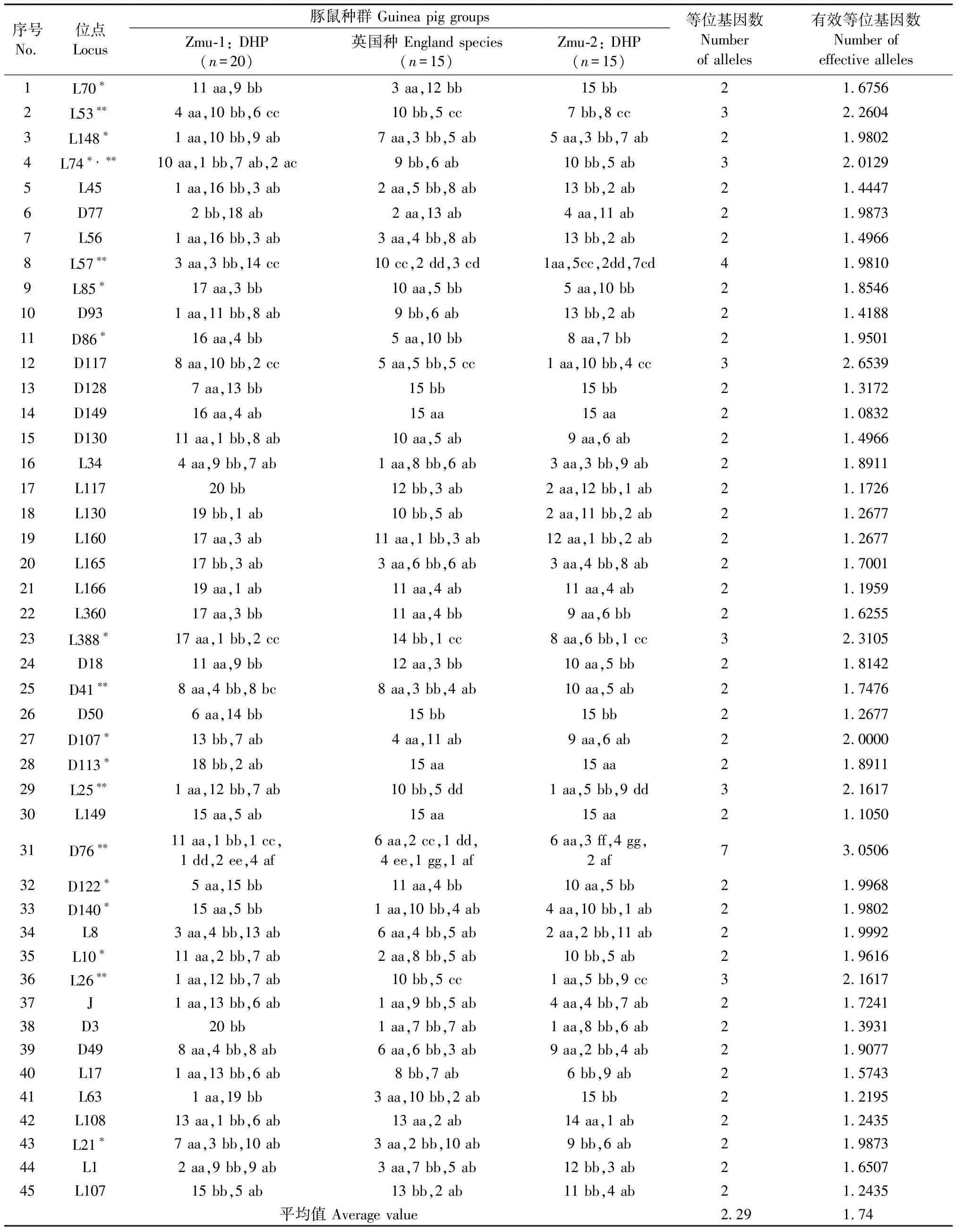
表1 三个豚鼠种群微卫星位点的基因型频率、等位基因数、有效等位基因数Table 1 Genotypic frequencies, number of alleles, and number of effective alleles of the microsatellite loci of the three guinea pig groups
注:*表示种群间有差异的微卫星位点;**表示有特殊等位基因的位点。
Note.*Differential locus between guinea pig groups;**Specific locus.
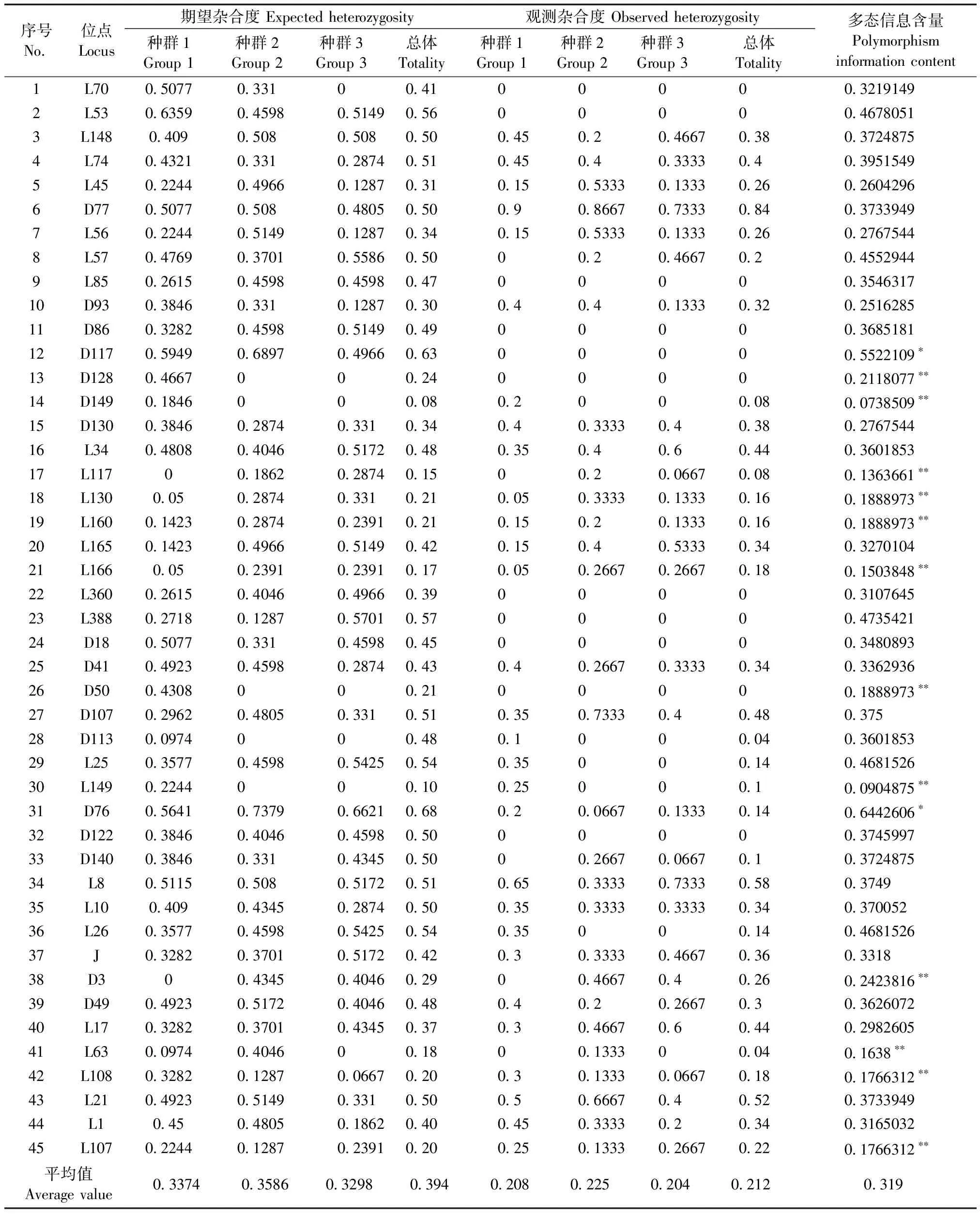
表2 三个品系豚鼠的观测杂合度、期望杂合度及PIC
注:种群1:Zmu-1: DHP豚鼠;种群2:英国种豚鼠;种群3:Zmu-2: DHP豚鼠。*高度多态性位点;**低度多态性位点;不标注:中度多态性位点。
Note. Group 1: Zmu-1: DHP; Group 2: England species; Group 3: Zmu-2: DHP.*High polymorphic locus;**Low polymorphic locus; No mark indicates a moderate polymorphic locus.
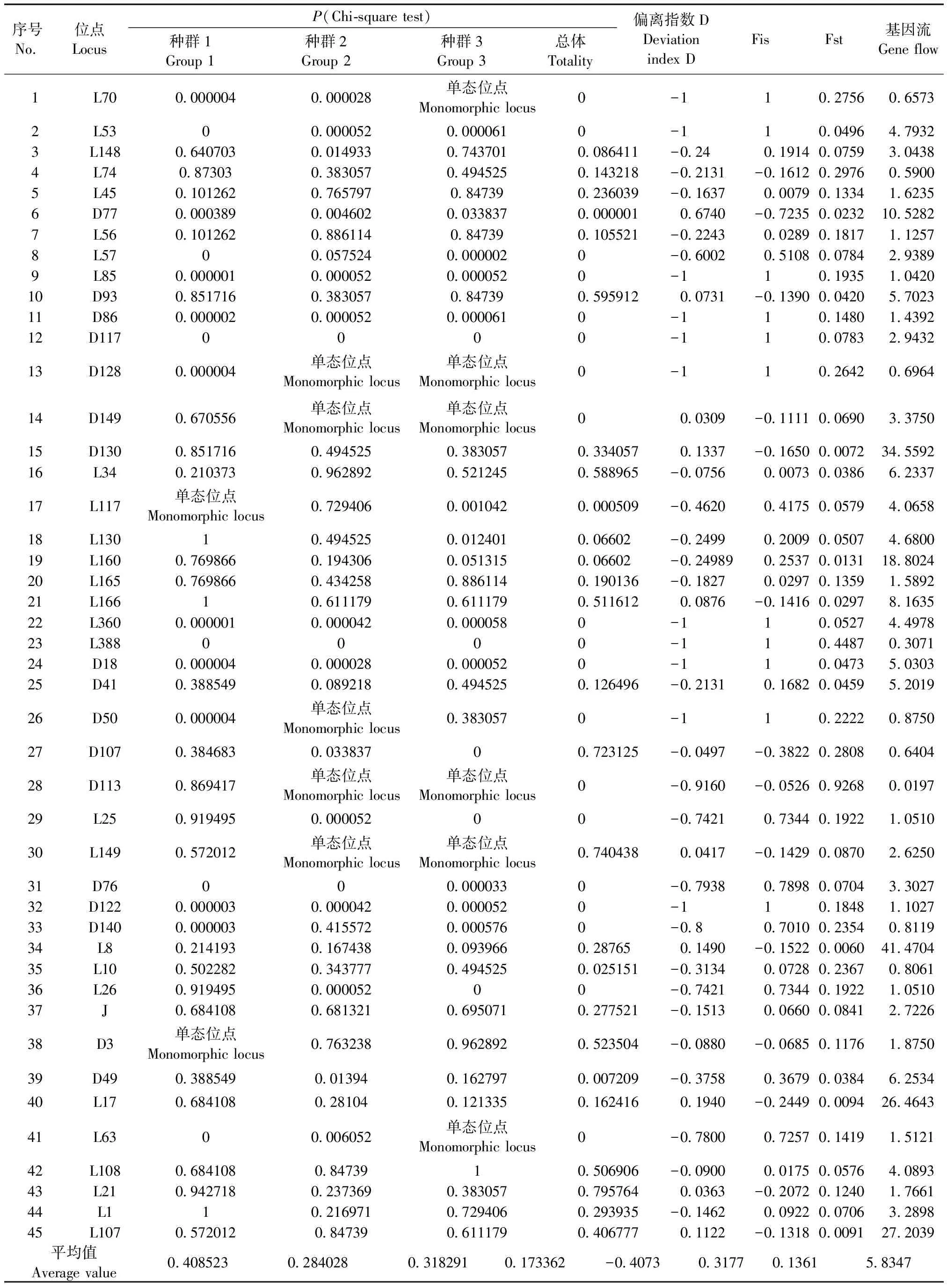
表3 豚鼠群体哈代-温伯格遗传平衡、Fis、Fst、基因流
注:星号以下为遗传距离,星号以上为遗传相似性系数。
Note. Under the asterisk: genetic distance; Above the asterisk: genetic similarity coefficient.
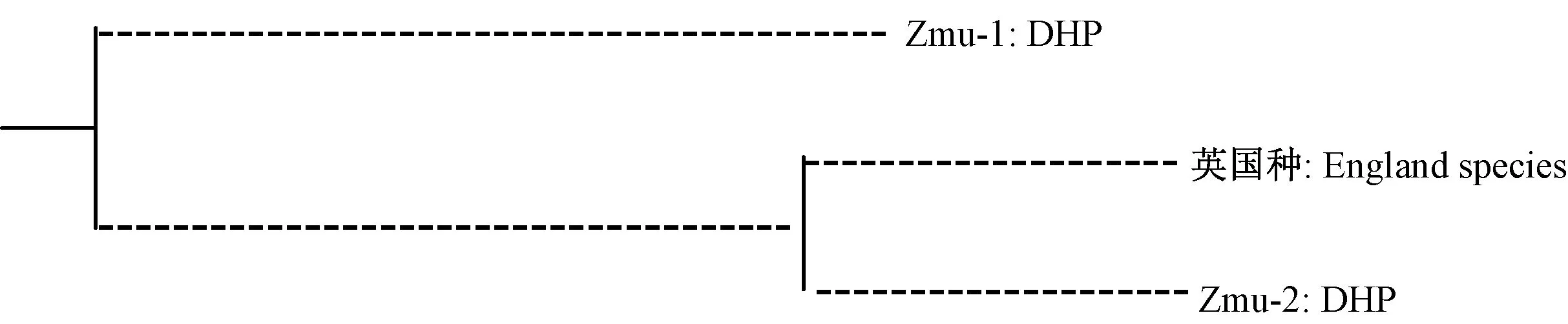
图2 基于遗传距离的三个种群的UPGMA聚类图Figure 2 UPGMA cluster graph of genetic distance
2.5 遗传相似度及遗传距离
三个种群间的Nei遗传距离及遗传相似度见表4,图2是三个种群的聚类图。从表图可见Zmu-1: DHP的遗传结构与英国种较近,与Zmu-2: DHP较远,英国种与Zmu-2: DHP两者最近。
3 讨论
实验动物的遗传类型决定了动物的用途及实验效果。从遗传学角度来说,近交系动物基因位点上所具有的基因单一,质量检测评定比较简单,而远交系动物遗传呈多样性,不同个体的同一位点上存在多种基因,使得其遗传质量评定变得比较复杂。远交系动物的遗传质量一般通过位点各等位基因的发生频率来评价,质量较好的远交系动物其基因位点杂合性较高,但群体同一位点多次测定的基因频率基本能够保持稳定。微卫星DNA通常是插入性状基因,与性状基因连锁的基因片段,可作为性状基因的遗传标记,通过检测微卫星的遗传信息,一般就能获知某些性状基因的遗传信息,或阐明个体及群体的遗传构成。本文探讨了我校三个远交系豚鼠种群的数量遗传特性,从表1、2可见,各微卫星位点平均基因数、总杂合度及多态信息量等指标判断,这些种群的遗传多态性较低,处于中等偏下水平。查其原因可能与种群数量较小、初期曾采用近亲繁殖及选种呈局限性等因素有关。朱亮等曾报道,他们的豚鼠群体处于中度遗传多态性水平。事实上,实验动物远交系动物不同于其他动物,群体较小,长期封闭繁殖,容易形成近亲繁殖。但本结果中多数微卫星位点的PIC较高,这些位点可用于远交系豚鼠遗传检测。
通过比较远交系动物种群间遗传结构,及鉴定其多态性变化是由群体内个体变异,还是群体间遗传分化引起的差异,对判断种群是同种还是异种很有必要。我校三个豚鼠种群中,Zmu-1: DHP(白色)及Zmu-2: DHP(黑色)种群都源自英国种豚鼠(花色),从表3中Fst、Fis、基因流值及表4遗传距离可见,三个种群的遗传相似度较高,并且纯合子个体较多,缺少杂合子个体,虽然种群间遗传处于中等分化水平,但通过基因交流阻止了遗传分化的影响,说明三个群体豚鼠属于一个种属下的不同亚群。相对来讲,英国种与Zmu-2: DHP遗传距离最近,与Zmu-1: DHP较近,而Zmu-1: DHP及Zmu-2: DHP相对较远。这种情况似乎与毛色相似度有关,说明花色和黑色都是有色豚鼠,遗传结构较近;白色与有色豚鼠属于完全不同的毛色,其群体间遗传结构则较远。从表3三个种群的具体位点看,少数位点极显著偏离遗传平衡,多数位点遗传偏离平衡不显著,但结合偏离指数看,整个种群仍处于不平衡水平。Klöting等[12]曾指出,实验动物封闭群Hardy-Weinberg平衡偏离是常见的问题。因此,为了提高本远交系豚鼠种群的遗传质量,今后必须扩大每个种群的个体数量,原先每个种群为30盒,现起码要扩充至50盒以上;要尽量遍布所有笼盒内选择豚鼠种子,每个笼盒只选择单一性别的个体留种,避免同胞兄妹繁殖;如有必要可引入其他远交系豚鼠,与现有豚鼠杂交后再采取远交封闭4代以上育种,以渗入其他基因,丰富种群的遗传多样性。
除了评价群体遗传结构外,某些微卫星位点还可作为性状基因的风向标,用于性状基因定位。课题组曾发现不同种群豚鼠在多个性状方面呈显著差异性[4, 6],例如白色豚鼠多为近视及对某些病毒敏感,而有色尤其是黑色豚鼠多为远视,并对某些病毒呈现抗性,说明这些性状差异与豚鼠品系遗传间存在相关性。微卫星是性状基因内含子中的重复序列,多态性微卫星位点的不同等位基因,常与某些相对性状或基因,如近视及远视性状及基因分别连锁。如果已知该微卫星的染色体位置,通过进一步种群间连锁分析,就可以定位性状基因,已有多篇文献报道了微卫星基因定位的研究结果[13-14]。因此,筛选到符合基因定位要求,即与相对性状连锁的多态性微卫星标记是关键。本文根据表1的豚鼠微卫星位点基因型频率,筛选出12个呈种群差异的位点(带*标记),可能作为基因定位的分子标记。另外,还筛选出7个白色豚鼠或有色豚鼠种群的特征性微卫星标记(带**标记),例如L26位点上两个有色豚鼠携带c基因,而白色豚鼠则缺少c基因,有可能作为特征性状基因的指示标记。
总之,本文报道了我校三个远交系豚鼠种群的遗传多态性,评估了这些豚鼠种群的遗传质量,同时报道了与不同种群或毛色性状相关的多态性微卫星位点,为今后豚鼠育种及遗传鉴定,优势性状基因定位及其遗传机理研究等提供基础资料。
