山西花生地方品种芽期耐寒性鉴定及SSR遗传多样性
2018-10-10白冬梅薛云云赵姣姣田跃霞权宝全姜慧芳
白冬梅 薛云云 赵姣姣 黄 莉 田跃霞 权宝全 姜慧芳,*
山西花生地方品种芽期耐寒性鉴定及SSR遗传多样性
白冬梅1,*薛云云1赵姣姣2黄 莉2田跃霞1权宝全1姜慧芳2,*
1山西省农业科学院经济作物研究所, 山西汾阳 032200;2中国农业科学院油料作物研究所, 湖北武汉 430062
低温寒害是引起花生产量和品质下降的主要因素之一, 培育和种植高产稳产耐寒性强的品种是降低低温寒害的理想途径。然而高耐寒性种质的缺乏和耐寒性鉴定的困难, 是限制耐寒性育种取得突破的主要原因。本研究对72份山西花生地方品种进行芽期耐寒性鉴定, 以其相对发芽率和相对发芽指数作为耐寒性评价指标, 初步将其分为高耐寒、耐寒、中感、敏感、高感5级。为了解山西花生地方品种耐寒性遗传多样性, 合理、高效利用耐寒型花生资源, 用多态性好的90对SSR引物评价了不同耐寒性花生品种, 结果显示, 参试品种遗传多样性存在较大差异, 在遗传距离为0.4时, 被聚类为三大类群。3份高耐寒品种和7份耐寒品种分别聚类到不同的3个类群中, 说明耐寒性花生遗传多样性丰富。
花生; 地方品种; 芽期耐寒性; 遗传多样性
花生(L)是我国重要的油料和经济作物, 在国内大宗油料作物中, 单位面积产量、产油量、种植效益以及国际市场竞争力等均具有明显优势[1]。目前国内食用油过度依赖进口, 油脂供给安全问题凸显, 进一步发展我国花生生产, 拓宽花生种植区域, 是满足不断增长的市场需求、提高农业生产效益、增加农民收入的迫切需要[2]。然而, 在花生播种后常常遭遇低温寒害, 种子活力受到损害, 出苗率明显降低, 轻者延缓花生萌发和幼苗生长发育, 重者发生大面积低温烂种, 缺苗断垄, 导致严重减产。高耐寒性种质的缺乏和耐寒性鉴定的困难, 是限制耐寒性育种取得突破的主要原因之一。
山西省地处黄土高原, 大部分地区海拔在1500 m以上, 年平均气温3~14℃。山西省花生种植历史悠久, 经过漫长的自然训化和人工选择, 孕育了丰富的变异类型和较强的抗逆性[3]。我们对山西省地方品种的农艺性状和品质性状详细分析表明, 山西省花生地方品种具有丰富的遗传多样性[4-5]。本研究对72份山西省地方花生品种进行了芽期耐寒性鉴定及SSR多态性检测, 分析研究其耐寒性遗传多样性, 为花生耐寒性育种及其相关研究提供了丰富的遗传资源和重要的理论依据。
1 材料与方法
1.1 参试花生品种
本试验选取山西省农业科学院经济作物研究所花生课题组征集的72份山西花生地方品种, 其中普通型51份、多粒型11份、珍珠豆型10份, 其编号、品名、类型见表1。

表1 72份山西供试花生地方品种编号、品名、类型
1.2 芽期耐寒性鉴定
采用BIC-300人工气候箱模拟大田气象条件, 进行芽期耐寒性筛选鉴定。选取2016年和2017年成熟饱满、种皮完整、大小一致的种子20粒, 采用纸间发芽, 3次重复, 常温浸种8 h后放入12℃人工气候箱培养72 h, 然后2℃低温胁迫暗培养96 h, 再调到10℃暗培养72 h, 最后置于25℃人工气候箱恒温发芽, 以常温浸种8 h后在25℃发芽为对照, 每天计录发芽种子数, 18 d后计算各品种的相对发芽率(%)和相对发芽指数(GI), GI = ∑(Gt/Dt)。Gt为第天的发芽种子数, Dt为相对应的发芽日数。
1.3 SSR引物及PCR扩增
本研究在中国农业科学院油料作物研究所花生生物技术实验室进行, 所用的154对SSR引物由该实验室提供。选取花生健壮幼叶, 采用优化的CTAB法[6]提取基因组DNA, 用1.0%琼脂糖凝胶电泳检测DNA质量, 紫外分光光度计测定其浓度并统一调整DNA浓度至100 ng L–1。PCR反应体系为10 μL, 含Mix 2.5 μL (由北京全式金生物技术有限公司生产)、ddH2O 5 μL、10~40 pmol L–1引物对0.5 μL、10~20 ng模板DNA 2 μL。扩增条件为94℃预变性3 min; 93℃变性30 s, 55~65℃(不同引物退火温度不同)退火30 s, 72℃延伸1 min, 共32个循环; 72℃延伸10 min。PCR产物经6%变性聚丙烯酰胺凝胶电泳检测, 硝酸银染色, 显影, 扫描保存。
1.4 数据统计分析
根据PCR扩增结果, 以0、1、C统计SSR扩增带型, 在相同迁移率位置上, 有带记为“1”, 无带记为“0”, 缺失记为“C”, 建立相应的数据库, 用Microsoft Excel 2013处理基本数据。再根据不同分析软件的格式要求作相应转换。用Powermarker-V 3.25 软件[7-11]计算每对引物的多样性参数, 包括等位基因数()、主基因频率(MAF)、基因多样性指数()、多态性信息含量指数(PIC), 利用非加权组平均法(UPGMA)进行聚类分析, 生成聚类图; 用Popgene Ver.1.32 分析Shannon’s信息指数(I)。
2 结果与分析
2.1 芽期耐寒性品种分析
吸胀后突遇低温胁迫, 各种质材料间表现出明显的差异, 有的材料受低温影响小, 能正常发芽, 生长状况良好。有的材料耐低温能力很差, 低温胁迫后, 种子霉烂, 不能正常发芽生长。其他材料介于这两种类型之间, 萌发情况参差不齐(图1)。
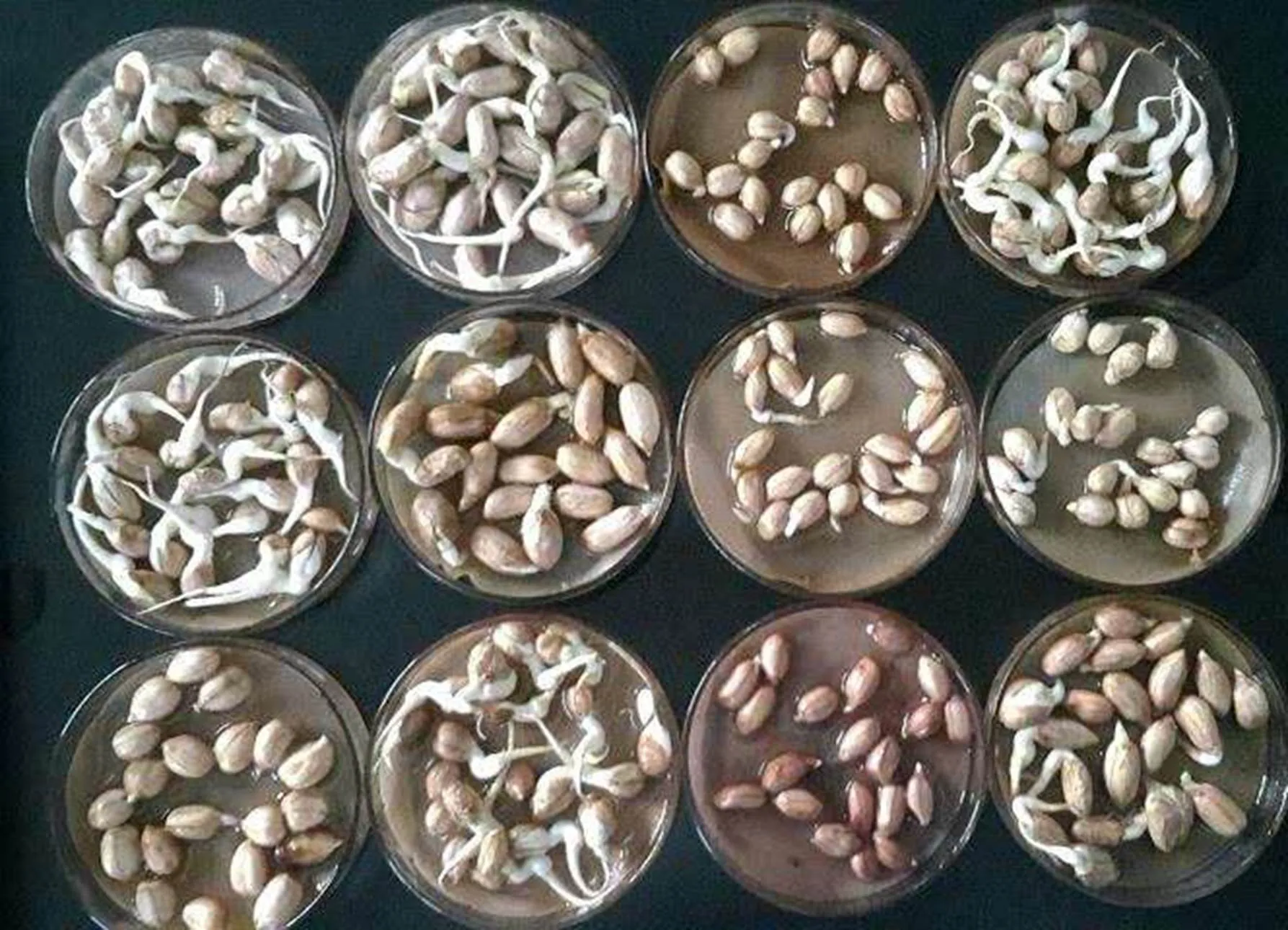
图1 低温胁迫后部分花生品种萌发情况
由表2可见, 72份材料相对发芽率变幅范围为6.86%~96.50%, 相对发芽指数变幅范围为12.87%~ 94.46%, 表现最好的临县多粒、新降大花生、榆次花生3份材料的相对发芽率与相对发芽指数均>90%, 可作为高耐寒材料; 而表现最差的2份材料侯马大粒和文水多粒的相对发芽率低于10%, 相对发芽指数低于20%, 可作为高感材料。按照相对发芽率和相对发芽指数的变幅范围, 将参试的72份材料耐寒性分为5级。一级为高耐寒材料, 相对发芽率与相对发芽指数均>90%, 有3份, 占总材料的4.17%, 其中珍珠豆型1份, 普通型2份; 二级为耐寒材料, 相对发芽率>90%, 80%<相对发芽指数<90%, 有7份, 占总材料的9.72%, 其中珍珠豆型1份, 多粒型3份, 普通型3份; 三级为中间材料, 50%<相对发芽率<90%, 50%<相对发芽指数<80%, 有17份, 占总材料的23.61%, 其中有珍珠豆型1份, 多粒型2份, 普通型14份; 四级为敏感材料, 10%<相对发芽率<50%, 20%<相对发芽指数<50%, 有43份, 占总材料的59.72%, 其中珍珠豆型7份, 多粒型5份, 普通型31份; 五级为高感材料, 相对发芽率<10%, 相对发芽指数<20%, 有2份, 占总材料的2.78%, 其中多粒型1份, 普通型1份。四级和五级材料受低温胁迫, 种子霉烂, 活力丧失, 不能正常发芽生长, 严重影响出苗, 形成田间缺苗断垄影响产量。鉴定筛选出的高耐寒和耐寒材料中, 包括2份珍珠豆型, 3份多粒型, 5份普通型, 说明花生耐寒性与品种植物学属性关系不大。
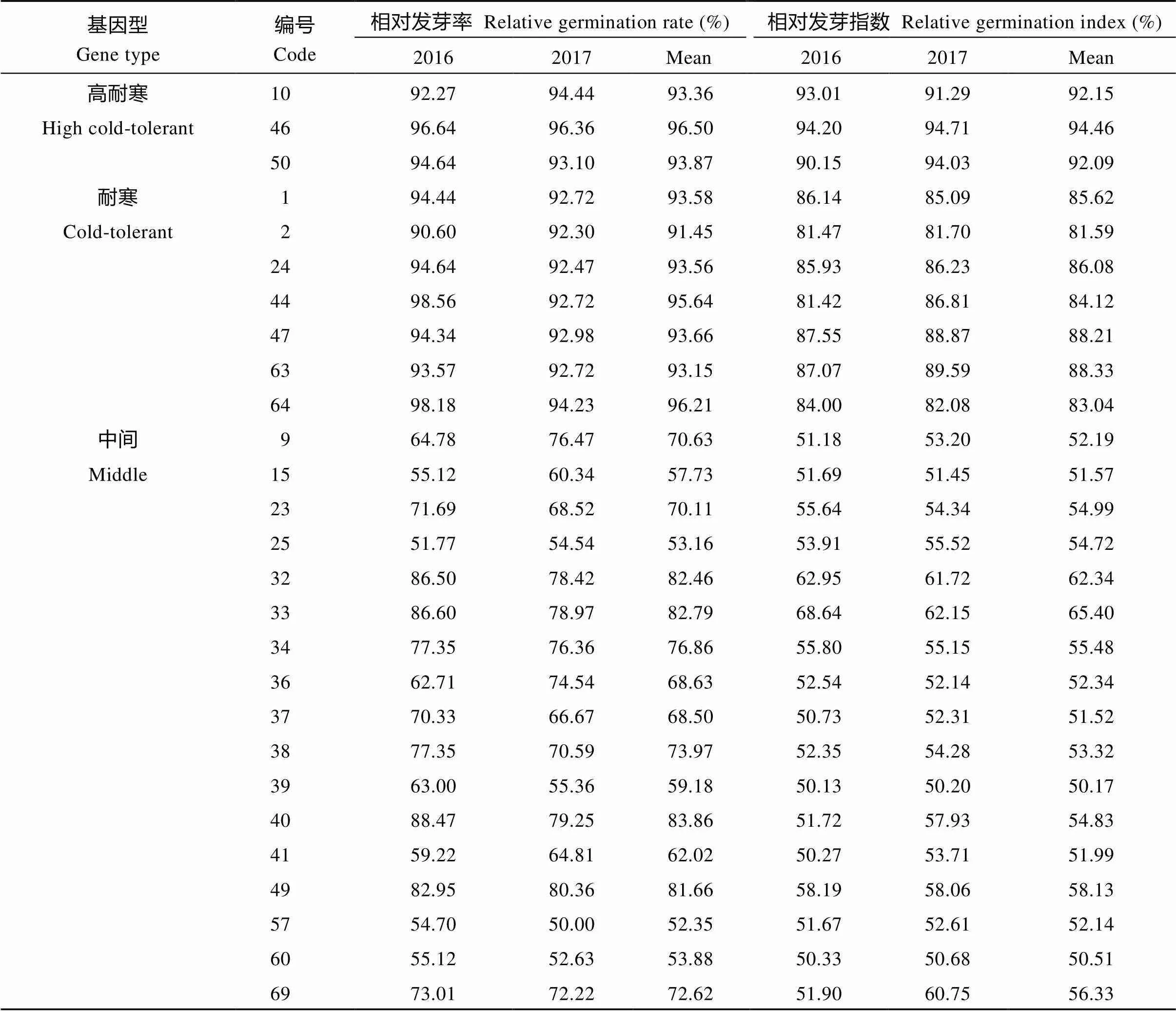
表2 供试品种的耐寒性鉴定
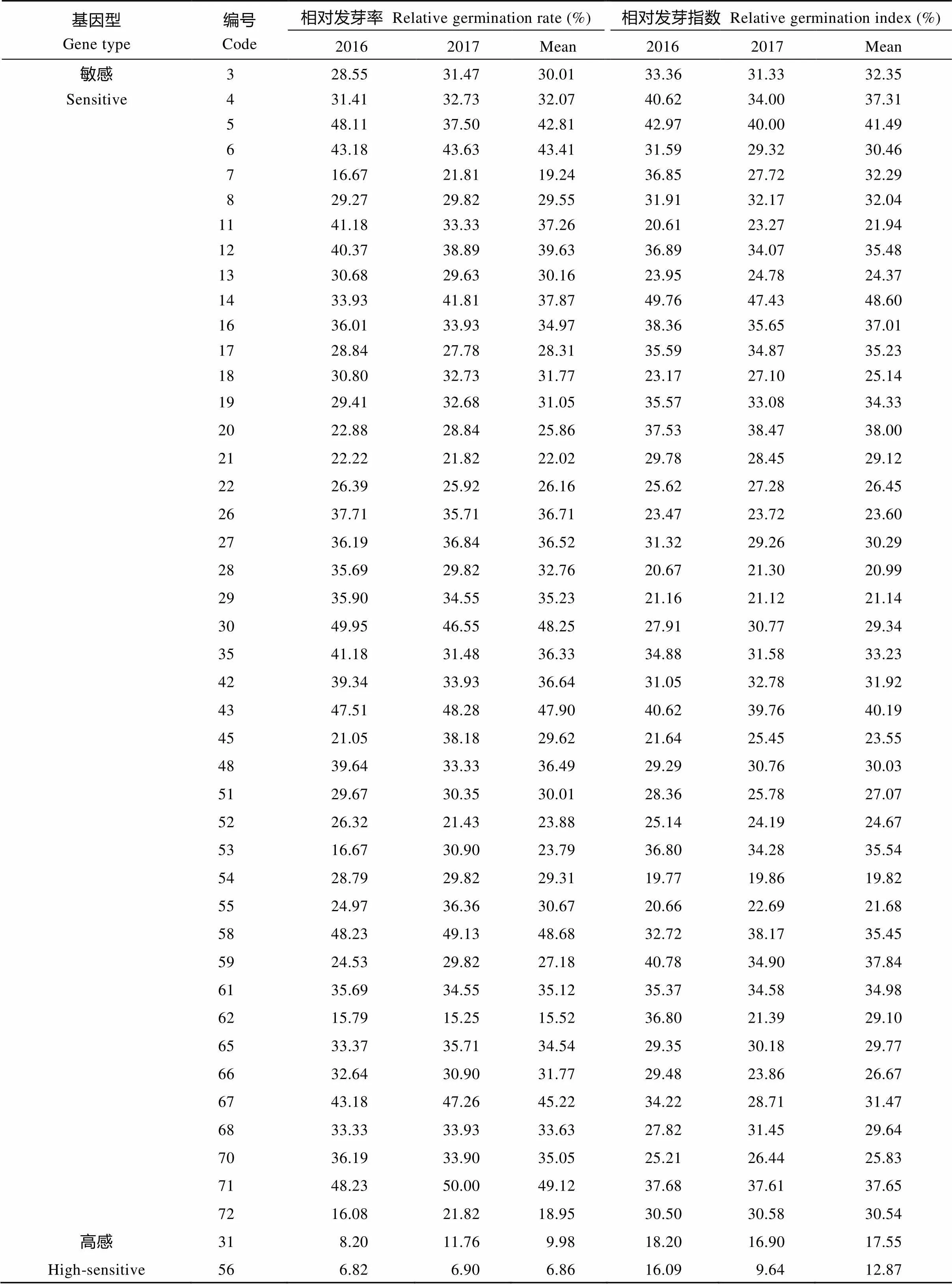
(续表2)
2.2 SSR标记多态性分析
选用8份不同地理来源、不同类型的花生品种基因组DNA, 用154对SSR引物进行PCR扩增。有101对引物扩增出稳定、清晰可辨的谱带, 有效扩增比率为47.3%, 其中90对具有多态性, 多态性引物比率为89.1% (图2)。最终确定了谱带清晰稳定且具多态性的90对SSR引物用于本研究。
从表3可以看出, 72份山西省地方品种在90个SSR标记中扩增出317个等位基因数(), 每对引物平均扩增出3.5222个等位基因, 不同引物每个位点等位基因数差异很大, 变化范围2~8个, 检测到的等位基因最多的引物是A07B516和A08A90A。主基因频率(MAF)变化幅度为0.2917 (A07B34)~ 0.8986 (A02B62), 平均0.6834。基因多样性指数(GD)变化幅度为0.1823 (A02B62)~0.7711 (A08A90), 平均0.4537; 多态信息含量指数(PIC)变异范围为0.1657 (A02B62)~0.7378 (A08A90), 平均0.4047; Shannon’s信息指数(I)变异范围为0.3283 (A02B62)~ 1.6734 (A08A90), 平均0.8092。表明本研究所用的SSR位点的遗传多样性丰富, 显示山西省花生地方品种遗传多样性方面存在较大差异。其中16对引物最有效, Shannon’s信息指数均在1以上, 综合比较各参数, 等位基因数值多的SSR位点, 基因多样性指数、多态信息含量指数和Shannon’s信息指数的值也大, 三者变化趋势一致, 因此, 基因多样性指数、多态信息含量指数和Shannon’s信息指数对于遗传多样性分析, 更具有可靠的实际意义。
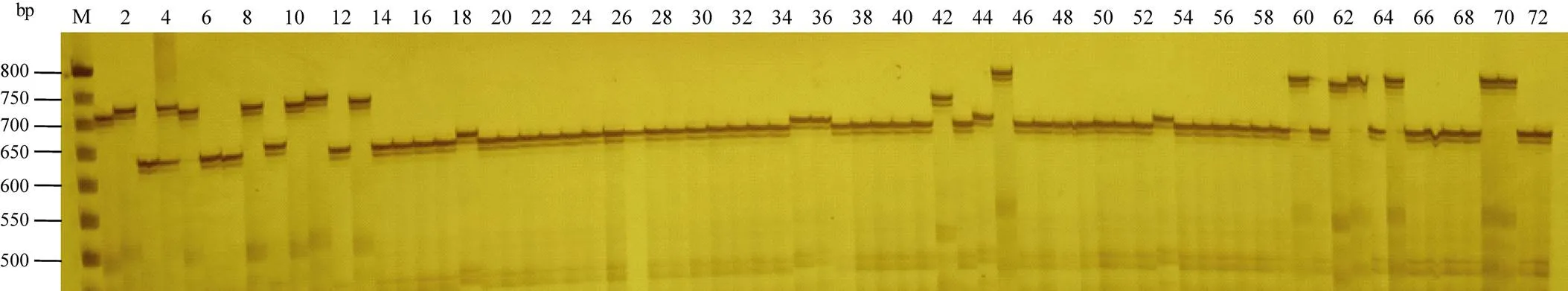
图2 引物A06B209在72份花生品种中的扩增
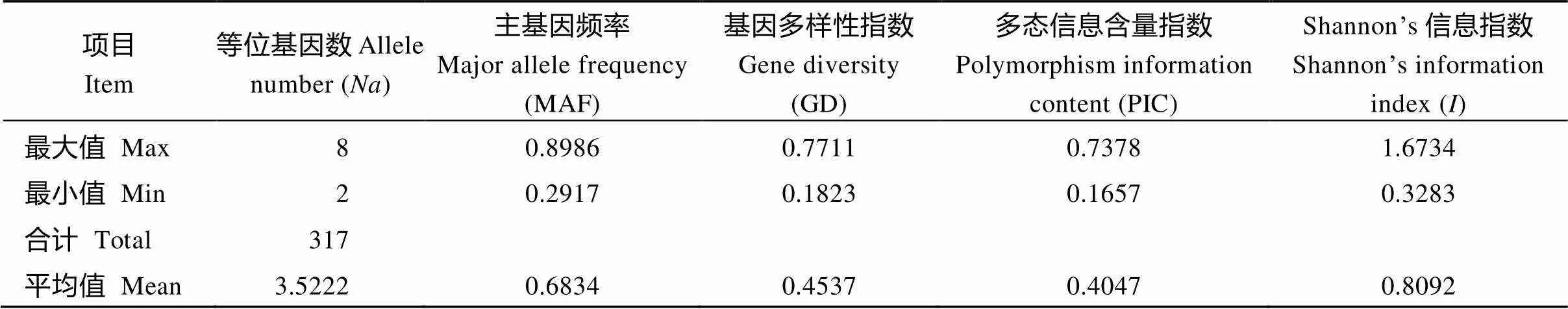
表3 90个SSR标记的遗传参数
2.3 不同耐寒性花生品种的遗传多样性
根据SSR标记数据, 利用Powermarker-V3.25软件, 采用非加权组平均法(UPGMA)聚类分析表明, 72份参试材料在遗传距离为0.4时, 被分为三大类群。第I类群是以多粒型为主的10个品种, 其中包括2份耐寒材料; 第II类是以普通型为主51个品种, 其中包括2份高耐寒材料和3份耐寒材料; 第III类是以珍珠豆型为主的11个品种, 其中包括1份高耐寒材料和2份耐寒材料, 说明花生耐寒性遗传多样性丰富(图3)。耐寒品种汾西小粒和长子花生亲缘关系最远, 而高耐寒品种新降大花生和高感品种侯马大粒亲缘关系最近, 说明高耐品种和高感品种并不是亲缘关系最远, 耐寒品种间也不是亲缘关系最近。
3 讨论
花生芽期寒害是引起花生产量和品质下降的主要因素之一, 山西地处黄土高原高海拨区, 其地方品种具有丰富的遗传多样性, 可能蕴含着高耐寒型基因。因此, 本研究以山西花生地方品种为试验材料, 采用人工气候箱中模拟大田气象条件, 可以不受季节气候条件等因素的限制而开展鉴定工作, 从而加快鉴定进度。而且人工气候箱设备的培养条件(光照、温度、水分等)易于控制且重复性好, 鉴定出的耐寒性花生品种在田间更具有适应性。本研究中耐寒性鉴定设置了2℃低温胁迫, 这是经过多次的、几个温度梯度的试验得出来的结果, 温度低于2℃, 多数试材会被淘汰, 温度高会出现大部分材料的正常发芽出苗而达不到选择的目的, 这与封海胜[12]所做的花生种子吸胀期间耐低温性鉴定是一致的。刘海龙等[13]利用花生种质资源耐低温表型方法鉴定花生种质资源耐低温属性。吕建伟等[14]以花生相对出苗率将花生种质资源划分为不耐、低耐、中耐、高耐4个等级。唐月异等[15]以露白率及芽长/种长作为鉴定花生吸胀期耐寒性的指标。目前对于花生不同阶段耐寒性评价没有统一的标准, 本研究结合前人研究基础, 把相对发芽率和相对发芽指数作为鉴定花生品种芽期耐寒性的指标, 初步将72份花生品种耐寒性分为高耐寒、耐寒、中感、敏感、高感5级。本研究鉴定筛选出的高耐寒和耐寒材料中, 包括2份珍珠豆型、3份多粒型和5份普通型, 表明花生耐寒性与品种植物学属性关系不大, 这与唐月异等[15]研究得出的结论一致。常硕其等[16]对杂交品种亲本及后代进行耐寒性鉴定时, 发现水稻的耐寒性是可以通过杂交稳定遗传的, 后代与亲本的耐寒性呈正相关。康旭梅等[17]提出, 在综合考虑后代优良性状的同时, 只要保证亲本之一的耐寒性较强, 就有可能保证F1耐寒性强且具有优良的综合性状。这些研究足以证明筛选耐寒性资源作为杂交亲本培育耐寒性杂交后代的育种方法是完全可行的。本研究筛选出的临县多粒、新降大花生、榆次花生等耐寒品种可以作为亲本进行杂交培养耐寒性后代, 为选育高产高油酸耐寒性强新品种及其相关研究提供材料基础和理论依据。
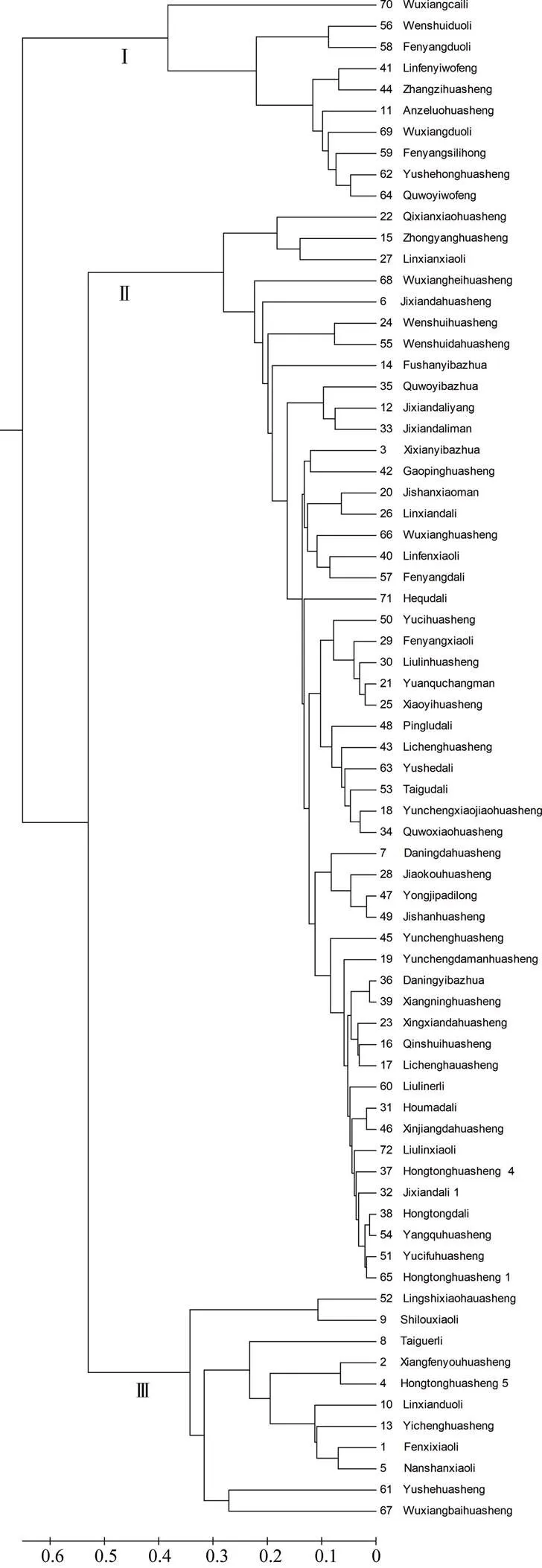
图3 基于SSR标记的72份山西花生地方品种的聚类分析图
分子标记是揭示花生遗传多样性的有效手段, 国内外研究者曾用RAPD[18]、SSR[19-26]、AFLP[27-28]等分子标记对花生种质资源遗传多样性广泛研究。其中, SSR 以共显性好、多态性丰富成为花生最实用的检测标记。本研究所用的SSR位点的遗传多样性丰富, 显示出山西省花生地方种质资源遗传多样性方面存在较大差异。综合比较各遗传参数, 等位基因数值多的SSR位点, 基因多样性指数(GD)、多态信息含量指数(PIC)和Shannon’s信息指数()的值也大, 三者变化趋势一致。因此, 基因多样性指数、多态信息含量指数和Shannon’s信息指数对于遗传多样性分析, 更具有可靠的实际意义, 这一结果与花生[11]、豌豆[10,29-30]和小扁豆[31]上的研究结果一致。在遗传距离为0.4时, 参试品种被聚为三个类群, 类群I以多粒型为主, 类群II以普通型为主, 类群III是珍珠豆型为主, 说明SSR 标记的聚类与花生植物属性关系密切, 与花生耐寒性特性和地域特性关系不大, 3份高耐寒品种和7份耐寒品种分布于3个不同的类群中, 这可能与控制耐寒性的遗传因子在不同品种间存在差异有关。本研究所用材料仅限于山西省花生地方品种, 分析花生耐寒性遗传特性的分子基础, 还需要更多的验证分析和丰富的育种材料, 以取得更加可靠的结果, 从而为花生耐寒性育种及其相关研究提供理论依据和材料基础。
4 结论
通过对72份山西地方品种芽期耐寒性鉴定, 初步筛选出3份高耐寒品种和7份耐寒品种, 分布在三个不同的类群中, 说明花生耐寒品种遗传多样性丰富。
[1] 禹山林. 中国花生品种及其系谱. 上海: 上海科学技术出版社, 2008.pp 1–5 Yu S L. Peanut Varieties and Pedigree in China. Shanghai: Shanghai Scientific and Technical Publishers, 2008. pp 1–5 (in Chinese)
[2] 万书波. 中国花生栽培学. 上海: 上海科学技术出版社, 20013. pp 20–21 Wan S B. Peanut Cultivation in China. Shanghai: Shanghai Scientific and Technical Publishers, 2013. pp 20–21 (in Chinese)
[3] 沈浩, 刘登义. 遗传多样性概述. 生物学杂志, 2001, 18(2): 4–7 Shen H, Liu D Y. Summary of genetic diversity., 2001, 18(2): 4–7 (in Chinese with English abstract)
[4] 白冬梅, 王国桐, 薛云云, 田跃霞, 权宝全. 山西省地方花生品种农艺性状的遗传多样性分析. 山西农业科学, 2014, 42: 542–547 Bai D M, Wang G T, Xue Y Y, Tian Y X, Quan B Q. Analysis of genetic diversity of Shanxi peanut landrace based on agronomic traits., 2014, 42: 542–547 (in Chinese with English abstract)
[5] 白冬梅, 王国桐, 薛云云, 田跃霞, 权宝全. 山西省地方花生种质资源品质性状的综合评价. 中国农学通报, 2014, 30(24): 187–193 Bai D M, Wang G T, Xue Y Y, Tian Y X, Quan B Q. Comprehensive evaluation of peanut germplasm resources in Shanxi province based on quality traits., 2014, 30(24): 187–193 (in Chinese with English abstract)
[6] Cuc L M, Mace E S, Crouch J H, Quang V D, Long T D, Varshney R K. Isolation and characterization of novel microsatellite markers and their application for diversity assessment in cultivated ground–nut (L)., 2008, 8: 55
[7] 陈本银, 姜慧芳, 廖伯寿, 任小平, 黄家权, 雷永, 王圣玉. 野生花生种质的 SSR 遗传多样性. 热带亚热带植物学报, 2008, 16: 296–303 Chen B Y, Jiang H F, Liao B S, Ren X P, Huang J Q, Lei Y, Wang S Y. Genetic diversity analysis ofgerplasm by SSR.,2008, 16: 296–303 (in Chinese with English abstract)
[8] Lin K. Muse S V. PowerMarker: an integrated analysis environment for genetic marker analysis., 2005, 21: 2128–2129
[9] Ferguson M E, Burow M D, Schulze S R, Bramel P J, Paterson A H, Kresovich S, Mitchell S. Microsatellite identification and characterization in peanut (L.)., 2004, 108: 1064–1070
[10] He G H, Meng R H, Newman M, Gao G Q, Pittman R N, Prakash C S. Microsatellites as DNA markers in cultivated peanut (L.)., 2003, 3: 3
[11] 任小平, 张晓杰, 廖伯寿, 雷永, 黄家权, 陈玉宁, 姜慧芳. ICRISAT 花生微核心种质资源SSR 标记遗传多样性分析. 中国农业科学, 2010, 43: 2848–2858 Ren X P, Zhang X J, Liao B S, Lei Y, Huang J Q, Chen Y N, Jiang H F. Analysis of genetic diversity in ICRISAT mini core collection of peanut (L.) by SSR markers.2010, 43: 2848–2858 (in Chinese with English abstract)
[12] 封海胜. 花生种子吸胀期间耐低温性鉴定. 中国油料, 1991, (1): 67–70 Feng H S. Identification on low temperature resistance of peanut seed during imbibition stage., 1991, (1): 67–70 (in Chinese)
[13] 刘海龙, 陈小姝, 杨富军, 白冬梅, 孙晓苹, 吕永超, 任小平, 姜慧芳, 高华援. 花生种质资源耐低温表型鉴定方法研究. 花生学报, 2017, 46: 20–25 Liu H L, Chen X S, Yang F J, Bai D M, Sun X P, Lyu Y C, Ren X P, Jiang H F, Gao H Y. Research of identification method on low temperature resistance of peanut germpasm resources phenotype., 2017, 46: 20–25 (in Chinese with English abstract)
[14] 吕建伟, 马天进, 李正强, 陈锋, 姜慧芳. 花生种质资源出苗期耐低温性鉴定方法及应用. 花生学报, 2014, 43(3): 13–18 Lyu J W, Ma T J, Li Z Q, Chen F, Jiang H F. Identification method of low temperature tolerance on peanut germplasm resource., 2014, 43(3): 13–18 (in Chinese with English abstract)
[15] 唐月异, 王传堂, 高华媛, 凤桐, 张树伟, 王秀贞, 张建成, 禹山林. 花生种子吸胀期间耐低温性及其与品质性状的相关研究. 核农学报, 2011, 25: 436–442 Tang Y Y, Wang C T, Gao H Y, Feng T, Zhang S W, Wang X Z, Zhang J C, Yu S L. Low temperature tolerance during seed imbibition and its relationship to main quality in peant.2011, 25: 436–442 (in Chinese with English abstract)
[16] 常硕其, 邓启云, 罗祎, 陈小龙. 超级杂交稻及其亲本的耐冷性研究. 杂交水稻, 2015, 30(1): 51–57 Chang S Q, Deng Q Y, Luo W, Chen X L. Research on low temperature tolerance in super hybrid rice and parent., 2015, 30(1): 51–57 (in Chinese with English abstract)
[17] 康旭梅, 李小湘, 潘孝武, 刘文强. 普通野生稻耐冷性研究进展. 杂交水稻, 2017, 32(2): 1–5 Kang X M, Li X X, Pan X W, Liu W Q. Research progress on low temperature tolerance inGriff.2017, 32(2): 1–5 (in Chinese with English abstract)
[18] Subramanian V, Gurtu S, Rao R C N, Nigam S N. Identification of DNA polymorphism in cultivated groundnut using random amplified polymorphic DNA (RAPD) assay., 2000, 43: 656–660
[19] Tang R H, Gao G Q, He L Q, Han Z Q, Shan S H, Zhong R C, Zhou C Q, Jiang J, Li Y R, Zhuang W J. Genetic diversity in cultivated groundnut based on SSR markers., 2007, 34: 449–459
[20] 林茂, 李正强, 郑治洪, 吕建伟, 马天进, 李超, 阚健全. 贵州省花生地方品种的遗传多样性. 作物学报, 2012, 38: 1387–1396 Lin M, Li Z Q, Zheng Z H, Lyu J W, Ma T J, Li C, Kan J Q. Genetic diversity of peanut landraces in Guizhou province., 2012, 38: 1387–1396 (in Chinese with English abstract)
[21] 姜慧芳, 任小平, 廖伯寿, 黄家权, 雷永, 陈本银, Guo B Z, Holbrook C C, Upadhyaya H D. 中国花生核心种质的建立及与ICRISAT 花生微核心种质的比较. 作物学报, 2008, 34: 25–30Jiang H F, Ren X P, Liao B S, Huang J Q, Lei Y, Chen B Y, Guo B Z, Holbrook C C, Upadhyaya H D. Peanut core collection established in China and compared with ICRISAT mini core collection., 2008, 34: 25–30 (in Chinese with English abstract)
[22] Han Z Q, Gao G Q, Wei P X, Tang R H, Zhong R C. Analysis of DNA polymorphism and genetic relationships in cultivated peanut (L.) using micro-satellite markers., 2004, 30: 1097–1101
[23] 洪彦彬, 梁炫强, 陈小平, 林坤耀, 周桂元, 李少雄, 刘海燕. 花生栽培种(L.)类型间遗传差异的SSR分析. 分子植物育种, 2008, 6: 71–78 Hong Y B, Liang X Q, Chen X P, Lin K Y, Zhou G Y, Li S X, Liu H Y. Genetic differences in peanut cultivated types (L.) revealed by SSR polymorphism.2008, 6: 71–78 (in Chinese with English abstract)
[24] Jiang H F, Liao B S, Ren X P, Lei Y, Mace E, Fu T D, Crouch J H. Comparative assessment of genetic diversity of peanut (L.) genotypes with various levels of resistance to bacteria wilt through SSR and AFLP analyses., 2007, 34: 544–554
[25] 黄莉, 任小平, 张晓杰, 陈玉宁, 姜慧芳. ICRISAT 花生微核心种质农艺性状和黄曲霉抗性关联分析. 作物学报, 2012, 38: 935–946 Huang L, Ren X P, Zhang X J, Chen Y N, Jiang H F. Association analysis of agronomic traits and resistance to aspergillus flavus in the ICRISAT peanut mini-core collection., 2012, 38: 935–946 (in Chinese with English abstract)
[26] Krishna G K, Zhang J, Burow M, Pittman R N, Delikostadinov S G, Lu Y Z, Puppala N. Genetic diversity analysis in peanut (L.) using microsatellite markers., 2004, 9: 685–697
[27] He G H, Channapatna P. Evaluation of genetic relationships among botanical varieties of cultivated peanut (L.) using AFLP markers., 2001, 48: 347–352
[28] He G H, Meng R H, Gao H, Guo B Z, Gao G Q, Melanie N, Roy N P, Prakash C S. Simple sequence repeat markers for botanical varieties of cultivated peanut (L.)., 2005, 142: 131–136
[29] 宗绪晓, 关建平, 王述民, 刘庆昌. 中国豌豆地方品种SSR标记遗传多样性分析. 作物学报, 2008, 34: 1330–1338 Zong X X, Guan J P, Wang S M, Liu Q C. Genetic diversity among Chinese pea (L.) landraces revealed by SSR markers.2008, 34: 1330–1338 (in Chinese with English abstract)
[30] 宗绪晓, Ford R, Robert R R, 关建平, 王述民. 豌豆属() SSR标记遗传多样性结构鉴别与分析. 中国农业科学, 2009, 42: 36–46 Zong X X, Ford R, Robert R R, Guan J P, Wang S M. Identification and analysis of genetic diversity structure within Pisum genus based on microsatellite markers., 2009, 42: 36–46 (in Chinese with English abstract)
[31] 刘金, 关建平, 徐东旭, 张晓艳, 顾竟, 宗绪晓. 小扁豆种质资SSR标记遗传多样性及群体结构分析. 作物学报. 2008, 34: 1901–1909 Liu J, Guan J P, Xu D X, Zhang X Y, Gu J, Zong X X. Analysis of genetic diversity and population structure in Lentil (Medik.) germplasm by SSR markers., 2008, 34: 1901–1909 (in Chinese with English abstract)
Identification of Cold-tolerance During Germination Stage and Genetic Diversity of SSR Markers in Peanut Landraces of Shanxi Province
BAI Dong-Mei1,*, XUE Yun-Yun1, ZHAO Jiao-Jiao2, HUANG Li2, TIAN Yue-Xia1, QUAN Bao-Quan1, and JIANG Hui-Fang2,*
1Industrial Crops Research Institute, Shanxi Academy of Agricultural Sciences, Fenyang 032200, Shanxi, China;2Oil Crops Research Institute, Chinese Academy of Agricultural Sciences, Wuhan 430062, Hubei, China
Cold injury is one of the main factors causing yield and quality decline in peanut. Cultivating and planting varieties with high and stable yield and strong cold tolerance is an ideal way to reduce cold injury, However, the lack of high cold tolerant germplasm and the difficulty of cold tolerance identification are the main reasons to limit the breakthrough of cold tolerance breeding. In this study, 72 local peanut varieties in Shanxi province were identified for cold tolerance at germination stage. Based on their relative germination rate and relative germination index, the cold tolerance of 72 peanut cultivars we preliminarily divided the 72 peanut cultivars into five grades, namely high-cold-tolerant type, cold-tolerant type, middle type, sensitive type, and high sensitive type. Ninety pairs of SSR primers with good polymorphism were used to evaluate the peanut cultivars with different cold-tolerance levels, and to examine the genetic diversity of cold-tolerant peanut landraces in Shanxi province making rational and efficient use of cold tolerant peanut resources. The tested cultivars were highly different in genetic diversity and were clustered into three groups with genetic distance of 0.4. Three high-cold-tolerant cultivars and seven cold-tolerant cultivars were clustered into three different groups, indicating that the cold-tolerant peanut varieties are rich in genetic diversity.
peanut; landraces; cold-tolerance at germination stage; genetic diversity
2018-02-24;
2018-06-12;
2018-07-03.
10.3724/SP.J.1006.2018.01459
白冬梅, E-mail: baidm1221@163.com; 姜慧芳, E-mail: peanut @oilcrops.com, Tel: 027-86711550
本研究由国家现代农业产业技术体系建设专项(CARS-13), 山西省农业科学院生物育种工程项目(17yzgc051)和山西省农业科学院科技自主创新能力提升工程项目(2017zzlx-07)资助。
This study was supported by the China Agricultural Research System (CARS-13), the Biological Breeding Project of Shanxi Academy of Agricultural Sciences (17yzgc051), and the Science and Technology Independent Innovation Ability Enhancement Project of Shanxi Academy of Agricultural Sciences (2017zzlx-07).
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20180702.1803.008.html
