玉米ZmNAOD基因的克隆与功能分析
2018-10-10马晨雨詹为民李文亮张梦迪席章营
马晨雨 詹为民 李文亮 张梦迪 席章营
玉米基因的克隆与功能分析
马晨雨 詹为民 李文亮 张梦迪 席章营*
河南农业大学农学院, 河南郑州 450046
玉米籽粒发育和光周期特性是影响产量的关键因素。本文通过RT-PCR方法, 从玉米骨干自交系昌7-2籽粒的cDNA中克隆得到一个乙酰鸟氨酸脱酰酶基因, 命名为。的CDS (coding DNA sequence)长1344 bp, 编码447个氨基酸。qRT-PCR分析表明,基因在玉米雄穗中的表达量最高, 其次是在籽粒、叶、茎、根中; 该基因在授粉后不同天数籽粒中的表达趋势为, 0~15 d快速上升, 之后迅速下降。对该基因的过表达转基因拟南芥的研究表明,基因在转基因拟南芥的根中表达量最高; 经暗处理10 d后, 转基因株系根的长度显著长于野生型; 转基因拟南芥的生育期明显提前, 其粒长和千粒重显著大于野生型拟南芥。这些结果表明,基因的表达可能与籽粒发育和光周期调控有关。
玉米; 基因克隆; 转基因; 籽粒发育; 光周期
玉米(L.)是全世界总产量最高的作物, 具有粮食、饲料、工业原料等多种用途, 在农业生产中占据重要地位。据联合国粮农组织(http://www. fao.org/)统计, 在全球范围内, 1998年以后, 玉米的总产量超过稻谷和小麦, 成为世界第一大粮食作物; 当前我国的玉米单位面积产量和发达国家相比还有较大的差距, 在玉米产量及相关性状功能基因的研究方面还相对落后, 用于分子设计育种的基因资源还比较少。因此, 挖掘玉米产量及相关性状的功能基因, 研究其遗传控制机制, 分析其基因功能和遗传调控网络对于进一步提高玉米单产具有重要意义。
籽粒大小是影响玉米产量的主要因素, 也是当代育种重点选育的性状之一[1]。相对而言, 大粒的种子能够提供较多的养分来应对发芽过程中的各种逆境, 从而确保苗齐苗壮, 为高产稳产奠定基础[2]。光周期的敏感性是影响玉米生育期和地理分布的主要性状[3], 研究光周期调控途径的内在机制是调控生育期和打破热带玉米种质利用障碍的关键, 对我国玉米种质资源的扩增和创新具有重要作用。
乙酰鸟氨酸脱酰酶(-Acetylornithine deacetylase, NAOD)是鸟氨酸合成途径中的关键酶, 参与鸟氨酸合成的线性途径(linear pathway)[4-6], 催化-乙酰鸟氨酸与水生成醋酸盐和鸟氨酸的反应, 在植物中参与果实发育、逆境应答、光周期调节等过程。鸟氨酸是重要的储氮化合物, 它是调节多种生长发育过程的多胺类化合物(polyamines, PAs)的前体物质之一。在植物中, PAs影响细胞分裂、花期、果实、根的形成以及个体衰老等过程[7-11]。
Molesini等[12]克隆得到了拟南芥的基因, 通过对基因缺失的突变体表型的调查发现, 其花期比野生型拟南芥提前, 不育荚果数目增多; 进一步研究发现, 在突变体中, 由于鸟氨酸合成途径的受阻, 花中的鸟氨酸含量比野生型低45%, 乙酰鸟氨酸的含量约比野生型高16%, 从而引起了精胺、亚精胺等PAs含量的变化, 并推测这是造成突变体花期提前和不育荚果数目增多的原因[13]; 在木瓜中, 与NAOD相似的酰化氨基酸水解酶调控果实成熟[14]; 番茄中, 单倍体果实中的基因与乙烯诱导花蕾形成相关[15]。
本研究通过RT-PCR的方法从玉米自交系昌7-2中克隆得到了一个乙酰鸟氨酸脱酰酶基因,拟从的序列、时空表达、转基因拟南芥的表型等方面来研究基因的功能。
1 材料与方法
1.1 试验材料
1.1.1 植物材料及处理 2016年6 月将玉米自交系昌7-2种植于人工气候室和郑州毛庄河南农业大学科教园区(34°51′54.66″N, 113°35′12.11″E)。在温室中分别于三叶期取叶片和根, 十叶期将整株幼苗剥开取茎, 3次重复; 对田间种植的材料, 标注每株的抽雄日期, 选3株生长良好且长势一致的植株, 于抽雄后第5天取其雄穗; 选长势一致的植株自交授粉, 标记授粉日期, 于授粉后第0、第5、第10、第15、第20、第25、第30、第35、第40天, 分别取3株果穗, 剥离果穗中部的籽粒, 液氮冷冻后保存于-80℃冰箱中备用。
1.1.2 主要试剂和菌株 RNA 提取试剂盒为北京天恩泽柱式植物RNAout 2.0; cDNA第1 链合成试剂盒为诺唯赞HiScript II Q RT Suppermix for qPCR; qRT-PCR试剂盒为Promega生物技术有限公司的GOqPCR Master Mix, 限制性核酸内切酶购自New England Biolabs公司, Premix酶、pMD18-T载体和T4连接酶均购自TaKaRa (大连)公司; 大肠杆菌感受态细胞DH5α和根癌农杆菌感受态细胞GV3101购自上海唯地生物技术有限公司, 其他生化试剂购自北京索莱宝科技有限公司。
1.2 玉米ZmNAOD基因的克隆与序列分析
基于B73的GRMZM2G181273的mRNA序列, 应用Oligo 7设计克隆引物NAOD-cDNA-F: 5¢-CCG ATCCTTTCCCCGTCTGA-3¢、NAOD-cDNA-R: 5¢-A GGGAGTATTGGTCTATCCCAT-3¢, 以昌7-2授粉后5 d籽粒的总RNA 反转录的cDNA 第1 链为模板进行RT-PCR 扩增。PCR 体系(25.0 μL)含cDNA 1 μL、LADNA 酶(0.1 U μL–1) 12.5 μL、上下游引物(10 μmol L–1)各1 μL、ddH2O 9.5 μL。PCR扩增程序为95℃预变性5 min; 95℃变性50 s, 55℃退火50 s, 72℃延伸90 s, 35个循环; 72℃再延伸10 min。PCR 产物经1%琼脂糖凝胶电泳检测及胶回收纯化后, 与pMD18-T克隆载体16℃隔夜连接(约10 h), 转化大肠杆菌DH5α 感受态细胞, 将PCR检测呈阳性的菌落送华大基因测序。
利用ProtParam tool在线软件分析基因编码的蛋白的分子量、等电点等理化性质; 利用Signalp 4.0 server软件[16]进行信号肽预测; 利用TMHMM Server v.2.0在线软件进行跨膜结构分析; 利用SOPMA软件进行蛋白的二级结构预测; 运用Netphos 2.0 server软件进行磷酸化位点预测; 用WoLF PSORTII Prediction进行亚细胞定位预测; 利用MEGA 7.0 (NJ, 1000 BootStrap)[17]构建系统进化树。
1.3 ZmNAOD基因启动子区域的克隆与序列分析
根据玉米参考基因组(B73 RefGen_v3)第5染色体GRMZM2G181273上游的基因组序列信息 (198 620 000~198 623 032), 应用Oligo 7设计重叠引物P--F1: 5¢-AATCTGCTGTTACCTTGC G-3¢、P--R1: 5¢-GTTGGAACTTGCTGCTT GGA-3¢, P--F2: 5¢-CTCCAACCCCTTACA TGC-3¢、P--R2: 5¢-CGGCCCAAATGTACA CCT-3¢, 以昌7-2叶片的DNA为模板进行扩增, 采用25.0 μL的PCR体系, PCR程序为95℃预变性5 min; 95℃变性50 s, 分别用55℃、57℃退火50 s, 72℃延伸1 min, 35个循环; 72℃再延伸10 min。PCR产物经电泳、胶回收、载体连接和转化大肠杆菌后测序, 将两段序列比对拼接去除冗余序列后获得基因的启动子序列, 利用PlantCARE[18]在线软件分析启动子序列。
1.4 ZmNAOD基因的时空表达分析
以昌7-2不同器官(根、茎、叶、雄穗、籽粒)和授粉后不同天数的籽粒中的cDNA 为模板, 以玉米18SrRNA为内参基因, 内参基因引物[19]为18S-F: 5¢-CCTGCGGCTTAATTGACTC-3¢、18S-R: 5¢-GTTA GCAGGCTGAGGTCTCG-3¢; 在基因的编码序列中跨内含子设计qRT-PCR引物Q-NAOD-F: 5¢-TTGAATACCCCGGCAC-3¢、Q-NAOD-R: 5¢-TCC AAGACAGTCGGTT-3¢。反应体系含荧光定量Mix GoqPCR Master Mix 2 × 12.5 μL, 引物(10 μmol L–1)各1 μL, cDNA 模板1 μL, 加DNase free水至终体积25 μL。使用罗氏LightCycler 480 II型实时荧光定量PCR仪扩增, 反应程序为94℃预变性 180 s; 94℃变性20 s, 58℃退火 20 s, 72℃延伸 40 s, 40个循环; 72℃读取荧光值; 循环结束后60℃保温60 s; 由熔解曲线判定PCR 反应的特异性。每个样品进行3次生物学重复及3次PCR 重复, 参照2–ΔΔCt法[20]计算相对表达量。
1.5 ZmNAOD基因过表达载体的构建与遗传转化
以含有目标基因的阳性菌液为模板, 利用引物O-NAOD-F: 5¢-GAAGATCTATGGCATCTCCGG-3¢, O-NAOD-R: 5¢-CGGACTAGTTCAGACCTCTTCT-3¢扩增带有II与I酶切位点的基因的CDS片段; 然后, 对胶回收片段及含有CaMV35S启动子的过表达载体质粒pCAMBIA1304 在37℃双酶切4 h, 用T4连接酶16℃连接过夜(约10 h), 构建重组质粒pCAMBIA 1304-。将菌液PCR呈阳性及测序正确的重组阳性质粒转入根癌农杆菌菌株GV3101 感受态细胞中, 涂布于含有卡那霉素(50 μg mL–1)和利福平(50 μg mL–1)的YEP固体培养基上, 经28℃培养72 h, 挑取单克隆点进行PCR 检测及质粒双酶切鉴定。将验证正确的阳性重组菌株GV3101 pCAMBIA1304-菌液加入含50 μg mL–1卡那霉素和50 μg mL–1利福平的YEP 液体培养基中, 于28℃振荡培养过夜(约16 h)。离心收集菌体后, 用含有200 μmol L–1Silwet L-77的MS液体培养基重悬菌体至OD值为0.8, 使用floral dip方法[21]侵染拟南芥, 得到T0代转基因种子。
1.6 纯合转基因拟南芥植株的筛选与表型鉴定
将T0代转基因种子用6.25%的NaClO溶液浸泡15 min后, 用灭菌ddH2O清洗5次, 然后平铺于含有35 μg mL–1潮霉素B的MS培养基上进行筛选; 在T1代提取拟南芥基因组DNA, 检测载体是否插入到拟南芥基因组中; 在T2和T3代, 取其叶片提取RNA进行qRT-PCR鉴定, 直至选出纯合转基因植株。
取T3代纯合转基因植株的根、茎、叶、花序、荚果, 用灭菌水清洗干净后, 用吸水纸吸干水分, 保存于-80℃冰箱备用。将在MS培养皿中正常生长14 d的转基因拟南芥和野生型拟南芥放置在黑暗环境下培养10 d, 并分别调查15株转基因株系和野生型拟南芥根的长度。选择基因表达量相对较高的3个转基因株系进行表型鉴定, 每个株系种植26株, 以野生型为对照, 记录每个单株出现花蕾的日期与第1朵花开花的日期。成熟时, 收获野生型拟南芥和转基因拟南芥的种子, 自然风干后, 从每个材料中随机选取1000粒净种子测定千粒重, 重复3次; 随机选取100粒净种子, 用Image J图像识别软件[22]测量每粒种子的长和宽, 重复3次。
2 结果与分析
2.1 玉米ZmNAOD基因的克隆与序列分析
玉米基因的CDS全长为1344 bp, 编码447个氨基酸(图1)。该蛋白质的分子式为C2195H3441N585O661S18, 相对分子质量为49.179 kD, 理论等电点为5.45, 其中赖氨酸和亮氨酸含量最高, 均为9.4%; 有59个带负电荷的氨基酸残基(Asp + Glu)、45个带正电荷的氨基酸残基(Arg + Lys), 总体带负电荷, 属于稳定的亲水蛋白。保守结构域预测显示, 该蛋白具有高度保守的peptidase_M20结构域中的M20_ArgE-ralated亚族结构, 该亚族可能与脱乙酰基酶活动相关; 没有信号肽序列及跨膜结构, 含有18个磷酸化位点。其蛋白质的二级结构中, 含有36.42%的α-螺旋, 34.90%的无规则卷曲, 19.91%的延伸链和8.95%的β-转角; 亚细胞定位预测显示, 该蛋白有可能位于细胞质中。将该蛋白的氨基酸序列在UniProt数据库中进行BLAST并构建系统进化树(图2)表明,编码的蛋白与高粱中的SORBI_3004G275600相似性最高, 为97.00%; 该蛋白与拟南芥中的AtNAOD (At4g17830)蛋白的相似度为60.54%, 在拟南芥中AtNAOD蛋白的表达与开花时间及籽粒发育有关, 其他蛋白功能尚无研究。
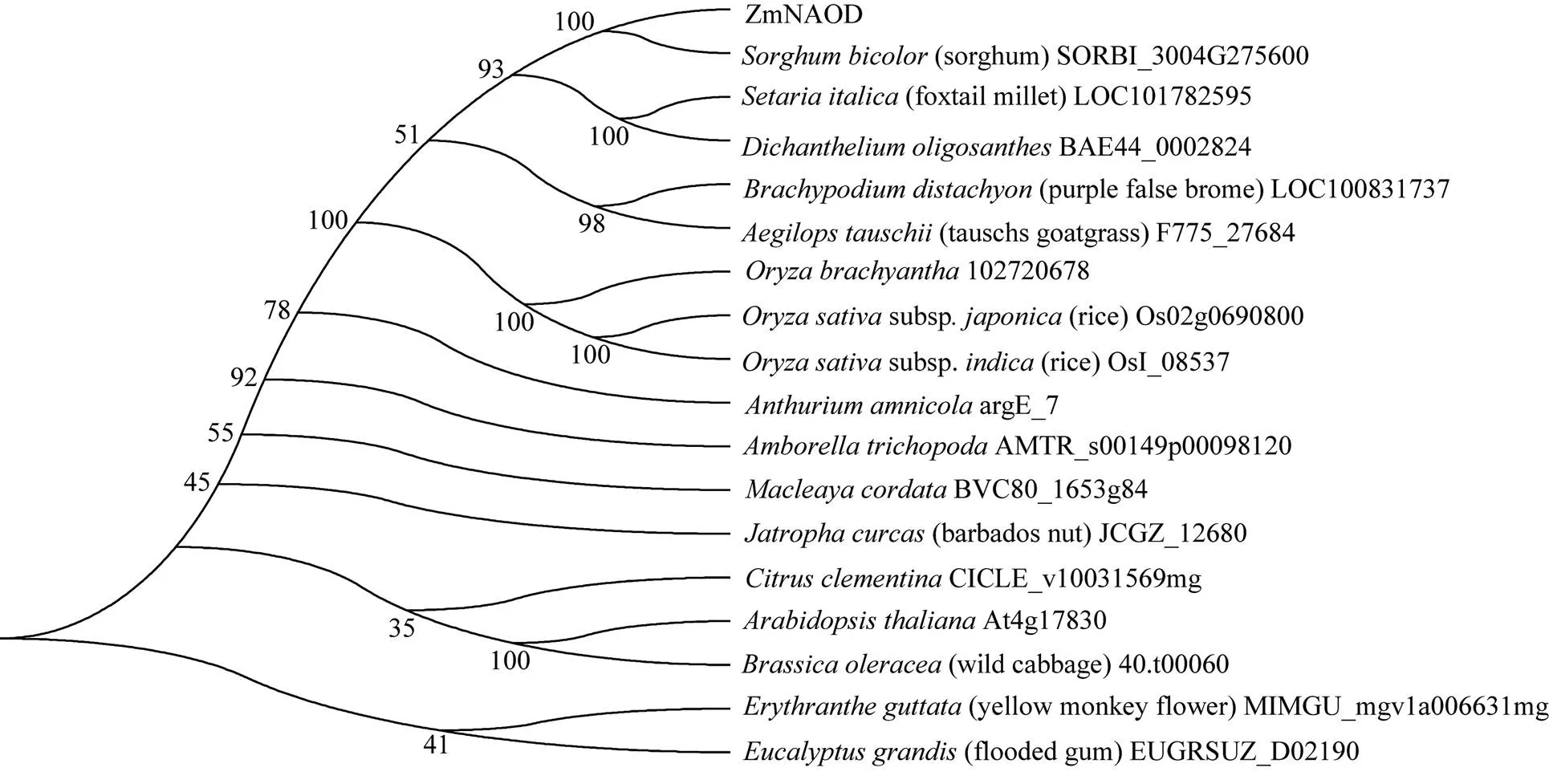
图2 植物中NAOD蛋白的系统进化树
2.2 玉米ZmNAOD基因启动子区域的克隆与分析
以昌7-2基因组DNA为模板进行启动子区域的TA克隆, 将PCR产物测序后分别得到一条867 bp和一条1658 bp的片段, 将两段序列比对拼接去除冗余序列后, 最终得到一条2401 bp的序列, 通过序列比对确认为基因的启动子区域。
PlantCARE分析显示, 在该启动子区域含有典型的TATA-box与CAAT-box, 还有胚乳表达元件Skn-1_motif、GCN4_motif, 昼夜节律调控元件circadian, 光响应元件G-box、Sp1、ATCT-motif、LAMP-element、Box II、CATT-motif、GAG-motif、GA-motif、I-box、TGGCA-motif, 与激素相关的顺式作用元件ABRE、AuxRR-core、CGTCA-motif、TGACG-motif、TCA-element, 与逆境响应相关的HSE、LTR、MBS、TC-rich repeats, 及其他诱导型和组织特异性启动子元件等(表1)。
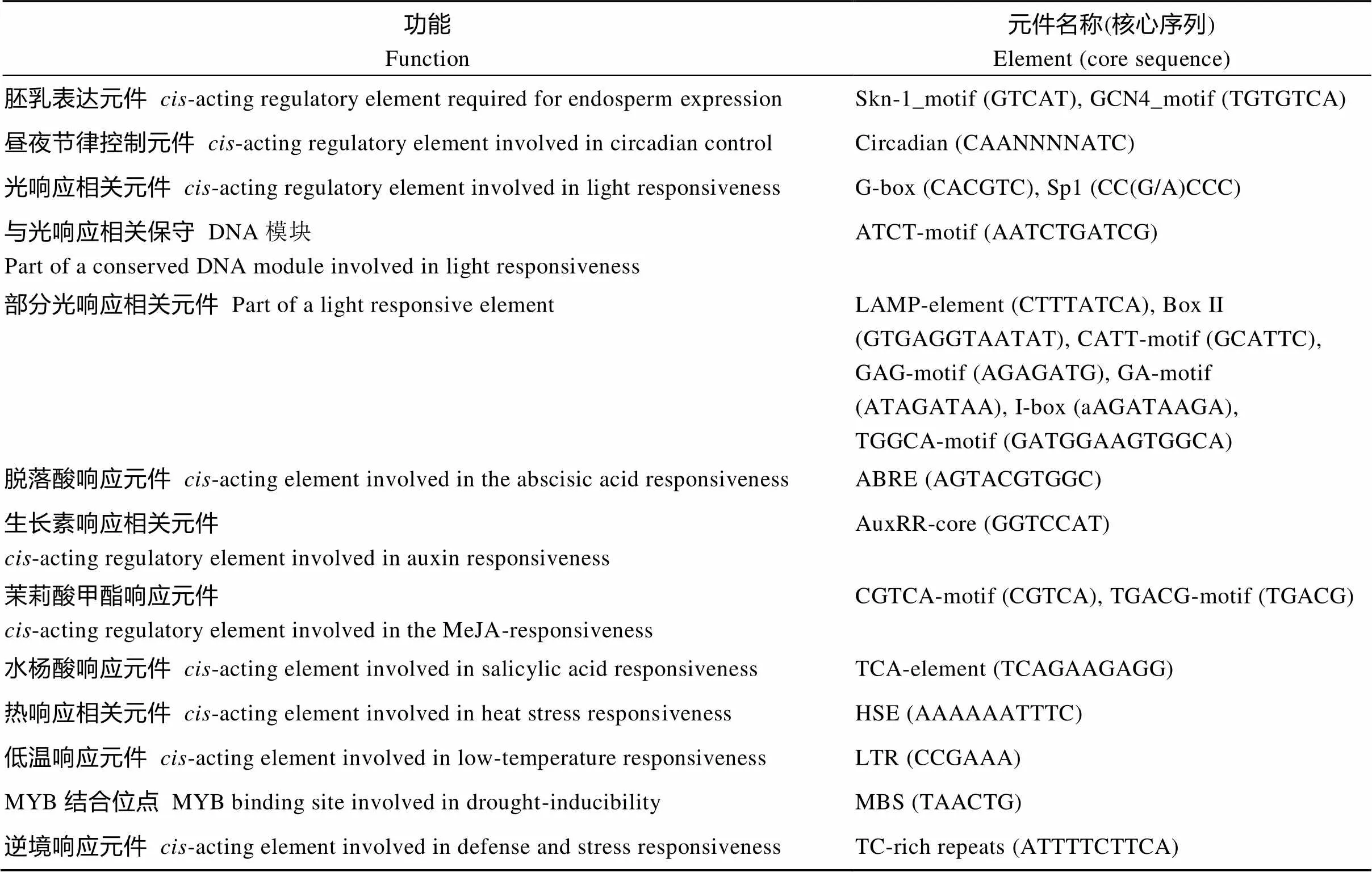
表1 ZmNAOD启动子区域调控元件预测
2.3 ZmNAOD基因的时空表达分析
基因的表达没有器官特异性, 在根、茎、叶、雄穗和籽粒中均有表达, 其中在雄穗中的表达量最高, 其次是籽粒, 然后是叶、茎和根(图3)。
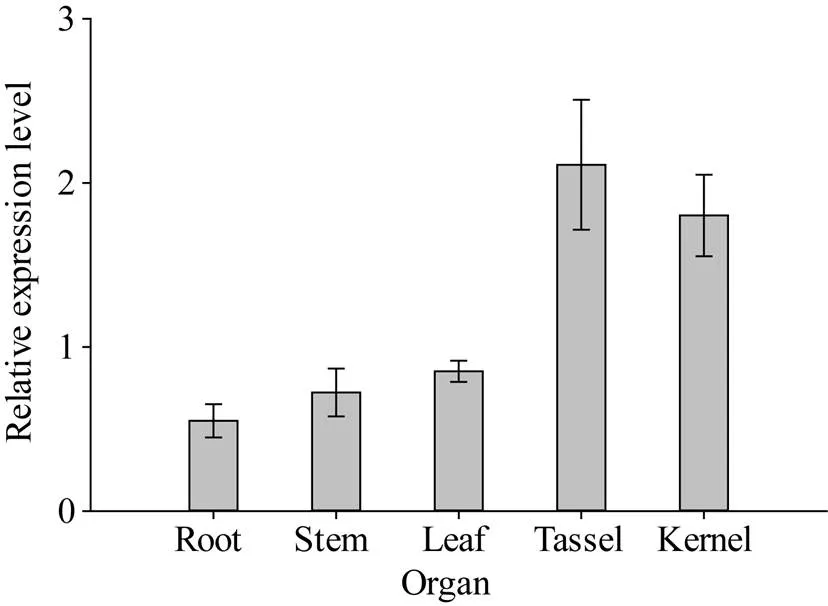
图3 ZmNAOD基因在昌7-2不同器官中的表达
在授粉后0~15 d内, 籽粒中基因的表达量呈快速上升趋势, 在授粉后15 d达到最高值, 之后迅速下降, 20 d后表达量很低(图4)。
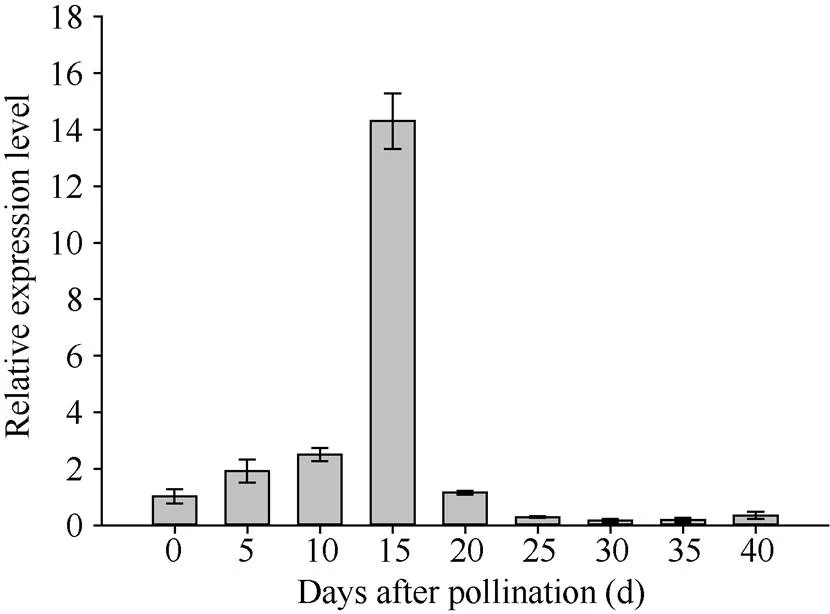
图4 ZmNAOD基因在授粉后不同天数的籽粒中的表达
2.4 ZmNAOD基因过表达载体的构建及转基因拟南芥的获得
将pCAMBIA1304载体和目的基因双酶切产物连接, 经转化大肠杆菌、菌液检测和测序后, 从阳性转化子提取质粒, 用II和I进行双酶切电泳检测, 电泳结果显示存在一条约10 000 bp的载体片段条带和一条约1500 bp的目的基因条带。用滴蘸法将基因转化拟南芥, 通过抗性筛选, 获得有潮霉素抗性的拟南芥, 在T1代提取拟南芥基因组DNA进行潮霉素基因的PCR鉴定, 收获具有抗性基因的种子; 从T2代取30株转基因拟南芥叶片提取RNA, 反转录为cDNA, 以基因在野生型拟南芥叶片中的表达量为基准进行qRT-PCR鉴定, 选择表达量相对较高的5个单株收获种子在T3代种植为株系; 在T3代提取5个株系的cDNA进行qRT-PCR鉴定表明, 获得了能够稳定转录的转基因植株(图5), 选择基因表达量相对较高的T3-17、T3-23、T3-26株系收获种子进行表型鉴定。
2.5 转基因拟南芥的表型鉴定
2.5.1基因在转基因拟南芥不同器官中的表达 选择表达量相对较高的T3-23株系, 以基因在转基因拟南芥叶片中的表达量为基准计算其在不同器官中的相对表达量。结果显示,基因在根中的表达量最高, 随后是果荚、叶、花序, 在茎中的表达量最低(图6)。
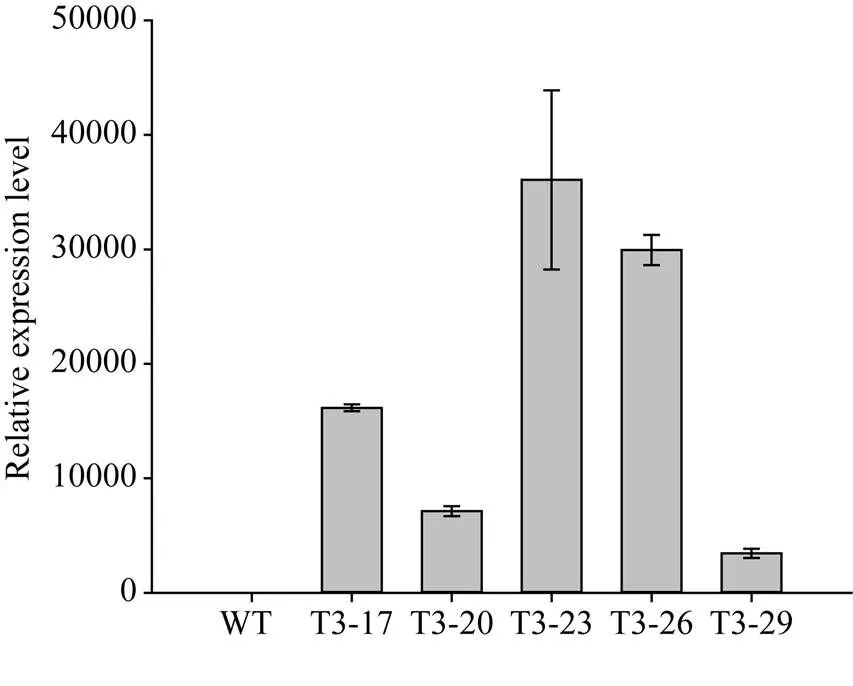
图5 ZmNAOD基因在拟南芥野生型和转基因株系中的表达
2.5.2 暗处理后拟南芥的根长鉴定 将野生型拟南芥种子和T3代转基因种子点播于MS培养基中, 在22℃、16 h光照/8 h黑暗的条件下培养14 d, 转移至黑暗条件下培养10 d后, 转基因株系的根长为(5.71±0.16) cm, 野生型拟南芥的根长为(4.37±0.31) cm, 前者显著大于后者。
2.5.3 转基因拟南芥的花期鉴定 野生型拟南芥及3个转基因株系(T3-17、T3-23、T3-26)用于生育期调查的有效株数依次为26、24、24、15。野生型拟南芥出现第1朵花序的天数为(37.60±0.47) d, 第1朵花开花的天数为(48.23±0.55) d; 3个转基因拟南芥株系出现第1朵花序的天数分别为(36.08±0.42) d、(34.80±1.08) d、(36.00±0.53) d, 第1朵花开花的天数分别为(46.96±0.64) d、(43.00±0.72) d、(46.00±0.65) d (表2)。转基因拟南芥株系出现第1朵花序的天数及第1朵花开花的天数均显著早于野生型拟南芥(图7)。
2.5.4 转基因拟南芥籽粒性状的鉴定 野生型拟南芥籽粒的长度为(0.7857±0.0380) mm, 宽度为(0.6682±0.0259) mm, 千粒重为(13.353±0.122) mg; 转基因拟南芥T3-17株系种子的长度为(0.8936± 0.0449) mm, 宽度为(0.6798±0.0128) mm, 千粒重为(14.233±0.031) mg; 转基因拟南芥T3-23株系种子的长度为(0.9004±0.0527) mm, 宽度为(0.6656± 0.0315) mm, 千粒重为(14.249±0.137) mg; 转基因拟南芥T3-26株系种子的长度为(0.9029±0.0564) mm, 宽度为(0.6646±0.0220) mm, 千粒重为(14.222±0.152) mg (表3)。转基因拟南芥的粒长和千粒重均显著大于野生型, 粒宽差异不显著。
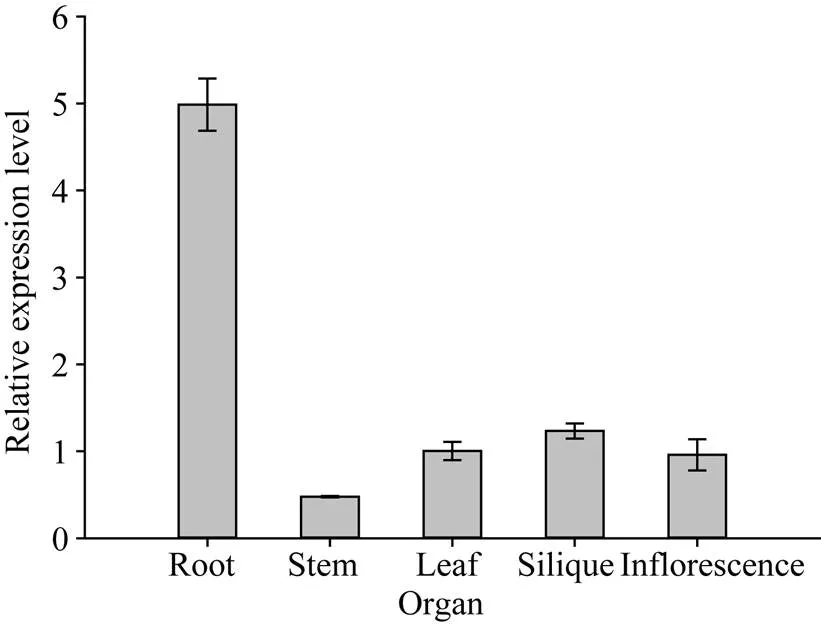
图6 ZmNAOD基因在转基因拟南芥不同器官中的表达

表2 野生型拟南芥和转基因株系的生育期
**表示0.01概率水平差异显著;*表示0.05概率水平上差异显著。
**: significance at the 0.01 probability level;*: significance at the 0.05 probability level.

图7 种植45 d后转基因拟南芥和野生型拟南芥的表型对比
1, 2: 转基因株系T3-23; 3, 4: 野生型植株。
1, 2: transgenic lines T3-23; 3, 4: wild type lines.
3 讨论
乙酰鸟氨酸脱酰酶是鸟氨酸合成途径中的一种关键酶, 参与鸟氨酸合成中的线性途径, 催化-乙酰鸟氨酸与水生成醋酸盐和鸟氨酸的反应。在植物中有很多推测的NAOD蛋白的编码序列, 其中的一些蛋白质与生长发育调节相关。Molesini等[8]克隆得到拟南芥的基因, 该基因的缺失使不育荚果数增加, 使花期提前。番茄单倍体果实中基因与花蕾形成有关[15]。
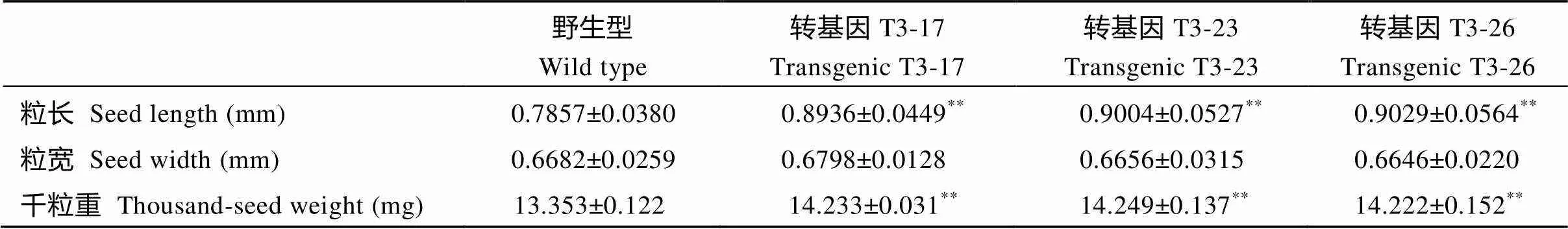
表3 转基因拟南芥籽粒的表型分析
**表示0.01概率水平差异显著。**: significance at the 0.01 probability level.
本研究显示,基因在玉米雄穗和籽粒中的表达量均较高, 在授粉后不同天数的籽粒中的表达呈现0~15 d迅速上升之后快速下降的趋势,基因的启动子区域含有多种与胚乳表达相关的元件, 过表达转基因拟南芥籽粒的千粒重显著大于野生型。这些结果表明,基因可能参与玉米籽粒发育的调控。雄穗和雌穗花序均是由花序分生组织(inflorescence meristem, IM)分化而来的[23], 有很多基因同时对雄穗和雌穗性状起着调控作用, 例如()基因对玉米雄花小穗的数量产生影响, 同时还影响雌穗的行数[24-25];()和()基因的双缺失突变体的雄穗分枝数和穗行数都会增加[26]。本研究中,基因在雄穗和籽粒中的表达量均较高, 可能是该基因同时参与雄穗和雌穗发育的调控。在基因启动子区域发现的GCN4-motif和E-box顺式作用元件是许多籽粒特异表达基因的启动子部分常有的元件[27]。张冬梅等[28]的研究结果表明, 玉米自交系昌7-2籽粒的平均灌浆速率在授粉后前15 d迅速增快, 随后开始逐渐降低, 这与本研究中基因在授粉后不同天数的籽粒中的表达趋势基本一致。可能是因为基因表达量的增加, 催化了乙酰鸟氨酸与水生成醋酸盐和鸟氨酸的反应, 使得鸟氨酸快速积累, 进而引起植物体内PAs含量的变化, 导致干物质积累速率改变[10,13], 灌浆速率增加。过表达转基因拟南芥籽粒的千粒重显著大于野生型, 可能与NAOD参与植物果实发育的机制[29]有关。
本研究中,基因的启动子区域含有大量的光响应元件,基因过表达的转基因拟南芥的花期与野生型拟南芥相比显著提前,基因在转基因拟南芥根中的表达量最高, 经过10 d的黑暗处理后的转基因拟南芥根长显著大于野生型拟南芥。Qiao等[30]克隆得到一个玉米肌动蛋白解聚因子基因, 并构建过表达载体1302将其转入拟南芥植株, 暗培养8 d后发现转基因株系的下胚轴长度比野生型增加55.8%, 在的启动子区域也发现了许多与光照有关的调控元件, 推测基因的功能与光响应相关。本研究发现黑暗条件能够促进转基因拟南芥根的伸长, 说明光照可能对基因的表达起着调控作用。基因的启动子区域的G-box、GA motif、GAG motif、I-box等元件常在光调控基因的启动子区域被发现, 它们对光调控的转录活动至关重要[31-33]。这些结果表明,基因的表达可能与玉米光周期的调节相关。
4 结论
克隆了玉米乙酰鸟氨酸脱酰酶基因, 该基因启动子区域含有与胚乳发育、光响应等有关的顺式作用元件。基因在昌7-2的不同器官中均有表达, 在授粉后不同天数的籽粒中的表达量先上升后下降, 与昌7-2的灌浆速率的变化基本一致;基因在拟南芥中的过表达使花期提前, 使转基因株系种子长度和千粒重显著增加, 推测基因的表达可能与玉米的籽粒发育和光周期有关。
[1] Shomura A, Izawa T, Ebana K, Ebitani T, Kanegae H, Konishi S, Yano M. Deletion in a gene associated with grain size increased yields during rice domestication., 2008, 40: 1023–1028
[2] Moles A T, Ackerly D D, Webb C O, Tweddle J C, Dickie J B, Westoby M. A brief history of seed size., 2005, 307: 576–580
[3] Andres F, Coupland G. The genetic basis of flowering responses to seasonal cues.,2012, 13: 627–639
[4] Slocum R D. Genes, enzymes and regulation of arginine biosynthesis in plants., 2005, 43: 729–745
[5] Shargool D, Jain J C, Mckay G. Ornithine biosynthesis, and arginine biosynthesis and degradation in plant cells., 1988, 27: 1571–1574
[6] Caldovic L, Tuchman M.-acetylglutamate and its changing role through evolution., 2003, 372: 279–290
[7] Carbonell J, Navarro J L. Correlation of spermine levels with ovary senescence and with fruit set and development inL.,1989, 178: 482–487
[8] Evans P T, Malmberg R L. Do polyamines have roles in plant development?, 1989, 40: 235–269
[9] Imai A, Matsuyama T, Hanzawa Y, Akiyama T, Tamaoki M, Saji H, Shirano Y, Kato T, Hayashi H, Shibata D, Tabata S, Komeda Y, Takahashi T. Spermidine synthase genes are essential for survival of Arabidopsis., 2004, 135: 1565–1573
[10] Liu J, Nada K, Pang X, Honda C, Kitashiba H, Moriguchi T. Role of polyamines in peach fruit development and storage., 2006, 26: 791–798
[11] Tiburcio A F, Altabella T, Bitrián M, Alcázar R. The roles of polyamines during the lifespan of plants: from development to stress., 2014, 240: 1–18
[12] Molesini B, Mennella G, Martini F, Francese G, Pandolfini T. Involvement of the putative-acetylornithine deacetylase fromin flowering and fruit development., 2015, 56: 1084–1096
[13] Molesini B, Zanzoni S, Mennella G, Francese G, Losa A, L Rotino G, Pandolfini T. The-acetylornithine deacetylase controls ornithine biosynthesis via a linear pathway with downstream effects on polyamine levels., 2017, 58: 130–144
[14] Fabi J P, Seymour G B, Graham N S, Broadley M R, May S T, Lajolo F M, Cordenunsi B R, Oliveira do Nascimento J R. Analysis of ripening-related gene expression in papaya using an Arabidopsis-based microarray., 2012, 12: 242–261
[15] Molesini B, Rotino G L, Spena A, Pandolfini T. Expression profile analysis of early fruit development in iaaM-parthenocarpic tomato plants., 2009, 2: 143–149
[16] Petersen T N, Brunak S, von Heijne G, Nielsen H. SignalP 4.0: discriminating signal peptides from transmembrane regions., 2011, 8: 785–786
[17] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets., 2016, 33: 1870–1874
[18] Lescot M, Déhais P, Thijs G, Marchal K, Moreau Y, Van de Peer Y, Rouzé P, Rombauts S. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences., 2002, 30: 325–327
[19] Wang X T, Wu L J, Zhang S F, Wu L C, Ku L X, Wei X M, Xie L L, Chen Y H. Robust expression and association ofwith circadian., 2011, 30: 1261–1272
[20] Rajeevan M S, Ranamukhaarachchi D G, Vernon S D, Unger E R. Use of real-time quantitative PCR to validate the results of cDNA array and differential display PCR technologies., 2001, 25: 443–451
[21] Clough S J, Bent A F. Floral dip: a simplified method for-mediated transformation of., 1998, 16: 735–743
[22] Williams K, Munkvold J, Sorrells M. Comparison of digital image analysis using elliptic Fourier descriptors and major dimensions to phenotype seed shape in hexaploid wheat (L.)., 2013, 190: 99–116
[23] Upadyayula N, da Sliva H S, Bohn M O, Rocheford T R. Genetic and QTL analysis of maize tassel and ear inflorescence architecture., 2006, 112: 592–606
[24] Taguchi-Shiobara F, Yuan Z, Hake S, Jackson D. Thegene encodes a leucine-rich repeat receptor-like protein that regulates shoot meristem proliferation in maize., 2001, 15: 2755–2766
[25] Bommert P, Nagasawa N S, Jackson D. Quantitative variation in maize kernel row number is controlled by thelocus., 2013, 45: 334–337
[26] Chuck G S, Brown P J, Meeley R, Hake S. Maizetranscription factorsandaffect yield traits by regulating the rate of lateral primordia initiation.,2014, 111: 18775–18780
[27] Thomas T L. Gene expression during plant embryogenesis and germination: an overview.,1993, 5: 1401–1410
[28] 张冬梅, 刘洋, 赵永锋, 祝丽英, 黄亚群, 郭晋杰, 陈景堂. 不同杂种优势群玉米籽粒灌浆速率分析. 中国农业科学, 2014, 47: 3323–3335 Zhang D M, Liu Y, Zhao Y F, Zhu L Y, Huang Y Q, Guo J J, Chen J T. Analysis of maize grain filling rate in different heterotic groups., 2014, 47: 3323–3335 (in Chinese with English Abstract)
[29] Pandolfini T, Molesini B, Spena A. Fruit development and seed dispersal. In: Østergaard L, ed. Annual Plant Reviews. UK: Wiley Blackwell Press, 2009, Vol 38, pp 326–345
[30] Qiao D H, Dong Y B, Zhang L, Zhou Q, Hu C H, Ren Y L, Li Y L. Ectopic expression of the maizegene inrevealing its functions in kernel development., 2016, 126: 239–253
[31] Millar A J, Kay S A. Integration of circadian and phototransduction pathways in the network controllinggene transcription in, 1996, 93: 15491–15496
[32] Terzaghi W B, Cashmore A R. Light-regulated transcription., 1995, 46: 445–474
[33] Tobin E M, Kehoe D M. Phytochrome-regulated gene expression., 1994, 5: 335–346
Cloning and Function Analysis ofGene in Maize
MA Chen-Yu, ZHAN Wei-Min, LI Wen-Liang, ZHANG Meng-Di, and XI Zhang-Ying*
Agronomy College, Henan Agricultural University, Zhengzhou 450046, Henan, China
The development of kernels and the photoperiod characters are key elements that impact yield in maize. A-acetylornithine deacctylase (NAOD) gene namedwas cloned from maize inbred line Chang 7-2 by the method of RT-PCR in our work. The length of CDS (coding DNA sequence) ingene is 1344 bp, coding a polypeptide of 447 amino acids. The expression ofwas abundant in tassel, kernel, leaf, stem and root successively, and was up-regulated in seed from 0 to 15 d, and down-regulated after 15 d. The over-expression recombinant plasmid, pCAMBIA1304-, was transformed intousingmediated method, and homogeneous transgenic lines were obtained. Thegene expression was abundant in roots of transgenic, and the root length was significantly longer than that of the wild type (WT) after a 10-day dark treatment. Flowering days of transgenic lines were earlier, the seeds were longer and the thousand seed weight was heavier than those of the WT. These results demonstrated that themight be involved in seed development and photoperiod.
maize; gene cloning; genetic modification; seed development; photoperiod
2018-03-13;
2018-06-12;
2018-07-02.
10.3724/SP.J.1006.2018.01433
席章营, E-mail: xizhangying@163.com
E-mail: ma18736076042@163.com
本研究由国家自然科学基金项目(31371629)资助。
This study was supported by the National Natural Science Foundation of China (31371629).
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20180629.1906.014.html
