甘蓝型油菜PIN家族基因的鉴定与生物信息学分析
2018-09-11华营鹏宋海星官春云张振华
高 堃 华营鹏 宋海星 官春云 张振华 周 婷,*
甘蓝型油菜PIN家族基因的鉴定与生物信息学分析
高 堃1,2华营鹏1,2宋海星1,2官春云3张振华1,2周 婷1,2,*
1湖南农业大学资源环境学院, 湖南长沙 410128;2南方粮油作物协同创新中心, 湖南长沙 410128;3国家油料作物改良中心湖南分中心, 湖南长沙 410128
PIN家族基因是一类调控植物生长素极性运输的重要载体元件, PIN基因编码生长素输出蛋白, 介导生长素在植物体的运输, 然而在基因组较复杂的甘蓝型油菜中缺乏系统研究。本研究运用生物信息学方法在甘蓝型油菜全基因组数据库筛选甘蓝型油菜PIN家族基因, 对鉴定出的29个基因开展拷贝数变异、分子特征、跨膜结构域、保守基序、染色体定位、系统进化树构建、PIN蛋白二级结构及三级结构预测等研究, 结合高通量转录组测序进行低氮胁迫下的转录水平分析。结果表明, 甘蓝型油菜PIN家族基因拷贝数明显多于拟南芥、甘蓝和白菜所具有的PIN家族基因数量; BnPINs蛋白多属于由碱性氨基酸组成的稳定蛋白, 含有保守的N末端结构域, 二级结构与拟南芥PIN蛋白相似; 系统进化选择能力分析表明,基因与甘蓝和白菜PIN家族基因进化关系相近。转录组测序表明,、、基因主要在甘蓝型油菜根部表达且受长期低氮(72 h)诱导,和基因主要在地上部表达, 低氮会抑制表达。本研究结果为进一步研究甘蓝型油菜PIN家族基因生物学功能尤其是在响应低氮胁迫中的功能奠定基础, 为已知大量数据的其他物种家族基因生物信息学研究提供参考。
甘蓝型油菜; 生长素;; 家族基因; 生物信息学
生长素是具有极性运输特点的植物激素, 主要在植物叶原基、幼叶、根和发育的种子中合成, 通过极性运输到靶细胞调节作物一系列生理反应和生长发育[1]。生长素在细胞间的运输依赖于细胞膜上定位的AUXIN RESISTANT1/LIKE AUX1(AUX/ LAX)和PIN-FORMED(PIN)以及ATP-Binding Cassette subfamily B/P-glycoprotein (PGP/ABCB)生长素运输载体, AUX/LAX属于生长素内向型运输蛋白, 在拟南芥中有AUXIN1(AUX1)、LIKE AUX1 (LAX1)、LAX2和LAX3共4个成员; 而PIN/ PGP/ABCB属于生长素外向型运输蛋白[2]。基因编码的蛋白是最重要的生长素外输载体, 几乎存在于所有单子叶和双子叶植物中[3]。植物基因最早在拟南芥中被发现[4], AtPIN1蛋白在细胞膜上呈极性分布, 负责将生长素从胞内运输到胞外[5]。随后发现拟南芥PIN家族蛋白共有8个成员, 命名为PIN1~PIN8[6]。PIN1蛋白在植物发育中具有重要作用, 主要参与胚的发育、叶序和叶脉的形成及维管组织分化, 负责生长素在茎尖分生组织环流及从地上部到根的运输[7]; PIN2蛋白主要参与根的向重力生长,突变体表型为向重力生长缺陷[8];基因主要参与植物的向性生长, 组织化学染色表明其主要在维管组织和黄化苗顶端弯钩表达[9]; PIN4蛋白极性定位在内皮层、皮层和维管组织细胞膜上, 参与生长素向根尖静止中心运输[10]; PIN5蛋白定位在内质网上, 主要负责生长素从胞质到内质网腔的运输[11]; Benková等[12]研究发现,基因在整个侧根发育过程中均有表达;基因在胚发育早期就有表达, 根尖柱状细胞定位的PIN7蛋白主要参与根的向重力生长[13];基因在花药中特异表达, 调控花粉发育, PIN8蛋白与PIN5蛋白都定位在内质网上, 二者在调控配子体、孢子体发育和植物体内生长素平衡等方面具有拮抗作用[14]; PIN1、PIN2和PIN7蛋白在异源表达系统被证明有向胞外运输生长素的活性, 且该过程不需要其他因子参与[15]。生长素在植物根部有2种运输方式, 一是通过中柱向根尖的向顶运输, 二是根尖生长素经表皮和皮层细胞回运的向基运输[16], PIN1和PIN4蛋白共同调控向顶运输, PIN2和PIN3蛋白参与向基运输[17]。除PIN家族蛋白外, 药物抗性糖蛋白(multidrug-resistance- p-glycoproteins, MDR/PGPs)家族成员MDR1、PGP1、PGP2、PGP4和PGP19也有向胞外输出生长素的功能[18]。
迄今为止, 在玉米、水稻、大豆、小麦等作物上克隆了许多同源基因, 但在甘蓝型油菜中尚未报道。油菜是世界上重要的油料作物, 提供人类所需的主要植物油, 其在保证食用油供给、改善食物结构、促进养殖业和轻纺工业发展等方面具有重要作用[19]。甘蓝型油菜(, AnAnCnCn, ~ 1345 Mb, 2=4x=38)属于十字花科芸薹属油料作物[20], 是白菜(, ArAr, ~485 Mb, 2=2x=20)和甘蓝(, CoCo, ~630 Mb, 2=2x=18)2个二倍体基本种在7500年前通过天然远缘杂交形成的异源四倍体作物, 全基因组包含约十万多个蛋白编码基因[21], 进化、遗传的复杂性在一定程度上影响了各种功能基因在甘蓝型油菜生长发育中的作用。氮是植物生长发育所必需的大量营养元素, 被誉为“生命元素”, 同时, 油菜对氮缺乏极其敏感[22]。已有研究表明, 生长素的极性运输在植物的氮胁迫反应中发挥重要作用[23-24]。目前并不清楚PIN家族基因是如何响应氮素缺乏的。基于上述研究背景, 本研究也开展了甘蓝型油菜PIN家族基因对不同氮素供应水平的转录响应研究。
本研究以拟南芥PIN家族基因为参考序列, 在甘蓝型油菜全基因组数据库中进行了BnPINs家族基因的鉴定, 借助生物信息学方法研究了甘蓝型油菜PIN家族基因的序列信息、分子特征、保守基序特征、染色体定位、系统进化关系、蛋白质结构及低氮胁迫下的表达, 旨在了解甘蓝型油菜PIN家族基因的遗传信息和编码的BnPINs蛋白结构, 为进一步揭示该家族基因的生物学功能奠定研究基础。
1 材料与方法
1.1 试验材料
本研究水培试验所用甘蓝型油菜品种为“湘油15”。
1.2 试验方法
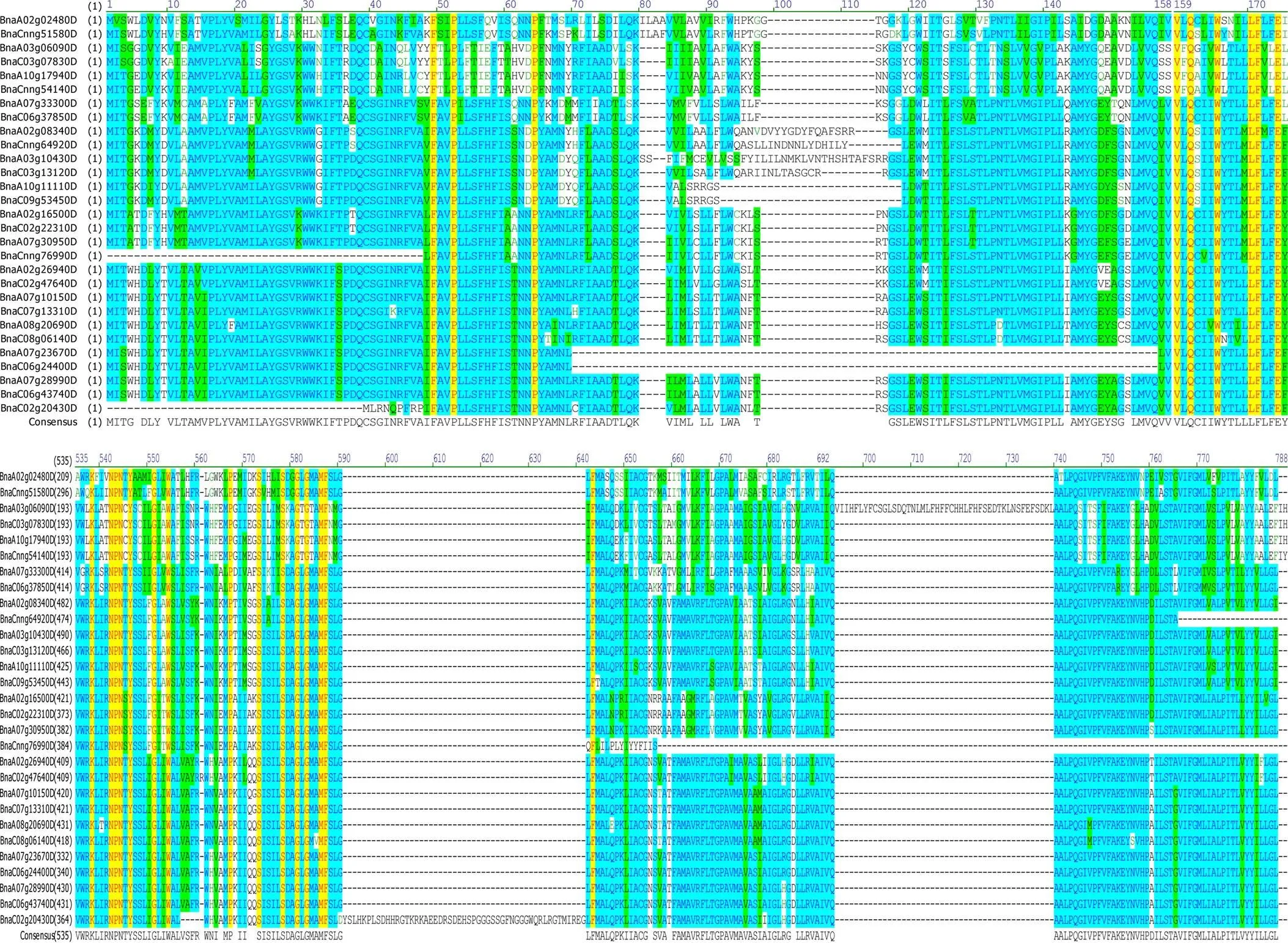
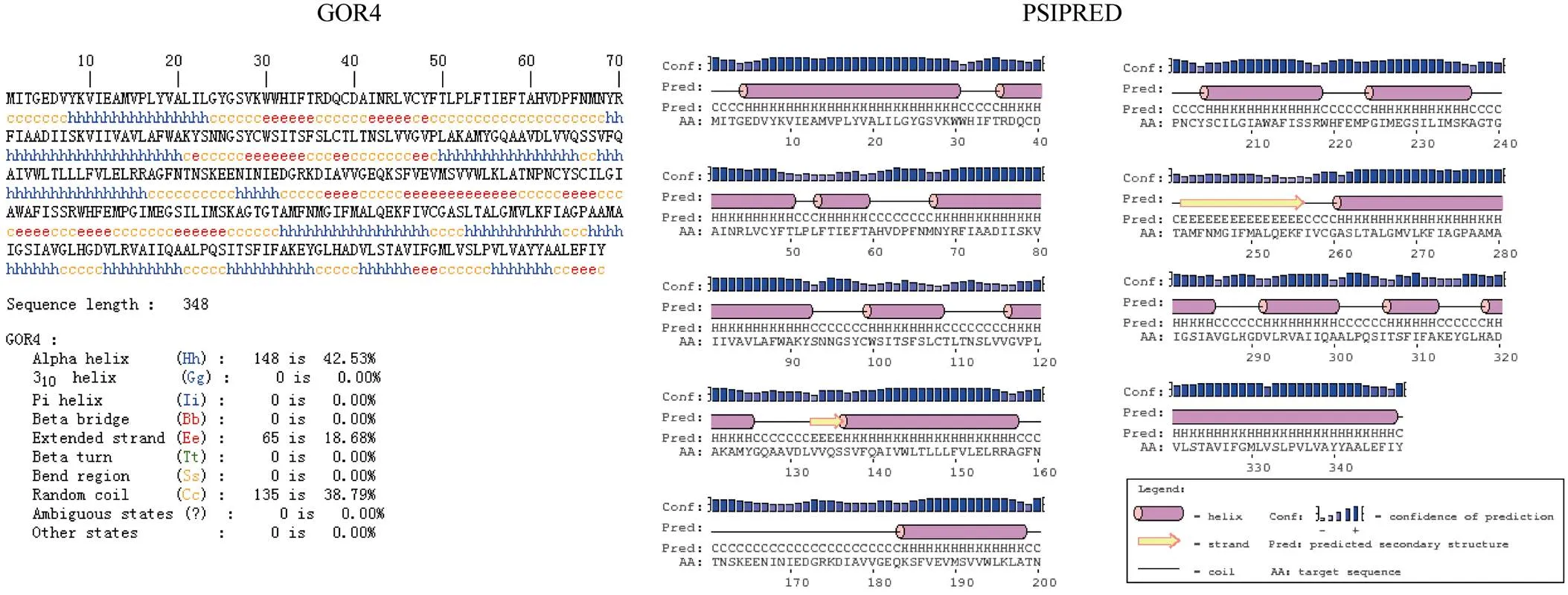
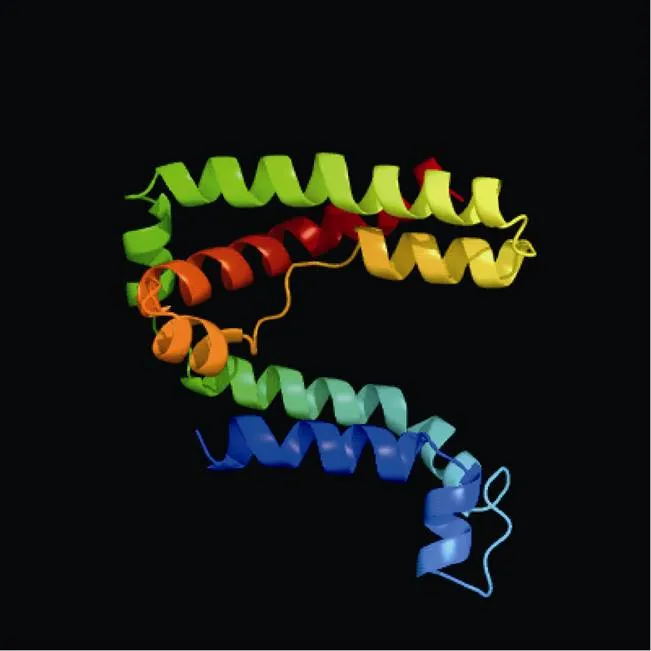
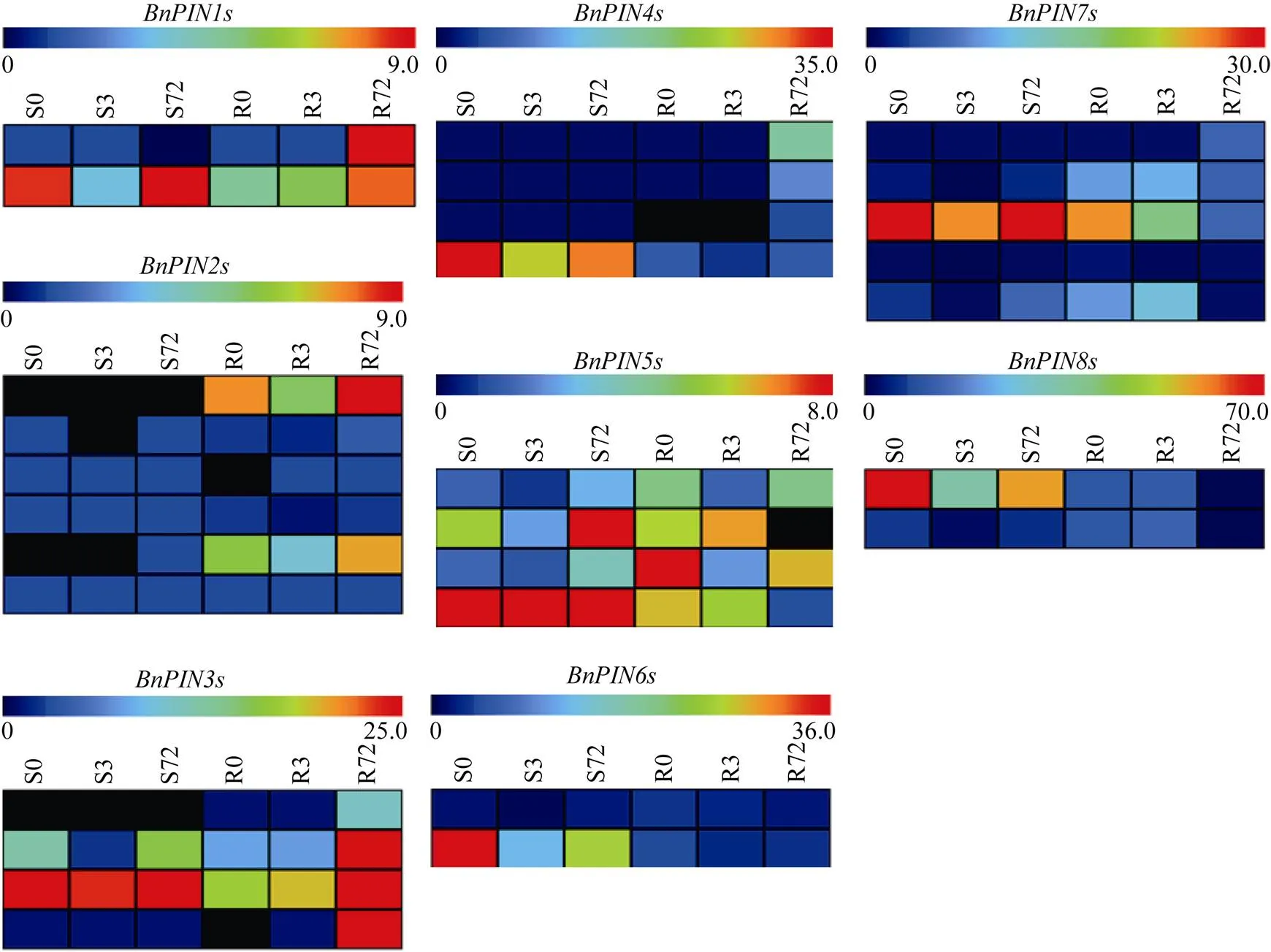
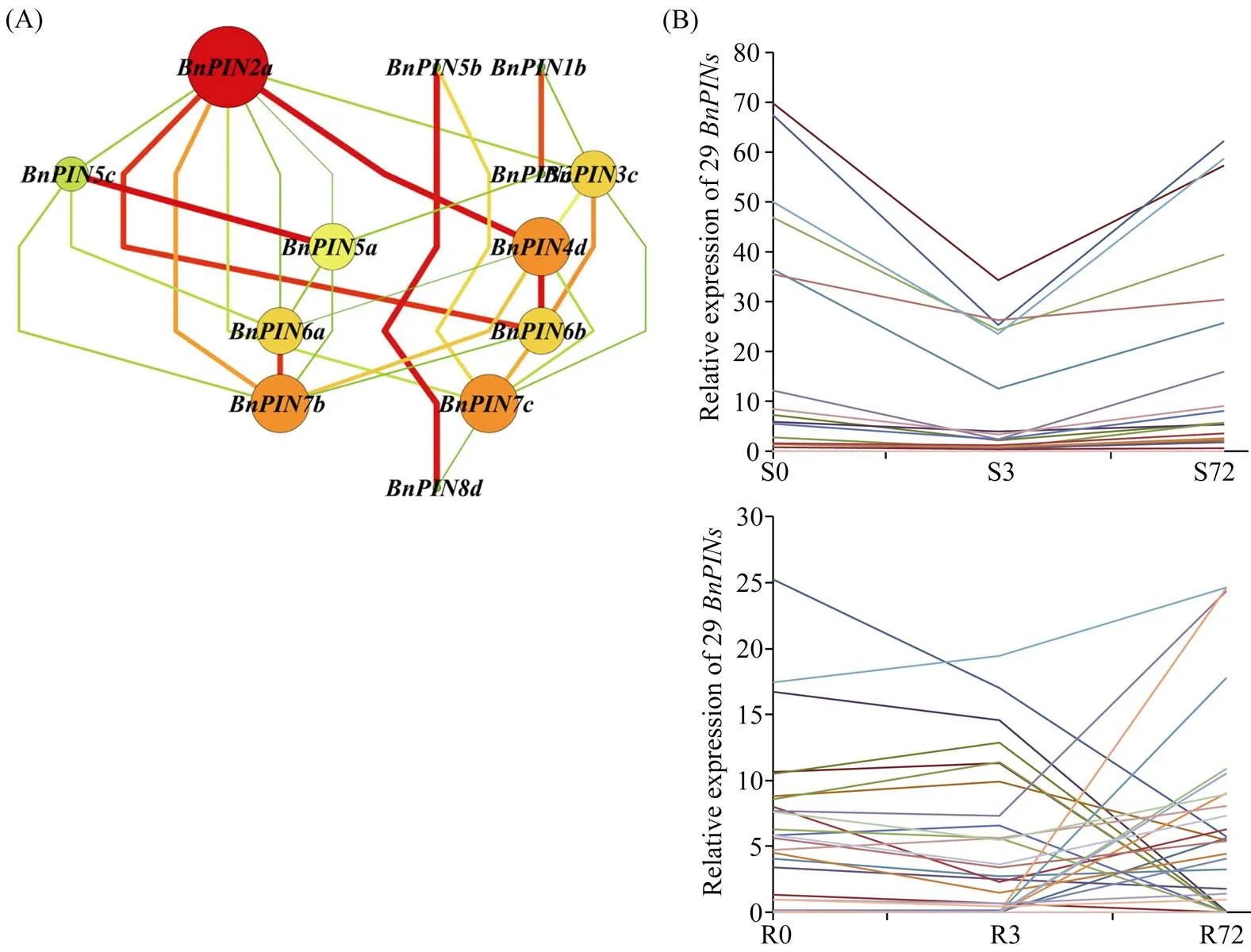
1.2.1 甘蓝型油菜PIN家族基因的鉴定 本研究中拟南芥PIN家族蛋白信息参考Paponov[3]分析结果。首先利用AtPINs为原始序列, 使用在线工具Pfam[25](http://pfam.xfam.org/)和SMART[26](http:// smart.emblheidelberg.de/)分析AtPINs序列, 分析结果显示AtPINs具有跨膜结构域。其次下载甘蓝型油菜全基因组数据(http://brassicadb.org/brad/), 并利用Bio Edit软件构建本地数据库, 用含有跨膜结构域的AtPINs序列进行本地数据库BLAST检索, 设定检索参数E值 1.2.2 甘蓝型油菜PIN家族基因生物信息学分析 使用ExPASy (http://www.expasy.org/tools)[27]中Prot-Param和Prot Scale工具分析BnPINs蛋白的氨基酸组分和理化性质, 应用TMHMM (http://www. cbs.dtu.dk/services/TMHMM/)[28]预测BnPINs蛋白的跨膜结构。 1.2.3 保守结构域和保守残基的鉴定 使用SMART (http://smart.emblheidelberg.de/)[26]鉴定保守结构域, 并使用Pfam (http://pfam.xfam.org/)注释[25]。使用Vector NTI对保守残基进行多重比对分析。 1.2.4 甘蓝型油菜PIN蛋白保守基序分布信息获取 运用MEME Version 4.9.1 (http://meme.nbcr.net/ meme/cgi-bin/meme.cgi)[29]在线工具分析BnPINs蛋白的保守基序信息, 设置最大基序检索值为10。 1.2.5 甘蓝型油菜PIN家族基因系统进化树构建 运用Clustal X version 2.1[30]对拟南芥、甘蓝、白菜PIN家族蛋白的氨基酸序列和本研究鉴定的BnPINs蛋白氨基酸序列进行多重比对, 使用MEGA6.0[31]分析比对结果并采用邻接法(neighbor- joining, NJ)构建系统进化树, 设定泊松校正法计算进化距离和重复1000次的自展法(bootstrap)检验。 1.2.6 甘蓝型油菜PIN家族基因染色体定位分析 利用Pfam[25](http://pfam.xfam.org/)对本研究筛选到的基因进行结构域判定, 去除冗余基因,确定位置信息, 利用Circos 0.6[32]构建PIN家族基因染色体分布图谱。 1.2.7 甘蓝型油菜PIN蛋白二级结构分析及三级结构预测 使用2种不同的工具进行二级结构分析: GOR4 (https://npsa-prabi.ibcp.fr/cgibin/page=npsagor4. html/)[33]和PSIPRED (http://bioinf.cs.ucl.ac.uk/ PSIPRED/)[34-35]。使用Phyre2 (http://www.sbg.bio.ic. ac.uk/phyre2/html)[36]预测BnPINs蛋白的三级结构。 1.2.8 低氮胁迫下甘蓝型油菜PIN家族基因时空表达和共表达网络分析 1.2.8.1 营养液培养试验 培养试验在湖南农业大学植物营养与养分高效利用课题组光照培养室进行, 温度设置为22ºC, 光照周期为14 h (光照)/10 h (黑暗), 光照强度为300~320 μmol m–2s–1, 湿度为60%~75%。选取大小一致的油菜种子, 用1%的NaClO灭菌10 min, 将种子表面冲洗干净后, 4ºC下用灭菌超纯水(>18.25 MΩ)浸泡24 h, 然后将种子播种到塑料育苗盘固定的纱布上, 育苗盘中加适量超纯水。1周后将长势一致的幼苗移栽至盛有10 L营养液的黑色塑料盆中, 营养液采用Hoagland配方[37]。试验设置2个氮水平: 正常氮(9 mmol L–1NO3–)和低氮(0.3 mmol L–1NO3–)。 1.2.8.2 高通量转录组测序 油菜幼苗先在正常氮水平培养10 d, 然后将其移至低氮处理, 地上部(S)和根(R)在0、3和72 h取样, 每个处理准备3个独立的生物学重复。测序平台为Illumina Hiseq 2000[38], 每个样品产生6.0-Gb测序数据, 测序方式为双末端(PE)测序, 读长为150 bp。使用Multiple Experiment Viewer (http://www.tm4.org/mev.html)[39]绘制基因表达谱的热图, 使用错误发现率(FDR)≤0.05和倍数变化(log2)≥1作为鉴定差异表达基因的阈值。 1.2.8.3 基因共表达网络分析 构建基因共表达网络鉴定BnPINs家族基因相互作用的强弱并定位低氮胁迫下连接最相邻的核心基因, 相关值阈值设置为默认参数(http://plantgrn.noble.org/DeGNServer/ Analysis.jsp), 然后用Cytoscapev.3.2.1 (http://www. cytoscape.org/)[40]进行基因共表达网络图谱的绘制。 以AtPINs的氨基酸序列为查询对象, 对NCBI数据库进行BLAST搜索, 在异源四倍体甘蓝型油菜中鉴定了29个, 明显多于拟南芥、甘蓝和白菜具有的PIN家族基因数量。然而甘蓝型油菜中基因数量远少于甘蓝和白菜中的总数, 表明在异源多倍体过程中遭受严重的基因损失。29个BnPINs家族基因包括2个、6个、4个、4个、4个、2个、5个和2个(图1)。 图1 甘蓝型油菜、拟南芥、甘蓝、白菜PIN家族基因拷贝数变异 柱状图顶部数值为该物种拷贝的基因数目。 The number at the top of the histogram is the number of genes copied for that species. 利用AtPINs数据在甘蓝型油菜全基因组数据库中共检索到29条基因序列, 鉴定结果和序列分析表明, 29个基因编码的蛋白质平均含有527个氨基酸。氨基酸数目最多的是BnPIN2e蛋白, 长度为643个氨基酸, 预测分子量是69.44 kDa, 理论等电点(pI)为9.24; 氨基酸数目最少的是BnPIN5a蛋白, 长度为348个氨基酸, 预测分子量是38.25 kDa, 理论等电点(pI)为5.96; 29条编码的蛋白质等电点变化范围为5.96 (BnPIN5a)~9.38 (BnPIN2f), 大部分编码氨基酸为碱性氨基酸, 仅有BnPIN4d、BnPIN5a、BnPIN5b、BnPIN5c和BnPIN5d蛋白主要由酸性氨基酸组成; BnPIN2e蛋白的外显子/内含子数量最多, 含有13个外显子/12个内含子; 根据亲水性指数介于-0.5~ +0.5为两性蛋白(GRAVY为负值表示亲水性, 正值表示疏水性)的原则[41], 发现仅有BnPIN5a、BnPIN5b、BnPIN5c、BnPIN5d和BnPIN8a、BnPIN8b是疏水性蛋白, 其余均为两性蛋白(表1)。 使用SMART分析BnPINs蛋白保守结构域, 由图2可以看出, 即使BnPIN蛋白保守结构域在同一个类别, 其位置也有差异。29个BnPINs蛋白都具有跨膜结构域, 都在第7~641位的氨基酸残基片段上, BnPIN1a蛋白具有最少的跨膜结构域(5个); BnPIN5dBnPIN7bBnPIN7cBnPIN8b蛋白具有最多的跨膜结构域(均含10个); 绝大部分BnPINs蛋白还有1~2个位于第132~453位氨基酸残基片段的低容量跨膜结构域(除了BnPIN7a蛋白位于第76~88位残基片段上外), 但BnPIN2d、BnPIN3b、BnPIN5a、BnPIN5b、BnPIN5c、BnPIN7c、BnPIN8b蛋白均不含有。通过PIN家族蛋白氨基酸系列比对, 鉴定出BnPINs蛋白高度保守的氨基酸残基, Phe-49、Pro-52、Pro-64、Val-159、Gln-161、Trp-165、Leu-170、Phe-171、Glu-174、Lys-538、Asn-542、Pro-543、Asn-544、Tyr-546、Gly-551、Trp-554、Phe-567、Ser-573、Ser-578、Gly-581、Gly-583、Met-586、Phe-587、Phe-643, 这24个氨基酸残基在所有BnPINs蛋白系列中均高度保守(图3)。这些结果表明, BnPINs蛋白具有参与生长素外向型运输的跨膜结构域。 染色体定位发现,基因并不是均匀分布在甘蓝型油菜基因组中, 29个分布于A/C亚基因组, 13个基因分别位于A亚基因组的A2、A3、A7、A8、A10染色体, 10个基因分别位于C亚基因组的C2、C3、C6、C7、C8、C9染色体。、、、位于A2染色体,、位于A3染色体,、、、位于A7染色体,位于A8染色体,、位于A10染色体, 而、位于C2染色体,、位于C3染色体,、、位于C6染色体,位于C7染色体,位于C8染色体,位于C9染色体, 其余6个基因由于基因组拼接及注释不完整具体物理位置尚未确定(图4)。 保守基序分析发现, BnPINs蛋白N末端具有保守1 (motif 1)基序。甘蓝型油菜PIN家族蛋白保守基序数量变化具有一定规律, 数量为1~10, 除了BnaC02g20430D、BnaCnng76990D外, 其他序列都含有10个保守基序。BnaCnng54140D、BnaA10g 17940D、BnaC03907830D、BnaA03g06090D、Bna Cnng51580D和BnaA02g02480D缺失8 (Motif 8)基序(图5)。 利用邻接法构建甘蓝型油菜、拟南芥、甘蓝和白菜PIN家族基因系统进化树。由图6可知: (1)整个进化树可分为两大组, 第一组是~和基因, 第二组为、和基因, 该结果与Cazzonelli等[42]将拟南芥PIN蛋白分为PIN1组(AtPIN1~AtPIN4和AtPIN7)和PIN5组(AtPIN5、AtPIN6和AtPIN8)结果一致。(2) BnPINs家族基因与甘蓝和白菜基因进化关系较近,和、和、和、和均位于同一个进化分支,和进化关系也相近。、和分别与、和基因进化关系密切。 蓝色代表跨膜结构域, 粉色代表低复杂度结构。 Transmembrane regions are in blue and low complexities are in pink. 使用GOR4和SPIPRED进行BnPINs蛋白的二级结构分析, GOR4和SPIPRED预测结构类似(图7)。运用Phyre2数据库、分析的保守氨基酸残基和PIN蛋白二级结构对BnPINs蛋白进行三级结构预测(图8)。由BnPIN5b蛋白二级结构和BnPIN2a蛋白三级预测结构可知, α螺旋是BnPINs蛋白的主要结构, BnPIN5b蛋白二级结构特征为, α螺旋>延伸链>无规则卷曲>β转角, 这些结果与PIN蛋白向胞外运输生长素的功能紧密联系。 图3 甘蓝型油菜PIN家族蛋白氨基酸序列比对和保守结构域分析 黄色区域为高度保守的氨基酸位点。 The yellow area is a highly conserved amino acid site. 图4 23个甘蓝型油菜PIN基因染色体分布 图5 甘蓝型油菜PIN家族蛋白保守基序分布 左侧为蛋白系列名称, 右侧不同颜色分别对应不同的保守基序在系列上的位置。 The different colors on the right correspond to the positions of different conserved motifs on the series, respectively. 由图9可以看出,、、基因主要在甘蓝型油菜根部表达且受长期低氮(72 h)诱导;和基因主要在地上部表达, 低氮会抑制表达。为了进一步确定29个中哪一个是核心基因, 又构建了基因共表达网络,被鉴定为核心基因, 在低氮胁迫下, 29个基因在地上部的表达有一定规律(图10-A); 基因表达量几乎都随着低氮胁迫时间延长而先下降后上升, 且在3 h出现最低值, 但在根部未出现这一规律(图10-B)。 生长素的极性运输与植物生长发育密切相关, 如植物的向光性、向地性、顶端优势、根形成、花发育、胚胎形态建成、维管组织形成等[43]。随着分子生物学和遗传学发展, 在生长素结合蛋白、外输载体和相关基因克隆等领域已取得一系列进展。鉴定了多种拟南芥生长素运输载体, 对其功能和运输机制进行了研究, 而在其他作物中的此类研究十分有限。本研究发现BnPINs家族基因与甘蓝和白菜基因进化关系相近, 但在遗传和进化过程中基因遭受严重损失,基因拷贝数丢失。BnPINs蛋白多为碱性氨基酸组成的稳定蛋白, 定位在细胞质膜上, 具有保守的N末端结构域和5~6次跨膜折叠, 二级结构与AtPIN蛋白相似, 这些结构与BnPINs蛋白向胞外运输生长素的功能紧密相关。因此, 鉴定并获得不同物种PIN家族基因的遗传和进化信息, 了解其编码的PIN蛋白结构, 对于进一步研究PIN家族基因生物学功能和探索生长素外向型运输载体(PIN蛋白)定位机制、影响定位的因素和运输载体之间的相互作用具有重要意义。 图6 甘蓝型油菜PIN家族基因系统进化分析 不同颜色代表不同的进化分支, I、II代表家族2个亚组。 Different clades are in different colors, I and II above clades indicate the two subgroups offamily. 图7 甘蓝型油菜BnPIN5b蛋白二级结构分析 GOR4和PSIPRED分别是进行二级结构分析的2种不同的工具。 GOR4 and PSIPRED are two tools for the secondary structure analysis. 图8 甘蓝型油菜BnPIN2a蛋白三级结构预测 三级结构预测该晶体为转运子。 The three-dimensional structure predicts that the crystal is a transporter. 近年来, PIN蛋白通过调控生长素调控植物逆境胁迫的研究层出不穷, 且在植物响应营养元素缺乏或毒害中也有报道。缺硼时, 拟南芥通过调控基因的表达调控生长素在根系的分布从而影响根系生长[44]; 磷镁互作时可能通过介导、和基因在根中的表达调控根系构型[45]; 超表达水稻基因缓解了水稻根系铝毒[46], 延迟了体内磷素由营养器官向生殖器官的转移[47]。 油菜对氮素养分具有较高的营养需求, 但其氮素利用率很低, 缺氮会严重抑制油菜产量和品质的提高[22]。已有研究表明, 缺氮会增加拟南芥主茎中生长素的极性运输水平, 从而增强顶端优势, 造成地上部分枝数减少[23]。氮素缺乏可能通过改变生长素极性运输基因的表达, 从而影响植株的生长表型, 最终影响产量。由此可见,基因与氮素营养的互作在植物生长发育过程中发挥重要作用。本研究利用转录组测序发现,基因的表达具有较强组织特异性,、、基因主要在油菜根部表达且受长期低氮(72 h)诱导,和基因主要在地上部表达, 低氮会抑制表达, 反映了PIN家族基因时空表达的特性[48]。上述研究结果为进一步揭示甘蓝型油菜PIN家族基因在低氮胁迫下调控生长素的极性运输作用奠定了研究基础, 为发展氮高效油菜新品种选育提供了新方向。 图9 低氮胁迫下甘蓝型油菜PIN家族基因转录组分析 S: 地上部; R: 根; 0、3、72 (h)代表缺氮胁迫的时间。图中结果表示3次生物学重复的平均值。 S: shoot; R: root; 0, 3, and 72 (h) represent the time course of nitrogen deficiency. The results represent the average of three biological replicates. 图10 低氮胁迫下甘蓝型油菜PIN家族基因共表达网络分析和表达量分析 A: 共表达网络分析; B: 基因表达量分析; 圆圈节点代表基因, 节点的大小代表节点之间相互作用的能力, 两个节点之间的连线代表基因之间的相互作用; S: 地上部; R: 根; 0、3、72 (h)代表缺氮胁迫的时间。图中结果表示3次生物学重复的平均值。 A: co-expression network analysis; B: expression of PIN family genes; cycle nodes represent genes, and the size of the nodes represents the power of the interrelation among the nodes by degree value, edges between two nodes represent interactions between genes; S: shoot; R: root; 0, 3, and 72 (h) represents the time course of nitrogen deficiency. The results represent the average of three biological replicates. 十字花科作物PIN家族基因拷贝数存在差异, 甘蓝型油菜比与它进化关系相近的甘蓝和白菜具有更多同源基因,基因分别位于A/C亚基因组的A2、A3、A7、A8、A10、C2、C3、C6、C7、C8、C9染色体。BnPINs蛋白定位在细胞质膜上, 主要是α螺旋结构, 多为两性蛋白, 具有高度保守的氨基酸残基位点、N端保守基序和多个跨膜结构域, 表明BnPINs蛋白可作为细胞质膜上的运输载体参与生长素向胞外运输。基因表达具有组织特异性, 低氮胁迫显著改变了一些基因的表达丰度, 进而可能影响生长素在作物体内的分配, 最终影响作物生长发育。 [1] 刘士平, 王璐, 王继荣, 薛艳红, 寿惠霞. 高等植物的基因家族. 植物生理学通讯, 2009, 45: 833–841 Liu S P, Wang L, Wang J R, Xue Y H, Shou H X.gene family in higher plants., 2009, 45: 833–841 (in Chinese) [2] Petrášek J, Friml J. Auxin transport routes in plant development., 2009, 136: 2675–2688 [3] Paponov I A, Teale W D, Trebar M, Blilou I, Palme K. The PIN auxin efflux facilitators: evolutionary and functional perspectives., 2005, 10: 170–177 [4] Abas L, Benjamins R, Malenica N, Paciorek T, Wiśniewska J, Anzola J M, Sieberer T, Friml J, Luschnig C. Intracellular trafficking and proteolysis of the Arabidopsis auxin efflux facilitator PIN2 are involved in root gravitropism., 2006, 8: 249–256 [5] Schnabel E L, Frugoli J. Theandfamilies of auxin transport genes in., 2004, 272: 420–432 [6] Krecek P, Skupa P, Libus J, Naramoto S, Tejos R, Friml J, Zažímalová E. The PIN-FORMED(PIN) protein family of auxin transporters., 2009, 10: 249–254 [7] Gälweiler L, Guan C, Müller A, Wisman E, Mendgen K, Yephremov A, Palme K. Regulation of polar auxin transport by AtPIN1 in Arabidopsis vascular tissue., 1998, 282: 2226–2230 [8] Blilou I, Xu J, Wildwater M, Willemsen V, Paponov I, Friml J, Heidstra R, Aida M, Palme K, Scheres B. The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsisroots., 2005, 433: 39–44 [9] Ding Z, Galván-Ampudia C S, Demarsy E, Łangowski L, Kleine-Vehn J, Fan Y, Morita M T, Tasaka M, Fankhauser C, Offringa R. Light-mediated polarization of the PIN3 auxin transporter for the phototropic response in., 2011, 13: 447–452 [10] Friml J, Benková E, Blilou I, Wisniewska J, Hamann T, Ljung K, Woody S, Sandberg G, Scheres B, Jürgens G. AtPIN4 mediates sink-driven auxin gradients and root patterning in Arabidopsis., 2002, 108: 661–673 [11] Mravec J, Skupa P, Bailly A, Hoyerová K, Krecek P, Bielach A, Petrásek J, Zhang J, Gaykova V, Stierhof Y D, Dobrev P, Schwarzerová K, Rolcík J, Seifertová D, Luschnig C, Benková E, Zazimalová E, Geisler M, Friml J. Subcellular homeostasis of phytohormone auxin is mediated by the ER-localized PIN5 transporter., 2009, 459: 1133–1140 [12] Benková E, Michniewicz M, Sauer M, Teichmann T, Seifertová D, Jürgens G, Friml J. Local, efflux-dependent auxin gradients as a common module for plant organ formation., 2003, 115: 591–602 [13] Kleine-Vehn J, Ding Z, Jones A R, Tasaka M, Morita M T, Friml J. Gravity-induced PIN transcytosis for polarization of auxin fluxes in gravity-sensing root cells., 2010, 107: 22344–22349 [14] Ding Z, Wang B, Moreno I, Dupláková N, Simon S, Carraro N, Reemmer J, Pěnčík A, Chen X, Tejos R, Skupa P, Pollmann S, Mravec J, Petrášek J, Zažímalová E, Honys D, Rolčík J, Murphy A, Orellana A, Geisler M, Friml J. ER-localized auxin transporter PIN8 regulates auxin homeostasis and male gametophyte development in Arabidopsis., 2012, 3: 941 [15] Chen R, Hilson P, Sedbrook J, Rosen E, Caspar T, Masson P H. Thegene encodes a component of the polar-auxin-transport efflux carrier., 1998, 95: 15112–15117 [16] 李俊华, 种康. 植物生长素极性运输调控机理的研究进展. 植物学通报, 2006, 23: 466–477 Li J H, Chong K. Current research advances on polar auxin transport in plant., 2006, 23: 466–477 (in Chinese with English abstract) [17] 刘进平. 生长素运输机制研究进展. 中国农学通报, 2007, 23(5): 432–433Liu J P. Research advances on auxin transport mechanism., 2007, 23(5): 432–433 (in Chinese with English abstract) [18] 李运合, 孙光明, 吴蓓. 植物生长素的极性运输载体研究进展. 西北植物学报, 2009, 29: 1714–1722 Li Y H, Sun G M, Wu B. Advances on carriers of plant polar auxin transport.-,2009, 29: 1714–1722(in Chinese with English abstract) [19] 朱德进, 张辉, 黄卉, 宁运旺, 张永春. 不同施肥处理对不同地力水平油菜产量和经济效益的影响. 江苏农业科学, 2013, 41(10): 73–76 Zhu D J, Zhang H, Huang H, Ning Y W, Zhang Y C. Different fertilization treatments on rape yield at different soil fertility levels and the impact of economic benefits.,2013, 41(10): 73–76(in Chinese with English abstract) [20] Wang X, Wang H, Wang J, Sun R, Wu J, Liu S, Bai Y, Mun J H, Bancroft I, Cheng F. The genome of the mesopolyploid crop species., 2011, 43: 1035–1039 [21] Bayer P E, Hurgobin B, Golicz A, Chan C K, Yuan Y, Lee H T, Renton M, Meng J, Li R, Long Y, Zou J, Bancroft I, Chalhoub B, King G J, Batley J, Edwards D. Assembly and comparison of two closely relatedgenomes., 2017, 10: 1–9 [22] Rathke G W, Christen O, Diepenbrock W. Effects of nitrogen source and rate on productivity and quality of winter oilseed rape (L.) grown in different crop rotations., 2005, 94: 103–113 [23] de Jong M, George G, Ongaro V, Williamson L, Willetts B, Ljung K, Leyser O. Auxin and strigolactone signaling are required for modulation of Arabidopsis shoot branching by nitrogen supply., 2014, 166: 384–395 [24] Liu J X, An X, Cheng L, Chen F J, Bao J, Yuan L X, Zhang F S, Mi G H. Auxin transport in maize roots in response to localized nitrate supply., 2010, 106: 1019–1026 [25] Finn R D, Bateman A, Clements J, Coggill P, Eberhardt R Y, Eddy S R, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer E L L, Tate J, Punta M. Pfam: the protein families database., 2013, 27: 1–9 [26] Letunic I, Doerks T, Bork P. SMART: recent updates, new developments and status in 2015., 2015, 43: D257–D260 [27] Gasteiger E, Hoogland C, Gattiker A, Hoogland C, Ivanyi I, Appel R D, Bairoch A. ExPASy: the proteomics server for in-depth., 2003, 31: 3784–3788 [28] Hofmann K, Stoffel W. TM base-A database of membrane spanning protein segments., 1993, 374: 1-6 [29] Bailey T L, Elkan C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers., 1994, 2: 28–36 [30] Larkin M A, Blackshields G, Brown N P, Chenna R, McGettigan P A, McWilliam H, Valentin F, Wallace I M, Wilm A, Lopez R, Thompson J D, GibsonT J, Higgins D G. Clustal W and Clustal X version 2.0., 2007, 23: 2947–2948 [31] Kumar S, Nei M, Dudley J, Tamura K. MEGA: a biologist centric software for evolutionary analysis of DNA and protein sequences., 2008, 9: 299–306 [32] Krzywinski M, Schein J, Birol I, Connors J, Gascoyne R, Horsman D, Jones S J, Marra M A. Circos an information aesthetic for comparative genomics., 2009, 19: 1639–1645 [33] Combet C, Blanchet C, Geourjon C, Deléage G. Network protein sequence analysis., 2000, 25: 147–150 [34] Jones D T. Protein secondary structure prediction based on position-specific scoring matrices., 1999, 292: 195–202 [35] Buchan D W A, Minneci F, Nugent T C O, Bryson K, Jones D T. Scalable web services for the PSIPRED protein analysis workbench., 2013, 41: W340–W348 [36] Mezulis S, Sternberg M J E, Kelley L A. Phyre Storm: a web server for fast structural searches against the PDB., 2016, 428: 702–708 [37] Hoagland D R, Arnon D I. The water culture method for growing plants without soil., 1950, 347: 32 [38] Morin D, Bainbridge M, Fejes A, Hirst M, Krzywinski M, Pugh T J, McDonald H, Varhol R, Jones S J M, Marra M A. Profiling the He La S3 transcriptome using randomly primed cDNA and massively parallel short-read sequencing., 2008, 45: 81–94 [39] Eisen M B, Spellman P T, Brown P O, Botstein D. Cluster analysis and display of genome-wide expression patterns., 1998, 95: 14863–14868 [40] Kohl M, Wiese S, Warscheid B. Cytoscape: software for visualization and analysis of biological networks., 2011, 696: 291–303 [41] 王占军, 金伦, 徐忠东, 欧祖兰. 麻风树基因的生物信息学分析. 生物学杂志, 2014, 31(4): 68–72 Wang Z J, Jin L, Xu Z D, Ou Z L. Bioinformatics analysis of genefrom., 2014, 31(4): 68–72(in Chinese with English abstract) [42] Cazzonelli1 C I, Vanstraelen M, Simon S, Yin K, Carron-Arthur A, Nisar N, Tarle G, Cuttriss A J, Searle I R, Benkova E, Mathesius U, Masle J, Friml J, Pogson B J. Role of thePIN6 auxin transporter in auxin homeostasis and auxin-mediated development., 2013, 8: 1–14 [43] 倪迪安, 许智宏. 生长素的生物合成、代谢、受体和极性运输. 植物生理学通讯, 2001, 37: 346–352 Ni D A, Xu Z H. Auxin biosynthesis, metabolism, receptor and polar transport., 2001, 37: 346–352 (in Chinese) [44] Li K, Kamiya T, Fujiwara T. Differential roles of PIN1 and PIN2 in root meristem maintenance under low-B conditions in., 2015, 56: 1205–1214 [45] Niu Y F, Jin G L, Li X, Tang C X, Zhang Y S, Liang Y C, Yu J Q.Phosphorus and magnesium interactively modulate the elongation and directional growth of primary roots in(L.) Heynh., 2015, 66: 3–14 [46] Wu D, Shen H, Yokawa K, Baluška F. Alleviation of aluminium-induced cell rigidity by overexpression ofin rice roots., 2014, 65: 5305–5315 [47] 陈赢男. 生长素转运蛋白对水稻株型、根系生长和磷素营养的调控作用. 南京农业大学博士学位论文, 江苏南京, 2012Chen Y N. Regulation of Auxin Transporter on Plant Type, Root Growth and Phosphorus Nutrition in Rice. PhD Dissertation of Nanjing Agricultural University, Nanjing, Jiangsu, China, 2012(in Chinese with English abstract) [48] Peer W N, Bandyopadhyay A, Blakeslee J J, Makam S N, Chen R J, Masson P H, Murphy A S. Variation in expression and protein localization of the PIN family of auxin efflux facilitator proteins in flavonoid mutants with altered auxin transport in., 2004, 16: 1898–1911 Identification and Bioinformatics Analysis of thePIN Family Gene in GAO Kun1,2, HUA Ying-Peng1,2, SONG Hai-Xing1,2, GUAN Chun-Yun3, ZHANG Zhen-Hua1,2, and ZHOU Ting1,2,* 1College of Resource and Environment, Hunan Agricultural University, Changsha 410128, Hunan, China;2Southern Regional Collaborative Innovation Center for Grain and Oil Crops in China, Changsha 410128, Hunan, China;3Hunan Branch, National Oil Crops Improvement Center, Changsha 410128, Hunan, China The PIN family is a type of important carrier elements that regulate the polar transportation of auxin. Thegenes encode auxin efflux carriers with multiple transmembrane domains that mediate auxin transport in plants. However, there is a lack of systematic research in the genome of complexity. In this study, thegenes were screened from theDatabase using bioinformatics, and study on molecular characteristics of BnPIN proteins, such as copy number variations, transmembrane domains, conserved motifs, chromosomal locations, phylogenetic relationships, secondary and three-dimensional structures, and high-throughput transcriptome sequencing was used to analyze the transcriptional level under low nitrate stress. The results showed that most of the BnPIN proteins which is belonged to the stable protein consisting of basic amino acids. TheBnPIN family proteins contained secondary structures similar to those of Arabidopsis PINs accompanied by conserved N-terminal domains. The phylogenetic analysis showed thatgenes were similar to the corresponding homologs ofand. High-throughput transcriptome analysis showed that the,, andgenes were mainly expressed in roots ofunder long-term (72 h) low nitrate (NO3–) stress. Theandgenes were mainly expressed in the shoot and limited NO3–repressed theexpression. This study is valuable for the research that the roles of thefamily in the regulation of auxin transport. Our results also provide reference for the integrated genomic and transcriptomic studies of gene family in plant species with complex genomes. ; auxin;; gene family; bioinformatics 2018-01-30; 2018-06-12; 2018-06-29. 10.3724/SP.J.1006.2018.01334 周婷, E-mail: zhoutingplant@foxmail.com E-mail: gaokun0874@foxmail.com 本研究由国家重点研发计划项目(2017YFD0200103), 国家自然科学基金项目(31101596, 31372130), 湖南农业大学新进教师科研启动基金项目(30555|550100100021), 湖南农业大学校青年基金项目(17QN40)和国家现代农业产业技术体系建设专项资助。 This study was supported by the National Key R&D Program of China (2017YFD0200103), the National Natural Science Foundation of China (31101596, 31372130), the Research Starting Foundation for New Teachers of Hunan Agricultural University (30555|550100100021), the Youth Foundation of Hunan Agricultural University (17QN40), and the China Agriculture Research System. URL:http://kns.cnki.net/kcms/detail/11.1809.S.20180628.1740.004.html2 结果与分析
2.1 十字花科作物PIN家族基因拷贝数变异

2.2 甘蓝型油菜PIN家族基因分子特征
2.3 甘蓝型油菜PIN家族蛋白保守结构域和氨基酸系列比对分析
2.4 甘蓝型油菜PIN家族基因染色体的位置
2.5 甘蓝型油菜PIN家族蛋白保守基序分布
2.6 甘蓝型油菜PIN家族基因系统进化分析

2.7 甘蓝型油菜PIN蛋白二级结构分析和三级结构预测


2.8 甘蓝型油菜PIN家族基因在低氮胁迫下时空表达和基因共表达网络分析
3 讨论

4 结论
