大豆GmHDL57基因的克隆及抗盐功能鉴定
2018-09-11柯丹霞彭昆鹏张孟珂王净净
柯丹霞 彭昆鹏 张孟珂 贾 妍 王净净
大豆基因的克隆及抗盐功能鉴定
柯丹霞*彭昆鹏 张孟珂 贾 妍 王净净
信阳师范学院生命科学学院 / 大别山农业生物资源保护与利用研究院, 河南信阳 464000
HD-Zip I类转录因子在植物抵御非生物胁迫过程中发挥重要功能, 本研究克隆得到1个大豆HD-Zip I类基因(homeodomain-leucine zipper protein 57)。序列分析表明,基因包含1个1038 bp的开放读码框, 编码345个氨基酸, 具有HD-Zip类家族蛋白典型的保守结构域。基因时空表达分析表明, 大豆基因在大豆植株的各个不同时期及不同器官中均有表达, 在花中表达量最高。采用实时荧光定量PCR技术分析了4种非生物胁迫(脱落酸、NaCl、PEG、冷)对幼苗期大豆根中基因表达的影响。结果表明, 该基因表达量受高盐胁迫诱导显著升高, 在脱落酸及干旱胁迫下上升幅度较小, 但在冷胁迫下呈下降趋势。盐胁迫前后基因在根中的表达量明显高于茎和叶, 在盐胁迫48 h时达到峰值, 72 h和96 h时表达量缓慢下降。此外, 构建基因的植物超表达载体, 利用根癌农杆菌转化法获得转基因百脉根, 200 mmol L–1NaCl处理条件下, 转基因百脉根的株高、根长、叶绿素含量、根系活力以及阳离子K+、Ca2+含量显著高于野生型, 而丙二醛含量、相对质膜透性以及Na+的含量明显低于野生型。以上研究结果表明,基因参与了大豆对非生物胁迫的应答过程, 过量表达基因能够显著提高百脉根的抗盐能力。
大豆;基因; 非生物胁迫; 抗盐性
大豆是世界上重要的油料、粮食和经济作物, 高盐、干旱、低温等非生物胁迫严重影响了大豆的生长和产量。植物转录因子是一类重要的调控蛋白, 通过与靶基因互作以及不同转录因子之间形成复合物, 从而激活或抑制下游特定的信号转导通路。研究表明, 转录因子在作物抵抗非生物胁迫信号途径中发挥重要作用[1]。因此, 利用生物技术手段超量表达某一特定抗逆转录因子, 是培育大豆耐逆新品种, 提高大豆产量和品质的一条行之有效的途径。
同源异型域亮氨酸拉链蛋白HD-Zip (homeodomain leucine-zipper)是植物特有的一类转录因子, 包含一个由60个或61个氨基酸组成的同源异型结构域HD和一个亮氨酸拉链结构域LZ[2]。参与非生物胁迫应答的主要是HD-Zip I类蛋白[3]。拟南芥中有17个HD-Zip I类蛋白[4], 其中,和受干旱和外源ABA诱导表达, 盐胁迫和渗透胁迫能够诱导的表达[5-6]。烟草HD-Zip I类基因受失水胁迫诱导, 正调控ABA在叶片中的积累[7]。蒺藜苜蓿受NaCl胁迫诱导, 在根中表达量呈上调趋势[8]。紫花苜蓿受NaCl和ABA诱导, 负调控植株对盐胁迫的应答反应[9]。野生大豆HD-Zip I类基因受碱胁迫诱导在叶片和根中高表达, 超量表达提高了转基因拟南芥对碳酸盐的耐受性, 这一新发现为揭示野生大豆耐盐碱胁迫机制提供了线索[10]。
目前, 在大豆中鉴定到35个HD-Zip I类转录因子[11-12], 但是有关该类转录因子参与抗逆生理方面的研究报道甚少。早期研究发现1个大豆HD-Zip I类转录因子GmHZ-1正调控大豆花叶病毒(SMV)的感染[13]。我们在前期工作中克隆得到1个大豆HD-Zip I类盐胁迫应答基因, 超量表达显著增强了豆科模式植物百脉根的抗盐能力[14-15]。目前, HD-Zip I类转录因子在拟南芥中的抗逆分子机理研究日趋成熟, 而在大豆中的研究相对滞后。本研究克隆得到大豆的另一个HD-Zip I类基因, 从基因编码蛋白序列结构、系统进化树、基因的时空表达特性等方面进行了生物信息学分析。通过外源脱落酸、NaCl、PEG、冷等模拟非生物胁迫, 探究基因表达对非生物胁迫的响应特征。构建了Ubiquitin启动子驱动基因的植物表达载体, 获得稳定转化的模式豆科植物百脉根, 鉴定了该基因的抗盐功能, 揭示了基因在转基因百脉根抗盐胁迫中的调控机制, 为进一步利用基因创制抗盐大豆新种质奠定了基础。
1 材料与方法
1.1 材料及处理
栽培大豆Williams 82种子经消毒后, 于1/2 Hoagland培养基中萌发生长, 光照培养箱参数设置为温度25℃, 相对湿度60%, 18 h光照/6 h黑暗。待幼苗生长至V1期(第一复叶期)时进行非生物胁迫处理。其中, 3组幼苗分别使用含100 μmol L–1ABA、含100 mmol L–1NaCl、含30% PEG-6000的1/2 Hoagland培养液处理。另外1组幼苗移至1/2 Hoagland培养液中, 于4℃培养箱冷处理。每组试验处理18棵苗, 分别于胁迫前0 h、胁迫后1、6、12、24和48 h这6个时间点取样, 每次随机挑选3棵幼苗, 剪取根部组织, 液氮速冻后于–80℃冰箱储存备用。试验重复3次。
对另外一批生长至V1期的大豆幼苗, 用含100 mmol L–1NaCl的1/2 Hoagland培养液处理, 方法同上。设置3个重复, 每个重复18棵苗, 分别于盐胁迫前0 h、胁迫后12、24、48、72和96 h取样, 每个时间点随机挑选3棵幼苗, 分别收集幼苗根、茎、叶组织, 液氮速冻后于–80℃冰箱储存备用。
1.2 GmHDL57基因的克隆与重组质粒的构建
参照TaKaRa公司RNA抽提试剂盒操作说明, 提取各胁迫样品的总RNA。使用天根公司的反转录试剂盒制备cDNA第1链。根据NCBI网站公布的基因序列(GenBank: XM_006574409.2), 设计引物F-GmHDL57 (5¢-ATGGCGAGTGGCAAGC TTTATGC-3¢)和R-GmHDL57 (5¢-TCAATAGGGCCA GAAACAG-3¢), 以cDNA第1链为模板扩增目的片段, 大小为1038 bp。目的片段回收纯化后连接T载体测序验证。将测序正确的目的基因插入植物表达载体p1301U中, 上下游引物分别为F-GmHDL57- OX (5¢-CGggatccATGGCGAGTGGCAAG-3¢)和R- GmHDL57-OX (5¢-GGggtaccTCAATAGGGCCAGAA AC-3¢), 小写字母区域为酶切位点序列H I和I, 构建过表达载体p1301U- GmHDL57。
1.3 GmHDL57基因的生物信息学分析
利用DNAMAN软件确定基因的开放读码框以及编码蛋白的分子量大小; 利用SignaIP-4.1软件预测蛋白的信号肽及切割位点; 利用PSORT 软件分析蛋白的亚细胞定位情况; 利用在线软件ProtScale 预测蛋白的亲水、疏水氨基酸组成情况; 分别运用DictyOGlyc 1.1 Server、NetPhos 2.0 Server在线软件预测基因翻译后磷酸化修饰情况; 使用DNAMAN软件进行同源蛋白多序列比对分析以及系统进化树的构建; 利用SWISS MODEL Workspace 软件在线预测GmHDL57蛋白的三级结构模型。
1.4 GmHDL57基因的时空及胁迫表达检测
从转录组数据中[12]获取基因在幼叶、花、1 cm豆荚、开花后10、14 d荚壳、开花后10、14、21、25、28、35、42 d种子、根和根瘤共14个组织中的表达数据, 利用Excel表绘图分析基因的时空表达特征。
根据基因序列, 利用Primer 5.0软件设计1对实时荧光定量PCR引物, F-GmHDL57-rt (5¢-GAAAACCTTTTGATGACC-3¢)和R-GmHDL57- rt (5¢-TCAATAGGGCCAGAAACAG-3¢), 以各个胁迫样品的cDNA 第1链为模板, 按照TaKaRa公司PrimeScript RT reagent Kit操作说明进行。以大豆肌动蛋白11 ()为内参基因, 实时荧光定量PCR上游引物为F-ACT11 (5¢-ATTTTGACTGAGCGTG GTTATTCC-3¢); 下游引物为R-ACT11 (5¢-GCTGGT CCTGGCTGTCTCC-3¢)。根据相对定量法(DDCt)公式计算结果, 利用Microsoft Excel绘图分析基因在非生物胁迫下的表达特征。
1.5 百脉根稳定转化及盐胁迫处理
采用根癌农杆菌介导的子叶节转化法将基因导入百脉根中, 详细方法参见文献[16]。以转基因和野生型百脉根植株叶片DNA为模板, PCR扩增基因, 引物为F-Gus (5¢-ATGT TACGTCCTGTAGAAAC-3¢)和R-Gus (5¢-TCATT GTTTGCCTCCCTG-3¢), PCR产物大小为1812 bp。提取部分PCR阳性植株叶片RNA, 进行基因表达分析(引物同1.4 F-GmHDL57-rt和R-GmHDL57-rt), PCR产物大小为138 bp。以百脉根为内参基因, 引物为F-GPDH (5¢-GCCTCATT CAACATCATTCC-3¢)和R-GPDH (5¢-CTATGAACA ACAAAAGGTTGC-3¢), PCR产物大小为112 bp。
收集RT-PCR检测为阳性的植株种子即T0代转基因百脉根种子, 将其与野生型种子表面灭菌, 移入MS培养基上待种子萌发。1周后将幼苗移入含有基质的盆钵中, 3周后选取长势一致的幼苗, 将转基因和野生型植株各分成3组, 每组24 株, 分别用终浓度为0、100和200 mmol L–1NaCl 的1/8 Hoagland 营养液处理, 设置3个重复, 14 d后照相并测定相关生理生化指标。
1.6 转基因百脉根生理指标的测定
丙二醛含量测定、叶片质膜透性(相对电导率)测定、叶绿素含量测定、根系活力检测以及阳离子Na+、K+和Ca2+含量测定的具体操作方法见参考文献[14]。
2 结果与分析
2.1 GmHDL57基因的序列分析
根据基因的已知序列设计引物, 以Williams 82大豆cDNA为模板, 克隆得到与NCBI数据库中公布的基因序列完全一致的基因。该基因包含1个1038 bp的开放读码框, 编码345个氨基酸。GmHDL57蛋白的理论分子量为39.08 kD。利用SignaIP-4.1软件预测GmHDL57蛋白不存在信号肽及切割位点, 不属于分泌蛋白。经在线软件ProtScale 预测GmHDL57蛋白除了C端303~314位和中间215~241位氨基酸为疏水区, 其余大部分区域均为亲水区。运用DictyOGlyc 1.1 Server和NetPhos 2.0 Server 在线软件预测基因翻译后磷酸化修饰情况表明, GmHDL57蛋白有可能在24个位点发生磷酸化修饰, 并且磷酸化位点集中分布在疏水区域。利用PSORT软件预测GmHDL57蛋白的亚细胞定位情况, 发现其定位于细胞核。采用SWISS MODEL Workspace 软件在线预测蛋白的三级结构模型显示, GmHDL57蛋白主要由3个α螺旋、不规则卷曲与延伸链组成(图1)。3个α螺旋是由GmHDL57蛋白的HD区域折叠而成的三维空间结构, 这里是靶基因5¢上游区顺式作用元件的专一识别位点, 起着与DNA结合的功能, 因此α螺旋是该蛋白行使生物学功能的基础。LZ区的亮氨酸残基侧链伸出, 与α螺旋紧密相连, 二者共同作用形成一个稳定的拉链状的疏水作用区域。综上, GmHDL57是一个典型的HD-Zip类转录因子。
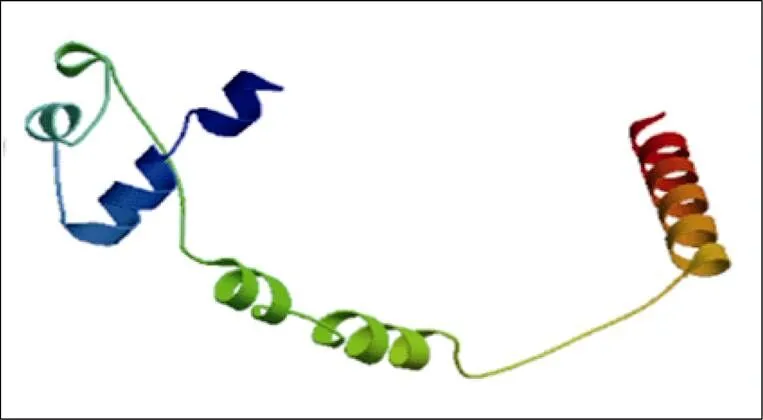
图1 GmHDL57蛋白质的三级空间结构预测
2.2 GmHDL57的同源蛋白比对及进化树分析
将大豆GmHDL57蛋白与另外10种豆科植物同源蛋白的保守结构域进行序列比对发现, GmHDL57蛋白与野生大豆、木豆、赤豆等物种的同源蛋白氨基酸序列相似性较高, 并且该类蛋白在不同物种中都有2个高度保守的区域, 分别为同源异型框结构域HD和亮氨酸拉链结构域LZ (图2)。
: 栽培大豆;: 野生大豆;: 木豆;: 赤豆;: 菜豆;: 绿豆;: 鹰嘴豆;: 狭叶羽扇豆;: 蒺藜苜蓿;: 蔓花生;: 落花生。黑线部分为同源异型框结构域序列, 虚线部分为同源异型框结合类亮氨酸拉链结构域序列。
The sequence marked with solid line demonstrates the homeobox domain, and the sequence marked with dotted line demonstrates the homeobox associated leucine zipper domain.
选取与基因编码的氨基酸序列相似性较高的几种植物的HD-Zip蛋白进行进化树分析。结果表明, 大豆GmHDL57蛋白与野生大豆亲缘关系最近, 其次是木豆、赤豆、菜豆和绿豆, 与鹰嘴豆、狭叶羽扇豆和蒺藜苜蓿的亲缘关系较远, 与蔓花生和落花生的亲缘关系最远(图3)。

图3 大豆GmHDL57与同系物的系统进化分析
: 栽培大豆;: 野生大豆;: 木豆;: 赤豆;: 菜豆;: 绿豆;: 鹰嘴豆;: 狭叶羽扇豆;: 蒺藜苜蓿;: 蔓花生;: 落花生。标尺代表遗传相似性, 表明不同物种间同系物进化关系的远近。
The scale represents genetic similarity, indicating the proximity relationships among species.
2.3 GmHDL57基因的时空表达分析
根据转录组数据[12]获取基因的时空表达情况, 从图4可以看出, 在幼叶、花、1 cm豆荚、根、根瘤以及开花后荚壳和种子中都能检测到基因的表达。在生殖器官花、豆荚和10 d种子中该基因表达量较高, 在营养器官幼叶中基因表达量较低。其中,基因在开花后10 d荚壳中的表达量高于14 d荚壳, 在开花后10 d种子中的表达量高于14 d、35 d和42 d种子, 在开花后25 d和28 d种子中表达量较低, 而在开花后21 d种子中未检测到该基因的表达。
2.4 GmHDL57基因在不同胁迫下的表达特征分析
NaCl和PEG胁迫处理1 h时,基因的表达量迅速上升, 且随处理时间的延长表达量持续升高, 至48 h 时达到最大值, 分别为胁迫前的8.5倍和7.2倍。而在ABA胁迫下,基因的表达量在1 h和6 h两个时间点增加缓慢, 至12 h时明显上升, 后又缓缓升高, 至48 h达到胁迫前的3.9倍。4℃冷胁迫处理时,基因的表达量持续下降, 至12 h达最小值, 为胁迫前的0.5倍, 之后趋于稳定, 表现出对冷胁迫的适应性(图5)。
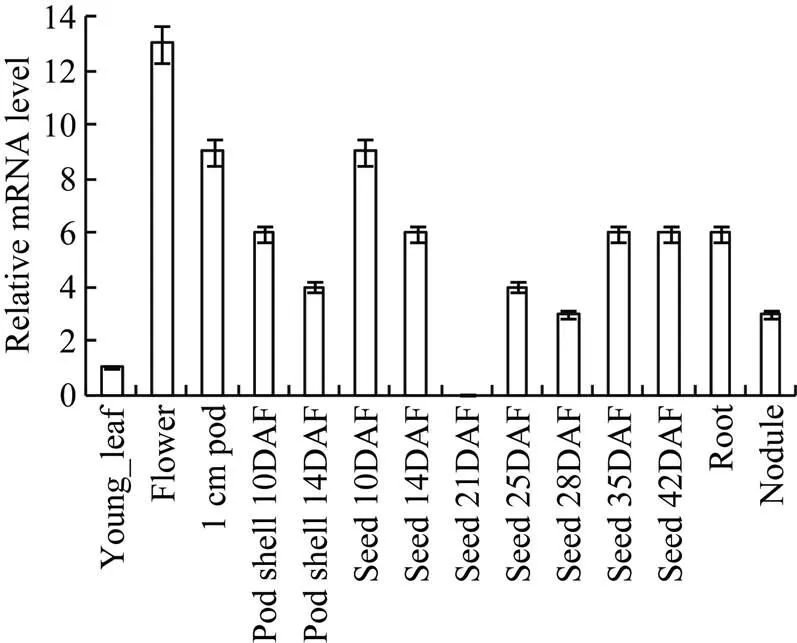
图4 大豆GmHDL57基因在不同器官中的表达分析
横坐标依次为: 幼叶、花、1 cm豆荚、开花后10 d、14 d荚壳、开花后10 d、14 d、21 d、25d、28 d、35 d、42 d种子、根和根瘤; DAF代表开花后的天数。
Abscissa represented young leaf, flower, 1 cm pod, pod shell at 10 d, pod shell at 14 d, seed at 10 d, seed at 14 d, seed at 21 d, seed at 25 d, seed at 28 d, seed at 3 d, seed at 42 d, root and nodule. DAF means days after flower.

图5 大豆GmHDL57基因在非生物胁迫下的表达分析
由图5可知, 大豆基因在响应高盐胁迫时基因表达量变化最为明显, 因此我们进一步对盐胁迫下幼苗期大豆根、茎、叶不同组织中基因的表达特征进行了分析。鉴于盐胁迫下该基因的表达量在48 h内一直持续上升, 并没有出现拐点, 因此, 我们延长胁迫处理取样时间至96 h。由图6可知, 盐胁迫前后基因在大豆根中的表达量明显高于茎和叶, 在盐胁迫处理48 h时,基因在根、茎、叶中的表达水平均达到峰值, 在72 h和96 h时表达量缓慢下降, 但仍高于盐胁迫处理前的表达水平。由此推断, 盐胁迫条件下基因具有组织表达特异性, 可能主要在根中参与盐胁迫应答反应。

图6 盐胁迫下GmHDL57基因在大豆不同组织中的表达特征分析
2.5 转基因百脉根的获得及抗盐表型分析
通过根癌农杆菌介导的百脉根子叶节转化法, 将构建成功的植物表达载体p1301U-GmHDL57导入百脉根中, 共获得46株卡那霉素抗性植株。经PCR鉴定, 获得28株PCR阳性植株(图7-A)。从中随机挑选5株进行RT-PCR检测, 均能检测到基因的表达(图7-B)。收集RT-PCR检测为阳性的植株种子, 不同株系的转基因种子与野生型种子经消毒在MS培养基上生长1周, 盆钵中生长3周后, 用0、100、200 mmol L–1NaCl进行盐胁迫处理。14 d后, 100 mmol L–1NaCl处理的野生型百脉根叶片开始发黄枯萎, 而转基因的2个株系生长状态优于对照; 随着盐浓度的升高, 200 mmol L–1NaCl处理的野生型百脉根生长受到明显抑制, 大部分植株茎叶枯黄, 生长矮小, 甚至死亡, 而转基因株系生长状态较好, 只有小部分植株叶片开始发黄干枯(图7-C)。
进一步对转基因和野生型百脉根的株高和根长测量统计发现, 在不含NaCl的基质中, 转基因株系和野生型百脉根株高和根长并无明显差异, 随着NaCl浓度的增加, 野生型株高和根长受到明显抑制, 而转基因株系株高和根长受抑制程度较轻(图8)。以上结果表明,基因的过表达能够减缓盐胁迫对植株的毒害作用, 植株的耐盐能力增强。
2.6 盐胁迫下转基因百脉根相关生理指标检测
从图9可以看出, 在正常条件下, 转基因和野生株系间丙二醛(MDA)含量、叶绿素含量、相对质膜透性以及根系活力的差异不显著。随着盐浓度的增加, 植株叶片MDA含量和相对质膜透性呈升高趋势, 但转基因株系升高幅度显著低于野生型。表明盐胁迫条件下, 转基因百脉根细胞膜的氧化损伤程度较轻, 能够维持较好的细胞膜生理活性。与之相反, 植株叶片叶绿素含量和根系活力随着盐浓度的增加而下降, 但转基因株系下降的幅度显著低于野生型, 表明转基因株系在盐胁迫条件下具备较强的光合能力以及较高的根系活力。

图7 转基因百脉根阳性植株检测及抗盐表型鉴定
A: PCR检测植株中基因的表达; B: RT-PCR检测植株中基因的表达; C: 不同盐浓度处理14 d后百脉根的生长状态。Lj 9-5, Lj 1-1: 转基因株系。M: DL2000 DNA marker。
A: detection ofgene expression in plants by PCR; B: detection ofgene expression in plants by RT-PCR; C: growth state ofin different salt concentration treatment for 14 d. Lj 9-5, Lj 1-1: transgenic lines. M: DL2000 DNA marker.

图8 盐胁迫对转基因百脉根株高(A)和根长(B)的影响
Lj 9-5, Lj 1-1: 转基因株系。*代表差异显著(<0.05), **代表差异极显著(<0.01)。
Lj 9-5, Lj 1-1: transgenic lines. * means significant difference (<0.05) and ** means extremely significant difference (<0.01).
2.7 盐胁迫下转基因百脉根中阳离子的含量分析
在无盐胁迫下, 转基因和野生型百脉根叶片和根系中Na+、K+和Ca2+含量无明显差异。随着盐浓度的升高, Na+和Ca2+含量均呈现升高趋势, 而K+含量呈下降趋势。与野生型对照相比, 转基因百脉根中Na+含量的增加幅度较小, Ca2+的增加幅度较大, 而K+的下降幅度较小(图10)。以上结果表明, 在高盐胁迫下,基因的过表达能够调节转基因百脉根体内的阳离子含量, 从而增强转基因百脉根对盐的耐受性。
3 讨论
近年来, 关于植物HD-Zip类转录因子参与非生物胁迫应答反应已成为研究热点。目前的研究主要集中在拟南芥、水稻[17-18]、玉米[19]、杨树[20]、黄瓜[21-22]等植物, 对于大豆中该类蛋白的功能研究报道较少, 尚处于起步阶段。本研究克隆得到1个HD-Zip I类基因, 其编码蛋白具有HD-Zip类家族蛋白典型的保守结构域。基因在大豆植株的各个不同生长阶段及不同器官中均有表达, 生殖生长较营养生长阶段表达量高。拟南芥和在营养生长和生殖生长阶段均有表达,主要在根和茎的皮层中表达,和主要在根、叶和花中表达, 而在根、茎、叶、花、子叶等各部位均有表达[23]。HD-Zip类家族基因表达规律的复杂性决定了该类转录因子功能的多样性。
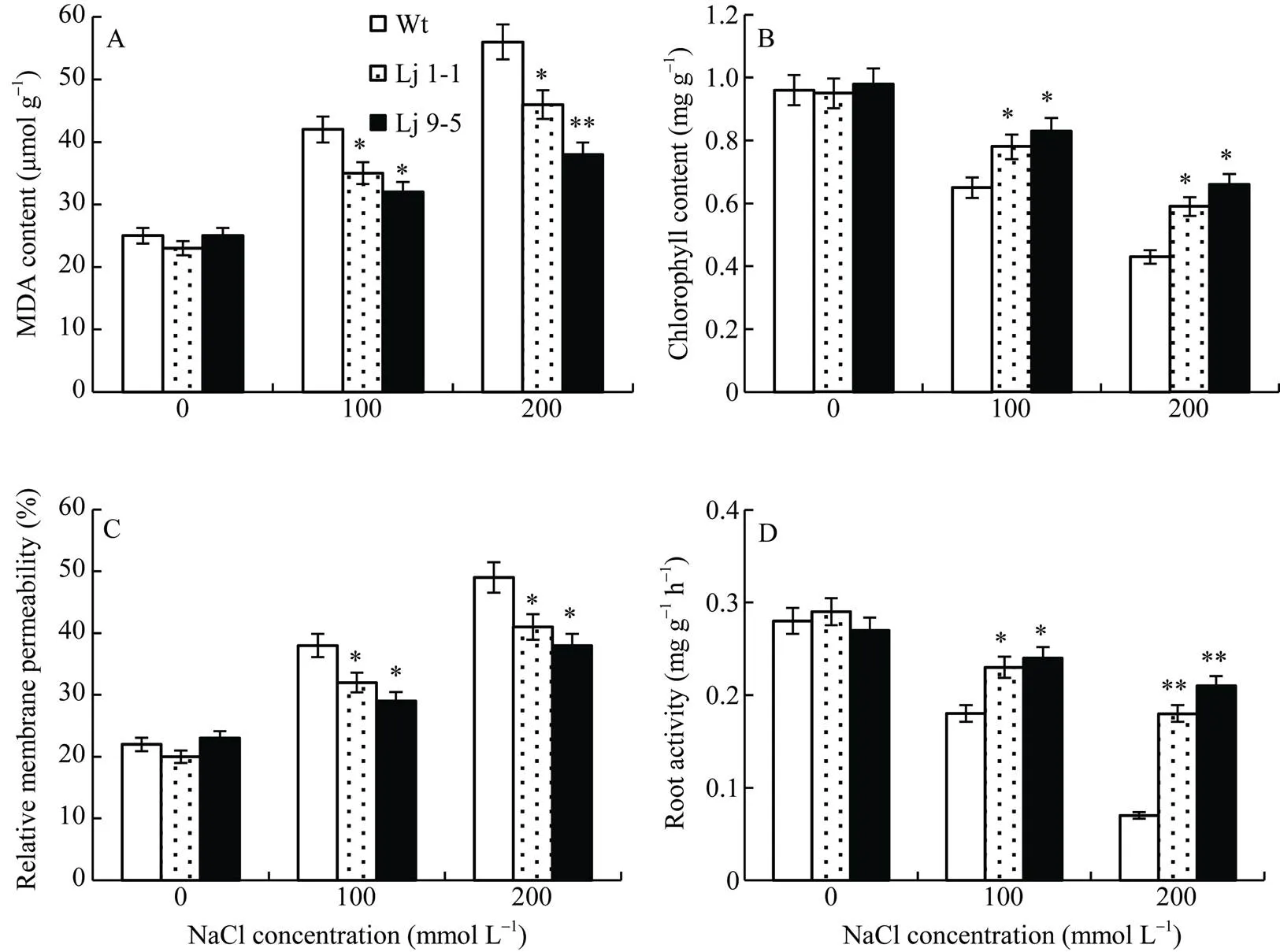
图9 盐胁迫下转基因百脉根的生理指标
A: 丙二醛含量; B: 相对质膜透性; C: 叶绿素含量; D: 根系活力。Lj 9-5, Lj 1-1: 转基因株系。*代表差异显著(<0.05), **代表差异极显著(<0.01)。
A: MDA content; B: relative membrane permeability; C: chlorophyll content; D: root activity. Lj 9-5, Lj 1-1: transgenic lines. * means significant difference (<0.05), ** means extremely significant difference (<0.01).
研究表明, 用ABA、NaCl或者低温处理拟南芥幼苗, HD-Zip类基因和的表达量上调至对照的2~25倍, 而基因的表达量减少为对照的一半。和受ABA或者NaCl影响, 其表达量下调为对照的0.5倍。和的表达不受外源ABA的影响, 经盐胁迫和低温处理后其表达量减少[24]。本研究中大豆基因在外源ABA、NaCl和PEG胁迫处理下, 表达量呈上升趋势, 48 h时达到对照的3.9~8.5倍。而在4℃冷胁迫处理时,基因的表达量整体呈下降趋势, 12 h时降至最低, 为对照的0.5倍。说明大豆基因的表达在盐和干旱胁迫下与ABA胁迫有着相似的应答模式, 而在冷胁迫下与ABA胁迫有着相反的应答模式。进一步对盐胁迫下大豆根、茎、叶不同组织中基因的表达特征分析发现, 盐胁迫条件下基因在根中表达量较高, 具有组织表达特异性, 可能主要在根中发挥功能。
本研究构建了基因的植物表达载体, 通过稳定转化获得了转基因百脉根植株, 并希望通过盐胁迫下转基因及野生型对照株系各项生理指标的测定揭示基因的抗盐功能及调控机制。通常高盐条件下, 植株的丙二醛含量和相对质膜透性会迅速增加, 导致质膜结构的破坏, 影响功能的发挥。本研究中转基因百脉根的丙二醛含量和相对质膜透性明显低于野生型对照, 说明盐胁迫下, 转基因百脉根的膜脂过氧化程度较低, 细胞膜受损程度较小。此外, 在高盐胁迫下, 转基因株系的叶绿素含量和根系活力显著高于野生型对照, 说明转基因百脉根在盐胁迫下具备较强的光合能力以及较高的根系活力。通过对以上生理指标的监测发现,基因的过表达减缓了盐胁迫对百脉根生长的抑制, 从而使转基因百脉根保持较好的生长状态。高盐胁迫影响植株对Na+、K+和Ca2+等阳离子的吸收。细胞质内Na+含量过高会对植物造成不利影响[25]。在植物细胞质内K+以相对较高的浓度存在, 参与调节离子平衡以及稳定细胞内环境[26-27]。Ca2+也是植物生长所必需的, 能够促进细胞对K+的吸收、调节水分平衡、充当第二信使等[28]。因此, 维持细胞质内较低的Na+水平以及较高的K+、Ca2+水平, 对植物抵抗盐胁迫具有重要意义[29]。本试验中, 在同一高盐浓度条件下, 转基因植株中Na+含量明显低于对照, 而K+和Ca2+含量显著高于对照, 说明在高盐胁迫下转基因植株维持较低的Na+水平, 较高的K+和Ca2+水平, 减轻了过量Na+对植株的毒害作用, 从而维持植株的正常生理功能。从盐胁迫诱导、组织表达特征和阳离子含量分析推断, 盐胁迫条件下基因主要在根中发挥功能, 控制Na+向地上部分运输和积累, 调节植物体内离子平衡, 进而降低盐胁迫对植株带来的伤害。

图10 盐胁迫下转基因百脉根的阳离子含量
A: 叶片中Na+含量; B: 根中Na+含量; C: 叶片中K+含量; D: 根中K+含量; E: 叶片中Ca2+含量; F: 根中Ca2+含量。Lj 9-5, Lj 1-1: 转基因株系。*代表差异显著(<0.05), **代表差异极显著(<0.01)。
A: Na+content in leaves; B: Na+content in roots; C: K+content in leaves; D: K+content in roots; E: Ca2+content in leaves; F: Ca2+content in roots. Lj 9-5, Lj 1-1: transgenic lines. * means significant difference (<0.05) and ** means extremely significant difference (<0.01).
我们推断基因的超量表达能够提高转基因百脉根的耐盐性。目前, 大豆遗传转化效率低仍然极大制约了大豆抗逆分子改良的发展[30-34], 因此优化大豆子叶节遗传转化体系, 获得转基因大豆植株, 进一步探究该基因在大豆体内的功能及其调控机制, 是我们下一步工作的重点。
4 结论
克隆得到一个大豆HD-Zip I类基因, 该基因在大豆植株的各个不同时期及不同器官中均有表达。基因的表达在盐和干旱胁迫下与ABA胁迫应答模式相似, 而在冷胁迫下与ABA胁迫应答模式相反。盐胁迫条件下基因在根中表达量较高, 具有组织表达特异性。在高盐胁迫下, 转基因百脉根的株高、根长、叶绿素含量、根系活力以及阳离子K+、Ca2+含量均显著高于野生型, 而丙二醛含量、相对质膜透性以及Na+的含量明显低于野生型。说明基因可能以不同机制参与了大豆对非生物胁迫的应答过程, 过量表达基因能够显著提高百脉根的抗盐能力。
[1] Ariel F D, Manavella P A, Dezar C A, Chan R L. The true story of the HD-Zip family., 2007, 12: 419–426
[2] Mukherjee K, Brocchieri L, Burglin T R. A comprehensive classification and evolutionary analysis of plant homeobox genes., 2009, 26: 2775–2794
[3] Harris J C, Hrmova M, Lopato S, Langridge P. Modulation of plant growth by HD-Zip class I and II transcription factors in response to environmental stimuli., 2011, 190: 823–837
[4] Henriksson E, Olsson A S B, Johannesson H, Johansson H, Hanson J, Engström P, Söderman E. Homeodomain leucine zipper class I genes inexpression patterns and phylogenetic relationships., 2005, 139: 509–518
[5] Olsson A S B, Engstrom P, SodermanE. The homeobox genesandencode potential regulators of growth in response to water deficit in., 2004, 55: 663–677
[6] Himmelbach A, Hoffmann T, Leube M, Höhener B, Grill E. Homeodomain protein ATHB6 is a target of the protein phosphatase ABI1 and regulates hormone responses in., 2002, 21: 3029–3038
[7] Ré D A, Dezar C A, Chan R L, Baldwin I T, Bonaventure G.NaHD20 plays a role in leaf ABA accumulation during water stress, benzylacetone emission from flowers, and the timing of bolting and flower transitions., 2011, 62: 155–166
[8] Ariel F, Diet A, Verdenaud M, Gruber V, Frugier F, Chan R, Crespi M. Environmental regulation of lateral root emergence inrequires the HD-Zip I transcription factor HB1., 2010, 22: 2171–2183
[9] 李明娜, 龙瑞才, 杨青川, 沈益新, 康俊梅, 张铁军. 紫花苜蓿盐诱导HD-Zip类转录因子的克隆及功能分析. 中国农业科学, 2014, 47: 622–632Li M N, Long R C, Yang Q C, Shen Y X, Kang J M, Zhang T J. Cloning and function analysis of a salt-stress-induced HD-Zip transcription factorfrom alfalfa., 2014, 47: 622–632 (in Chinese with English abstract)
[10] Cao L, Yu Y, Duanmu H Z,Chen C, Duan X B, Zhu P H, Chen R R, Li Q, Zhu Y M, Ding X D. A novelhomeodomain-leucine zipper (HD-Zip) I gene,, positively regulates bicarbonate tolerance and responds to osmotic stress in Arabidopsis., 2016, 16: 184
[11] Chen X, Chen Z, Zhao H, Zhao Y, Cheng B, Xiang Y. Genome-wide analysis of soybean HD-zip gene family and expression profiling under salinity and drought treatments., 2014, 9: e87156
[12] Belamkar V, Weeks N T, Bharti A K, Farmer A D, Graham M A, Cannon S B. Comprehensive characterization and RNA-Seq profiling of the HD-Zip transcription factor family in soybean () during dehydration and salt stress., 2014, 15: 950
[13] Wang YJ, Li Y D, Luo G Z, Tian A G, Wang H W, Zhang J S, Chen S Y.Cloning and characterization of an HD-Zip I genefrom soybean., 2005, 221: 831–843
[14] 柯丹霞, 李祥永, 王磊, 程琳, 刘永辉, 李小艳, 王慧芳. 大豆的克隆及其转基因百脉根的抗盐分析. 中国农业科学, 2017, 50: 1559–1570 Ke D X, Li X Y, Wang L, Cheng L, Liu Y H, Li X Y, Wang H F. Isolation offromand analysis of saline tolerance for transgenic., 2017, 50: 1559–1570 (in Chinese with English abstract)
[15] 柯丹霞, 李祥永. 结瘤信号途径中相关调控蛋白的研究进展. 信阳师范学院学报(自然科学版), 2015, 28: 621–626Ke D X, Li X Y. Research progress of key regulatory proteins in nodulation pathway.(Nat Sci Edn), 2015, 28: 621–626 (in Chinese with English abstract)
[16] Marquez A J.Handbook. The Netherlands: Springer, 2005. pp 279–284
[17] Agalou A, Purwantomo S, Overnas E, Johannesson H,Zhu X Y,Estiati A,de Kam R J,Engström P,Slamet-Loedin I H,Zhu Z,Wang M,Xiong L Z,Meijer A H, Ouwerkerk P B.A genome-wide survey of HD-Zip genes in rice and analysis of drought-responsive family members., 2008, 66: 87–103
[18] Zhang S X, Haider I, Kohlen W, Jiang L,Bouwmeester H,Meijer A H,Schluepmann H,Liu C M,Ouwerkerk P B.Function of the HD-Zip I genein ABA-mediated drought and salt tolerances in rice., 2012, 80: 571–585
[19] Zhao Y, Zhou Y Q, Jiang H Y, Li X Y,Gan D F,Peng X J,Zhu S W,Cheng B J. Systematic analysis of sequences and expression patterns of drought-responsive members of the HD-Zip gene family in maize., 2011, 6: e28488
[20] Hu R b, Chi X Y, Chai G H, Kong Y Z,He G,Wang X Y,Shi D C,Zhang D Y,Zhou G K.Genome-wide identification, evolutionary expansion, and expression profile of homeodomain- leucine zipper gene family in poplar ()., 2012, 7: e31149
[21] Liu W, Fu R, Li Q, Li J,Wang L N,Ren Z H.Genome-wide identification and expression profile of homeodomain-leucine zipper class I gene family in., 2013, 531: 279–287
[22] Fu R, Liu W, Li Q, Li J, Wang L N, Ren Z H. Comprehensive analysis of the homeodomain-leucine zipper IV transcription factor family in., 2013, 56: 395–405
[23] Elhiti M, Stasolla C. Structure and function of homeodomain-leucine zipper (HD-Zip) proteins., 2009, 4: 86–88
[24] Sahu B B, Shaw B P. Isolation, identification and expression analysis of salt-induced genes in, a natural halophyte, using PCR-based suppression subtractive hybridization., 2009, 9: 69
[25] 王臻昱, 才华, 柏锡, 纪巍, 李勇, 魏正巍, 朱延明. 野生大豆基因的克隆及其转基因苜蓿的耐盐碱性分析. 作物学报, 2013, 38: 971–979 Wang Z Y, Cai H, Bai X, Ji W, Li Y, Wei Z W, Zhu Y M. Isolation offromand analysis of saline- alkaline tolerance for transgenic., 2013, 38: 971–979 (in Chinese with English abstract)
[26] 魏正巍, 朱延明, 化烨, 才华, 纪巍, 柏锡, 王臻昱, 文益东. 转基因苜蓿植株的获得及其耐碱性分析. 作物学报, 2013, 39: 68–75 Wei Z W, Zhu Y M, Hua Y, Cai H, Ji W, Bai X, Wang Z Y, Wen Y D. Transgenic alfalfa withand its alkaline tolerance analysis., 2013, 39: 68–75 (in Chinese with English abstract)
[27] 赵阳, 朱延明, 柏锡, 纪巍, 吴婧, 唐立郦, 才华. 转双价基因苜蓿耐碱性及氨基酸含量分析. 作物学报, 2014, 40: 431–438 Zhao Y, Zhu Y M, Bai X, Ji W, Wu J, Tang L L, Cai H. Over-expressingenhances alkaline tolerance and methionine content in transgenic., 2014, 40: 431–438 (in Chinese with English abstract)
[28] Yang T, Poovaiah B W. Hydrogen peroxide homeostasis: activation of plant catalase by calcium/calmodulin., 2002, 99: 4097–4102
[29] 朱娉慧, 陈冉冉, 于洋, 宋雪薇, 李慧卿, 杜建英, 李强, 丁晓东, 朱延明. 碱胁迫相关基因的克隆及其转基因苜蓿的耐碱性分析. 作物学报, 2017, 43: 1319–1327Zhu P H, Chen R R, Yu Y, Song X W, Li H Q, Du J Y, Li Q, Ding X D, Zhu Y M. Cloning of generelated to alkaline stress and alkaline tolerance of transgenic plants., 2017, 43: 1319–1327 (in Chinese with English abstract)
[30] Olhoft P M, Flagel L E, Donovan C M. Efficient soybean transformation using hygromycin B selection in the cotyledonary-node method., 2003, 216: 723–735
[31] 王昌陵, 赵军, 李英慧, 范云六, 张丽娟, 刘章雄, 关荣霞, 吕淑霞, 常汝镇, 邱丽娟. 转录因子ABP9 转化大豆(L.)及遗传转化条件优化. 中国农业科学, 2008, 41: 1908–1916 Wang C L, Zhao J, Li Y H, Fan Y L, Zhang L J, Liu Z X, Guan R X, Lyu S X, Chang R Z, Qiu L J. Transforming transcription factor ABP9 into Soybean and optimization of the transformation system., 2008, 41: 1908–1916 (in Chinese with English abstract)
[32] Devi M K, Sakthivela G, Giridhar P. Protocol for augmented shoot organogenesis in selected variety of soybean., 2012, 50: 729–734
[33] 杨权, 王月月, 刘炎光, 蒋春志, 张孟臣, 张洪霞, 张洁, 王冬梅. 大豆子叶节遗传转化体系优化及抗逆基因的转化研究. 大豆科学, 2015, 34: 205–211 Yang Q, Wang Y Y, Liu Y G, Jiang C Z, Zhang M C, Zhang H X, Zhang J, Wang D M. Study on optimization of soybean cotyledonary node genetic transformation system and the transformation of resistance gene., 2015, 34: 205–211 (in Chinese with English abstract)
[34] 柯丹霞, 熊文真, 彭昆鹏, 李祥永. 抗盐基因大豆子叶节遗传转化研究. 信阳师范学院学报(自然科学版), 2017, 30(1): 46–51 Ke D X, Xiong W Z, Peng K P, Li X Y. Study on genetic transformation of salt resistant genein soybean.(Nat Sci Edn), 2017, 30(1): 46–51 (in Chinese with English abstract)
Cloning and Salt Resistance Function Identification ofGenefrom
KE Dan-Xia*, PENG Kun-Peng, ZHANG Meng-Ke, JIA Yan, and WANG Jing-Jing
College of Life Sciences / Institute for Conservation and Utilization of Agro-bioresources in Dabie Mountains, Xinyang Normal University, Xinyang 464000, Henan, China
TheHD-Zip I class transcription factor plays an important role in plant resistance to abiotic stresses. An HD-Zip I class gene(homeodomain-leucine zipper protein 57) was cloned from soybean in this study. Sequence analysis showed thatgene contained a 1038 bp ORF, encoding 345 amino acids, and featured with HD-Zip family proteins’ typical conserved domain.was expressed in different organs of soybean plants and the highest expression occurred in flowers. The effects of abiotic stresses (abscisic acid, NaCl, PEG, and cold) ongene expression in soybean seedling stage were analyzed by real-time quantitative PCR. The expression level ofgene was obviously increased under high salinity stress and less affected by ABA and drought stress but decreased by cold stress. The expression level ofgene in roots was significantly higher than that in stems and leaves before and after salt stress, and reached the peak at 48 h, then decreased slowly at 72 h and 96 h after salt stress. The overexpression vector ofwas constructed and transformed intostrain EHA105 to obtain the stable transgenicplants. After being treated with 200 mmol L–1NaCl for 14 d, the shoot height, root length, chlorophyll content, root activity as well as K+and Ca2+content increased significantly in transgenic plants compared with the wild type. The malondialdehyde content, relative membrane permeability and Na+content were obviously reduced in transgenic plants compared with the wild type. It is hypothesized that thegene participates in the abiotic stress response of soybean, and over-expression ofgene could enhance resistance to saline in.
;gene; abiotic stress; salt resistance
2017-11-03;
2018-06-12;
2018-06-30.
10.3724/SP.J.1006.2018.01347
柯丹霞, E-mail: kdx_029@163.com
本研究由国家自然科学基金项目(31400213), 河南省科技攻关计划项目(182102110448), 信阳师范学院青年骨干教师资助计划项目(2015), 信阳师范学院“南湖学者奖励计划”青年项目和信阳师范学院研究生科研创新基金资助。
This study was supported by the National Natural Science Foundation of China (31400213), the Science and Technology Research Projects of Henan Province (182102110448), the Funding Scheme for Young Core Teachers of Xinyang Normal University (2015), the Nanhu Scholars Program for Young Scholars of Xinyang Normal University, and the Scientific Research Foundation of Graduate School of Xinyang Normal University.
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20180629.1549.008.html
