神经氨酸酶抑制剂拉尼米韦抗流感病毒分子动力学模拟研究
2018-09-07潘大波姜德文张永豪吴明凯龙治峰
潘大波,姜德文,张永豪,吴明凯,龙治峰
(黔东南民族职业技术学院,贵州 凯里 556000)
0 引言
神经氨酸酶抑制剂是抗流感病毒一类重要的药物。目前神经氨酸酶抑制剂主要有拉尼米韦,拉尼米韦辛酸酯、奥司米韦、帕拉米韦和扎那米韦等。其中拉尼米韦辛酸酯是拉尼米韦的前药,在体内代谢成拉尼米韦而发挥抗流感活性[1-2]。研究显示拉尼米韦的抗病毒谱广,包括N1、N7等[3-4]。为了探讨拉尼米韦能否抵抗新型亚型流感病毒H17N10,我们采用分子模拟的方法考察拉尼米韦与各种亚型NA的结合特征来阐释它抗病毒的分子机制[5]。
1 资料与方法
1.1 实验材料
从PDB数据库中获得H1N1pdm和H7N9神经氨酸酶蛋白-LAN复合物(蛋白ID:3TI3和4MWU)。H17N10的神经氨酸酶拉尼米韦复合物由H17N10的神经氨酸酶(蛋白ID:4GDI)与H7N9神经氨酸酶蛋白-LAN复合物叠合获得。晶体中的Ca离子和水分子保留。删掉不合理的结构。
1.2 分子动力学模拟
分子动力学模拟在Amber软件中模拟完成。拉尼米韦分子参数用Amber的BCC方法拟合电荷,用GAFF方法获得其参数。蛋白运用14SB立场。体系加12 Å水盒子和抗离子。分子动力学模拟过程经历体系最小化、体系加热、平衡和生产相模拟四部分。每个体系模拟200 ns。
1.3 结合自由能计算与氨基酸残基能量分解
选取每条轨迹的250个结构运用MM-GBSA的方法计算神经氨酸酶与拉尼米韦之间的结合自由能。溶剂和溶质的介电常数分别为80和4。氨基酸残基能量分解的参数与结合自由能计算的参数一样。
2 结果
2.1 分子动力学模拟平衡考察
检测体系在200 ns模拟过程中配体周围8 Å以内的氨基酸碳α的RMSD随时间的变化,在整过模拟过程中,体系的RMSD趋于平衡确在1-1.5 Å之间波动,说明在整过模拟过程中,体系处于平衡的过程。然而,N10体系的波动比N1和N9的大,这可能是N10与拉尼米韦的结合不是特别稳定导致的。
2.2 拉尼米韦与神经氨酸酶的结合亲和力
运用MM-GBSA计算拉尼米韦与各种亚型神经氨酸酶的结合自由能如表1所示。拉尼米韦与H1N1pdm和H7N9的结合自由能(ΔGbind)分别为-36.96 kcal/mol和-33.07 kcal/mol,而拉尼米韦与H17N10的结合自由能为-18.48 kcal/mol。其中极性相互作用(ΔGpolar)和非极性相互作用(ΔGnonpolar)都是负值,均有利于拉尼米韦与神经氨酸酶的结合。
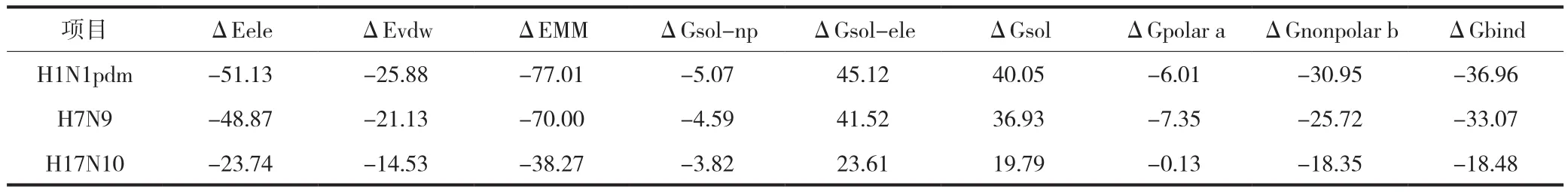
表1 拉尼米韦与神经氨酸酶的结合自由能(kcal/mol)
2.3 神经氨酸酶氨基酸对拉尼米韦的能量贡献
为了弄清H1N1pdm-LAN和H17N10与拉尼米韦的结合差异,运用MM-GBSA计算神经氨酸酶的氨基酸残基对拉尼米韦的能量贡献(图1)显示,在H1N1pdm-LAN体系中,对拉尼米韦的结合能量贡献大于1 kcal/mol的氨基酸为Arg292、Glu227、Glu119、Tyr406、Arg371、Arg152、Trp178、Asp151和Arg224。将H17N10的氨基酸能量贡献值减去H1N1pdm-LAN发现H17N10的氨基酸存在大于1 kcal/mol的氨基酸7个,这些氨基酸不利于H17N10与拉尼米韦的结合。

图1 (A)H1N1pdm-LAN体系NA中氨基酸残基能量贡献值
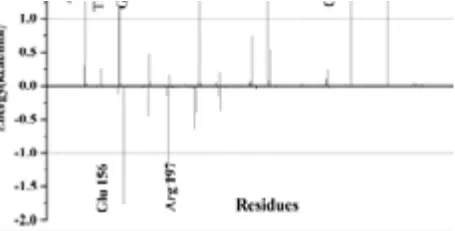
图1 (B)H17N10与H1N1pdm的氨基酸残基能量贡献值之差
3 讨论
由拉尼米韦与 H1N1pdm和H7N9和的结合自由能显示,拉尼米韦在抗H1N1pdm和H7N9的作用较好,这在前期很多的实验中得到验证。然而,对于新出现的H17N10,计算拉尼米韦与H17N10的神经氨酸酶的结合自由能比拉尼米韦与H1N1pdm或H7N9的结合亲和力弱约15 kcal/mol,这表明拉尼米韦抗H17N10的能力较弱[6-8]。
为什么会出现这样的情况,通过H1N1pdm和H17N10的神经氨酸酶序列比对发现,拉尼米韦与H1N1pdm结合的关键氨基酸在H17N10的序列不同,氨基酸性质各异导致与拉尼米韦的亲和力不一样,例如119为氨基酸,在H1N1pdm为Gln,能量贡献值为-2.24 kcal/mol,在H17N10为Arg,能量贡献值仅为-0.06 kcal/mol,氨基酸151位、152位、292位、371位和406位等均存在类似的不利于拉尼米韦与H17N10的神经氨酸酶结合的情况[9-10]。
通过运用分子动力学模拟的方法考察拉尼米韦与不同神经氨酸酶亚型的结合亲和力可以快速判断拉尼米韦对不同的神经氨酸酶亚型具有活性。同时通过能量分解可以阐释拉尼米韦与神经氨酸酶结合的详细作用机制。
