海岛棉CSSLs分子评价及纤维品质、产量性状QTL定位
2018-08-10李志坤谷淇深柯会锋吴立强王国宁吴金华张桂寅阎媛媛马峙英王省芬
李 超 李志坤 谷淇深 杨 君 柯会锋 吴立强 王国宁 张 艳 吴金华 张桂寅 阎媛媛 马峙英 王省芬,*
海岛棉CSSLs分子评价及纤维品质、产量性状QTL定位
李 超1,2李志坤1谷淇深1杨 君1柯会锋1吴立强1王国宁1张 艳1吴金华1张桂寅1阎媛媛1马峙英1王省芬1,*
1河北农业大学教育部华北作物种质资源研究与利用重点实验室, 河北保定 071001;2中国农业科学院棉花研究所 / 棉花生物学国家重点实验室, 河南安阳 455000
本课题组前期以陆地棉中棉所8号(CCRI8)为轮回亲本, 海岛棉Pima 90-53为供体亲本培育了一套陆地棉中棉所8号为背景的海岛棉染色体片段置换系(CSSLs), 本研究利用SSR标记对该置换系群体BC3F5进行基因型检测, 在3个不同环境下(河北保定、青县和新疆轮台)鉴定其纤维品质和产量相关性状并进行QTL定位。该置换系群体包含182个家系, 置换片段数在1~15个之间, 平均为6.6个; 导入片段长度在0.7~83.2 cM之间, 平均长度为16.8 cM; 置换片段总长度20 249.6 cM; 背景回复率在92.3%~99.6%之间, 平均为96.2%。共检测出59个相关的QTL, 其中与纤维品质性状相关的41个, 单个QTL的贡献率为1.27%~26.66%; 与产量性状相关的18个, 单个QTL的贡献率为2.03%~19.38%; 检测到14个稳定的QTL, 其中4个马克隆值和2个纤维伸长率相关的稳定QTL增效基因均来自高值亲本海岛棉Pima 90-53, 2个铃重相关的稳定QTL增效基因来自高值亲本陆地棉中棉所8号。研究结果为深入开展纤维品质和产量性状的QTL精细定位、QTL间互作和分子育种提供了理论依据。
陆地棉; 海岛棉; 染色体片段置换系; 纤维品质; 产量; QTL定位
棉花纤维品质和产量性状易受环境影响, 属于多基因控制的数量性状, 遗传机制比较复杂。通过常规育种方法改良纤维品质和产量性状已经不能满足现代农业生产的要求。随着分子生物学的快速发展, 将纤维品质和产量性状与分子标记结合起来统计分析, 在分子水平上探究多个环境下纤维品质和产量性状的遗传特性, 筛选出与纤维品质和产量性状紧密连锁且不受环境影响的标记, 从而用于分子标记辅助育种。
前人利用RIL、F2、F2:3等群体已经开展了很多与棉花纤维品质和产量性状相关的研究[1-8], 但是这些群体的遗传背景复杂, 严重制约了对目标性状QTL的精细解析。染色体片段置换系群体遗传背景只含有供体亲本的一个或几个染色体片段, 其他部分则与轮回亲本完全相同, 能够消除遗传背景的噪音, 是进行QTL定位研究的理想材料。朱亚娟等[9]以染色体片段置换系IL-15-5和IL-15-5-1构建的F2和F2:3分离群体为基础进行了衣分和子指QTL定位; Wang等[10]利用染色体片段置换系定位到43个加性QTL和6个上位性QTL; 付央等[11]培育了一套以TM-1为背景、含海岛棉3-79第18染色体不同长度片段的置换系, 定位到纤维品质和产量性状相关的7个加性QTL和5个上位性QTL; 沈超等[12]利用黄褐棉染色体片段导入系进行了产量和纤维品质性状QTL定位; 解信美[13]培育了一套以陆地棉遗传标准系TM-1为背景的阔叶棉染色体片段渐渗系; Guo等[14]利用海岛棉染色体片段置换系鉴定出3个纤维品质相关的QTL位于第1和第11染色体, 单个QTL贡献率为6.23%~10.73%; 何蕊等[15]利用陆地棉‘中棉所36’和海岛棉‘海1’构建的染色体片段置换系, 对BC5F3、BC5F3:4、BC5F3:5开展了关于株高的QTL定位研究, 单个QTL的贡献率为7.35%~13.17%。
本课题组前期培育了一套陆地棉‘中棉所8号’为背景的海岛棉染色体片段置换系(CSSLs)[16], 本研究将利用SSR标记对该置换系群体BC3F5进行基因型检测, 在3个生态环境差异较大的条件下鉴定其纤维品质、铃重和衣分, 并定位其QTL。研究结果对于深入开展QTL的精细定位、QTL间互作和分子育种具有重要意义。
1 材料与方法
1.1 试验材料
置换系群体为包含182个材料的BC3F5。轮回亲本为陆地棉品种中棉所8号(CCRI8), 非轮回亲本为海岛棉品种Pima 90-53。CCRI8产量高但纤维品质不如海岛棉, Pima 90-53为产量低但纤维品质优良。
1.2 田间种植和表型鉴定
2016年采用随机区组方式在生态环境差异很大的河北保定、青县和新疆轮台种植置换系群体。其中, 在河北保定种植点设置2次重复, 新疆轮台和河北青县种植点均设置3次重复, 每个材料1行, 行长7 m, 行距77 cm, 在青县和保定株距约35 cm, 新疆株距约10 cm。在成熟期分别收取亲本及BC3F5群体各株系吐絮正常的20个棉铃, 晾干后称重, 计算单铃重。轧花后称取皮棉重, 计算衣分(衣分=皮棉重/籽棉重×100%), 用USTER HVI 1000全自动纤维品质测定仪测定纤维品质。
1.3 棉花基因组DNA的提取
参考Paterson等[17]方法, 略作修改, 提取棉花叶片DNA。
1.4 SSR引物筛选及反应体系
王云鹏等[16]以陆地棉中棉所8号为轮回亲本, 海岛棉Pima 90-53为供体亲本培育了一套海岛棉染色体片段置换系。本研究在该图谱的基础上, 按照10~20 cM为标准挑选205个SSR标记, 包含221个多态性位点, 标记序列来自Cottongen (https://www. cottongen.org/)。SSR引物由苏州金唯智生物科技有限公司合成。
SSR扩增体系为20 μL, 含2×MasterMix II 10 μL, 上、下游引物(10 μmol L–1)各1 μL, DNA (50 ng μL–1)模板1 μL, ddH2O 7 μL。扩增程序为94℃预变性5 min; 94℃变性45 s, 55℃退火45 s, 72℃延伸60 s, 30个循环; 72℃延伸5 min, 4℃保存。PCR产物用QIAxcel DNA High Resolution Kit (1200)检测单株的基因型。
1.5 染色体片段置换系基因型检测
染色体片段置换系扩增产物带型如图1所示, 与CCRI8一致的记作“a”, 与Pima 90-53一致的记作“b”, 杂合带型记作“h”, 缺失的记录为“-”。
1.6 染色体片段置换系遗传组成分析
结合182个BC3F5株系基因型结果, 利用GGT 2.0软件分析株系基因型, 计算染色体片段置换系的背景回复率和导入片段长度。
置换系遗传背景回复率:() = [+()]/(2); 理论遗传背景回复率:[()] = 1–(1/2)+1。其中,()代表回交代后基因型为受体亲本的标记数量,代表标记总数。
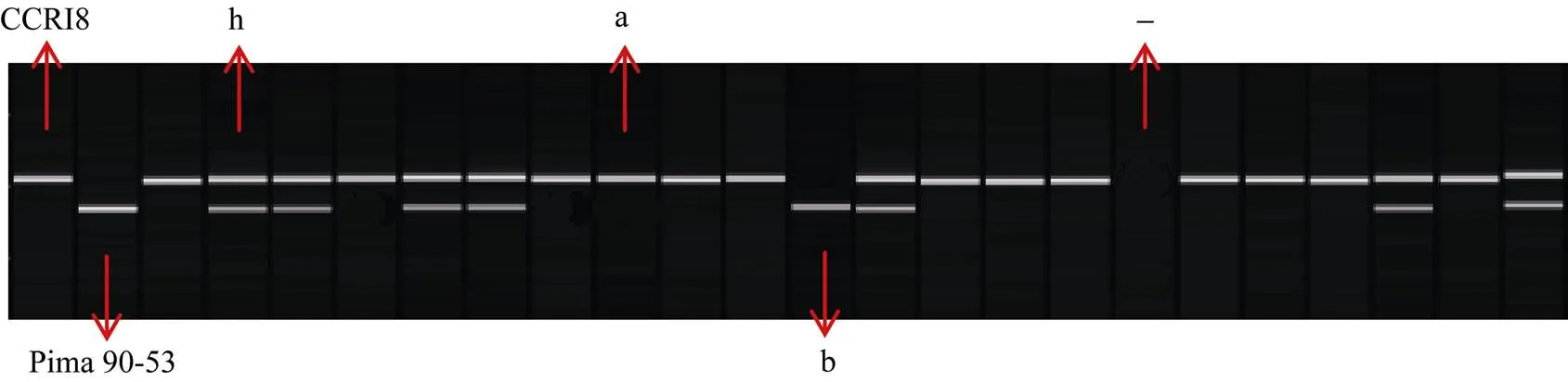
图1 SSR引物扩增型
a: CCRI8基因型; b: Pima 90-53基因型; h: 杂合带型; –: 缺失带型。
a: CCRI8 genotype; b: Pima 90-53 genotype; h: heterozygous genotype; –: missing genotype.
参照Young等[18]的方法分析导入片段长度,代表A、B位点之间的遗传距离,代表B、C位点之间的遗传距离,代表B位点导入片段长度(图2), 则= 1/2(+)。
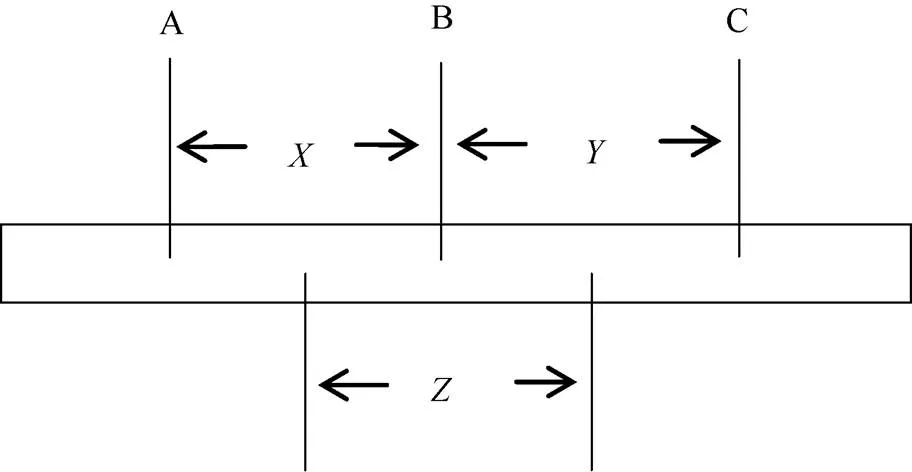
图2 导入片段长度分析示意图
1.7 数据统计分析方法和QTL定位
使用SPSS 19.0软件对置换系BC3F5群体的纤维品质和产量性状进行描述统计分析和相关性分析。利用QTL IciMapping V4.0软件, 结合完备区间作图法对置换系BC3F5群体纤维品质和产量性状进行QTL定位分析, 设置步长为1 cM, LOD大于3.0。QTL以“小写字母q-目标性状英文缩写名-染色体代号-QTL数目-环境代号”命名, 比如与纤维伸长率相关的QTL命名为、和, 其中, 1、2、3分别代表第1、第2、第3染色体, 4、5、6分别代表与纤维伸长率相关的QTL个数, 7、8、9分别代表保定、轮台、青县种植点。
2 结果与分析
2.1 染色体片段置换系基因型分析
利用作图软件GGT2.0对182个置换系做基因型分析, 结果显示, 染色体片段置换系大部分目标片段是纯合供体亲本片段, 杂合率为1.60%, 置换片段数量较少, 长度较短, 且相邻标记相互重叠(图3)。
2.1.1 置换片段数量分布 置换系置换片段总数量为1203个, 置换片段数最少为1个, 最多为15个, 平均为6.6个。置换片段数量最少的株系仅有1个, 占0.5%; 置换片段数量为2~4个的株系共41个, 占22.5%; 置换片段数量为5~7个的株系最多, 共82个, 占45.1%; 置换片段数量8个以上的株系共58个, 占31.9% (图4-A)。
2.1.2 置换片段长度分布 该置换系置换片段总长度为20 249.6 cM, 置换片段长度最长为83.2 cM, 最短为0.7 cM, 平均16.8 cM。单个置换片段长度小于15 cM的最多, 有697个, 占57.8%; 置换片段长度介于15.1~30.0 cM之间有384个, 占31.9%; 置换片段长度介于30.1~45.0 cM之间有79个, 占9.4%; 置换片段长度超过60.0 cM最少, 有11个, 占0.9% (图4-B)。
2.1.3 置换系背景回复率分析 该置换系的背景回复率最低为92.3%, 最高为99.6%, 平均96.2%。高于背景回复率平均值的株系有100个, 占整个群体的55%, 而低于理论背景回复率93.7%的株系很少, 有18个, 占整个群体的9.9% (图4-C)。
2.1.4 置换片段覆盖情况 从表1可知, 置换片段覆盖总长度为2842.1 cM, 用于构建置换系的染色体总长为2929.4 cM, 覆盖率为97.0%。但是, 仍存在个别染色体缺失海岛棉的置换片段, 其中, A4、A7、A12、D2、D7、D12未能完全覆盖, 覆盖率分别为80.3%、95.1%、88.9%、91.3%、91.3%和88.1%。A12缺失片段长度最长, 为19.1 cM, A7缺失片段长度最短, 为5.4 cM。
上述研究结果表明, 本研究所用的置换系其置换片段的含量在整个基因组中的比例已经很低, 适合进行重要性状的QTL定位研究。
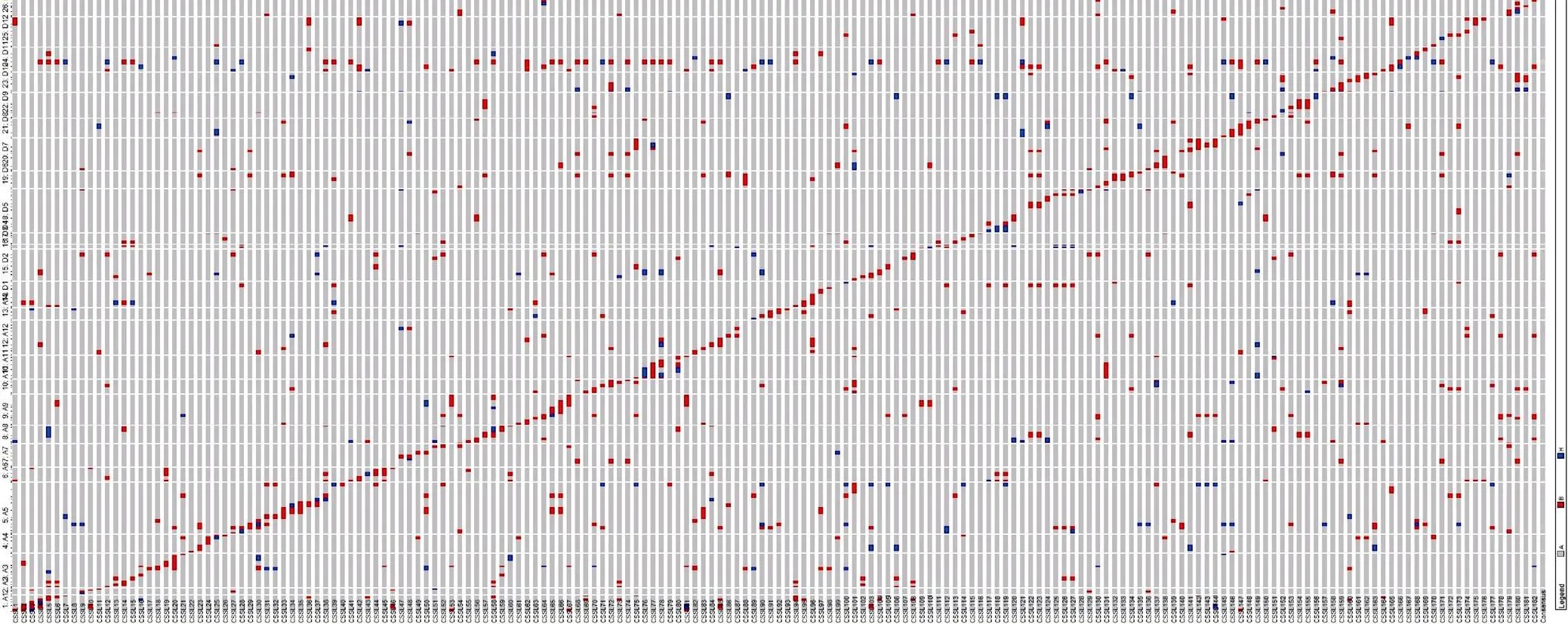
图3 染色体片段置换系基因型
左侧数字代表染色体编号(从下至上依次为A1、A2、……、A12、A13、D1、D2、……、D12、D13), 底部数字代表置换系编号(从左至右依次为CSSL1、CSSL2、……、CSSL181、CSSL182), 灰色、红色、蓝色分别代表受体亲本基因型、纯合供体亲本基因型和杂合基因型。
The left number represents the chromosome number (from the bottom to the top is A1, A2, ..., A12, A13, D1, D2, ..., D12, D13), the bottom number represents the number of CSSLs (from left to right is CSSL1, CSSL2, ..., CSSL181, CSSL182), gray, red, blue represent the genotypes of recipient parent, homozygous donor parent, and heterozygous individual, respectively.
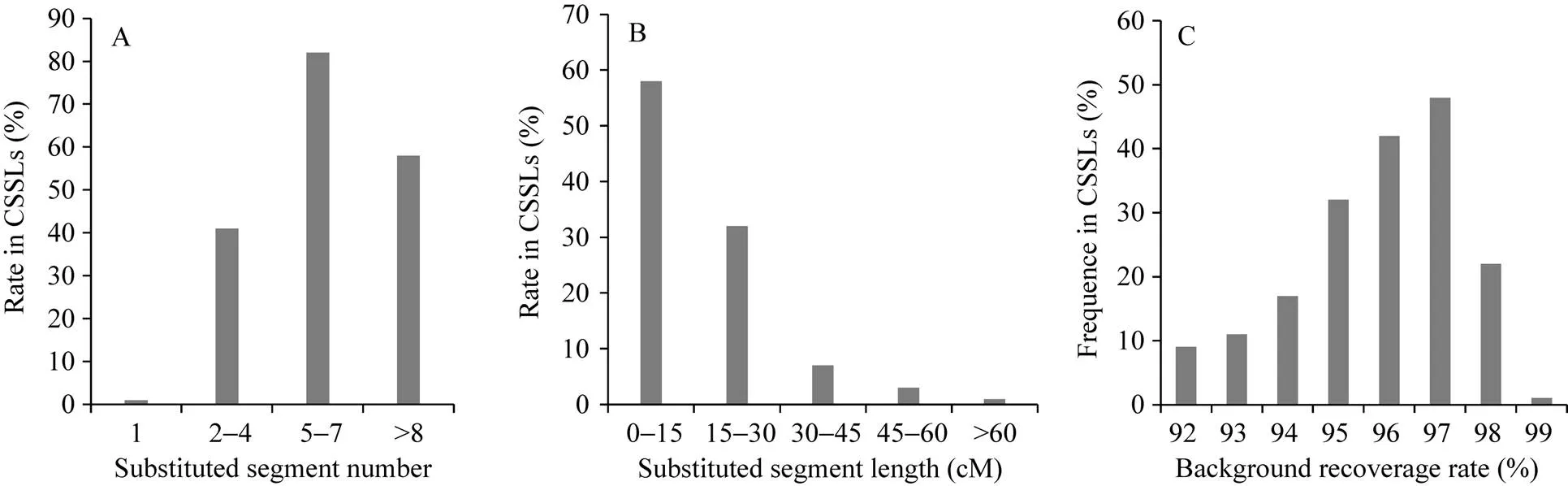
图4 染色体片段置换系置换片段分析
A、B、C分别表示置换片段数量、置换片段长度、背景回复率。
A, B, C represent substituted segment number, substituted segment length, and background recoverage rate, respectively.
2.2 染色体片段置换系表型性状分析
2.2.1 亲本纤维品质和产量性状统计分析 从表2可知, 陆地棉CCRI8的纤维品质各项指标均不如海岛棉Pima 90-53表现优异, 但CCRI8的衣分和铃重均优于Pima 90-53。
2.2.2 染色体片段置换系纤维品质和产量性状统计分析 从表3可以看出, 保定种植点的纤维强度、马克隆值、纤维整齐度, 青县种植点的纤维伸长率, 轮台种植点的马克隆值、纤维伸长率峰度均略大于1, 近似正态分布; 其他性状的峰度和偏度均小于1, 符合正态分布, 均适合进行QTL定位分析。
2.2.3 染色体片段置换系纤维品质和产量性状相关分析 用SPSS 19.0软件对染色体片段置换系的纤维品质和产量性状各项指标进行相关分析表明, 保定、轮台和青县种植点纤维长度与强度均呈极显著正相关, 整齐度与长度、强度均呈极显著正相关; 保定和轮台种植点纤维长度与伸长率均呈显著负相关, 保定和青县种植点纤维强度与马克隆值分别呈极显著和显著正相关; 轮台和青县种植点衣分与铃重均呈极显著正相关(表4)。说明性状间可能存在一因多效现象, QTL定位时可能会出现QTL区间包含多个性状的情况。
2.2.4 纤维品质和产量性状QTL定位 共定位到59个纤维品质和产量性状相关的QTL, 包括41个纤维品质相关的QTL和18个产量性状相关的QTL, 分布在20条染色体上, 贡献率为1.27%~26.66%。其中, 在第11染色体检测到8个相关的QTL, 第3、第15染色体均检测到6个相关的QTL, 第5、第23染色体均检测到5个相关的QTL, 第9染色体检测到4个相关的QTL, 第1、第14、第16染色体均检测到3个相关的QTL, 第4、第12、第21、第24染色体均检测到2个相关的QTL, 第2、第6、第10、第13、第17、第19、第22、第26染色体均检测到1个相关的QTL (表5)。从图5可以看出, 13个与马克隆值相关的QTL中, 有8个增效基因来源于高值亲本Pima 90-53; 11个与铃重相关的QTL中, 有8个增效基因来源于高值亲本CCRI8。在3个环境中共定位到14个纤维品质和产量性状相关的稳定QTL, 其中, 6个能在3个环境中重复检测到, 分别是马克隆值相关的QTL (、、)和纤维伸长率相关的QTL (、、); 有8个QTL能在2个环境中被检测到, 分别是4个马克隆值相关的QTL (、;、)、2个纤维伸长率相关的QTL (、)和2个铃重相关的QTL (、)。发现马克隆值相关的稳定QTL (、;、)和纤维伸长率相关的稳定QTL (、)增效基因均来自高值亲本海岛棉Pima 90-53, 铃重相关的稳定QTL (、)增效基因来自高值亲本陆地棉中棉所8号。
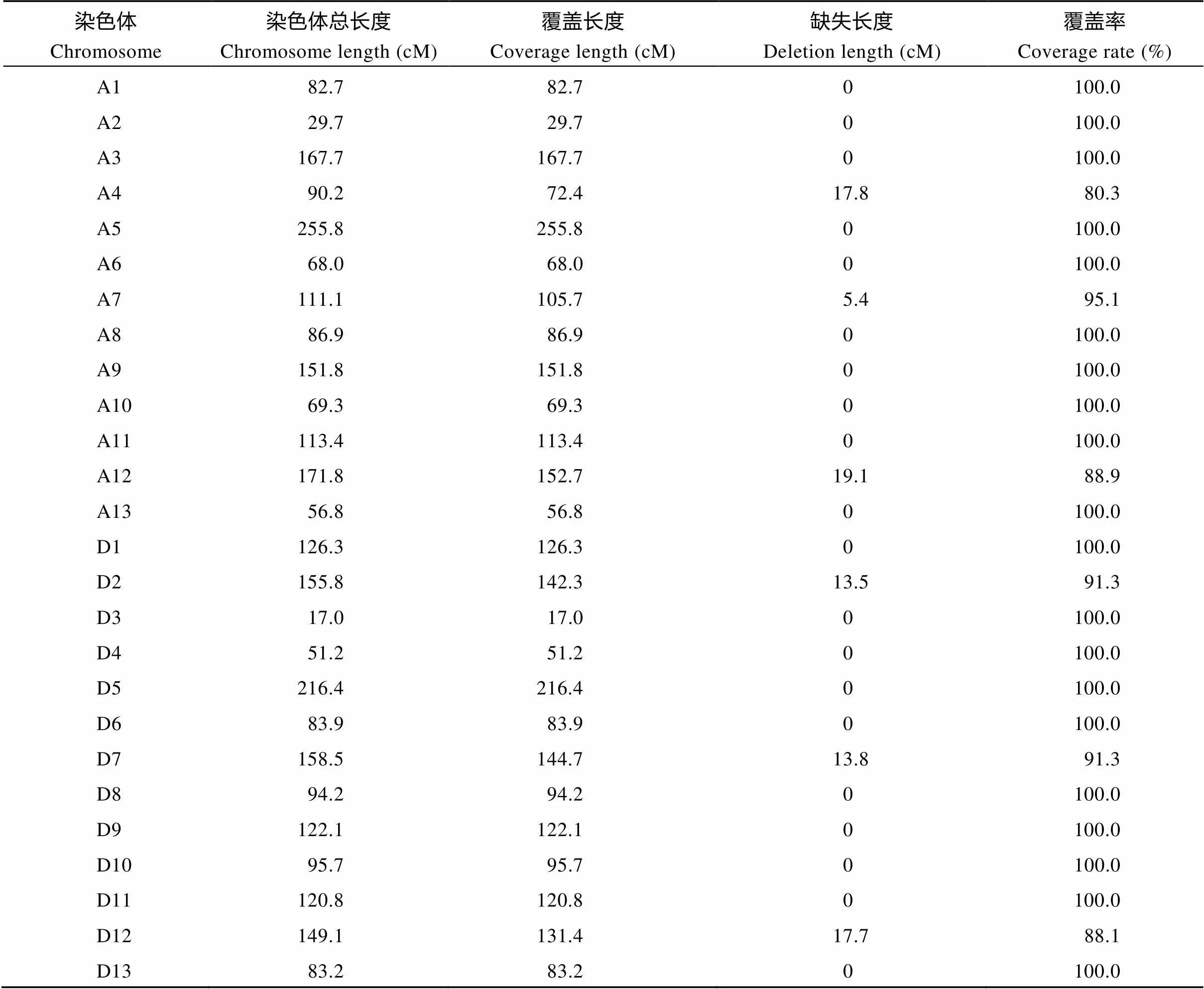
表1 染色体片段置换系置换片段覆盖情况
2.2.5 遗传图谱与物理图谱的共线性分析 以南京农业大学版本(AD1) genome NAU为参考序列, 利用MapChart 2.2软件对存在稳定QTL的染色体分析发现, 其遗传图谱与物理图谱存在共线性关系(图6)。A5 (c5)与V5之间存在明显的共线性排列模式; 而A3 (c3)与V3、A11 (c11)与V11、D7 (c16)与V7、D9 (c23)与V9之间标记存在明显的重排。标记共线性分析比较对提高基因克隆效率和棉花育种有重要意义。
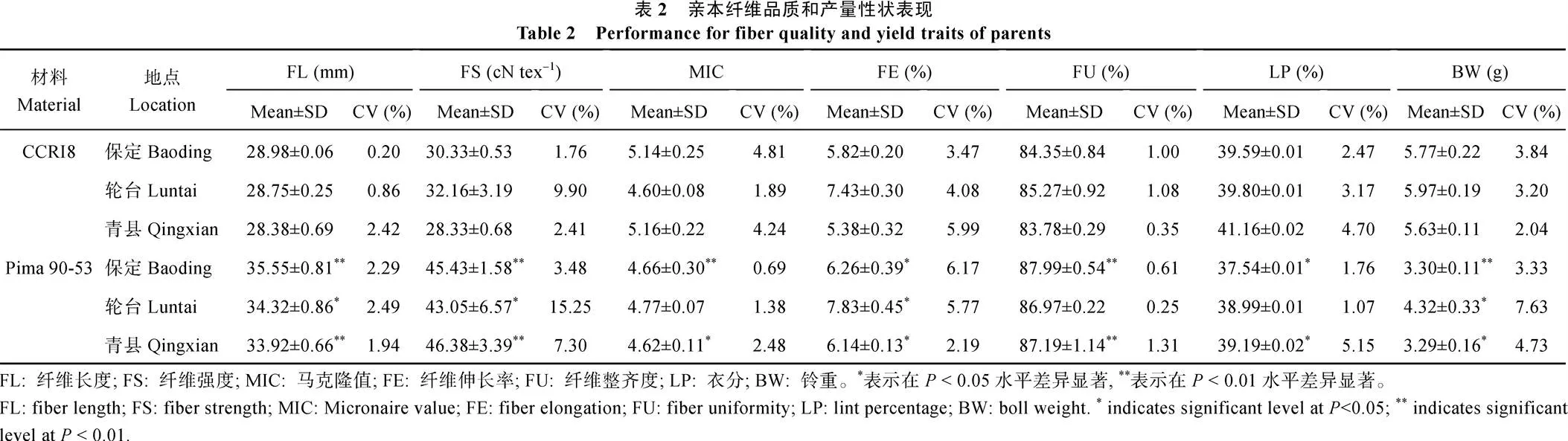
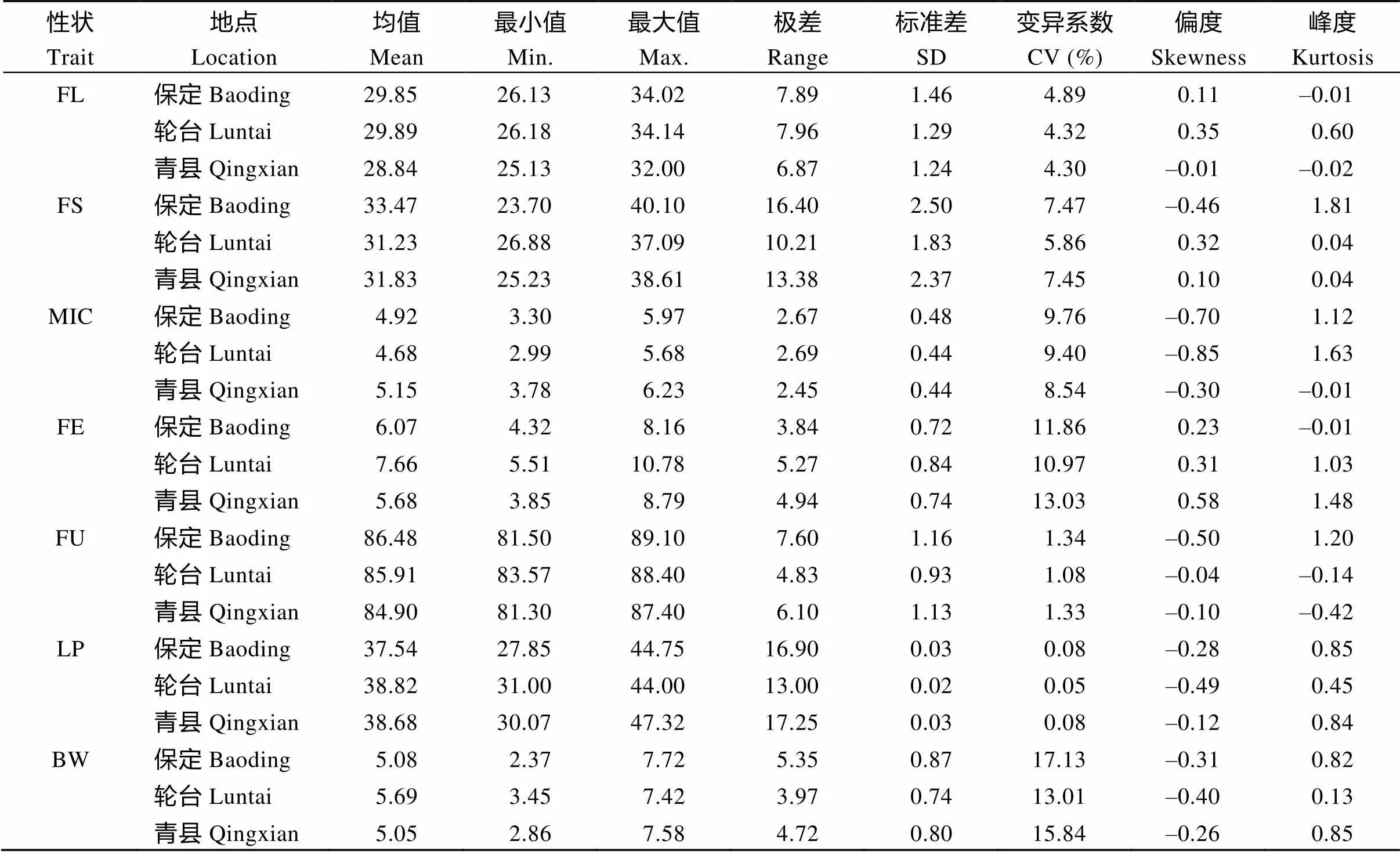
表3 染色体片段置换系纤维品质和产量性状统计分析
>FL: 纤维长度; FS: 纤维强度; MIC: 马克隆值; FE: 纤维伸长率; FU: 纤维整齐度; LP: 衣分; BW: 铃重。
FL: fiber length; FS: fiber strength; MIC: Micronaire value; FE: fiber elongation; FU: fiber uniformity; LP: lint percentage; BW: boll weight.
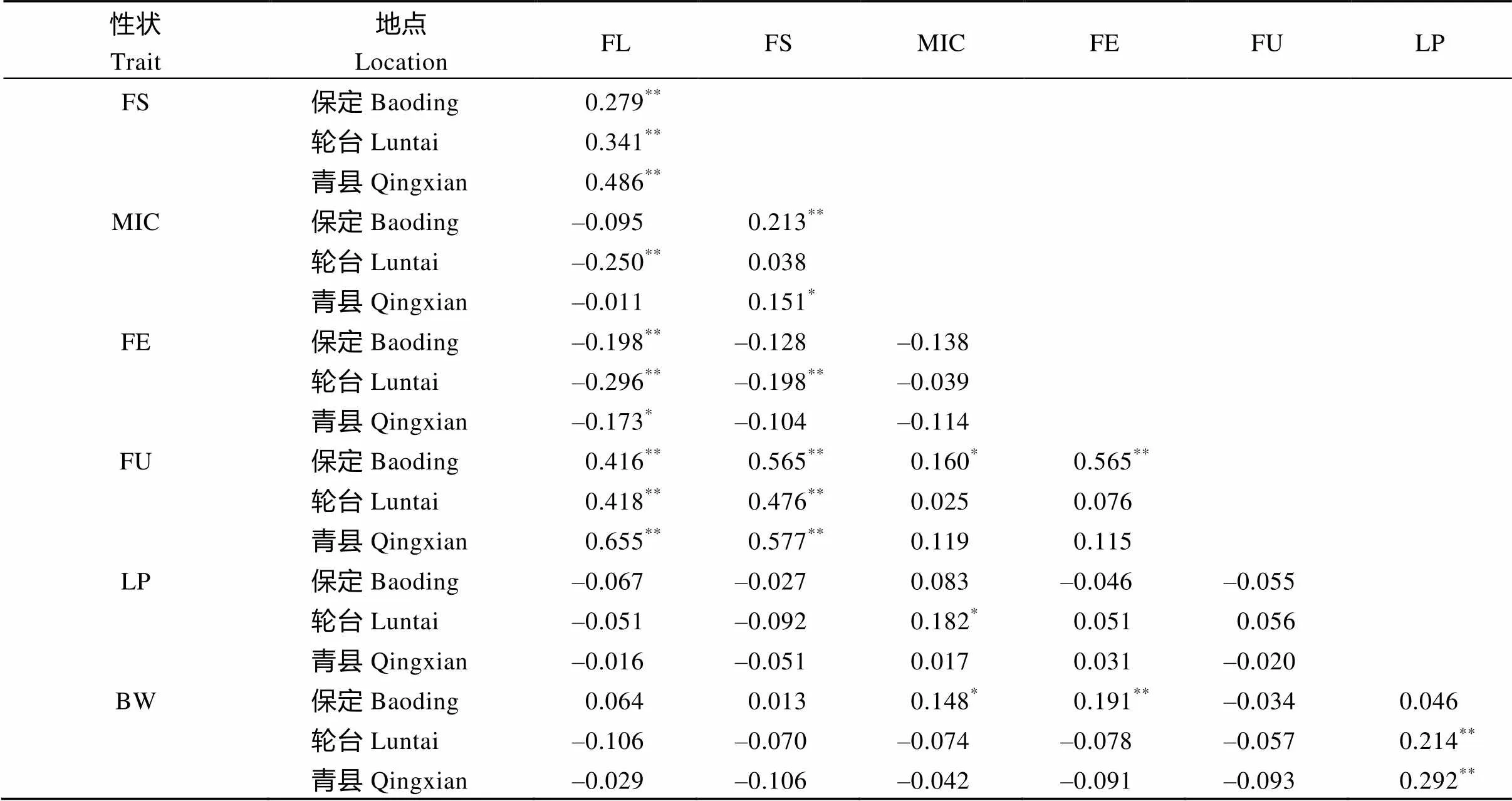
表4 染色体片段置换系纤维品质、产量性状间的相关系数
FL: 纤维长度; FS: 纤维强度; MIC: 马克隆值; FE: 纤维伸长率; FU: 纤维整齐度; LP: 衣分; BW: 铃重。*表示在< 0.05水平差异显著,**表示在< 0.01水平差异显著。
FL: fiber length; FS: fiber strength; MIC: Micronaire value; FE: fiber elongation; FU: fiber uniformity; LP: lint percentage; BW: boll weight.*indicates significant level at< 0.05;**indicates significant level at< 0.01.
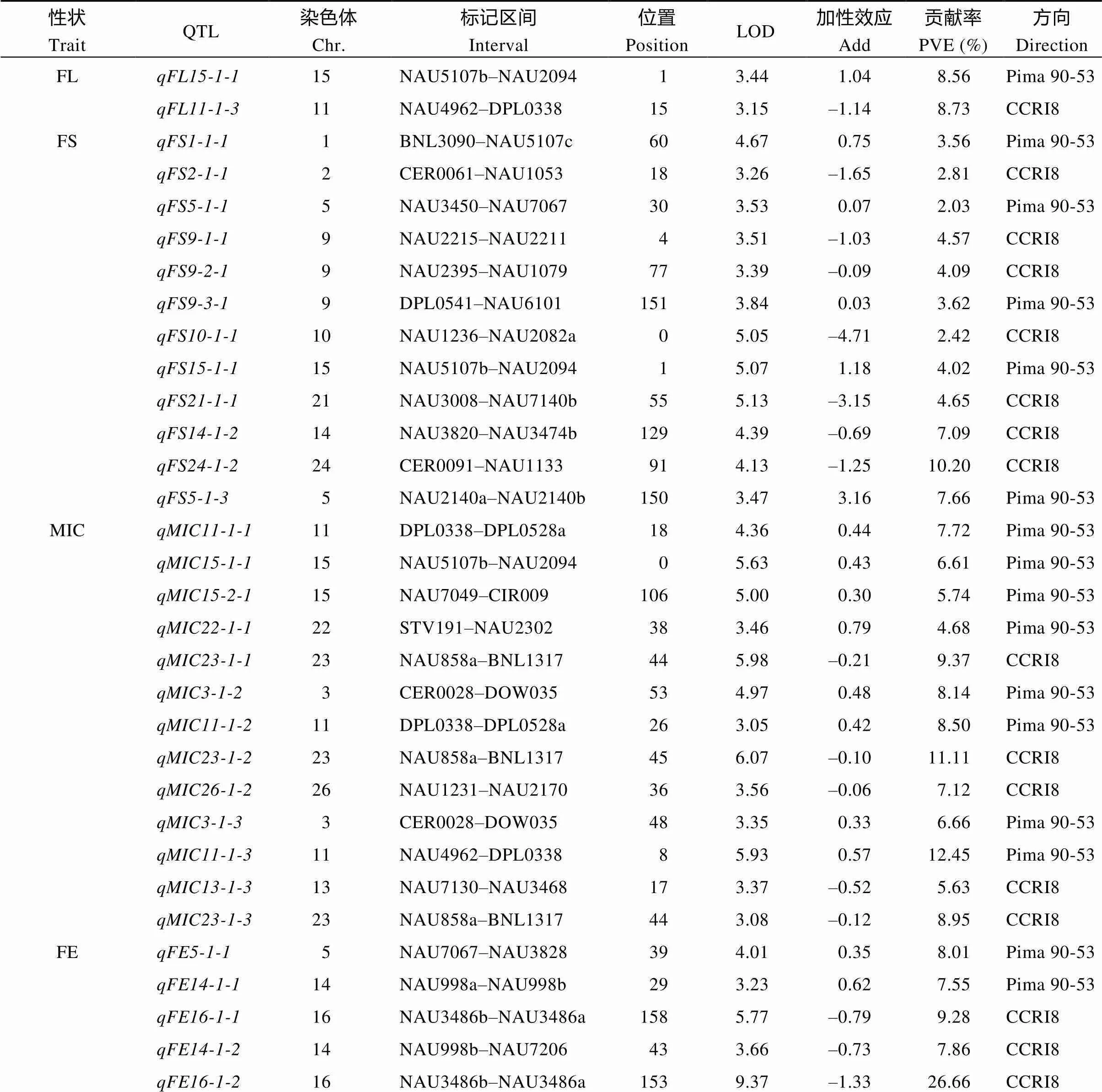
表5 染色体片段置换系纤维品质和产量性状相关QTL
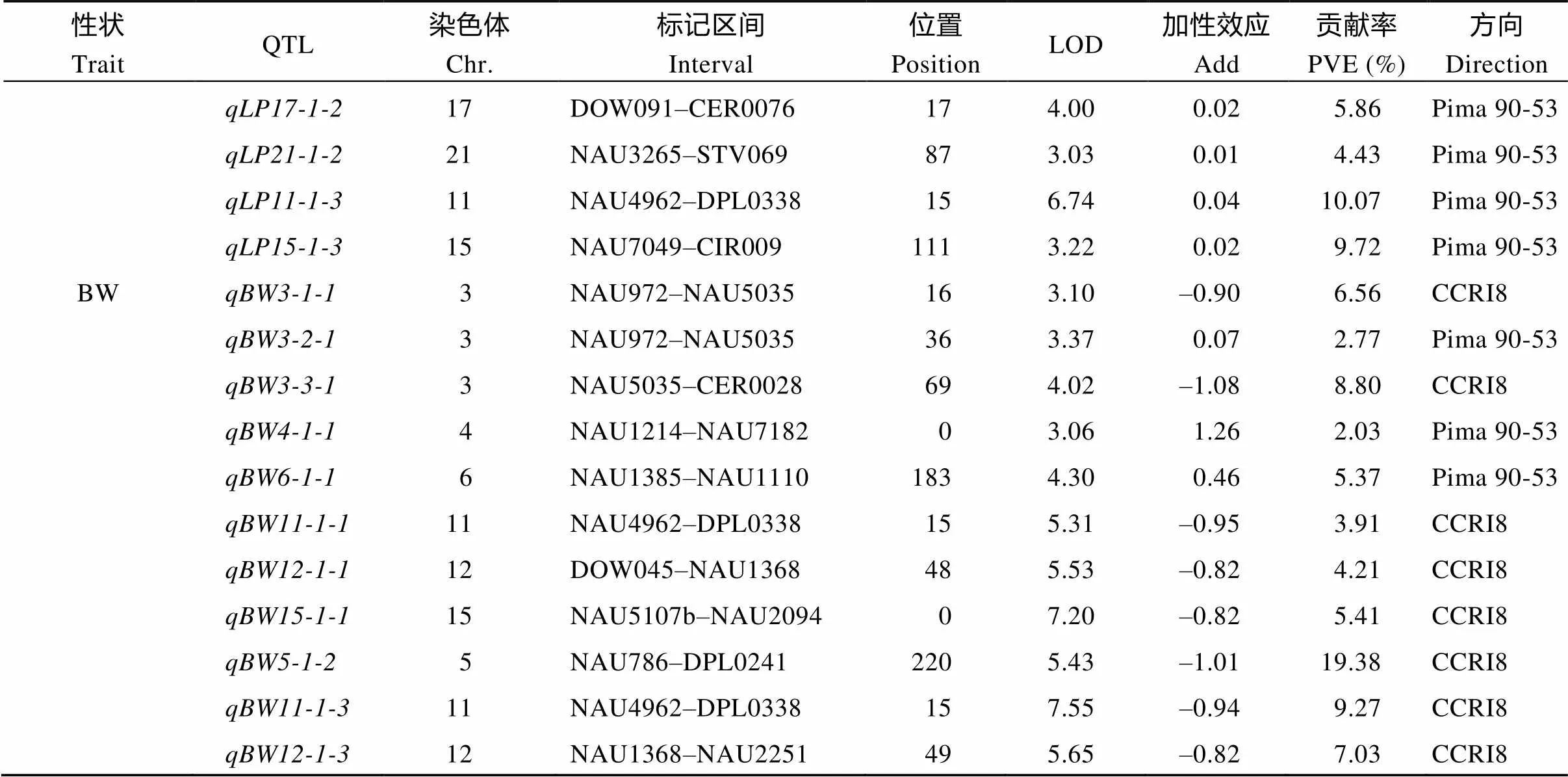
(续表5)
FL: 纤维长度; FS: 纤维强度; MIC: 马克隆值; FE: 纤维伸长率; FU: 纤维整齐度; LP: 衣分; BW: 铃重。
FL: fiber length; FS: fiber strength; MIC: Micronaire value; FE: fiber elongation; FU: fiber uniformity; LP: lint percentage; BW: boll weight.
3 讨论
染色体片段置换系只在个别区段与受体亲本有差异, 所有与受体亲本不同的性状都可能和这些区段有关, 本研究使用的染色体片段置换系的遗传背景与中棉所8号一致, 因此它与受体亲本之间的差异理论上来源于导入片段海岛棉Pima 90-53上的等位基因。在本研究中共检测出41个纤维品质相关QTL, 其中有21个QTL的增效基因来源于海岛棉Pima 90-53, 占总体比例的51.21%; 共检测出18个产量性状相关的QTL, 其中9个QTL的增效基因来源于陆地棉CCRI8, 这与前人研究结果较一致[19-20], 即不论是纤维品质性状还是产量性状, 高值亲本和低值亲本中都存在增效基因, QTL的增效基因既可能来源于高值亲本, 也可能来源于低值亲本。QTL是数量性状位点, 除了受本身基因控制以外, 也受试验环境的影响[21]。虽然在棉花纤维品质和产量性状方面进行了很多QTL定位研究, 但是真正能够用于标记辅助选择的却很少, 大多数还无法有效应用, 因此还需要继续发掘在不同群体、不同环境下都能稳定检测到的QTL。本研究在3个环境中共定位到14个纤维品质和产量性状相关的稳定的QTL, 其中6个能在3个环境中被重复检测到, 8个能在2个环境中被检测到, 说明有的QTL相对比较稳定, 受外界环境影响较小; 而有的QTL在不同环境中检测到的位置不稳定, 受环境影响较大。QTL成簇分布现象是指同一条染色体上有多个不同性状的QTL, 并且这些性状与相同的标记连锁或者分布在相同的标记区间。在棉花以及其他作物的研究中[22-25]都已发现控制不同性状的QTL集中连续分布的现象, 并且认为这可能与多基因连锁或者一因多效有密切关系。本研究中也发现20个QTL成簇分布。其中, 位于第11染色体的有7个QTL, 位于第15染色体的有6个QTL。这些成簇分布的QTL对于改良棉花纤维品质和产量具有重要意义。
关于棉花纤维品质和产量性状相关的QTL定位在国内外已有很多报道[26-29]。但是, 在前人研究中难以判断是否存在与本研究结果一致的QTL, 目前发现一个纤维强度相关的QTL ()与本课题组杨鑫雷等[30]利用的F2:3群体获得的结果一致, 均位于第9染色体–区间, 推断可能是同一个QTL。一致性QTL较少的主要原因可能: 一是分子标记种类较多, 不同研究机构选用的标记种类不同; 二是不同研究者选用的亲本不同; 三是遗传图谱构建工作量较大, 高密度遗传图谱较少。本研究使用的海岛棉染色体片段置换系群体是一个永久性分离群体, 可以提供大量的种子进行多年多点试验。根据初步定位结果, 将QTL-CSSLs与轮回亲本杂交衍生出F2或F2:3群体, 可用于目标性状的精细定位[31]。
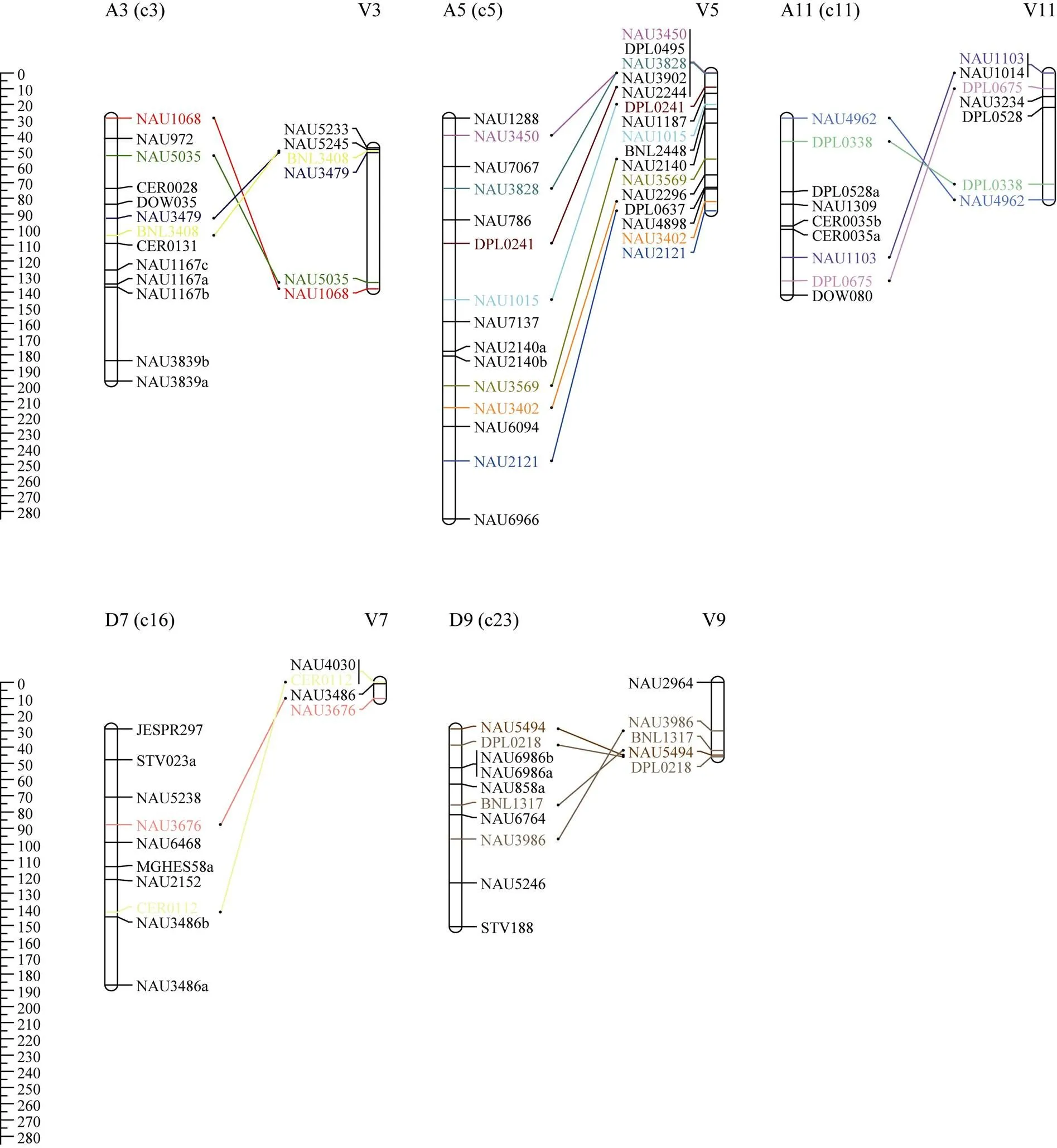
A3 (C3)、A5 (C5)、A11 (C11)、D7 (C7)、D9 (C9)为遗传图谱, V3、V5、V11、V7、V9为物理图谱。
A3 (C3), A5 (C5), A11 (C11), D7 (C7), and D9 (C9) are genetic map, and V3, V5, V11, V7, and V9 are physical map.
4 结论
该染色体片段置换系群体包含182个家系, 置换片段数在1~15个之间, 平均为6.6个; 导入片段长度在0.7~83.2 cM之间, 平均长度为16.8 cM; 置换片段总长度为20 249.6 cM; 背景回复率在92.3%~99.6%之间, 平均为96.2%。共检测到59个纤维品质和产量性状相关的QTL, 其中与纤维品质相关的41个, 单个QTL贡献率为1.27%~26.66%, 增效基因来源于海岛棉Pima 90-53的21个; 与产量性状相关的18个, 单个QTL贡献率为2.03%~19.38%, 增效基因来源于陆地棉中棉所8号的9个。检测到14个马克隆值、纤维伸长率、铃重相关的稳定QTL, 均可在多个环境中被检测到, 其中, 马克隆值相关的稳定QTL (、;、)和纤维伸长率相关的稳定QTL (、)增效基因均来自高值亲本海岛棉Pima 90-53, 铃重相关的稳定QTL (、)增效基因来自高值亲本陆地棉中棉所8号。
[1] Shen X L, Guo W Z, Lu Q X, Zhu X F, Yuan Y L, Zhang T Z. Genetic mapping of quantitative trait loci for fiber quality and yield trait by RIL approach in upland cotton., 2007, 155: 371–380
[2] 林忠旭, 冯常辉, 郭小平, 张献龙. 陆地棉产量、纤维品质相关性状主效QTL和上位性互作分析. 中国农业科学, 2009, 42: 3036–3047 Lin Z X, Feng C H, Guo X P, Zhang X L. Genetic analysis of major QTLs and epistasis interaction for yield and fiber quality in upland cotton., 2009, 42: 3036–3047 (in Chinese with English abstract)
[3] Tan Z Y, Fang X M, Tang S Y, Zhang J, Liu D J, Teng Z H, Li L, Ni H J, Zheng F M, Liu D X, Zhang T F, Paterson A H, Zhang Z S. Genetic map and QTL controlling fiber quality traits in upland cotton (L.)., 2015, 203: 615–628
[4] Zhi Y N, Chen H, Mei H X, Zhang T Z. Molecular tagging of QTLs for fiber quality and yield in the upland cotton cultivar Acala-Prema., 2014, 195: 143–156
[5] 李朋波, 曹美莲, 刘惠民, 杨六六, 陈耕. 陆地棉遗传图谱构建与纤维品质性状QTL定位. 西北植物学报,2006, 26: 1098–1104 Li P B, Cao M L, Liu H M, Yang L L, Chen G. Genetic map construction and QTL mapping of fibre quality in upland cotton (L.)., 2006, 26: 1098–1104
[6] 吴茂清, 张献龙, 聂以春, 贺道华. 四倍体栽培棉种产量和纤维品质性状的QTL定位. 遗传学报, 2003, 30: 443–452 Wu M Q, Zhang X L, Nie Y C, He D H. Localization of QTLs for yield and fiber quality traits of tetraploid cotton cultivar., 2003, 30: 443–452 (in Chinese with English abstract)
[7] Li C, Dong Y T, Zhao T L, Li L, Li C, Yu E, Mei L, Daud M K, He Q L, Chen J H, Zhu S J. Genome-wide SNP linkage mapping and QTL analysis for fiber quality and yield traits in the upland cotton recombinant inbred lines population., 2016, 7: 1356
[8] Wang H T, Huang C, Zhao W X, Dai B S, Shen C, Zhang B B, Li D G, Lin Z X. Identification of QTL for fiber quality and yield traits using two immortalized backcross populations in upland cotton., 2016, 11: e0166970
[9] 朱亚娟, 王鹏, 郭旺珍, 张天真. 利用海岛棉染色体片段导入系定位衣分和籽指QTL. 作物学报, 2010, 36: 1318–1323 Zhu Y J, Wang P, Guo W Z, Zhang T Z. Mapping QTLs for lint percentage and seed index usingchromosome segment introgression lines., 2010, 36: 1318–1323 (in Chinese with English abstract)
[10] Wang P, Zhu Y J, Song X L, Cao Z B, Ding Y Z, Liu B L, Zhu X F, Wang S, Guo W Z, Zhang T Z. Inheritance of long staple fiber quality traits ofinbackground using CSILs., 2012, 124: 1415–1428
[11] 付央, 苑冬冬, 胡文静, 蔡彩平, 郭旺珍. 陆地棉背景下海岛棉第18染色体片段置换系的培育及相关农艺性状QTL定位. 作物学报, 2013, 39: 21–28 Fu Y, Yuan D D, Hu W J, Cai C P, Guo W Z. Development ofchromosome 18 segment substitution lines in the genetic standard line TM-1 ofand mapping of QTLs related to agronomic traits., 2013, 39: 21–28 (in Chinese with English abstract)
[12] 沈超, 李定国, 聂以春, 林忠旭. 利用黄褐棉染色体片段导入系定位产量和纤维品质性状QTL. 作物学报, 2017, 43: 1733–1745 Shen C, Li D G, Nie Y C, Lin Z X, QTL mapping for yield and fiber quality traits usingchromosome segment introgression lines., 2017, 43: 1733–1745 (in Chinese with English abstract)
[13] 解信美. 陆地棉遗传标准系TM-1背景的阔叶棉TX-48染色体片段渐渗系的培育. 南京农业大学硕士学位论文, 江苏南京, 2013 Xie X M. Development of Chromosome Segment Introgression Lines fromacc. TX-48 in Genetic Standard Line,cv. TM-l.MS Thesis of Nanjing Agricultural University, Nanjing, Jiangsu, China, 2013 (in Chinese with English abstract)
[14] Guo Y P, Guo X, Wang F, Wei Z, Zhang S Q, Wang L Y, Yuan Y C, Zeng W G, Zhang G H, Zhang T Z, Song X L, Sun X Z. Molecular tagging andmarker-assisted selection of fiber quality traits using chromosome segment introgression lines (CSILs) in cotton., 2014, 200: 239–250
[15] 何蕊, 石玉真, 张金凤, 梁燕, 张保才, 李俊文, 王涛, 龚举武, 刘爱英, 商海红, 巩万奎, 白志川, 袁有禄. 利用染色体片段代换系定位陆地棉株高QTL. 作物学报, 2014, 40: 457–465 He R, Shi Y Z, Zhang J F, Liang Y, Zhang B C, Li J W, Wang T, Gong J W, Liu A Y, Shang H H, Gong W K, Bai Z C, Yuan Y L. QTL mapping for plant height using chromosome segment substitution lines in upland cotton., 2014, 40: 457–465 (in Chinese with English abstract)
[16] 王云鹏, 王省芬, 李志坤, 杨鑫雷, 张艳, 吴立强, 吴金华, 张桂寅, 马峙英. 陆地棉背景的Pima棉染色体片段置换系创制. 植物遗传资源学报, 2016, 17: 114–119 Wang Y P, Wang X F, Li Z K, Yang X L, Zhang Y, Wu L Q, Wu J H, Zhang G Y, Ma Z Y. Development of Pima cotton chromosome segment substitution lines withbackground.,2016, 17: 114–119 (in Chinese with English abstract)
[17] Paterson A H, Brubaker C L, Wendel J F. A rapid method for extraction of cotton (spp.) genomic DNA suitable for RFLP or PCR analysis., 1993, 11: 122–127
[18] Young N D, Tanksley S D. Restriction fragment length polymorphism maps and the concept of graphical genotypes.1989, 77: 95–101
[19] Wang B H, Guo W Z, Zhu X F, Wu Y T, Huang N T, Zhang T Z. QTL mapping of fiber quality in an elite hybrid derived-RIL population of upland cotton., 2006, 152: 367–378
[20] Zhang Z S, Hu M C, Zhang J, Liu D J, Zheng J, Zhang K, Wang W, Wan Q. Construction of a comprehensive PCR-based marker linkage map and QTL mapping for fiber quality traits in upland cotton (L.)., 2009, 24: 49–61
[21] 王琳, 刘方, 黎绍惠, 王春英, 张香娣, 王玉红, 华金平, 王坤波. 鲁棉研15号纤维品质性状QTL定位研究. 棉花学报, 2012, 24: 97–105 Wang L, Liu F, Li S H, Wang C Y, Zhang X D, Wang Y H, Hua J P, Wang K B. QTL mapping for fiber quality properties in lumianyan 15.2012, 24: 97–105 (in Chinese with English abstract)
[22] 杨晓军, 谢传晓, 李新海, 张世煌. 低氮逆境下玉米产量及相关性状QTL整合与一致性分析. 玉米科学, 2010, 18(4): 32–39 Yang X J, Xie C X, Li X H, Zhang S H. Analysis of consensus QTL for grain yield and other traits under low nitrogen conditions on maize.2010, 18(4): 32–39 (in Chinese with English abstract)
[23] Causse M, Salibacolombani V, Lecomte L, Duffé P, Rousselle P, Buret M. QTL analysis of fruit quality in fresh market tomato: a few chromosome regions control the variation of sensory and instrumental traits., 2002, 53: 2089–2098
[24] Moncada P, Martinez C P,Borrero J,Chatel M,Gauch H Jr,Guimaraes E,Tohme J,McCouch S R. Quantitative trait loci for yield and yield components in an×BC2F2population evaluated in an upland environment., 2001, 102: 41–52
[25] 胡文静, 张晓阳, 张天真, 郭旺珍. 陆地棉优质纤维QTL的分子标记筛选及优质来源分析. 作物学报, 2008, 34: 578–586 Hu W J, Zhang X Y, Zhang T Z, Guo W Z. Molecular tagging and source analysis of QTL for elite fiber quality in upland cotton., 2008, 34: 578–586 (in Chinese with English abstract)
[26] Gore M A, Fang D D, Poland J A. Linkage map construction and quantitative trait locus analysis of agronomic and fiber quality traits in cotton., 2014, 7: 1–10
[27] Zhang K, Zhang J, Ma J, Tang S Y, Liu D J, Teng Z H, Liu D X, Zhang Z S. Genetic mapping and quantitative trait locus analysis of fiber quality traits using a three-parent composite population in upland cotton (L.).2012, 29: 335–348
[28] Akash M W. Quantitative trait loci mapping for agronomic and fiber quality traits in upland cotton (L.) using molecular markers. PhD Dissertation of Louisiana State University, Louisiana, USA, 2003. https://digitalcommons.lsu. edu/gradschool_dissertations/1356
[29] Zhang T Z, Qian N, Zhu X F, Chen H, Wang S, Mei H X, Zhang Y M. Variations and transmission of QTL alleles for yield and fiber qualities in upland cotton cultivars developed in china., 2013, 8: e57220
[30] 杨鑫雷, 王志伟, 张桂寅, 潘玉欣, 吴立强, 李志坤, 王省芬, 马峙英. 棉花分子遗传图谱构建和纤维品质性状QTL分析. 作物学报, 2009, 35: 2159–2166 Yang X L, Wang Z W, Zhang G Y, Pan Y X, Wu L Q, Li Z K, Wang X F, Ma Z Y. Construction of molecular genetic map and QTL analysis of fiber quality in cotton., 2009, 35: 2159–2166 (in Chinese with English abstract)
[31] Zhou L J, Chen L M, Jiang L, Zhang W W, Liu L L, Liu X, Zhao Z G, Liu S J, Zhang L J, Wang J K, Wan J M. Fine mapping of the grain chalkiness QTLin rice (L.)., 2009, 118: 581–590
Molecular Evaluation for Chromosome Segment Substitution Lines ofand QTL Mapping for Fiber Quality and Yield
LI Chao1,2, LI Zhi-Kun1, GU Qi-Shen1, YANG Jun1, KE Hui-Feng1, WU Li-Qiang1, WANG Guo-Ning1, ZHANG Yan1, WU Jin-Hua1, ZHANG Gui-Yin1, YAN Yuan-Yuan1, MA Zhi-Ying1, and WANG Xing-Fen1,*
1North China Key Laboratory for Crop Germplasm Resources of Education Ministry, Hebei Agricultural University, Baoding 071001, Hebei, China;2Institute of Cotton Research, Chinese Academy of Agricultural Sciences / State Key Laboratory of Cotton Biology, Anyang 455000, Henan, China
In the previous study, we developed a set of chromosome segment substitution lines (CSSLs) usingCCRI8 as the recipient parent ande Pima 90-53 as the donor parent. In this study, we genotyped the BC3F5generation of CSSLs with SSR markers, conducted QTL mapping for the fiber quality and yield traits and identified the stable QTLs in three different environments (Baoding, Qingxian, Luntai). The substituted segment number of the 182 CSSLs varied from one to fifteen, averaged 6.6. The length of introgressed segments ranged from 0.7 cM to 83.2 cM, and averaged 16.8 cM. The total length of the substituted fragment was 20 249.6 cM, background recoverage rate varied from 92.3% to 99.6%, and the average background recoverage rate was 96.2%. Fifty-nine QTLs related to fiber quality and yield traits were detected. Among them, 41 QTLs were related to fiber quality traits and each QTL explained 1.27% to 26.66% of the phenotypic variation. Eighteen QTLs for fiber yield-traits including boll weight and lint percentage were detected and each QTL explained 2.03% to 19.38% of the phenotypic variation. Fourteen stable QTLs were detected in multiple environments. Among them, four QTLs related to micronaire value and two QTLs related to fiber elongation both had enhancing alleles fromPima 90-53. Two boll weight QTLs had enhancing alleles fromCCRI8. The results provide a theoretical basis for QTL fine mapping, QTL interaction and molecular breeding for fiber quality and yield traits.
upland cotton; sea island cotton; CSSLs; fiber quality; yield; QTL mapping
2017-11-06;
10.3724/SP.J.1006.2018.01114
王省芬, E-mail: cotton@hebau.edu.cn
李超, E-mail: leechao816@163.com
2018-06-12;
2018-06-20.
本研究由国家重点研发计划项目(2016YFD0101006)和河北省科技支撑计划项目(16226307D)资助。
The study was supported by the National Key Research and Development Program of China (2016YFD0101006) and Hebei Science and Technology Support Program (16226307D).
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20180620.0000.000.html
