定点突变提高土曲霉Aspergillus terreus脂肪酶的催化活性
2018-07-30张晓凤喻晓蔚徐岩
张晓凤,喻晓蔚,徐岩
定点突变提高土曲霉Aspergillus terreus脂肪酶的催化活性
张晓凤,喻晓蔚,徐岩
江南大学 生物工程学院 工业生物技术教育部重点实验室,江苏 无锡 214122

金城. 2018酶工程专刊序言. 生物工程学报, 2018, 34(7): 1021−1023.Jin C. Preface for special issue on enzyme engineering (2018). Chin J Biotech, 2018, 34(7): 1021−1023.
本研究旨在利用理性设计的方法来提高来源于土曲霉的酸性脂肪酶ATL的催化活力。通过同源比对,选择脂肪酶盖子区域和底物结合口袋域中的位点进行定点突变,得到8种ATL的突变脂肪酶。结果发现,盖子区域突变酶ATLLid与底物结合口袋域突变酶ATLV218W的催化活性显著提高。ATLLid和ATLV218W对底物对硝基苯酚月桂酸酯-nitrophenyl laurate (-NPL) 的催化活性最高,cat值较ATL分别提高了39.37倍和50.79倍,cat/m值较ATL分别提高了2.85倍和8.48倍。与ATL相比,ATLLid和ATLV218W的热稳定性略有下降,最适pH为5.0,pH 4.0–8.0具有较好的稳定性,说明突变未对ATL的嗜酸耐酸特性产生影响。通过同源建模模拟及分子对接技术分析底物-NPL与酶分子间的相互作用,解析了ATLLid和ATLV218W催化活性提高的机理。
土曲霉脂肪酶,毕赤酵母,同源建模,定点突变,催化活性,分子对接
脂肪酶 (EC 3.1.1.3) 又称甘油三酯水解酶,广泛分布于自然界中,是一类能在油水界面或水不溶系统进行酯类水解或酯类合成的酶[1]。脂肪酶在食品、化工、医药、饲料等领域得到了广泛的应用,因而对脂肪酶的催化活性提出了更高的要求。多数脂肪酶的最适作用pH在中偏碱性条件下,然而,研究人员发现在酸性条件下具有较高催化活性的酸性脂肪酶具有重要的工业应用价值。例如,在医药行业中,酸性脂肪酶可用于治疗胃肠紊乱、消化不良等疾病[2];在食品行业中,酸性脂肪酶可在适当条件下产生低级脂肪酸等风味物质,增强食品香味[3];在饲料行业中,在幼畜幼禽的饲料中添加脂肪酶可以有效解决幼畜因消化机能尚未发育健全、内源性脂肪酶分泌量不足而导致的早期断奶产生的明显应激,避免对消化系统发育和消化酶分泌产生不良影响,并能提高饲料内脂肪类物质的利用率[4]。
曲霉属脂肪酶在酸性环境中表现出良好的催化活性和耐受性,是获取高性能酸性脂肪酶的重要来源之一[5-8]。其中对于黑曲霉来源的酸性脂肪酶研究更为广泛,如产自黑曲霉AN0512的脂肪酶在pH 5.0时催化活性最高,且在pH 3.0–7.0范围内保持良好的稳定性[9];产自NICM 1207的脂肪酶在pH 2.5和45–50 ℃条件下水解活力最高,且在pH 2.5–9.0范围内可保持较高活性[5]。然而,黑曲霉来源的酸性脂肪酶生产工艺[10]和工业应用领域[11]存在专利壁垒,因此亟待开发其他来源的酸性脂肪酶,为工业应用提供更多的酶源选择。Shi等[7]发现了一种来自土曲霉A的酸性脂肪酶ATL,但仅对其进行了酶学特性研究。ATL具有良好的耐酸性能,其最适pH为4.0,且在pH 3.0–12.0范围内活力稳定。但是发酵脂肪酶活力较低,仅为13.7 U/mL,而且该酶催化活性低,比活力为24 U/mg[7]。
脂肪酶的盖子区域对酶活具有重要的影响。研究表明,对脂肪酶盖子区域进行替换或定点突变等蛋白质工程改造可使脂肪酶的催化活性和底物特异性发生变化。对来自sp.的脂肪酶基因LipK107的盖子区域进行定点突变,发现突变酶E130L+K131I和T138V的盖子区域疏水性增强,酶活性显著增强;突变酶I128E+V129D却使得盖子区域疏水性减弱,相应的酶活也有所降低[12]。说明盖子的氨基酸亲疏水性对酶活具有重要影响作用。Skjøt等将南极假丝酵母脂肪酶B (CALB) 的“盖子”替换成与其同源的来自粗糙链孢霉和玉米赤霉脂肪酶的相应的“盖子”,使得突变酶底物特异性发生改变,提高了对短链酯的水解活性,并且水解外消旋2-苯甲酸氨基乙酯的立体选择性增强[13]。说明盖子的结构可以改变脂肪酶的活性、底物特异性等方面的性质。此外,脂肪酶的某些保守氨基酸也会对酶活产生影响。Mosbah等为了研究Gly311位点对脂肪酶的作用,将该位点突变成Leu、Trp、Asp和Lys,结果突变酶的酶活都降低了,且突变酶的最适pH也发生了改变[14]。
本研究将土曲霉来源的酸性脂肪酶基因进行密码子优化,并在真核外源蛋白表达系统毕赤酵母中进行表达,通过ATL三维结构模拟及多序列同源比对,利用理性设计的方法在脂肪酶的盖子区域和底物结合口袋域选择关键位点进行定点突变,以期提高ATL的催化活性。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
大肠杆菌JM109和巴斯德毕赤酵母GS115由本实验室保存,分别用于载体扩增和目的蛋白外源表达。表达载体pPIC9K由本实验室保存。土曲霉脂肪酶基因ATL序列(GenBank Accession No. XP_001218444.1)根据毕赤酵母的密码子偏好性优化后,由南京金斯瑞公司合成。
1.1.2 培养基
大肠杆菌培养基LB:0.5% (/) 酵母浸出粉,1% (/) 胰蛋白胨,1% (/) NaCl,根据需要添加100 μg/mL氨苄青霉素 (Amp)。酵母培养基YPD、YPD-G418、MD、BMGY和BMMY根据“Invitrogen 公司操作手册”方法配制。
1.1.3 酶与试剂
限制性内切酶、T4 DNA连接酶、PrimeSTAR HS DNA聚合酶、PCR试剂购自TaKaRa 宝生物公司。引物由生工生物工程(上海)股份有限公司合成。PCR Purification Kit、Gel Extraction Kit、Plasmid Mini Kit I购自OMEGA BIO-TEK。对硝基苯酚酯-nitrophenyl系列底物:对硝基苯酚乙酸酯-nitrophenyl acetate (-NPA)、对硝基苯酚丁酸酯-nitrophenyl butyrate (-NPB)、对硝基苯酚戊酸酯-nitrophenyl valerate (-NPV)、对硝基苯酚辛酸酯-nitrophenyl caprylate (-NPC)、对硝基苯酚月桂酸酯-nitrophenyl laurate (-NPL)、对硝基苯酚豆蔻酸酯-nitrophenyl myristate (-NPM) 和对硝基苯酚棕榈酸酯-nitrophenyl palmitate (-NPP) 购自Sigma-Aldrich中国公司。其余试剂均为国产或进口分析纯。
1.2 方法
1.2.1 定点突变、重组表达质粒及工程菌的构建
从NCBI基因数据库中检索到ATL基因(GenBank登录号No. XP_001218444.1),以毕赤酵母密码子偏好性进行优化,将优化基因送南京金斯瑞公司合成,并在GS115中表达。利用限制性内切酶Ⅰ和Ⅱ分别对合成的ATL基因DNA片段和pPIC9K空载体进行双酶切,利用T4 DNA连接酶16 ℃连接过夜,转化至JM109感受态细胞,涂布于LB平板 (含100 μg/mL的Amp),筛选出阳性转化子,并提取得到重组质粒pPIC9K-ATL。pPIC9K-ATL经限制性内切酶Ⅰ线性化后,利用电转化法 (1 500 V,5 ms) 把重组质粒转化到宿主菌GS115中,并将转化物涂布于MD平板 (含250 μg/mL的G418),30 ℃培养2–3 d后,可挑取较大的阳性转化子菌落,提取酵母基因组利用引物3ʹAOX1和5ʹAOX1进行PCR验证,得到正确的重组菌株GS115/pPIC9K-ATL。具体电转化及筛选方法参见Invitrogen公司操作手册。
以pPIC9K-ATL为模板,分别以Lid-F和Lid-R上下游引物 (引物序列见表1) 进行全质粒PCR,得到突变后的质粒pPIC9K-ATLLid。反应体系为:模板1 μL;PrimerSTAR HS 25 μL;20 mmol/L的上下游引物各1 μL;加无菌水至总体积为50 μL。反应条件为:98 ℃预变性30 s;98 ℃变性10 s,58 ℃退火45 s,72 ℃延伸11 min,共30个循环;72 ℃延伸10 min。将PCR扩增产物经PCR Purification Kit试剂盒纯化后,限制性内切酶Ⅰ对PCR产物消化以降解模板质粒,再经过PCR Purification Kit试剂盒纯化后,转化至JM109感受态细胞。涂布于LB平板 (含100 μg/mL的Amp),筛选出阳性转化子,可获得突变质粒pPIC9K-ATLLid。其余7种突变质粒均按照该方法进行构建(引物序列见表1),分别得到不同的突变质粒:pPIC9K-ATLV218F、pPIC9K-ATLV218W、pPIC9K-ATLV218L、pPIC9K- ATLV218D、pPIC9K-ATLV218M、pPIC9K- ATLV218A、pPIC9K-ATLBP2。将突变后的质粒参考上述构建重组菌株GS115/pPIC9K-ATL的方法,得到8株突变株,分别为GS115/pPIC9K-ATLLid、GS115/ pPIC9K-ATLV218F、GS115/pPIC9K-ATLV218W、GS115/pPIC9K-ATLV218L、GS115/pPIC9K-ATLV218D、GS115/pPIC9K-ATLV218M、GS115/pPIC9K-ATLV218A和GS115/pPIC9K-ATLBP2。

表1 文中所用引物
The mutation sites are highlighted by bold.
1.2.2 脂肪酶的表达及蛋白纯化
将重组菌株在含有250 μg/mL G418的YPD平板上划线,30 ℃培养2–3 d。挑取一环平板上的单菌落,接种至25 mL BMGY培养基中,30 ℃、200 r/min振荡培养16–20 h至600为2–6。 6 000 r/min 离心10 min,收集菌体并用100 mL的BMMY培养基重悬,28 ℃、200 r/min振荡培养120 h,每隔24 h添加1%的甲醇诱导表达并且取样检测600、发酵液上清的蛋白浓度和酶活性。蛋白浓度的检测使用Bradford蛋白浓度测定试剂盒进行定量[15]。
利用Ni-NTA镍柱亲和层析和AKTA purifier蛋白纯化系统进行重组蛋白的分离纯化。发酵液4 ℃、6 000 r/min离心10 min后取上清液,用 0.22 μm微孔滤膜过滤处理后方可上样。上样缓冲液为Buffer A (0.5 mol/L NaCl,20 mmol/L Tris-HCl,pH 8.0),粗酶液上样到已用Buffer A平衡的镍柱上,再用含不同浓度咪唑 (0–0.25 mol/L) 的上样缓冲液梯度洗脱吸附的蛋白,收集各洗脱峰流出液,SDS-PAGE进行通过十二烷基磺酸钠-聚丙烯酰胺凝胶电泳。SDS-PAGE的制备和电泳方法参见《蛋白技术手册》[16],浓缩胶和分离胶浓度分别为5%和12%。
1.2.3 酶活测定
比色法:根据Kordel等[17]的方法,以对硝基苯酚月桂酸酯 (-NPL) 为底物进行酶活测定。酶活的定义:一定反应条件下每分钟产生1 μmol对硝基苯酚的酶量为一个脂肪酶水解酶活国际 单位。
碱滴定法:参考国家标准GB/T23535-2009方法[18],橄榄油与4% (/) 聚乙烯醇 (PVA) 以1∶3 (/)的比例混合,用高速匀浆机处理后得到乳白色的乳化液。以乳化后的橄榄油作为脂肪酶水解底物。每个反应体系包括4 mL橄榄油乳化液、5 mL柠檬酸-磷酸盐缓冲液和1 mL酶液。该反应被95%乙醇溶液终止,反应释放的脂肪酸由0.1 mol/L NaOH中和。酶活的定义:一定反应条件下每分钟产生1 μmol脂肪酸的酶量。
1.2.4 脂肪酶酶学性质研究
配制不同pH梯度的50 mmol/L柠檬酸-磷酸盐缓冲液 (pH 2.0–8.0) 和碳酸盐缓冲液 (pH 9.0–10.0),采用以橄榄油为底物的碱滴定法,测定脂肪酶的最适pH。测定pH稳定性时,将酶液加入上述不同梯度的缓冲液,并放于4 ℃,每隔3 h利用碱滴定法检测脂肪酶的残存活力,以初始酶活为100%相对酶活。
将酶液在不同的温度条件 (20–60 ℃) 下反应,利用碱滴定法检测脂肪酶的最适温度。测定温度稳定性时,将酶液在不同温度 (20–50 ℃) 中保温,每隔0.5–1 h利用碱滴定法检测脂肪酶的残存活力,以初始酶活为100%相对酶活。
将纯化后的脂肪酶与不同碳链长度对硝基苯酚酯底物混合进行显色反应,反应条件为40 ℃、50 mmol/L的柠檬酸-磷酸盐缓冲液 (pH 7.0),以检测脂肪酶对底物链长的特异性。使用的对硝基苯酚酯底物根据碳链长度由短至长依次为-NPA(C2)、-NPB(C4)、-NPV(C5)、-NPC(C8)、-NPL(C12)、-NPM(C14)和-NPP(C16)。
脂肪酶酶促反应动力学参数max、m值、cat值的计算采用双倒数作图法 (Lineweaver-Burk plot) 和米氏方程 (Michaelis-Menten equation)。以对硝基苯酚酯-NPL和-NPP作为反应底物,浓度范围是0.1–0.9 mmol/L,反应条件为40 ℃,50 mmol/L的柠檬酸-磷酸盐缓冲液 (pH 7.0)。
1.2.5 脂肪酶的同源建模
利用在线软件对脂肪酶进行同源建模(https://swissmodel.expasy.org/),根据数据库中已报道的同源性较高的蛋白结构,模拟出发脂肪酶的三级结构。计算机软件Discovery Studio 3.1 viewer 和PyMOL对酶的三级结构进行观察与分析。
1.2.6 脂肪酶的分子对接
利用软件AutoDock Vina进行分子对接研究,以脂肪酶蛋白为受体,底物-NPL为配体构建分子对接系统,分析突变前后的脂肪酶分子构象的变化对脂肪酶催化活性的影响。对接参数设置为:配体可旋转键12个;将ATL口袋区域设为格子中心;格子大小设置为X,Y,Z:62,43,43;晶格的中心(Center) 坐标设为(–21.5,18.1,82.9),其他参数为系统默认。
2 结果与分析
2.1 理性设计确定ATL突变位点
2.1.1 ATL的氨基酸序列与模拟三维结构分析
已有研究表明,来自黑曲霉NICM 1207的酸性脂肪酶ANL在固态发酵和液态发酵条件下脂肪酶酶活分别为630 U/g (固态底物) 和18 U/mL[5],纯化后的比活力为1 373.13 U/mg[19],酶活力远高于ATL已报道的数值 (24 U/mg)。ANL最适pH 和温度分别为2.5和45–50 ℃,且在pH 2.5–9.0范围内稳定性较好[5]。因此,本研究将以黑曲霉脂肪酶ANL为参考依据,对土曲霉脂肪酶ATL进行理性改造。ATL与ANL (GenBank Accession No. XP_001397501.1) 的氨基酸序列一致性为53.05% (图1A)。鉴于还未有报道ANL和ATL的蛋白三维结构,需利用在线工具模拟获得这两个蛋白的三维模拟结构。将ATL和ANL分别进行同源搜索比对,发现这两种脂肪酶预测的结构与来自疏棉状嗜热丝孢菌脂肪酶TLL (PDB登录号1DT5) 相似性最高,分别为51.7%和50.37%,因此1DT5可作为模板结构对ATL进行同源建模。由于1DT5是闭盖结构,无法进行底物与蛋白的分子对接,结合相似度较高的米黑根毛霉脂肪酶RML的开盖结构 (PDB登录号4TGL) 信息模拟ATL的开盖结构 (图1B)。
初中教育阶段,所有学生每周都可以获得由艺术家或经过专业培训的教师开展的1小时造型艺术和1小时音乐培训。
2.1.2 ATL突变位点的确定
由于ATL在氨基酸序列和三维模拟结构上都与ANL拥有较高的相似度,而且ANL具有较高的比活,因此通过参照ANL的氨基酸序列对ATL的催化活性区域的部分位点进行选择性突变设计。如图1A所示,参考ANL的盖子区域(92STIKNWIADLDFI104) 对ATL的盖子区域(92RSPANWIANLDFI104) 进行改造,利用全质粒PCR的方法,将ATL的盖子区域前4个氨基酸替换为Ser-Thr-Ile-Lys,得到突变酶ATLLid。ANL含有两个底物结合口袋域(Binding pocket 1 domain和Binding pocket 2 domain)。在Binding pocket 1 domain中,ATL仅在Val218位点上不同于ANL中的相应位点Phe219。将ATL中的Val218替换成Phe,得到突变酶ATLV218F。在Binding pocket 2 domain中,将ATL氨基酸区段265HSWYFGDISECQ276替换为ANL中的266HLWYFFAISECLL278得到突变酶ATLBP2。具体突变位点分布如图1B所示。
2.1.3 ATL第218位点与ANL第219位点保守性分析
分别对ANL的Phe219和ATL的Val218的氨基酸保守性进行分析(http://gremlin.bakerlab. org/submit.php)。结果表明(图2),ANL在219位点的保守性排序依次为亮氨酸Leu(L)、色氨酸Trp(W)、苯丙氨酸Phe(F) 和甲硫氨酸Met(M),均为疏水性氨基酸。但也有较少情况下,该位点可能为天冬氨酸Asp(D) 和谷氨酰胺Asn(Q)。ATL在相应218位点的保守性同样为Leu(L)、Trp(W)、Phe(F) 和Met(M)。此外,将ATL序列提交在线比对软件(https://blast.ncbi.nlm.nih.gov/),分析同源性较高的前100个序列的保守性,该位点还可能为丙氨酸Ala(A)、异亮氨酸Ile(I)。考虑到L与I性质相仿,选择其一作为突变点即可。综上,选择在ATL的第218位点除构建ATLV218F外继续构建5种突变酶:ATLV218W、ATLV218L、ATLV218D、ATLV218M和ATLV218A。
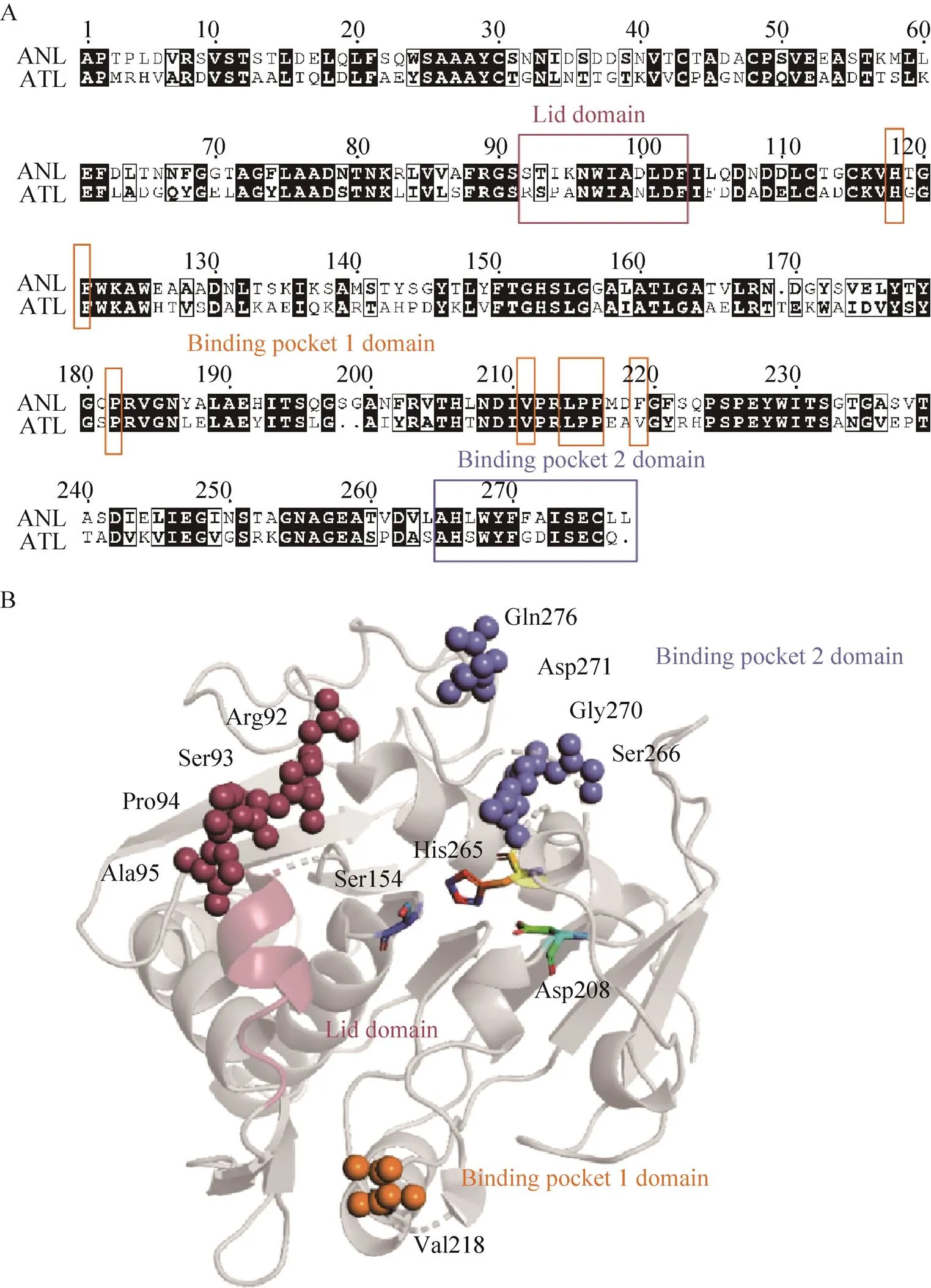
图1 氨基酸序列与三维结构分析(A:ANL和ATL的氨基酸序列比对;B:ATL三维结构模拟图)
2.2 重组菌株的诱导表达及分离纯化
将表达野生型脂肪酶的菌株GS115/pPIC9K- ATL与8株表达突变酶的菌株GS115/pPIC9K- ATLLid、GS115/pPIC9K-ATLV218F、GS115/pPIC9K- ATLV218W、GS115/pPIC9K-ATLV218L、GS115/ pPIC9K-ATLV218D、GS115/ pPIC9K-ATLV218M、GS115/pPIC9K-ATLV218A和GS115/pPIC9K- ATLBP2进行摇瓶发酵。设置3个平行,定时取样,检测发酵过程中菌体的生长情况、胞外总蛋白分泌情况和上清液中酶活的变化。如图3所示,当甲醇诱导发酵至96 h时,以-NPL为底物,突变菌株GS115/pPIC9K-ATLLid和GS115/pPIC9K- ATLV218W在pH 7.0、40 ℃条件下的酶活分别达到0.075 U/mL和0.141 U/mL,较出发菌株GS115/pPIC9K-ATL的酶活分别提高了1.95倍和3.66倍。将发酵96 h时收集的发酵上清液进行蛋白纯化,得到纯化的蛋白,并通过SDS-PAGE检测 (图4)。突变酶与野生型酶在蛋白分子量大小上没有显著区别,各样品均呈现约44 kDa大小的条带 (黑色箭头所示)。将纯化后的蛋白制备成约0.80 mg/mL蛋白浓度的酶液后,以-NPL为底物,在pH 7.0、40 ℃条件下,分别检测突变酶和野生型酶的酶活。结果表明,ATLLid和ATLV218W对-NPL的水解活性与野生型相比显著提高,比酶活分别为0.77 U/mg和1.32 U/mg,分别为野生型ATL活力 (0.34 U/mg) 2.26倍和3.38倍 (图5)。因此,将对ATLLid和ATLV218W进行酶学性质的测定和分析突变位点对蛋白催化活力影响的机制。

图2 ANL Phe219和ATL Val218的氨基酸保守性分析

图3 突变菌株的摇瓶发酵曲线(A:菌体浓度OD600;B:蛋白浓度;C:酶活)
2.3 突变酶和野生型酶的酶学性质
2.3.1 最适pH及pH稳定性
如图6所示,突变酶ATLLid和ATLV218W与野生型酶ATL的最适pH均为5.0,当pH为2.0时酶迅速失活,酶活力几乎为0。在碱性环境中,当pH高于7.0,酶活力明显下降,活力低于30%。可见脂肪酶起水解活性作用的pH范围较窄,一般在pH 4.5–6.0之间。如图6所示,在pH 4.0–8.0范围内ATLLid、ATLV218W与ATL均具有较好的稳定性,残余活力保持在90%以上。当在pH 3.0缓冲液中放置24 h后,ATLLid、ATLV218W和ATL分别残留约78.26%、76.19%和72.19%的相对酶活,而在pH 2.0时,分别残留45.07%、42.40%和33.86%的相对酶活。可见,ATL在毕赤酵母中重组表达后并没有改变其耐酸的酶学特性[7],且突变酶ATLLid和ATLV218W也同样具有良好的耐酸特性。
如图7A所示,突变酶ATLLid和ATLV218W的最适温度为35 ℃,而原始酶ATL则在40 ℃时活性最高。当温度上升至40 ℃,ATLLid酶活迅速下降,仅保留52.53%左右,ATLV218W则仍保留88.46%的酶活;当温度高于40 ℃时,3种蛋白的酶活均迅速下降,并在60 ℃完全失活;当温度降为20 ℃,ATLLid和ATLV218W分别保留69.62%和76.67%的活力,而ATL仅保留18.69%的活力。由图7可见,在20 ℃温浴6.5 h后,ATLLid、ATLV218W和ATL分别保留了74.79%、84.51%和85.01%的残存活力;在30 ℃温浴6.5 h后,分别测得55.70%、55.84%和68.87%的相对酶活;在40 ℃温浴6.5 h后,分别测得36.04%、40.85%和60.28%的相对酶活;而在50 ℃温浴,酶活性均迅速下降,温浴1 h后仅分别剩余6.77%、10.87%和11.69%的相对酶活。可见,突变使得ATL的最适温度向低温 (35 ℃) 略微偏移,对温度的稳定性也有所降低(ATLLid<ATLV218W<ATL)。

图4 野生型脂肪酶ATL和突变型脂肪酶的SDS-PAGE图
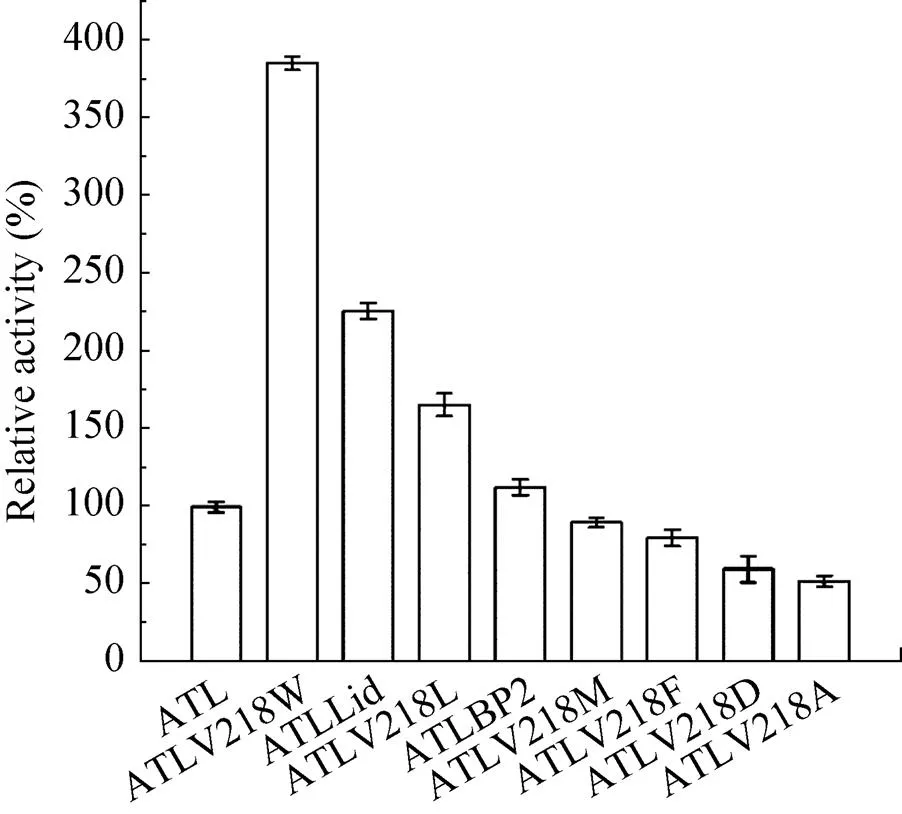
图5 野生型脂肪酶与突变型脂肪酶的水解活性对比
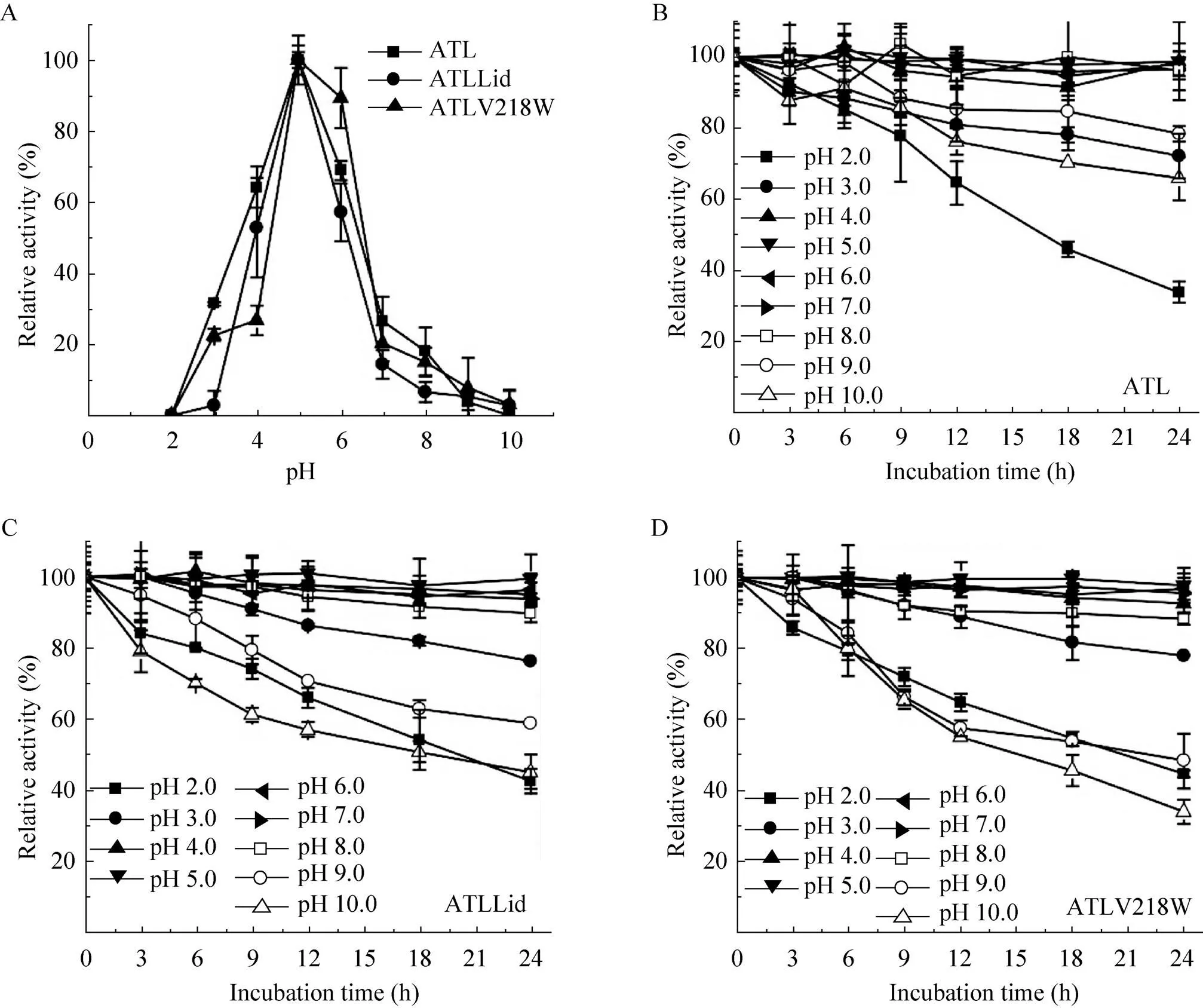
图6 pH对脂肪酶活力和稳定性的影响
2.3.3 底物特异性
对酶活较高的突变酶ATLLid和ATLV218W进行了底物碳链长度特异性研究,并与野生型酶ATL进行对比。如图8所示,突变酶与野生型酶均对12个碳的对硝基苯酚月桂酸酯-NPL特异性最高。然而,ATLLid对C14-C16的相对酶活显著高于ATLV218W和ATL,说明ATLLid提高了对长链脂肪酸的底物特异性。该结果表明,ATL盖子区域改造后可识别含长链脂肪酸的底物并使其更好地结合到活性中心。可见,脂肪酶的盖子区域部分位点的改变会对酶的底物特异性产生影响,正如Santarossa等[20]将莓实假单胞菌脂肪酶的盖子区域的Thr137和Thr138分别突变为Val和Asp后显示出了对不同碳链长度底物的不同偏好性。
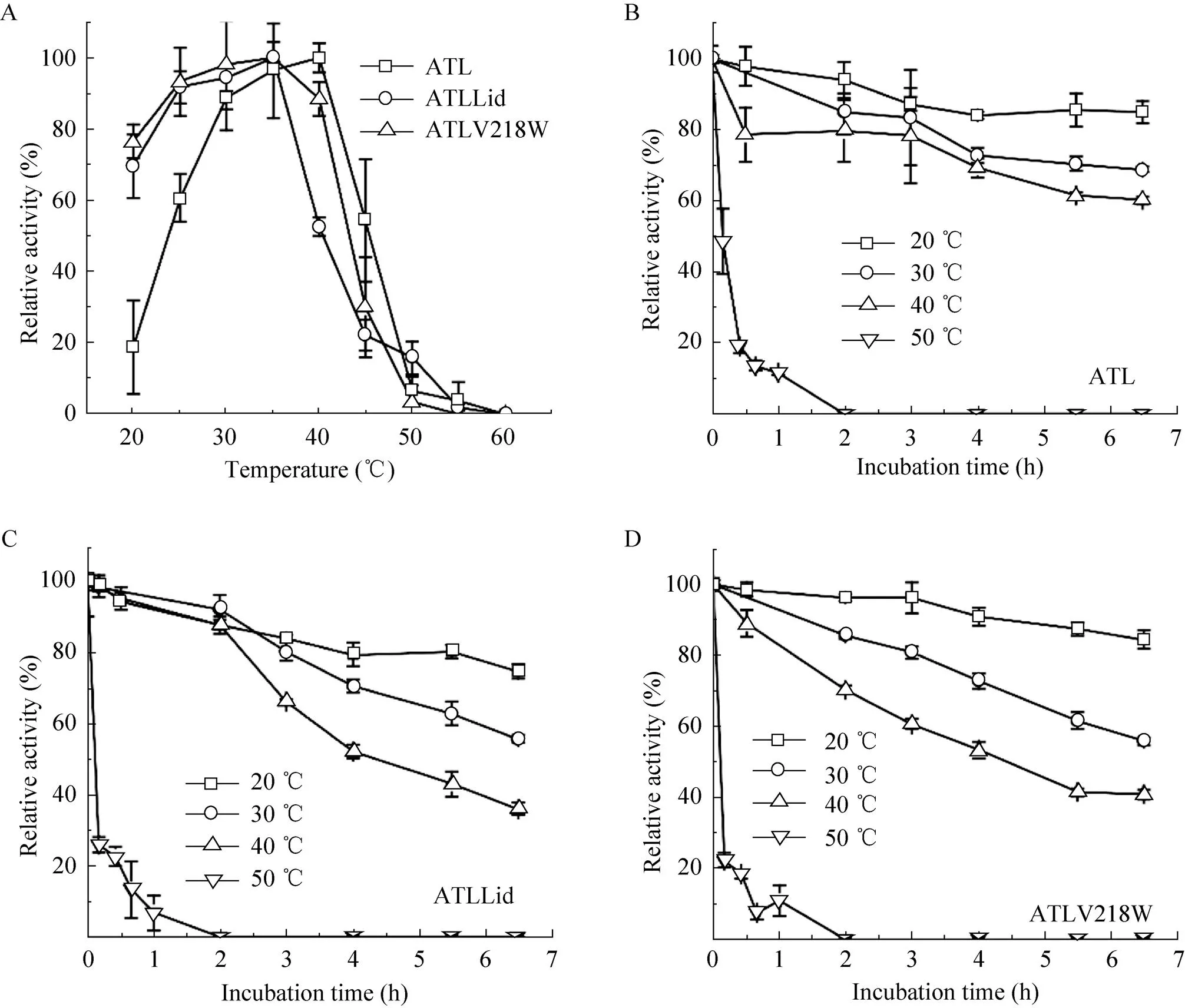
图7 温度对脂肪酶活力和稳定性的影响
2.3.4 酶促反应的动力学常数
以对硝基苯酚酯-NPP和-NPL为底物,测定突变酶ATLLid和ATLV218W与野生型酶ATL的反应动力学 (表2)。ATLLid对两种底物-NPL和-NPP的m值从原来的0.15 mmol/L和0.73 mmol/L分别提高到了1.53 mmol/L和1.69 mmol/L,说明突变的盖子结构阻碍底物进入酶的活性中心,导致突变酶对底物的亲和力变差,酶反应所需的底物浓度增大。但是,脂肪酶的cat值却比野生型酶分别提高了约39.37倍和9.55倍,催化活性显著增强。由此,cat/m值分别提高了2.85倍和3.57倍,该结果表明酶的催化效率提高的主要原因是盖子构型的变化导致的酶活性中心催化能力的提高,但随着对底物亲和力的降低,会影响催化效率的提高程度。
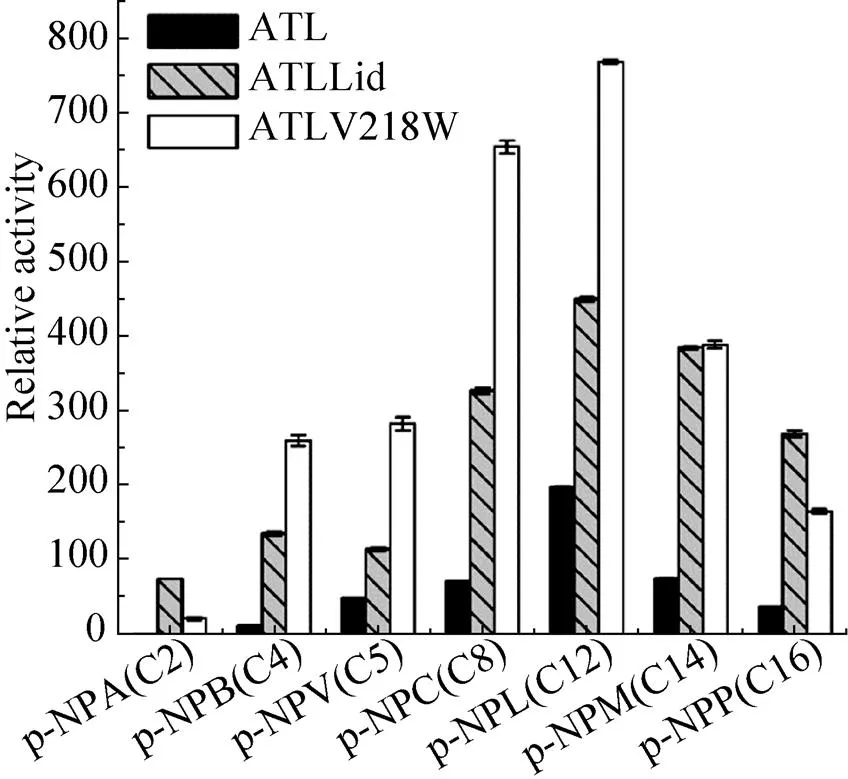
图8 突变型脂肪酶ATLLid、ATLV218W和野生型脂肪酶ATL的底物特异性
底物结合口袋域突变后,脂肪酶ATLV218W对两种底物-NPL和-NPP的m值比野生型酶ATL分别提高了4.33倍和0.95倍,说明将底物结合口袋域中的缬氨酸Val突变为色氨酸Trp后,对底物的亲和力也显著降低,酶反应需要的底物浓度变大。然而,脂肪酶的cat值却比野生型酶分别提高了约50.79倍和14.67倍,说明突变影响了酶的活性中心与底物的相互作用,导致催化活性显著增强。由此,cat/m值分别提高了8.48倍和7.01倍,该结果表明酶的催化效率提高的主要原因是底物结合口袋域构型变化导致的酶活性中心催化能力的提高。
综合上述两种突变酶的动力学参数的测定结果发现,选定的突变位点虽然使得脂肪酶对底物的亲和能力降低,但显著提高了酶的催化速度,从而提高了脂肪酶的催化活性。
2.4 突变酶的结构分析
本研究通过对ATL的盖子区域和底物结合口袋域的部分位点进行定点突变,得到两种对底物对硝基苯酚月桂酸酯-NPL的催化活性显著提高的突变酶ATLLid和ATLV218W。通过对蛋白质同源建模模拟及分子对接,分别分析导致ATLLid和ATLV218W对底物-NPL催化活性提高的原因。
2.4.1 突变酶ATLLid
解折叠自由能是蛋白质折叠态和伸展态之间的吉布斯自由能差,反映的是蛋白质解聚及解链过程中受到的氢键、范德华力、熵效应等的综合影响,是反映蛋白质热力学稳定性的重要参数[21]。如图1所示突变酶ATLLid的突变位点位于盖子区域的前4个氨基酸,利用FoldX[22]预测突变结构的解折叠自由能,得到的解折叠自由能差(ΔΔ) 约为–0.92 kcal/mol (ΔΔ=Δmutant–Δwild,ΔΔ>0代表正效应,ΔΔ<0代表负效应[21,23]),说明盖子区域的突变降低了整个结构的解折叠自由能,可能在一定程度上提高了ATL盖子区域的柔性,进而使得底物更易进入活性中心。
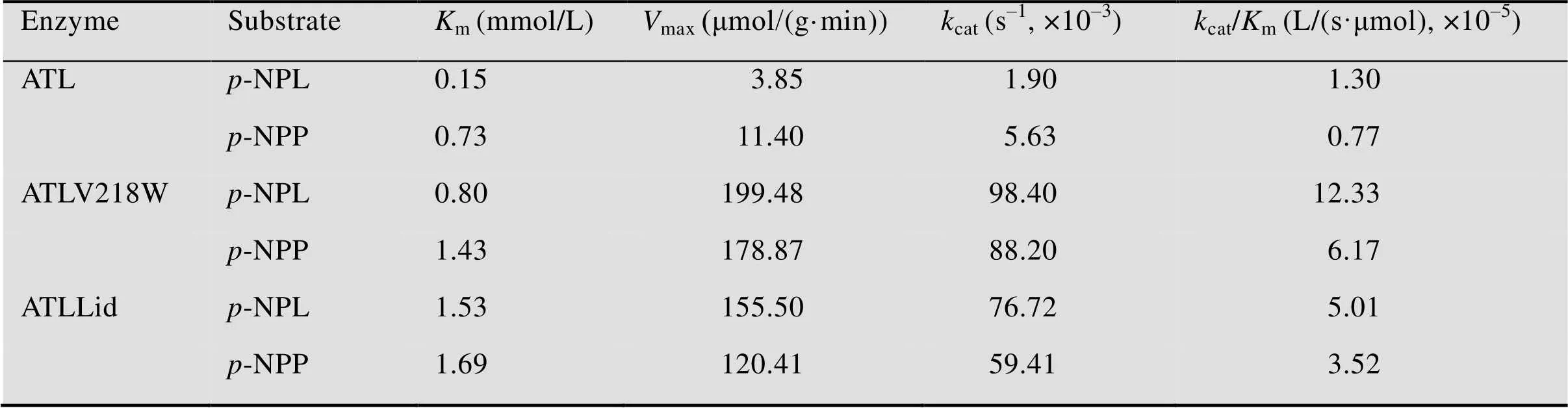
表2 野生型ATL与突变型酶ATLLid和ATLV218W动力学参数
蛋白的亲疏水相互作用是维持蛋白质空间构象的重要因素,通常根据蛋白质的总平均亲水性 GRAVY值 (Grand average of hydropathy) 来预测疏水性的变化[24]。GRAVY值是指序列中所有氨基酸亲水值的总和与氨基酸数量的比值,负值越大表示亲水性越好,正值越大表示疏水性越强。通过Protparam工具(http://web.expasy.org/ protparam/)分析结果显示,出发酶ATL的盖子序列的GRAVY为–0.300,而突变酶ATLLid盖子序列的GRAVY为0.050。这说明突变后的ATLLid盖子的疏水性增加,这可能导致盖子与疏水性底物的亲和力增加,底物进入活性中心的阻力减小,从而催化效率提高。
利用PyMOL中的Mutagenesis模块来构建突变酶ATLLid的模拟结构,并分别将ATL和ATLLid的开盖模拟结构与-NPL进行分子对接。如图9所示,-NPL所结合的狭长凹陷区域为底物结合口袋区域,红色代表催化活性中心,砖红色为盖子区域。突变酶ATLLid的底物结合口袋(图9A) 与野生型 (图9B) 相比,位于对硝基苯所在方向的口袋区域显著增大,与野生型相比空间位阻减少 (黄色箭头所示),使对硝基苯酚酯类底物更好地进入口袋并与活性中心结合,从而提高ATLLid对该类底物的偏好性。另外,ATLLid盖子的疏水性增加,能够更好地稳定底物中疏水性长链脂肪酸基团,因此ATLLid对长链底物如-NPL,催化活力更高。分子对接为ATLLid的底物特异性研究结果 (图8) 提供了有力的证据。
2.4.2 突变酶ATLV218W
如图10A所示,Val218处于Binding pocket 1 domain,位于β转角,介于α螺旋与无规卷曲之间,其N端通过α螺旋与催化三联体中的Asp208相连。使用PyMOL中的Mutagenesis模块进行虚拟突变,将218位点的V突变为W后,该β转角的二面角构型发生变化,使得该区域的空间构象发生改变,同时影响到Asp208的空间位置。如图10所示,突变后,ATLV218W中Asp208与His265的间距由3.2 Å (图10A) 缩短至2.8 Å (图10B),从而影响催化三联体的整体构象。由于催化三联体的空间位置发生极小的改变都将会影响酶的催化活性,在本研究中ATLV218W对于底物-NPL的cat相比野生型提高了49.9倍。
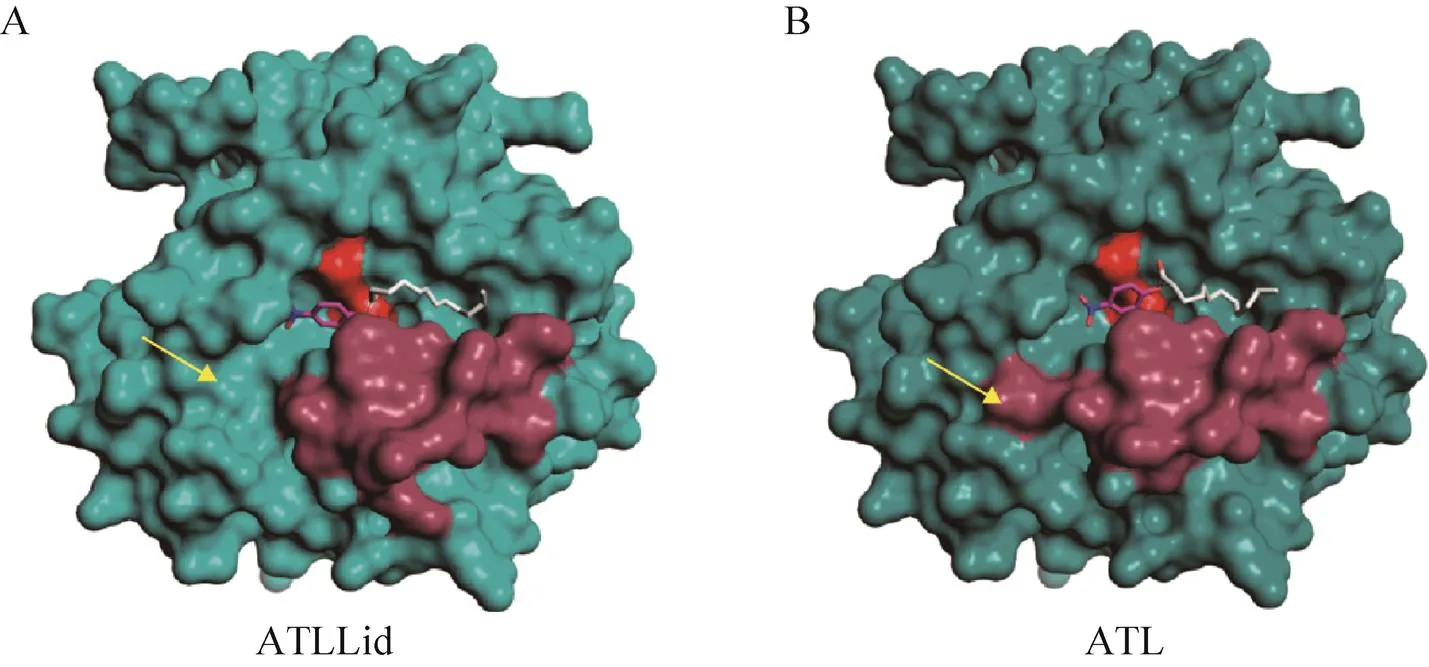
图9 ATLLid与ATL底物对接三维模拟图
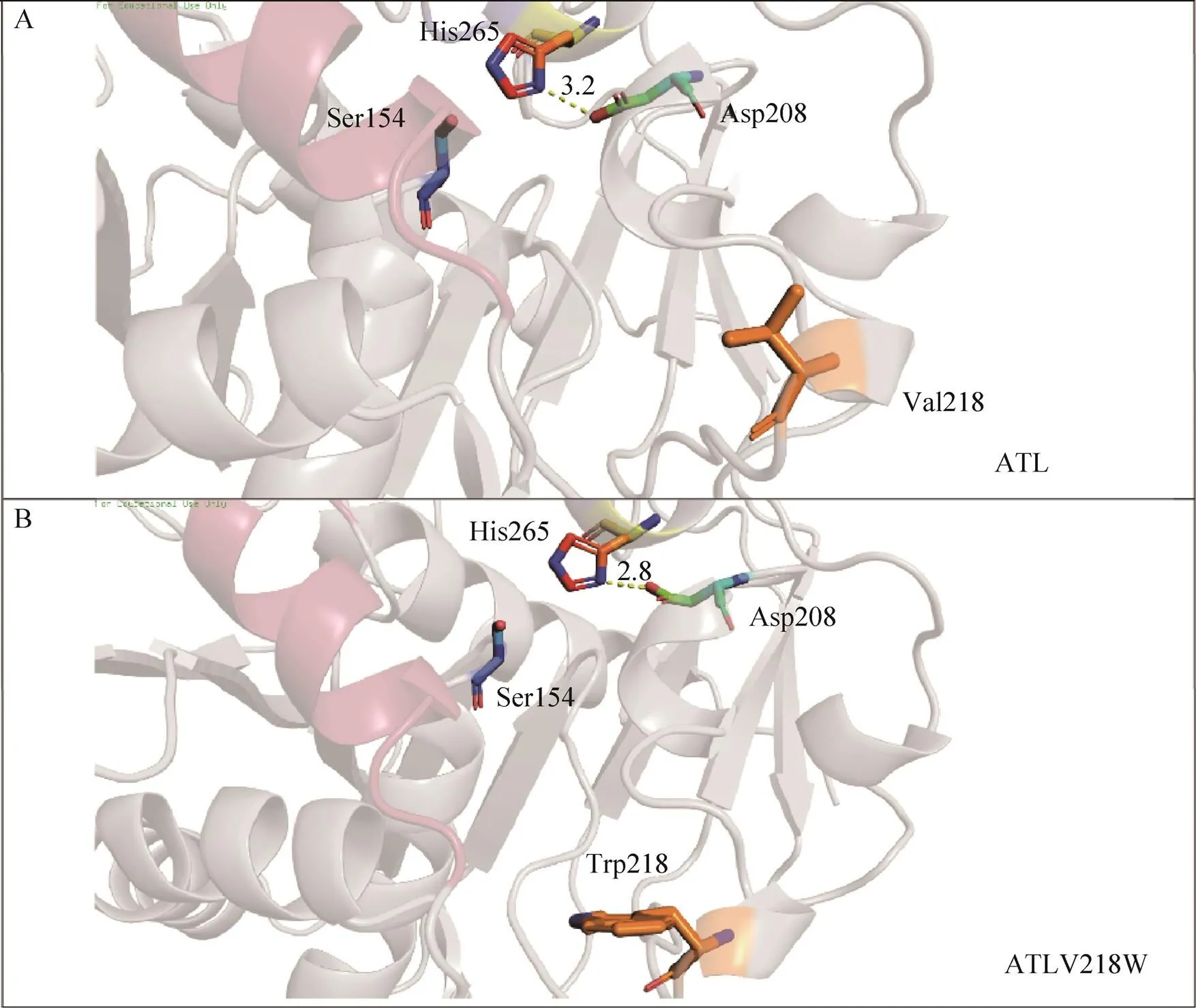
图10 ATL的突变点Val218的三维结构模拟图
3 结论
综上所述,本研究采用理性设计的方法,对来自的脂肪酶ATL的盖子区域和底物结合口袋域中的位点进行定点突变,得到了对对硝基苯酚酯类底物催化活性显著提高的两种突变酶ATLLid和ATLV218W。利用同源建模模拟及分子对接等手段分析得出,ATLLid盖子区域的突变可使盖子柔性增强、疏水性提高和底物结合口袋变大,使得底物更易进入催化活性中心,提高对长链底物的催化活力。ATLV218W底物结合口袋域的突变则影响了催化三联体中的Asp208的空间位置,进而影响酶的催化活性。因此,本研究的后续工作将在此研究工作基础上对盖子区域和底物结合口袋域进行饱和突变或组合突变,以期获得催化活性更高的脂肪酶。
[1] Sharma R, Chisti Y, Banerjee UC. Production, purification, characterization, and applications of lipases. Biotechnol Adv, 2001, 19(8): 627–662.
[2] Ramani K, Chockalingam E, Sekaran G. Production of a novel extracellular acidic lipase fromusing slaughterhouse waste as a substrate. J Ind Microbiol Biotechnol, 2010, 37(5): 531–535.
[3] Yan LJ, Xie ZR, Zhao CL, et al. A research on screening and fermentation conditions of thermostable lipase strain NJY-I-3. Food Sci Technol, 2010, 35(3): 11–15 (in Chinese). 闫丽娟, 谢振荣, 赵春雷, 等. 耐高温酸性脂肪酶菌株NJY-1-3的选育及发酵条件的研究. 食品科技, 2010, 35(3): 11–15.
[4] Wang HY, Li FH, Gao XH. Advances in lipase research and its application in animal feed. Feed Ind, 2007, 28(4): 14–17 (in Chinese). 王海燕, 李富华, 高秀华. 脂肪酶的研究进展及其在饲料中的应用. 饲料工业, 2007, 28(4): 14–17.
[5] Mahadik ND, Puntambekar US, Bastawde KB, et al. Production of acidic lipase byin solid state fermentation. Proc Biochem, 2002, 38(5): 715–721.
[6] Zhou J, Chen WW, Jia ZB, et al. Purification and characterization of lipase produced byCJLU-31 isolated from waste cooking oily soil. Am J Food Technol, 2012, 7(10): 596–608.
[7] Shi H, Meng Y, Yang M, et al. Purification and characterization of a hydrolysis-resistant lipase from. Biotechnol Appl Biochem, 2014, 61(2): 165–174.
[8] Mahadik ND, Bastawde KB, Puntambekar US, et al. Production of acidic lipase by a mutant ofNCIM 1207 in submerged fermentation. Proc Biochem, 2004, 39(12): 2031–2034.
[9] Liu G, Hu SQ, Li L, et al. Purification and characterization of a lipase with high thermostability and polar organic solvent-tolerance fromAN0512. Lipids, 2015, 50(11): 1155–1163.
[10] Mahadik ND, Gokhale DV, Bastawde KB, et al. Process for the preparation of acidic lipase: US, 6534303B2. 2003-03-18.
[11] Davidson JG, Medhekar R, Moore J, et al. Protease composition and method for treating a digestive disorder: US, 7067124B2. 2006-06-27.
[12] Gao B, Xu T, Lin JP, et al. Improving the catalytic activity of lipase LipK107 fromsp. by site-directed mutagenesis in the lid domain based on computer simulation. J Mol Cataly B: Enzym, 2011, 68(3/4): 286–291.
[13] Skjøt M, de Maria L, Chatterjee R, et al. Understanding the plasticity of the α/β hydrolase fold: lid swapping on thelipase B results in chimeras with interesting biocatalytic properties. Chembiochem, 2009, 10(3): 520–527.
[14] Mosbah H, Sayari A, Horchani H, et al. Involvement of Gly 311 residue on substrate discrimination, pH and temperature dependency of recombinantlipase: a study with emulsified substrate. Protein Expr Purif, 2007, 55(1): 31–39.
[15] Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72(1/2): 248–254.
[16] Wang JZ, Fan M. Manual of protein technique. Beijing: Science Press, 2000 (in Chinese).汪家政, 范明. 蛋白质技术手册. 北京: 科学出版社, 2000.
[17] Kordel M, Hofmann B, Schomburg D, et al. Extracellular lipase ofsp. strain ATCC 21808: purification, characterization, crystallization, and preliminary X-ray diffraction data. J Bacteriol, 1991, 173(15): 4836–4841.
[18] China Food Fermentation Industry Research Institute. Novozymes (China) Biotechnology Co., Ltd. GB/T 23535-2009 Lipase preparations. People's Republic of China State Administration of quality supervision, inspection and quarantine, China National Standardization Management Committee (in Chinese).中国食品发酵工业研究院. 诺维信(中国)生物技术有限公司. GB/T 23535-2009 脂肪酶制剂. 中华人民共和国国家质量监督检验检疫总局中国国家标准化管理委员会; 2009.
[19] Mhetras NC, Bastawde KB, Gokhale DV. Purification and characterization of acidic lipase fromNCIM 1207. Bioresour Technol, 2009, 100(3): 1486–1490.
[20] Santarossa G, Lafranconi PG, Alquati C, et al. Mutations in the "lid" region affect chain length specificity and thermostability of alipase. FEBS Lett, 2005, 579(11): 2383–2386.
[21] Benedix A, Becker CM, de Groot BL, et al. Predicting free energy changes using structural ensembles. Nat Methods, 2009, 6(1): 3–4.
[22] Schymkowitz J, Borg J, Stricher F, et al. The FoldX web server: an online force field. Nucleic Acids Res, 2005, 33(S2): W382–W388.
[23] Tian J. Improvement of the protein thermal stability by the computer aided molecular design[D]. Beijing: Chinese Academy of Agricultural Sciences, 2011 (in Chinese). 田健. 计算机辅助分子设计提高蛋白质热稳定性的研究[D]. 北京: 中国农业科学院, 2011.
[24] Kyte J, Doolittle RF. A simple method for displaying the hydropathic character of a protein. J Mol Biol, 1982, 157(1): 105–132.
(本文责编 郝丽芳)
Improvement of catalytic activity oflipase by site-directed mutagenesis
Xiaofeng Zhang, Xiaowei Yu, and Yan Xu
The Key Laboratory of Industrial Biotechnology, Ministry of Education, School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu, China
The catalytic activity oflipase (ATL) was improved by rational design. According to the sequence analysis and homologous modeling, several amino acids involved in the lid domain and substrate binding pocket domains of the acidic lipase ATL were mutated by site-directed mutagenesis, and eight mutants were constructed. These mutants and the wild type lipase ATL were expressed inGS115 and the enzymatic properties were characterized. The mutants ATLLid and ATLV218W exhibited higher hydrolytic activity than ATL towards-nitrophenyl laurate. Thecatvalues of ATLLid and ATLV218W towards-nitrophenyl laurate were 39.37- and 50.79-fold higher, and thecat/mvalues were 2.85- and 8.48-fold higher than the wild type, respectively. Although thermostability of these mutants decreased slightly, ATLLid and ATLV218W still exhibited the maximum activity at pH 5.0 and high stability in a broad range of pH (4.0–8.0), which were similar to the wild type. Using homologous modeling and molecular docking technology the mechanism for the improvement of catalytic activity was analyzed. These findings not only shed light on the relationship between the lid domain/substrate binding pocket domain and catalytic activity but also provided comprehensive scheme for further engineering to gain more efficient lipases.
lipase,, homologous modeling, site-directed mutagenesis, catalytic activity, molecular docking
January 24, 2018;
April 3, 2018
National Natural Science Foundation of China (No. 31671799), Six Talent Peaks Project in Jiangsu Province (No. NY-010), 333 Project in Jiangsu Province (No. BRA2015316), National High Technology Research and Development Program of China (863 Program) (No. 2012AA022207).
Xiaowei Yu. Tel/Fax: +86-510-85918201; E-mail: bioyuxw@aliyun.com
国家自然科学基金(No. 31671799),江苏省“六大人才高峰”项目 (No. NY-010),江苏省“333工程”项目 (No. BRA2015316),国家高技术研究发展计划 (863计划) (No. 2012AA022207) 资助。
10.13345/j.cjb.180040
2018-04-25
http://kns.cnki.net/kcms/detail/11.1998.q.20180424.1007.002.html
